Diferencia entre revisiones de «Evolución biológica»
→Microbiología y transferencia horizontal de genes: cambios de redacción |
Sin resumen de edición |
||
| Línea 1: | Línea 1: | ||
{{About|evolution in biology|related articles|Outline of evolution|other uses}} |
|||
{{pp-semi-protected|small=yes}} |
|||
{{short description|Change in the heritable characteristics of biological populations over successive generations }} |
|||
{{See introduction}} |
|||
{{Evolutionary biology}} |
|||
{{Use British English|date=January 2014}} |
|||
<!--NOTE: Please do not change the lead sentence(s) without consulting the discussion page first. This lead has been discussed and there is general consensus that this is the best one for now. Thanks.--> |
|||
'''Evolution''' is change in the [[Heredity|heritable]] [[Phenotypic trait|characteristics]] of biological [[population]]s over successive [[generation]]s.<ref>{{harvnb|Hall|Hallgrímsson|2008|pp=[https://books.google.com/books?id=jrDD3cyA09kC&pg=PA4 4–6]}}</ref><ref>{{cite web |title=Evolution Resources |location=Washington, DC |publisher=[[National Academies of Sciences, Engineering, and Medicine]] |year=2016 |url=http://www.nas.edu/evolution/index.html |deadurl=no |archiveurl=https://web.archive.org/web/20160603230514/http://www.nas.edu/evolution/index.html |archivedate=2016-06-03 |df= }}</ref> These characteristics are the expressions of [[gene]]s that are passed on from parent to offspring during [[reproduction]]. Different characteristics tend to exist within any given population as a result of [[mutation]], [[genetic recombination]] and other sources of [[Genetic variability|genetic variation]].<ref name="Futuyma2017c">{{cite book | last1=Futuyma | first1=Douglas J. | last2=Kirkpatrick | first2=Mark | year = 2017 | chapter = Mutation and variation | title=Evolution | pages = 79–102 | edition = Fourth | publisher = Sunderland, Massachusetts: Sinauer Associates, Inc | isbn=978-1-60535-605-1}}</ref> Evolution occurs when evolutionary processes such as [[natural selection]] (including [[sexual selection]]) and [[genetic drift]] act on this variation, resulting in certain characteristics becoming more common or rare within a population.<ref name="Scott-Phillips">{{cite journal |last1=Scott-Phillips |first1=Thomas C. |last2=Laland |first2=Kevin N. |last3=Shuker |first3=David M. |last4=Dickins |first4=Thomas E. |last5=West |first5=Stuart A. |author-link5=Stuart West |date=May 2014 |title=The Niche Construction Perspective: A Critical Appraisal |journal=[[Evolution (journal)|Evolution]] |volume=68 |issue=5 |pages=1231–1243 |doi=10.1111/evo.12332 |issn=0014-3820 |pmid=24325256 |pmc=4261998 |deadurl=no |df= |quote=Evolutionary processes are generally thought of as processes by which these changes occur. Four such processes are widely recognized: natural selection (in the broad sense, to include sexual selection), genetic drift, mutation, and migration (Fisher 1930; Haldane 1932). The latter two generate variation; the first two sort it.}}</ref> It is this process of evolution that has given rise to [[biodiversity]] at every level of [[biological organisation]], including the levels of [[species]], individual [[organism]]s and [[molecular evolution|molecules]].<ref>{{harvnb|Hall|Hallgrímsson|2008|pp=3–5}}</ref><ref name="Voet2016a">{{cite book | last1 = Voet | first1 = Donald | last2 = Voet | first2 = Judith G. | last3 = Pratt | first3 = Charlotte W. | year = 2016 | chapter = Introduction to the chemistry of life | title = Fundamentals of Biochemistry: Life at the molecular level | pages = 1–22 | edition = Fifth | publisher = Hoboken, New Jersey: Wiley | isbn=1-11-891840-1}}</ref> |
|||
The [[scientific theory]] of evolution by natural selection was proposed by [[Charles Darwin]] and [[Alfred Russel Wallace]] in the mid-19th century and was set out in detail in Darwin's book ''[[On the Origin of Species]]'' (1859).<ref name=Darwin>{{cite book |last=Darwin |first=Charles |authorlink = Charles Darwin |year=1860 |title=On the Origin of Species |place=London |publisher=John Murray |edition=2nd |pages=490 |url=http://darwin-online.org.uk/content/frameset?itemID=F376&viewtype=text&pageseq=508}}</ref> Evolution by natural selection was first demonstrated by the observation that more offspring are often produced than can possibly survive. This is followed by three observable [[fact]]s about living organisms: 1) traits vary among individuals with respect to their morphology, [[physiology]] and behaviour ([[Phenotype#Phenotypic variation|phenotypic variation]]), 2) different traits confer different rates of survival and [[reproduction]] (differential [[Fitness (biology)|fitness]]) and 3) traits can be passed from generation to generation ([[heritability]] of fitness).<ref name="Lewontin70">{{cite journal |last=Lewontin |first=Richard C. |authorlink=Richard Lewontin |date=November 1970 |title=The Units of Selection |url=http://joelvelasco.net/teaching/167/lewontin%2070%20-%20the%20units%20of%20selection.pdf |journal=[[Annual Review of Ecology, Evolution, and Systematics|Annual Review of Ecology and Systematics]] |volume=1 |pages=1–18 |doi=10.1146/annurev.es.01.110170.000245 |jstor=2096764 |issn=1545-2069 |deadurl=no |archiveurl=https://web.archive.org/web/20150206172942/http://joelvelasco.net/teaching/167/lewontin%2070%20-%20the%20units%20of%20selection.pdf |archivedate=2015-02-06 |df= }}</ref> Thus, in successive generations members of a population are more likely to be replaced by the [[offspring|progenies]] of parents with favourable characteristics that have enabled them to survive and reproduce in their respective [[biophysical environment|environments]]. In the early 20th century, other [[Alternatives to evolution by natural selection|competing ideas of evolution]] such as [[mutationism]] and [[orthogenesis]] were [[Superseded scientific theories|refuted]] as the [[Modern synthesis (20th century)|modern synthesis]] reconciled [[Darwinism|Darwinian evolution]] with [[classical genetics]], which established [[Adaptation|adaptive evolution]] as being caused by natural selection acting on [[Mendelian inheritance|Mendelian]] genetic variation.<ref name="Futuyma2017a">{{cite book | last1=Futuyma | first1=Douglas J. | last2=Kirkpatrick | first2=Mark | year = 2017 | chapter = Evolutionary Biology | title=Evolution | pages = 3–26 | edition = Fourth | publisher = Sunderland, Massachusetts: Sinauer Associates, Inc | isbn=978-1-60535-605-1}}</ref> |
|||
All [[life]] on Earth shares a [[last universal common ancestor]] (LUCA)<ref name="Kampourakis2014">{{harvnb|Kampourakis|2014|pp=[https://books.google.com/books?id=RKroAgAAQBAJ&pg=PA127 127–129]}}</ref><ref name="Doolittle_2000">{{cite journal |last=Doolittle |first=W. Ford |authorlink=Ford Doolittle |date=February 2000 |title=Uprooting the Tree of Life |url=http://shiva.msu.montana.edu/courses/mb437_537_2004_fall/docs/uprooting.pdf |format=PDF |journal=[[Scientific American]] |issn=0036-8733 |volume=282 |issue=2 |pages=90–95 |doi=10.1038/scientificamerican0200-90 |pmid=10710791 |archiveurl=https://web.archive.org/web/20060907081933/http://shiva.msu.montana.edu/courses/mb437_537_2004_fall/docs/uprooting.pdf |archivedate=2006-09-07 |accessdate=2015-04-05|bibcode=2000SciAm.282b..90D }}</ref><ref>{{cite journal |last1=Glansdorff |first1=Nicolas |author2=Ying Xu |last3=Labedan |first3=Bernard |date=July 9, 2008 |title=The Last Universal Common Ancestor: emergence, constitution and genetic legacy of an elusive forerunner |journal=[[Biology Direct]] |volume=3 |page=29 |doi=10.1186/1745-6150-3-29 |issn=1745-6150 |pmc=2478661 |pmid=18613974}}</ref> that lived approximately 3.5–3.8 billion years ago.<ref name="Origin1" /> The [[fossil|fossil record]] includes a progression from early [[Biogenic substance|biogenic]] [[graphite]],<ref name="NG-20131208" /> to [[microbial mat]] fossils,<ref name="AP-20131113" /><ref name="TG-20131113-JP" /><ref name="AST-20131108" /> to fossilised [[multicellular organism]]s. Existing patterns of biodiversity have been shaped by repeated formations of new species ([[speciation]]), changes within species ([[anagenesis]]) and loss of species ([[extinction]]) throughout the [[evolutionary history of life]] on Earth.<ref name="Futuyma04">{{harvnb|Futuyma|2004|p=33}}</ref> [[morphology (biology)|Morphological]] and [[biochemistry|biochemical]] traits are more similar among species that share a more [[Most recent common ancestor|recent common ancestor]], and can be used to reconstruct [[phylogenetic tree]]s.<ref name="The Cell by Panno">{{harvnb|Panno|2005|pp=xv-16}}</ref><ref>[[#NAS 2008|NAS 2008]], [http://www.nap.edu/openbook.php?record_id=11876&page=17 p. 17] {{webarchive|url=https://web.archive.org/web/20150630042457/http://www.nap.edu/openbook.php?record_id=11876&page=17 |date=2015-06-30 }}</ref> |
|||
[[Evolutionary biology|Evolutionary biologists]] have continued to study various aspects of evolution by forming and testing [[Hypothesis|hypotheses]] as well as constructing theories based on [[empirical evidence|evidence]] from the field or laboratory and on data generated by the methods of [[mathematical and theoretical biology]]. Their discoveries have influenced not just the development of [[biology]] but numerous other scientific and industrial fields, including [[agriculture]], [[medicine]] and [[computer science]].<ref name="Futuyma99">{{cite web |url=http://www.rci.rutgers.edu/~ecolevol/fulldoc.pdf |title=Evolution, Science, and Society: Evolutionary Biology and the National Research Agenda |year=1999 |editor-last=Futuyma |editor-first=Douglas J. |editor-link=Douglas J. Futuyma |publisher=Office of University Publications, [[Rutgers University|Rutgers, The State University of New Jersey]] |location=New Brunswick, New Jersey |type=Executive summary |format=PDF |oclc=43422991 |archiveurl=https://web.archive.org/web/20120131174727/http://www.rci.rutgers.edu/~ecolevol/fulldoc.pdf |archivedate=2012-01-31 |accessdate=2014-11-24}}</ref> |
|||
== History of evolutionary thought == |
|||
[[File:Lucretius Rome.jpg|thumb|upright|[[Lucretius]]]] |
|||
[[File:Alfred-Russel-Wallace-c1895.jpg|thumb|upright|[[Alfred Russel Wallace]]]] |
|||
[[File:Thomas Robert Malthus Wellcome L0069037 -crop.jpg|thumb|upright|[[Thomas Robert Malthus]]]] |
|||
[[File:Charles Darwin aged 51.jpg|thumb|upright|In 1842, [[Charles Darwin]] penned his first sketch of ''[[On the Origin of Species]]''.<ref>{{harvnb|Darwin|1909|p=53}}</ref>]] |
|||
{{Main|History of evolutionary thought}} |
|||
{{further|History of speciation}} |
|||
=== Classical times === |
|||
The proposal that one type of organism could descend from another type goes back to some of the first [[pre-Socratic philosophy|pre-Socratic]] Greek [[philosopher]]s, such as [[Anaximander#Origin of humankind|Anaximander]] and [[Empedocles#Cosmogony|Empedocles]].<ref>{{harvnb|Kirk|Raven|Schofield|1983|pp=100–142, 280–321}}</ref> Such proposals survived into Roman times. The [[poet]] and philosopher [[Lucretius]] followed Empedocles in his masterwork ''[[De rerum natura]]'' (''On the Nature of Things'').<ref>{{harvnb|Lucretius}}</ref><ref>{{cite journal|last=Sedley |first=David |authorlink=David Sedley |year=2003 |title=Lucretius and the New Empedocles |url=http://lics.leeds.ac.uk/2003/200304.pdf |format=PDF |journal=Leeds International Classical Studies |volume=2 |issue=4 |issn=1477-3643 |accessdate=2014-11-25 |deadurl=yes |archiveurl=https://web.archive.org/web/20140823062637/http://lics.leeds.ac.uk/2003/200304.pdf |archivedate=2014-08-23 |df= }}</ref> |
|||
=== Medieval === |
|||
In contrast to these [[Materialism|materialistic]] views, [[Aristotelianism]] considered all natural things as [[potentiality and actuality|actualisations]] of fixed natural possibilities, known as [[Theory of forms|forms]].<ref name="Torrey37">{{cite journal |last1=Torrey |first1=Harry Beal |last2=Felin |first2=Frances |date=March 1937 |title=Was Aristotle an Evolutionist? |journal=[[The Quarterly Review of Biology]] |volume=12 |issue=1 |pages=1–18 |doi=10.1086/394520 |issn=0033-5770 |jstor=2808399}}</ref><ref name="Hull67">{{cite journal |last=Hull |first=David L. |authorlink=David Hull |date=December 1967 |title=The Metaphysics of Evolution |journal=[[The British Journal for the History of Science]] |volume=3 |issue=4 |pages=309–337 |doi=10.1017/S0007087400002892 |jstor=4024958}}</ref> This was part of a medieval [[teleology|teleological]] understanding of [[Nature (philosophy)|nature]] in which all things have an intended role to play in a [[divinity|divine]] [[cosmos|cosmic]] order. Variations of this idea became the standard understanding of the [[Middle Ages]] and were integrated into [[Christianity|Christian]] learning, but Aristotle did not demand that real types of organisms always correspond one-for-one with exact metaphysical forms and specifically gave examples of how new types of living things could come to be.<ref>{{harvnb|Mason|1962|pp=43–44}}</ref> |
|||
=== Pre-Darwinian === |
|||
In the 17th century, the new [[scientific method|method]] of [[History of science#Modern science|modern science]] rejected the Aristotelian approach. It sought explanations of natural phenomena in terms of [[physical law]]s that were the same for all visible things and that did not require the existence of any fixed natural categories or divine cosmic order. However, this new approach was slow to take root in the biological sciences, the last bastion of the concept of fixed natural types. [[John Ray]] applied one of the previously more general terms for fixed natural types, "species," to plant and animal types, but he strictly identified each type of living thing as a species and proposed that each species could be defined by the features that perpetuated themselves generation after generation.<ref>{{harvnb|Mayr|1982|pp=256–257}} |
|||
* {{harvnb|Ray|1686}}</ref> The biological classification introduced by [[Carl Linnaeus]] in 1735 explicitly recognised the hierarchical nature of species relationships, but still viewed species as fixed according to a divine plan.<ref>{{cite web |url=http://www.ucmp.berkeley.edu/history/linnaeus.html |title=Carl Linnaeus (1707-1778) |last=Waggoner |first=Ben |date=July 7, 2000 |website=Evolution |publisher=[[University of California Museum of Paleontology]] |location=Berkeley, California |type=Online exhibit |accessdate=2012-02-11 |deadurl=no |archiveurl=https://web.archive.org/web/20110430160025/http://www.ucmp.berkeley.edu/history/linnaeus.html |archivedate=2011-04-30 |df= }}</ref> |
|||
Other [[Natural history|naturalists]] of this time speculated on the evolutionary change of species over time according to natural laws. In 1751, [[Pierre Louis Maupertuis]] wrote of natural modifications occurring during reproduction and accumulating over many generations to produce new species.<ref>{{harvnb|Bowler|2003|pp=73–75}}</ref> [[Georges-Louis Leclerc, Comte de Buffon]] suggested that species could degenerate into different organisms, and [[Erasmus Darwin]] proposed that all warm-blooded animals could have descended from a single microorganism (or "filament").<ref>{{cite web |url=http://www.ucmp.berkeley.edu/history/Edarwin.html |title=Erasmus Darwin (1731-1802) |date=October 4, 1995 |website=Evolution |publisher=University of California Museum of Paleontology |location=Berkeley, California |type=Online exhibit |accessdate=2012-02-11 |deadurl=no |archiveurl=https://web.archive.org/web/20120119004316/http://www.ucmp.berkeley.edu/history/Edarwin.html |archivedate=2012-01-19 |df= }}</ref> The first full-fledged evolutionary scheme was [[Jean-Baptiste Lamarck]]'s "transmutation" theory of 1809,<ref>{{harvnb|Lamarck|1809}}</ref> which envisaged [[spontaneous generation]] continually producing simple forms of life that developed greater complexity in parallel lineages with an inherent progressive tendency, and postulated that on a local level, these lineages adapted to the environment by inheriting changes caused by their use or disuse in parents.<ref name="Nardon_Grenier91">{{harvnb|Nardon|Grenier|1991|p=162}}</ref><ref name="Gould02">{{harvnb|Gould|2002}}{{page needed|date=December 2013}}</ref> (The latter process was later called [[Lamarckism]].)<ref name="Nardon_Grenier91" /><ref name="ImaginaryLamarck">{{cite journal |last=Ghiselin |first=Michael T. |authorlink=Michael Ghiselin |date=September–October 1994 |title=The Imaginary Lamarck: A Look at Bogus 'History' in Schoolbooks |url=http://www.textbookleague.org/54marck.htm |journal=The Textbook Letter |oclc=23228649 |accessdate=2008-01-23 |deadurl=no |archiveurl=https://web.archive.org/web/20080212174536/http://www.textbookleague.org/54marck.htm |archivedate=2008-02-12 |df= }}</ref><ref>{{harvnb|Magner|2002}}{{page needed|date=December 2013}}</ref><ref name="Jablonka07">{{cite journal |last1=Jablonka |first1=Eva |authorlink1=Eva Jablonka |last2=Lamb |first2=Marion J. |authorlink2=Marion J. Lamb |date=August 2007 |title=Précis of Evolution in Four Dimensions |journal=[[Behavioral and Brain Sciences]] |volume=30 |issue=4 |pages=353–365 |doi=10.1017/S0140525X07002221 |pmid=18081952 |issn=0140-525X}}</ref> These ideas were condemned by established naturalists as speculation lacking empirical support. In particular, [[Georges Cuvier]] insisted that species were unrelated and fixed, their similarities reflecting divine design for functional needs. In the meantime, Ray's ideas of benevolent design had been developed by [[William Paley]] into the ''[[Natural Theology or Evidences of the Existence and Attributes of the Deity]]'' (1802), which proposed complex adaptations as evidence of divine design and which was admired by Charles Darwin.<ref name="Darwin91">{{harvnb|Burkhardt|Smith|1991}} |
|||
* {{cite web |url=http://www.darwinproject.ac.uk/letter/entry-2532 |title=Darwin, C. R. to Lubbock, John |website=[[Correspondence of Charles Darwin#Darwin Correspondence Project website|Darwin Correspondence Project]] |publisher=[[University of Cambridge]] |location=Cambridge, UK |accessdate=2014-12-01 |deadurl=no |archiveurl=https://web.archive.org/web/20141215213940/http://www.darwinproject.ac.uk/letter/entry-2532 |archivedate=2014-12-15 |df= }} Letter 2532, November 22, 1859.</ref><ref name="Sulloway09">{{cite journal |last=Sulloway |first=Frank J. |authorlink=Frank Sulloway |date=June 2009 |title=Why Darwin rejected intelligent design |journal=[[Journal of Biosciences]] |volume=34 |issue=2 |pages=173–183 |doi=10.1007/s12038-009-0020-8 |issn=0250-5991 |pmid=19550032}}</ref><ref name="Dawkins90">{{harvnb|Dawkins|1990}}{{page needed|date=December 2014}}</ref> |
|||
=== Darwinian revolution === |
|||
The crucial break from the concept of constant typological classes or types in biology came with the theory of evolution through natural selection, which was formulated by [[Charles Darwin]] in terms of variable populations. Partly influenced by ''[[An Essay on the Principle of Population]]'' (1798) by [[Thomas Robert Malthus]], Darwin noted that population growth would lead to a "struggle for existence" in which favourable variations prevailed as others perished. In each generation, many offspring fail to survive to an age of reproduction because of limited resources. This could explain the diversity of plants and animals from a common ancestry through the working of natural laws in the same way for all types of organism.<ref name="Sober09">{{cite journal |last=Sober |first=Elliott |authorlink=Elliott Sober |date=June 16, 2009 |title=Did Darwin write the ''Origin'' backwards? |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=106 |issue=Suppl. 1 |pages=10048–10055 |bibcode=2009PNAS..10610048S |doi=10.1073/pnas.0901109106 |issn=0027-8424 |pmid=19528655 |pmc=2702806}}</ref><ref>{{harvnb|Mayr|2002|p=165}}</ref><ref>{{harvnb|Bowler|2003|pp=145–146}}</ref><ref>{{cite journal |last1=Sokal |first1=Robert R. |authorlink1=Robert R. Sokal |last2=Crovello |first2=Theodore J. |date=March–April 1970 |title=The Biological Species Concept: A Critical Evaluation |journal=[[The American Naturalist]] |volume=104 |issue=936 |pages=127–153 |doi=10.1086/282646 |issn=0003-0147 |jstor=2459191}}</ref> Darwin developed his theory of "natural selection" from 1838 onwards and was writing up his "big book" on the subject when [[Alfred Russel Wallace]] sent him a version of virtually the same theory in 1858. Their [[On the Tendency of Species to form Varieties; and on the Perpetuation of Varieties and Species by Natural Means of Selection|separate papers]] were presented together at an 1858 meeting of the [[Linnean Society of London]].<ref>{{cite journal |last1=Darwin |first1=Charles |authorlink1=Charles Darwin |last2=Wallace |first2=Alfred |authorlink2=Alfred Russel Wallace |date=August 20, 1858 |title=On the Tendency of Species to form Varieties; and on the Perpetuation of Varieties and Species by Natural Means of Selection |url=http://darwin-online.org.uk/content/frameset?itemID=F350&viewtype=text&pageseq=1 |journal=[[Zoological Journal of the Linnean Society|Journal of the Proceedings of the Linnean Society of London. Zoology]] |volume=3 |issue=9 |pages=45–62 |doi=10.1111/j.1096-3642.1858.tb02500.x |issn=1096-3642 |accessdate=2007-05-13 |deadurl=no |archiveurl=https://web.archive.org/web/20070714042318/http://darwin-online.org.uk/content/frameset?itemID=F350&viewtype=text&pageseq=1 |archivedate=2007-07-14 |df= }}</ref> At the end of 1859, Darwin's publication of his "abstract" as ''On the Origin of Species'' explained natural selection in detail and in a way that led to an increasingly wide acceptance of [[Darwinism|Darwin's concepts of evolution]] at the expense of [[Alternatives to evolution by natural selection|alternative theories]]. [[Thomas Henry Huxley]] applied Darwin's ideas to humans, using [[paleontology]] and [[comparative anatomy]] to provide strong evidence that humans and [[ape]]s shared a common ancestry. Some were disturbed by this since it implied that humans did not have a special place in the [[universe]].<ref>{{cite encyclopedia |last=Desmond |first=Adrian J. |authorlink=Adrian Desmond |encyclopedia=[[Encyclopædia Britannica Online]] |url=http://www.britannica.com/EBchecked/topic/277746/Thomas-Henry-Huxley |title=Thomas Henry Huxley |accessdate=2014-12-02 |date=July 17, 2014 |publisher=[[Encyclopædia Britannica, Inc.]] |location=Chicago, Illinois |deadurl=no |archiveurl=https://web.archive.org/web/20150119231241/http://www.britannica.com/EBchecked/topic/277746/Thomas-Henry-Huxley |archivedate=January 19, 2015 |df= }}</ref> |
|||
=== Pangenesis and heredity === |
|||
The mechanisms of reproductive heritability and the origin of new traits remained a mystery. Towards this end, Darwin developed his provisional theory of [[pangenesis]].<ref name="Liu09">{{cite journal |last1=Liu |first1=Y. S. |last2=Zhou |first2=X. M. |last3=Zhi |first3=M. X. |last4=Li |first4=X. J. |last5=Wang |first5=Q. L. |date=September 2009 |title=Darwin's contributions to genetics |journal=Journal of Applied Genetics |volume=50 |issue=3 |pages=177–184 |doi=10.1007/BF03195671 |issn=1234-1983 |pmid=19638672}}</ref> In 1865, [[Gregor Mendel]] reported that traits were inherited in a predictable manner through the [[Mendelian inheritance#Law of Independent Assortment (the "Second Law")|independent assortment]] and segregation of elements (later known as genes). Mendel's laws of inheritance eventually supplanted most of Darwin's pangenesis theory.<ref name="Weiling">{{cite journal |last=Weiling |first=Franz |date=July 1991 |title=Historical study: Johann Gregor Mendel 1822–1884 |journal=[[American Journal of Medical Genetics]] |volume=40 |issue=1 |pages=1–25; discussion 26 |doi=10.1002/ajmg.1320400103 |pmid=1887835}}</ref> [[August Weismann]] made the important distinction between [[germ cell]]s that give rise to [[gamete]]s (such as [[sperm]] and [[egg cell]]s) and the [[somatic cell]]s of the body, demonstrating that heredity passes through the germ line only. [[Hugo de Vries]] connected Darwin's pangenesis theory to Weismann's germ/soma cell distinction and proposed that Darwin's pangenes were concentrated in the [[cell nucleus]] and when expressed they could move into the [[cytoplasm]] to change the [[Cell (biology)|cell]]'s structure. De Vries was also one of the researchers who made Mendel's work well known, believing that Mendelian traits corresponded to the transfer of heritable variations along the germline.<ref name="Wright84">{{harvnb|Wright|1984|p=480}}</ref> To explain how new variants originate, de Vries developed [[Mutationism|a mutation theory]] that led to a temporary rift between those who accepted Darwinian evolution and biometricians who allied with de Vries.<ref name="Gould02" /><ref>{{harvnb|Provine|1971}}</ref><ref>{{cite journal |last1=Stamhuis |first1=Ida H. |last2=Meijer |first2=Onno G. |last3=Zevenhuizen |first3=Erik J. A. |date=June 1999 |title=Hugo de Vries on Heredity, 1889-1903: Statistics, Mendelian Laws, Pangenes, Mutations |volume=90 |issue=2 |pages=238–267 |journal=[[Isis (journal)|Isis]] |doi=10.1086/384323 |jstor=237050 |pmid=10439561}}</ref> In the 1930s, pioneers in the field of population genetics, such as [[Ronald Fisher]], [[Sewall Wright]] and [[J. B. S. Haldane]] set the foundations of evolution onto a robust statistical philosophy. The false contradiction between Darwin's theory, genetic mutations, and [[Mendelian inheritance]] was thus reconciled.<ref>{{harvnb|Quammen|2006}}{{page needed|date=December 2014}}</ref> |
|||
=== The 'modern synthesis' === |
|||
{{main|Modern synthesis (20th century)}} |
|||
In the 1920s and 1930s the so-called [[Modern synthesis (20th century)|modern synthesis]] connected natural selection and population genetics, based on Mendelian inheritance, into a unified theory that applied generally to any branch of biology. The modern synthesis explained patterns observed across species in populations, through [[Transitional fossil|fossil transitions]] in palaeontology, and complex cellular mechanisms in [[developmental biology]].<ref name="Gould02" /><ref>{{harvnb|Bowler|1989}}{{page needed|date=December 2013}}</ref> The publication of the structure of [[DNA]] by [[James Watson]] and [[Francis Crick]] with contribution of [[Rosalind Franklin]] in 1953 demonstrated a physical mechanism for inheritance.<ref name="Watson53">{{cite journal |last1=Watson |first1=J. D. |authorlink1=James Watson |last2=Crick |first2=F. H. C. |authorlink2=Francis Crick |date=April 25, 1953 |title=Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid |url=http://profiles.nlm.nih.gov/SC/B/B/Y/W/_/scbbyw.pdf |format=PDF |journal=[[Nature (journal)|Nature]] |volume=171 |issue=4356 |pages=737–738 |bibcode=1953Natur.171..737W |doi=10.1038/171737a0 |issn=0028-0836 |pmid=13054692 |accessdate=2014-12-04 |quote=It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material. |deadurl=no |archiveurl=https://web.archive.org/web/20140823063212/http://profiles.nlm.nih.gov/SC/B/B/Y/W/_/scbbyw.pdf |archivedate=2014-08-23 |df= }}</ref> [[Molecular biology]] improved understanding of the relationship between [[genotype]] and [[phenotype]]. Advancements were also made in phylogenetic [[systematics]], mapping the transition of traits into a comparative and testable framework through the publication and use of [[Phylogenetic tree|evolutionary trees]].<ref name="Hennig99">{{harvnb|Hennig|1999|p=280}}</ref><ref name="Wiley11">{{harvnb|Wiley|Lieberman|2011}}{{page needed|date=December 2013}}</ref> In 1973, evolutionary biologist [[Theodosius Dobzhansky]] penned that "[[Nothing in Biology Makes Sense Except in the Light of Evolution|nothing in biology makes sense except in the light of evolution]]," because it has brought to light the relations of what first seemed disjointed facts in natural history into a coherent [[Explanation|explanatory]] body of knowledge that describes and predicts many observable facts about life on this planet.<ref name="Dobzhansky73">{{cite journal|last=Dobzhansky|first=Theodosius|authorlink=Theodosius Dobzhansky|date=March 1973|title=Nothing in Biology Makes Sense Except in the Light of Evolution|url=http://www.phil.vt.edu/Burian/NothingInBiolChFina.pdf|journal=The American Biology Teacher|volume=35|issue=3|pages=125–129|doi=10.2307/4444260|via=|deadurl=no|archiveurl=https://web.archive.org/web/20151023161423/http://www.phil.vt.edu/Burian/NothingInBiolChFina.pdf|archivedate=2015-10-23|df=|jstor=4444260|citeseerx=10.1.1.324.2891}}</ref> |
|||
=== Further syntheses === |
|||
Since then, the modern synthesis has been further extended to explain biological phenomena across the full and integrative scale of the [[Biological organisation|biological hierarchy]], from genes to species. One extension, known as [[evolutionary developmental biology]] and informally called "evo-devo," emphasises how changes between generations (evolution) acts on patterns of change within individual organisms ([[Developmental biology|development]]).<ref name="Kutschera">{{cite journal |last1=Kutschera |first1=Ulrich |authorlink1=Ulrich Kutschera |last2=Niklas |first2=Karl J. |authorlink2=Karl J. Niklas |date=June 2004 |title=The modern theory of biological evolution: an expanded synthesis |journal=[[Naturwissenschaften]] |volume=91 |issue=6 |pages=255–276 |bibcode=2004NW.....91..255K |doi=10.1007/s00114-004-0515-y |issn=1432-1904 |pmid=15241603}}</ref><ref>{{harvnb|Cracraft|Bybee|2005}}{{page needed|date=December 2014}}</ref><ref name="Avise10">{{cite journal |last1=Avise |first1=John C. |authorlink1=John Avise |last2=Ayala |first2=Francisco J. |authorlink2=Francisco J. Ayala |date=May 11, 2010 |title=In the light of evolution IV: The human condition |url=http://faculty.sites.uci.edu/johncavise/files/2011/03/311-intro-to-ILE-IV.pdf |format=PDF |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=107 |issue=Suppl. 2 |pages=8897–8901 |doi=10.1073/pnas.1003214107 |pmid=20460311 |pmc=3024015 |issn=0027-8424 |accessdate=2014-12-29 |deadurl=no |archiveurl=https://web.archive.org/web/20140823063532/http://faculty.sites.uci.edu/johncavise/files/2011/03/311-intro-to-ILE-IV.pdf |archivedate=2014-08-23 |df= }}</ref> Since the beginning of the 21st century and in light of discoveries made in recent decades, some biologists have argued for an [[extended evolutionary synthesis]], which would account for the effects of non-genetic inheritance modes, such as [[epigenetics]], [[Maternal effect|parental effects]], ecological inheritance and [[Dual inheritance theory|cultural inheritance]], and [[evolvability]].<ref name="beyonddna">{{cite journal |last1=Danchin |first1=Étienne |last2=Charmantier |first2=Anne |last3=Champagne |first3=Frances A. |author-link3=Frances Champagne |last4=Mesoudi |first4=Alex |last5=Pujol |first5=Benoit |last6=Blanchet |first6=Simon |date=June 2011 |title=Beyond DNA: integrating inclusive inheritance into an extended theory of evolution |url=http://www.nature.com/nrg/journal/v12/n7/full/nrg3028.html |journal=[[Nature Reviews Genetics]] |volume=12 |issue=7 |pages=475–486 |doi=10.1038/nrg3028 |issn=1471-0056 |pmid=21681209}}</ref><ref name="eesbook">{{harvnb|Pigliucci|Müller|2010}}</ref> |
|||
== Heredity == |
|||
{{Further|Introduction to genetics|Genetics|Heredity|Reaction norm}} |
|||
[[File:ADN static.png|thumb|upright|[[DNA]] structure. [[nucleobase|Bases]] are in the centre, surrounded by phosphate–sugar chains in a [[Nucleic acid double helix|double helix]].]] |
|||
Evolution in organisms occurs through changes in heritable traits—the inherited characteristics of an organism. In humans, for example, [[Eye color|eye colour]] is an inherited characteristic and an individual might inherit the "brown-eye trait" from one of their parents.<ref>{{cite journal |last1=Sturm |first1=Richard A. |last2=Frudakis |first2=Tony N. |date=August 2004 |title=Eye colour: portals into pigmentation genes and ancestry |journal=[[Trends (journals)|Trends in Genetics]] |volume=20 |issue=8 |pages=327–332 |doi=10.1016/j.tig.2004.06.010 |issn=0168-9525 |pmid=15262401}}</ref> Inherited traits are controlled by genes and the complete set of genes within an organism's [[genome]] (genetic material) is called its genotype.<ref name="Pearson_2006">{{cite journal |last=Pearson |first=Helen |date=May 25, 2006 |title=Genetics: What is a gene? |journal=Nature |volume=441 |issue=7092 |pages=398–401 |bibcode=2006Natur.441..398P |doi=10.1038/441398a |issn=0028-0836 |pmid=16724031}}</ref> |
|||
The complete set of observable traits that make up the structure and behaviour of an organism is called its phenotype. These traits come from the interaction of its genotype with the environment.<ref>{{cite journal |last1=Visscher |first1=Peter M. |last2=Hill |first2=William G. |authorlink2=William G. Hill |last3=Wray |first3=Naomi R. |date=April 2008 |title=Heritability in the genomics era — concepts and misconceptions |journal=Nature Reviews Genetics |volume=9 |issue=4 |pages=255–266 |doi=10.1038/nrg2322 |issn=1471-0056 |pmid=18319743}}</ref> As a result, many aspects of an organism's phenotype are not inherited. For example, [[sun tanning|suntanned]] skin comes from the interaction between a person's genotype and sunlight; thus, suntans are not passed on to people's children. However, some people tan more easily than others, due to differences in genotypic variation; a striking example are people with the inherited trait of [[albinism]], who do not tan at all and are very sensitive to [[sunburn]].<ref>{{cite journal |last1=Oetting |first1=William S. |last2=Brilliant |first2=Murray H. |last3=King |first3=Richard A. |date=August 1996 |title=The clinical spectrum of albinism in humans |journal=[[Trends (journals)|Molecular Medicine Today]] |volume=2 |issue=8 |pages=330–335 |doi=10.1016/1357-4310(96)81798-9 |issn=1357-4310 |pmid=8796918}}</ref> |
|||
Heritable traits are passed from one generation to the next via DNA, a [[molecule]] that encodes genetic information.<ref name="Pearson_2006" /> DNA is a long [[biopolymer]] composed of four types of bases. The sequence of bases along a particular DNA molecule specify the genetic information, in a manner similar to a sequence of letters spelling out a sentence. Before a cell divides, the DNA is copied, so that each of the resulting two cells will inherit the DNA sequence. Portions of a DNA molecule that specify a single functional unit are called genes; different genes have different sequences of bases. Within cells, the long strands of DNA form condensed structures called [[chromosome]]s. The specific location of a DNA sequence within a chromosome is known as a [[locus (genetics)|locus]]. If the DNA sequence at a locus varies between individuals, the different forms of this sequence are called alleles. DNA sequences can change through mutations, producing new alleles. If a mutation occurs within a gene, the new allele may affect the trait that the gene controls, altering the phenotype of the organism.<ref name="Futuyma_2005">{{harvnb|Futuyma|2005}}{{page needed|date=December 2014}}</ref> However, while this simple correspondence between an allele and a trait works in some cases, most traits are more complex and are controlled by [[quantitative trait locus|quantitative trait loci]] (multiple interacting genes).<ref>{{cite journal |last=Phillips |first=Patrick C. |date=November 2008 |title=Epistasis—the essential role of gene interactions in the structure and evolution of genetic systems |journal=Nature Reviews Genetics |volume=9 |issue=11 |pages=855–867 |doi=10.1038/nrg2452 |issn=1471-0056 |pmc=2689140 |pmid=18852697}}</ref><ref name="Lin">{{cite journal |author1=Rongling Wu |author2=Min Lin |date=March 2006 |title=Functional mapping — how to map and study the genetic architecture of dynamic complex traits |journal=Nature Reviews Genetics |volume=7 |issue=3 |pages=229–237 |doi=10.1038/nrg1804 |issn=1471-0056 |pmid=16485021}}</ref> |
|||
Recent findings have confirmed important examples of heritable changes that cannot be explained by changes to the sequence of [[nucleotide]]s in the DNA. These phenomena are classed as [[Epigenetics|epigenetic]] inheritance systems.<ref name="Jablonka09">{{cite journal |last1=Jablonka |first1=Eva |last2=Raz |first2=Gal |date=June 2009 |title=Transgenerational Epigenetic Inheritance: Prevalence, Mechanisms, and Implications for the Study of Heredity and Evolution |journal=The Quarterly Review of Biology |volume=84 |issue=2 |pages=131–176 |doi=10.1086/598822 |issn=0033-5770 |pmid=19606595|url=http://compgen.unc.edu/wiki/images/d/df/JablonkaQtrRevBio2009.pdf |citeseerx=10.1.1.617.6333 }}</ref> [[DNA methylation]] marking [[chromatin]], self-sustaining metabolic loops, gene silencing by [[RNA interference]] and the three-dimensional [[Protein structure|conformation]] of [[protein]]s (such as [[prion]]s) are areas where epigenetic inheritance systems have been discovered at the organismic level.<ref name="Bossdorf10">{{cite journal |last1=Bossdorf |first1=Oliver |last2=Arcuri |first2=Davide |last3=Richards |first3=Christina L. |last4=Pigliucci |first4=Massimo |authorlink4=Massimo Pigliucci |date=May 2010 |title=Experimental alteration of DNA methylation affects the phenotypic plasticity of ecologically relevant traits in ''Arabidopsis thaliana'' |journal=Evolutionary Ecology |volume=24 |issue=3 |pages=541–553 |doi=10.1007/s10682-010-9372-7 |issn=0269-7653|url=http://doc.rero.ch/record/318722/files/10682_2010_Article_9372.pdf |type=Submitted manuscript }}</ref><ref>{{harvnb|Jablonka|Lamb|2005}}{{page needed|date=December 2014}}</ref> Developmental biologists suggest that complex interactions in [[gene regulatory network|genetic networks]] and communication among cells can lead to heritable variations that may underlay some of the mechanics in [[developmental plasticity]] and [[Canalisation (genetics)|canalisation]].<ref name="Jablonka02">{{cite journal |last1=Jablonka |first1=Eva |last2=Lamb |first2=Marion J. |date=December 2002 |title=The Changing Concept of Epigenetics |journal=[[Annals of the New York Academy of Sciences]] |volume=981 |issue=1 |pages=82–96 |bibcode=2002NYASA.981...82J |doi=10.1111/j.1749-6632.2002.tb04913.x |issn=0077-8923 |pmid=12547675}}</ref> Heritability may also occur at even larger scales. For example, ecological inheritance through the process of [[niche construction]] is defined by the regular and repeated activities of organisms in their environment. This generates a legacy of effects that modify and feed back into the selection regime of subsequent generations. Descendants inherit genes plus environmental characteristics generated by the ecological actions of ancestors.<ref name="Laland06">{{cite journal |last1=Laland |first1=Kevin N. |last2=Sterelny |first2=Kim |authorlink2=Kim Sterelny |date=September 2006 |title=Perspective: Seven Reasons (Not) to Neglect Niche Construction |journal=[[Evolution (journal)|Evolution]] |volume=60 |issue=9 |pages=1751–1762 |doi=10.1111/j.0014-3820.2006.tb00520.x |issn=0014-3820}}</ref> Other examples of heritability in evolution that are not under the direct control of genes include the inheritance of [[Dual inheritance theory|cultural traits]] and [[symbiogenesis]].<ref name="Chapman98">{{cite journal|last1=Chapman |first1=Michael J. |last2=Margulis |first2=Lynn |authorlink2=Lynn Margulis |date=December 1998 |title=Morphogenesis by symbiogenesis |url=http://www.im.microbios.org/04december98/14%20Chapman.pdf |format=PDF |journal=[[International Microbiology]] |volume=1 |issue=4 |pages=319–326 |issn=1139-6709 |pmid=10943381 |accessdate=2014-12-09 |deadurl=yes |archiveurl=https://web.archive.org/web/20140823062546/http://www.im.microbios.org/04december98/14%20Chapman.pdf |archivedate=2014-08-23 |df= }}</ref><ref name="Wilson07">{{cite journal |last1=Wilson |first1=David Sloan |authorlink1=David Sloan Wilson |last2=Wilson |first2=Edward O. |authorlink2=E. O. Wilson |date=December 2007 |title=Rethinking the Theoretical Foundation of Sociobiology |url=http://evolution.binghamton.edu/dswilson/wp-content/uploads/2010/01/Rethinking-sociobiology.pdf |format=PDF |journal=The Quarterly Review of Biology |volume=82 |issue=4 |pages=327–348 |doi=10.1086/522809 |issn=0033-5770 |pmid=18217526 |deadurl=yes |archiveurl=https://web.archive.org/web/20110511235639/http://evolution.binghamton.edu/dswilson/wp-content/uploads/2010/01/Rethinking-sociobiology.pdf |archivedate=2011-05-11 |df= }}</ref> |
|||
== Variation == |
|||
{{Multiple image|direction=vertical|align=right|image1=Biston.betularia.7200.jpg |image2=Biston.betularia.f.carbonaria.7209.jpg|width=200|caption1=White [[peppered moth]] |caption2=Black morph in [[peppered moth evolution]]}} |
|||
{{main|Genetic variation}} |
|||
{{Further|Genetic diversity|Population genetics}} |
|||
An individual organism's phenotype results from both its genotype and the influence from the environment it has lived in. A substantial part of the phenotypic variation in a population is caused by genotypic variation.<ref name="Lin" /> The modern evolutionary synthesis defines evolution as the change over time in this genetic variation. The frequency of one particular allele will become more or less prevalent relative to other forms of that gene. Variation disappears when a new allele reaches the point of [[fixation (population genetics)|fixation]]—when it either disappears from the population or replaces the ancestral allele entirely.<ref name="Amos">{{cite journal |last1=Amos |first1=William |last2=Harwood |first2=John |date=February 28, 1998 |title=Factors affecting levels of genetic diversity in natural populations |journal=[[Philosophical Transactions of the Royal Society B|Philosophical Transactions of the Royal Society B: Biological Sciences]] |volume=353 |issue=1366 |pages=177–186 |doi=10.1098/rstb.1998.0200 |issn=0962-8436 |pmc=1692205 |pmid=9533122}}</ref> |
|||
Natural selection will only cause evolution if there is enough [[genetic variation]] in a population. Before the discovery of Mendelian genetics, one common hypothesis was [[blending inheritance]]. But with blending inheritance, genetic variance would be rapidly lost, making evolution by natural selection implausible. The [[Hardy–Weinberg principle]] provides the solution to how variation is maintained in a population with Mendelian inheritance. The frequencies of alleles (variations in a gene) will remain constant in the absence of selection, mutation, migration and genetic drift.<ref name="Ewens W.J. 2004">{{harvnb|Ewens|2004}}{{page needed|date=December 2014}}</ref> |
|||
Variation comes from mutations in the genome, reshuffling of genes through [[sexual reproduction]] and migration between populations ([[gene flow]]). Despite the constant introduction of new variation through mutation and gene flow, most of the genome of a species is identical in all individuals of that species.<ref>{{cite journal |last1=Butlin |first1=Roger K. |last2=Tregenza |first2=Tom |date=February 28, 1998 |title=Levels of genetic polymorphism: marker loci versus quantitative traits |journal=Philosophical Transactions of the Royal Society B: Biological Sciences |volume=353 |issue=1366 |pages=187–198 |doi=10.1098/rstb.1998.0201 |issn=0962-8436 |pmc=1692210 |pmid=9533123}} |
|||
* {{cite journal |last1=Butlin |first1=Roger K. |last2=Tregenza |first2=Tom |date=December 29, 2000 |title=Correction for Butlin and Tregenza, Levels of genetic polymorphism: marker loci versus quantitative traits |journal=Philosophical Transactions of the Royal Society B: Biological Sciences |volume=355 |issue=1404 |doi=10.1098/rstb.2000.2000 |issn=0962-8436 |quote=Some of the values in table 1 on p. 193 were given incorrectly. The errors do not affect the conclusions drawn in the paper. The corrected table is reproduced below. |pages=1865}}</ref> However, even relatively small differences in genotype can lead to dramatic differences in phenotype: for example, chimpanzees and humans differ in only about 5% of their genomes.<ref>{{cite journal |last1=Wetterbom |first1=Anna |last2=Sevov |first2=Marie |last3=Cavelier |first3=Lucia |last4=Bergström |first4=Tomas F. |date=November 2006 |title=Comparative Genomic Analysis of Human and Chimpanzee Indicates a Key Role for Indels in Primate Evolution |journal=[[Journal of Molecular Evolution]] |volume=63 |issue=5 |pages=682–690 |doi=10.1007/s00239-006-0045-7 |issn=0022-2844 |pmid=17075697|bibcode=2006JMolE..63..682W }}</ref> |
|||
=== Mutation === |
|||
{{Main|Mutation}} |
|||
[[File:Gene-duplication.svg|thumb|upright|Duplication of part of a [[chromosome]]]] |
|||
Mutations are changes in the DNA sequence of a cell's genome. When mutations occur, they may alter the [[gene product|product of a gene]], or prevent the gene from functioning, or have no effect. Based on studies in the fly ''[[Drosophila melanogaster]]'', it has been suggested that if a mutation changes a protein produced by a gene, this will probably be harmful, with about 70% of these mutations having damaging effects, and the remainder being either neutral or weakly beneficial.<ref>{{cite journal |last1=Sawyer |first1=Stanley A. |last2=Parsch |first2=John |author3=Zhang Zhi |last4=Hartl |first4=Daniel L. |date=April 17, 2007 |title=Prevalence of positive selection among nearly neutral amino acid replacements in ''Drosophila'' |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=104 |issue=16 |pages=6504–6510 |bibcode=2007PNAS..104.6504S |doi=10.1073/pnas.0701572104 |issn=0027-8424 |pmc=1871816 |pmid=17409186}}</ref> |
|||
Mutations can involve large sections of a chromosome becoming [[gene duplication|duplicated]] (usually by [[genetic recombination]]), which can introduce extra copies of a gene into a genome.<ref>{{cite journal |last1=Hastings |first1=P. J. |last2=Lupski |first2=James R. |authorlink2=James R. Lupski |last3=Rosenberg |first3=Susan M. |last4=Ira |first4=Grzegorz |date=August 2009 |title=Mechanisms of change in gene copy number |journal=Nature Reviews Genetics |volume=10 |issue=8 |pages=551–564 |doi=10.1038/nrg2593 |issn=1471-0056 |pmc=2864001 |pmid=19597530}}</ref> Extra copies of genes are a major source of the raw material needed for new genes to evolve.<ref>{{harvnb|Carroll|Grenier|Weatherbee|2005}}{{page needed|date=December 2014}}</ref> This is important because most new genes evolve within [[gene family|gene families]] from pre-existing genes that share common ancestors.<ref>{{cite journal |last1=Harrison |first1=Paul M. |last2=Gerstein |first2=Mark |authorlink2=Mark Bender Gerstein |date=May 17, 2002 |title=Studying Genomes Through the Aeons: Protein Families, Pseudogenes and Proteome Evolution |journal=[[Journal of Molecular Biology]] |volume=318 |issue=5 |pages=1155–1174 |doi=10.1016/S0022-2836(02)00109-2 |issn=0022-2836 |pmid=12083509}}</ref> For example, the human [[eye]] uses four genes to make structures that sense light: three for [[Cone cell|colour vision]] and one for [[Rod cell|night vision]]; all four are descended from a single ancestral gene.<ref>{{cite journal |last=Bowmaker |first=James K. |title=Evolution of colour vision in vertebrates |date=May 1998 |journal=Eye |volume=12 |issue=3b |pages=541–547 |doi=10.1038/eye.1998.143 |issn=0950-222X |pmid=9775215}}</ref> |
|||
New genes can be generated from an ancestral gene when a duplicate copy mutates and acquires a new function. This process is easier once a gene has been duplicated because it increases the [[Gene redundancy|redundancy]] of the system; one gene in the pair can acquire a new function while the other copy continues to perform its original function.<ref>{{cite journal |last1=Gregory |first1=T. Ryan |authorlink1=T. Ryan Gregory |last2=Hebert |first2=Paul D. N. |authorlink2=Paul D. N. Hebert |date=April 1999 |title=The Modulation of DNA Content: Proximate Causes and Ultimate Consequences |url=http://genome.cshlp.org/content/9/4/317.full |journal=[[Genome Research]] |volume=9 |issue=4 |pages=317–324 |doi=10.1101/gr.9.4.317 |issn=1088-9051 |pmid=10207154 |accessdate=2014-12-11 |deadurl=no |archiveurl=https://web.archive.org/web/20140823063412/http://genome.cshlp.org/content/9/4/317.full |archivedate=2014-08-23 |df= |doi-broken-date=2018-11-14 }}</ref><ref>{{cite journal |last=Hurles |first=Matthew |title=Gene Duplication: The Genomic Trade in Spare Parts |date=July 13, 2004 |journal=PLOS Biology |volume=2 |issue=7 |page=e206 |doi=10.1371/journal.pbio.0020206 |issn=1545-7885 |pmc=449868 |pmid=15252449}}</ref> Other types of mutations can even generate entirely new genes from previously noncoding DNA.<ref>{{cite journal |last1=Liu |first1=Na |last2=Okamura |first2=Katsutomo |last3=Tyler |first3=David M. |last4=Phillips |first4=Michael D. |last5=Chung |first5=Wei-Jen |last6=Lai |first6=Eric C |date=October 2008 |url=http://www.nature.com/cr/journal/v18/n10/full/cr2008278a.html |title=The evolution and functional diversification of animal microRNA genes |journal=Cell Research |volume=18 |issue=10 |pages=985–996 |doi=10.1038/cr.2008.278 |issn=1001-0602 |pmc=2712117 |pmid=18711447 |display-authors=3 |accessdate=2014-12-11 |deadurl=no |archiveurl=https://web.archive.org/web/20150202063359/http://www.nature.com/cr/journal/v18/n10/full/cr2008278a.html |archivedate=2015-02-02 |df= }}</ref><ref>{{cite journal |last=Siepel |first=Adam |authorlink=Adam C. Siepel |date=October 2009 |title=Darwinian alchemy: Human genes from noncoding DNA |url=http://genome.cshlp.org/content/19/10/1693.full |journal=Genome Research |volume=19 |issue=10 |pages=1693–1695 |doi=10.1101/gr.098376.109 |issn=1088-9051 |pmc=2765273 |pmid=19797681 |accessdate=2014-12-11 |deadurl=no |archiveurl=https://web.archive.org/web/20140823063126/http://genome.cshlp.org/content/19/10/1693.full |archivedate=2014-08-23 |df= }}</ref> |
|||
The generation of new genes can also involve small parts of several genes being duplicated, with these fragments then recombining to form new combinations with new functions.<ref>{{cite journal |last1=Orengo |first1=Christine A. |last2=Thornton |first2=Janet M. |authorlink2=Janet Thornton |date=July 2005 |title=Protein families and their evolution—a structural perspective |journal=Annual Review of Biochemistry |volume=74 |pages=867–900 |doi=10.1146/annurev.biochem.74.082803.133029 |issn=0066-4154 |pmid=15954844}}</ref><ref>{{cite journal |last1=Long |first1=Manyuan |last2=Betrán |first2=Esther |last3=Thornton |first3=Kevin |last4=Wang |first4=Wen |date=November 2003 |title=The origin of new genes: glimpses from the young and old |journal=Nature Reviews Genetics |volume=4 |issue=11 |pages=865–875 |doi=10.1038/nrg1204 |issn=1471-0056 |pmid=14634634}}</ref> When new genes are assembled from shuffling pre-existing parts, [[protein domain|domains]] act as modules with simple independent functions, which can be mixed together to produce new combinations with new and complex functions.<ref>{{cite journal |last1=Wang |first1=Minglei |last2=Caetano-Anollés |first2=Gustavo |authorlink2=Gustavo Caetano-Anolles |date=January 14, 2009 |title=The Evolutionary Mechanics of Domain Organization in Proteomes and the Rise of Modularity in the Protein World |journal=[[Structure (journal)|Structure]] |volume=17 |issue=1 |pages=66–78 |doi=10.1016/j.str.2008.11.008 |issn=1357-4310 |pmid=19141283}}</ref> For example, [[polyketide synthase]]s are large [[enzyme]]s that make [[antibiotics]]; they contain up to one hundred independent domains that each catalyse one step in the overall process, like a step in an assembly line.<ref>{{cite journal |last1=Weissman |first1=Kira J. |last2=Müller |first2=Rolf |date=April 14, 2008 |title=Protein–Protein Interactions in Multienzyme Megasynthetases |journal=[[ChemBioChem]] |volume=9 |issue=6 |pages=826–848 |doi=10.1002/cbic.200700751 |issn=1439-4227 |pmid=18357594}}</ref> |
|||
=== Sex and recombination === |
|||
{{Further|Sexual reproduction|Genetic recombination|Evolution of sexual reproduction}} |
|||
In [[Asexual reproduction|asexual]] organisms, genes are inherited together, or ''linked'', as they cannot mix with genes of other organisms during reproduction. In contrast, the offspring of [[sex]]ual organisms contain random mixtures of their parents' chromosomes that are produced through independent assortment. In a related process called [[homologous recombination]], sexual organisms exchange DNA between two matching chromosomes.<ref>{{cite journal |last=Radding |first=Charles M. |date=December 1982 |title=Homologous Pairing and Strand Exchange in Genetic Recombination |journal=[[Annual Reviews (publisher)|Annual Review of Genetics]] |volume=16 |pages=405–437 |doi=10.1146/annurev.ge.16.120182.002201 |issn=0066-4197 |pmid=6297377}}</ref> Recombination and reassortment do not alter allele frequencies, but instead change which alleles are associated with each other, producing offspring with new combinations of alleles.<ref name=Agrawal>{{cite journal |last=Agrawal |first=Aneil F. |date=September 5, 2006 |title=Evolution of Sex: Why Do Organisms Shuffle Their Genotypes? |journal=[[Current Biology]] |volume=16 |issue=17 |pages=R696–R704 |doi=10.1016/j.cub.2006.07.063 |issn=0960-9822 |pmid=16950096|bibcode=1996CBio....6.1213A }}</ref> Sex usually increases genetic variation and may increase the rate of evolution.<ref>{{cite journal |last1=Peters |first1=Andrew D. |last2=Otto |first2=Sarah P. |date=June 2003 |title=Liberating genetic variance through sex |journal=[[BioEssays]] |volume=25 |issue=6 |pages=533–537 |doi=10.1002/bies.10291 |issn=0265-9247 |pmid=12766942}}</ref><ref>{{cite journal |last1=Goddard |first1=Matthew R. |last2=Godfray |first2=H. Charles J. |authorlink2=Charles Godfray |last3=Burt |first3=Austin |date=March 31, 2005 |title=Sex increases the efficacy of natural selection in experimental yeast populations |journal=Nature |volume=434 |issue=7033 |pages=636–640 |bibcode=2005Natur.434..636G |doi=10.1038/nature03405 |issn=0028-0836 |pmid=15800622}}</ref> |
|||
[[File:Evolsex-dia1a.png|thumb|upright=1.15|This diagram illustrates the ''twofold cost of sex''. If each individual were to contribute to the same number of offspring (two), ''(a)'' the [[sex]]ual population remains the same size each generation, where the ''(b)'' [[Asexual reproduction]] population doubles in size each generation.]] |
|||
The two-fold cost of sex was first described by [[John Maynard Smith]].<ref name="maynard">{{harvnb|Maynard Smith|1978}}{{page needed|date=December 2014}}</ref> The first cost is that in sexually dimorphic species only one of the two sexes can bear young. (This cost does not apply to hermaphroditic species, like most plants and many [[invertebrate]]s.) The second cost is that any individual who reproduces sexually can only pass on 50% of its genes to any individual offspring, with even less passed on as each new generation passes.<ref name="ridley">{{harvnb|Ridley|1993}}{{page needed|date=December 2014}}</ref> Yet sexual reproduction is the more common means of reproduction among eukaryotes and multicellular organisms. The [[Red Queen hypothesis]] has been used to explain the significance of sexual reproduction as a means to enable continual evolution and adaptation in response to [[coevolution]] with other species in an ever-changing environment.<ref name="ridley" /><ref name="red">{{cite journal |last=Van Valen |first=Leigh |authorlink=Leigh Van Valen |year=1973 |title=A New Evolutionary Law |url=https://dl.dropboxusercontent.com/u/18310184/evolutionary-theory/vol-01/Vol.1%2CNo.1%2C1-30%2CL.%20Van%20Valen%2C%20A%20new%20evolutionary%20law..pdf |format=PDF |journal=Evolutionary Theory |volume=1 |pages=1–30 |issn=0093-4755 |accessdate=2014-12-24 |deadurl=yes |archiveurl=https://web.archive.org/web/20141222094258/https://dl.dropboxusercontent.com/u/18310184/evolutionary-theory/vol-01/Vol.1%2CNo.1%2C1-30%2CL.%20Van%20Valen%2C%20A%20new%20evolutionary%20law..pdf |archivedate=2014-12-22 |df= }}</ref><ref name="parasite">{{cite journal |last1=Hamilton |first1=W. D. |authorlink1=W. D. Hamilton |last2=Axelrod |first2=Robert |authorlink2=Robert Axelrod |last3=Tanese |first3=Reiko |date=May 1, 1990 |title=Sexual reproduction as an adaptation to resist parasites (a review) |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=87 |issue=9 |pages=3566–3573 |bibcode=1990PNAS...87.3566H |doi=10.1073/pnas.87.9.3566 |issn=0027-8424 |pmid=2185476 |pmc=53943}}</ref><ref name="Birdsell">{{harvnb|Birdsell|Wills|2003|pp=113–117}}</ref> |
|||
=== Gene flow === |
|||
{{Further|Gene flow}} |
|||
Gene flow is the exchange of genes between populations and between species.<ref name="Morjan C, Rieseberg L 2004 1341–56">{{cite journal |last1=Morjan |first1=Carrie L. |last2=Rieseberg |first2=Loren H. |authorlink2=Loren H. Rieseberg |date=June 2004 |title=How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles |journal=[[Molecular Ecology]] |volume=13 |issue=6 |pages=1341–1356 |pmid=15140081 |doi=10.1111/j.1365-294X.2004.02164.x |issn=0962-1083 |pmc=2600545}}</ref> It can therefore be a source of variation that is new to a population or to a species. Gene flow can be caused by the movement of individuals between separate populations of organisms, as might be caused by the movement of mice between inland and coastal populations, or the movement of [[pollen]] between heavy-metal-tolerant and heavy-metal-sensitive populations of grasses. |
|||
Gene transfer between species includes the formation of [[Hybrid (biology)|hybrid]] organisms and [[horizontal gene transfer]]. Horizontal gene transfer is the transfer of genetic material from one organism to another organism that is not its offspring; this is most common among [[bacteria]].<ref>{{cite journal |last1=Boucher |first1=Yan |last2=Douady |first2=Christophe J. |last3=Papke |first3=R. Thane |last4=Walsh |first4=David A. |last5=Boudreau |first5=Mary Ellen R. |last6=Nesbo |first6=Camilla L. |last7=Case |first7=Rebecca J. |last8=Doolittle |first8=W. Ford |date=December 2003 |title=Lateral gene transfer and the origins of prokaryotic groups |journal=Annual Review of Genetics |volume=37 |pages=283–328 |doi=10.1146/annurev.genet.37.050503.084247 |issn=0066-4197 |pmid=14616063 |display-authors=3}}</ref> In medicine, this contributes to the spread of [[antibiotic resistance]], as when one bacteria acquires resistance genes it can rapidly transfer them to other species.<ref name=GeneticEvolution>{{cite journal |last=Walsh |first=Timothy R. |date=October 2006 |title=Combinatorial genetic evolution of multiresistance |journal=[[Current Opinion (Elsevier)|Current Opinion in Microbiology]] |volume=9 |issue=5 |pages=476–482 |doi=10.1016/j.mib.2006.08.009 |issn=1369-5274 |pmid=16942901}}</ref> Horizontal transfer of genes from bacteria to eukaryotes such as the yeast ''[[Saccharomyces cerevisiae]]'' and the adzuki bean weevil ''[[Callosobruchus chinensis]]'' has occurred.<ref>{{cite journal |last1=Kondo |first1=Natsuko |last2=Nikoh |first2=Naruo |last3=Ijichi |first3=Nobuyuki |last4=Shimada |first4=Masakazu |last5=Fukatsu |first5=Takema |date=October 29, 2002 |title=Genome fragment of ''Wolbachia'' endosymbiont transferred to X chromosome of host insect |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=99 |issue=22 |pages=14280–14285 |bibcode=2002PNAS...9914280K |doi=10.1073/pnas.222228199 |issn=0027-8424 |pmc=137875 |pmid=12386340 |display-authors=3}}</ref><ref>{{cite journal |last=Sprague |first=George F., Jr. |date=December 1991 |title=Genetic exchange between kingdoms |journal=[[Current Opinion (Elsevier)|Current Opinion in Genetics & Development]] |volume=1 |issue=4 |pages=530–533 |doi=10.1016/S0959-437X(05)80203-5 |issn=0959-437X |pmid=1822285}}</ref> An example of larger-scale transfers are the eukaryotic [[Bdelloidea|bdelloid rotifers]], which have received a range of genes from bacteria, [[fungus|fungi]] and plants.<ref>{{cite journal |last1=Gladyshev |first1=Eugene A. |last2=Meselson |first2=Matthew |authorlink2=Matthew Meselson |last3=Arkhipova |first3=Irina R. |date=May 30, 2008 |title=Massive Horizontal Gene Transfer in Bdelloid Rotifers |journal=Science |volume=320 |issue=5880 |pages=1210–1213 |bibcode=2008Sci...320.1210G |doi=10.1126/science.1156407 |issn=0036-8075 |pmid=18511688|url=http://nrs.harvard.edu/urn-3:HUL.InstRepos:3120157 |type=Submitted manuscript }}</ref> [[Virus]]es can also carry DNA between organisms, allowing transfer of genes even across [[Domain (biology)|biological domains]].<ref>{{cite journal |last1=Baldo |first1=Angela M. |last2=McClure |first2=Marcella A. |date=September 1999 |title=Evolution and Horizontal Transfer of dUTPase-Encoding Genes in Viruses and Their Hosts |journal=[[Journal of Virology]] |volume=73 |issue=9 |pages=7710–7721 |issn=0022-538X |pmc=104298 |pmid=10438861}}</ref> |
|||
Large-scale gene transfer has also occurred between the ancestors of [[Eukaryote|eukaryotic cells]] and bacteria, during the acquisition of [[chloroplast]]s and [[Mitochondrion|mitochondria]]. It is possible that eukaryotes themselves originated from horizontal gene transfers between bacteria and [[archaea]].<ref>{{cite journal |last1=Rivera |first1=Maria C. |last2=Lake |first2=James A. |authorlink2=James A. Lake |date=September 9, 2004 |title=The ring of life provides evidence for a genome fusion origin of eukaryotes |journal=Nature |volume=431 |issue=7005 |pages=152–155 |bibcode=2004Natur.431..152R |doi=10.1038/nature02848 |issn=0028-0836 |pmid=15356622}}</ref> |
|||
== Mechanisms == |
|||
[[File:Mutation and selection diagram.svg|thumb|upright=1.35|[[Mutation]] followed by natural selection results in a population with darker colouration.]] |
|||
From a [[Neo-Darwinism|neo-Darwinian]] perspective, evolution occurs when there are changes in the frequencies of alleles within a population of interbreeding organisms,<ref name="Ewens W.J. 2004" /> for example, the allele for black colour in a population of moths becoming more common. Mechanisms that can lead to changes in allele frequencies include natural selection, genetic drift, genetic hitchhiking, mutation and gene flow. |
|||
=== Natural selection === |
|||
{{Main|Natural selection}} |
|||
{{Further|Sexual selection}} |
|||
Evolution by means of natural selection is the process by which traits that enhance survival and reproduction become more common in successive generations of a population. It has often been called a "self-evident" mechanism because it necessarily follows from three simple facts:<ref name="Lewontin70" /> |
|||
* Variation exists within populations of organisms with respect to morphology, physiology, and behaviour (phenotypic variation). |
|||
* Different traits confer different rates of survival and reproduction (differential fitness). |
|||
* These traits can be passed from generation to generation (heritability of fitness). |
|||
More offspring are produced than can possibly survive, and these conditions produce competition between organisms for survival and reproduction. Consequently, organisms with traits that give them an advantage over their competitors are more likely to pass on their traits to the next generation than those with traits that do not confer an advantage.<ref name="Hurst">{{cite journal |last=Hurst |first=Laurence D. |authorlink=Laurence Hurst |title=Fundamental concepts in genetics: genetics and the understanding of selection |date=February 2009 |journal=Nature Reviews Genetics |volume=10 |issue=2 |pages=83–93 |doi=10.1038/nrg2506 |issn=1471-0056 |pmid=19119264}}</ref> This [[teleonomy]] is the quality whereby the process of natural selection creates and preserves traits that are [[teleology in biology|seemingly fitted]] for the [[function (biology)|functional]] roles they perform.<ref>{{harvnb|Darwin|1859|loc=[http://darwin-online.org.uk/content/frameset?itemID=F373&viewtype=text&pageseq=477 Chapter XIV]}}</ref> Consequences of selection include [[Assortative mating|nonrandom mating]]<ref>{{Cite book |last1=Otto |first1=Sarah P. |author-link1=Sarah Otto |last2=Servedio |first2=Maria R. |authorlink2=Maria Servedio|last3=Nuismer |first3=Scott L. |title=Frequency-Dependent Selection and the Evolution of Assortative Mating |journal=Genetics |date=August 2008 |volume=179 |issue=4 |pages=2091–2112 |issn=0016-6731 |doi=10.1534/genetics.107.084418 |pmc=2516082 |pmid=18660541|bibcode= }}</ref> and [[genetic hitchhiking]]. |
|||
The central concept of natural selection is the [[fitness (biology)|evolutionary fitness]] of an organism.<ref name="Orr">{{cite journal |last=Orr |first=H. Allen |authorlink=H. Allen Orr |date=August 2009 |title=Fitness and its role in evolutionary genetics |journal=Nature Reviews Genetics |volume=10 |issue=8 |pages=531–539 |doi=10.1038/nrg2603 |pmc=2753274 |pmid=19546856 |issn=1471-0056}}</ref> Fitness is measured by an organism's ability to survive and reproduce, which determines the size of its genetic contribution to the next generation.<ref name="Orr" /> However, fitness is not the same as the total number of offspring: instead fitness is indicated by the proportion of subsequent generations that carry an organism's genes.<ref name="Haldane">{{cite journal |last=Haldane |first=J.B.S. |authorlink=J. B. S. Haldane |date=March 14, 1959 |title=The Theory of Natural Selection To-Day |journal=Nature |volume=183 |issue=4663 |pages=710–713 |bibcode=1959Natur.183..710H |doi=10.1038/183710a0 |issn=0028-0836 |pmid=13644170}}</ref> For example, if an organism could survive well and reproduce rapidly, but its offspring were all too small and weak to survive, this organism would make little genetic contribution to future generations and would thus have low fitness.<ref name="Orr" /> |
|||
If an allele increases fitness more than the other alleles of that gene, then with each generation this allele will become more common within the population. These traits are said to be "selected ''for''." Examples of traits that can increase fitness are enhanced survival and increased [[fecundity]]. Conversely, the lower fitness caused by having a less beneficial or deleterious allele results in this allele becoming rarer—they are "selected ''against''."<ref name="Lande">{{cite journal |last1=Lande |first1=Russell |authorlink1=Russell Lande |last2=Arnold |first2=Stevan J. |date=November 1983 |title=The Measurement of Selection on Correlated Characters |journal=Evolution |volume=37 |issue=6 |pages=1210–1226 |doi=10.2307/2408842 |pmid=28556011 |issn=0014-3820 |jstor=2408842}}</ref> Importantly, the fitness of an allele is not a fixed characteristic; if the environment changes, previously neutral or harmful traits may become beneficial and previously beneficial traits become harmful.<ref name="Futuyma_2005" /> However, even if the direction of selection does reverse in this way, traits that were lost in the past may not re-evolve in an identical form (see [[Dollo's law of irreversibility|Dollo's law]]).<ref>{{cite journal |last1=Goldberg |first1=Emma E. |last2=Igić |first2=Boris |date=November 2008 |title=On phylogenetic tests of irreversible evolution |journal=Evolution |volume=62 |issue=11 |pages=2727–2741 |doi=10.1111/j.1558-5646.2008.00505.x |issn=0014-3820 |pmid=18764918}}</ref><ref>{{cite journal |last1=Collin |first1=Rachel |last2=Miglietta |first2=Maria Pia |date=November 2008 |title=Reversing opinions on Dollo's Law |journal=[[Trends (journals)|Trends in Ecology & Evolution]] |volume=23 |issue=11 |pages=602–609 |doi=10.1016/j.tree.2008.06.013 |issn=0169-5347 |pmid=18814933}}</ref> However, a re-activation of dormant genes, as long as they have not been eliminated from the genome and were only suppressed perhaps for hundreds of generations, can lead to the re-occurrence of traits thought to be lost like hindlegs in [[dolphin]]s, teeth in [[chicken]]s, wings in wingless stick [[insect]]s, tails and additional nipples in humans etc.<ref>{{cite journal |last1=Tomić |first1=Nenad |last2=Meyer-Rochow |first2=Victor Benno |year=2011 |title=Atavisms: Medical, Genetic, and Evolutionary Implications |journal=[[Perspectives in Biology and Medicine]] |volume=54 |issue=3 |pages=332–353 |doi=10.1353/pbm.2011.0034 |issn=0031-5982 |pmid=21857125}}</ref> "Throwbacks" such as these are known as [[atavism]]s. |
|||
[[File:Genetic Distribution.svg|thumb|left|upright=1.45|These charts depict the different types of genetic selection. On each graph, the x-axis variable is the type of [[phenotypic trait]] and the y-axis variable is the number of organisms. Group A is the original population and Group B is the population after selection.<br /> |
|||
'''·''' Graph 1 shows [[directional selection]], in which a single extreme [[phenotype]] is favoured.<br /> |
|||
'''·''' Graph 2 depicts [[stabilizing selection]], where the intermediate phenotype is favoured over the extreme traits.<br /> |
|||
'''·''' Graph 3 shows [[disruptive selection]], in which the extreme phenotypes are favoured over the intermediate.]] |
|||
Natural selection within a population for a trait that can vary across a range of values, such as height, can be categorised into three different types. The first is [[directional selection]], which is a shift in the average value of a trait over time—for example, organisms slowly getting taller.<ref>{{cite journal |last1=Hoekstra |first1=Hopi E. |last2=Hoekstra |first2=Jonathan M. |last3=Berrigan |first3=David |last4=Vignieri |first4=Sacha N. |last5=Hoang |first5=Amy |last6=Hill |first6=Caryl E. |last7=Beerli |first7=Peter |last8=Kingsolver |first8=Joel G. |date=July 31, 2001 |title=Strength and tempo of directional selection in the wild |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=98 |issue=16 |pages=9157–9160 |bibcode=2001PNAS...98.9157H |doi=10.1073/pnas.161281098 |issn=0027-8424 |pmc=55389 |pmid=11470913 |display-authors=3}}</ref> Secondly, [[disruptive selection]] is selection for extreme trait values and often results in [[bimodal distribution|two different values]] becoming most common, with selection against the average value. This would be when either short or tall organisms had an advantage, but not those of medium height. Finally, in [[stabilising selection]] there is selection against extreme trait values on both ends, which causes a decrease in [[variance]] around the average value and less diversity.<ref name="Hurst" /><ref>{{cite journal |last=Felsenstein |first=Joseph |authorlink=Joseph Felsenstein |date=November 1979 |title=Excursions along the Interface between Disruptive and Stabilizing Selection |journal=Genetics |volume=93 |issue=3 |pages=773–795 |issn=0016-6731 |pmc=1214112 |pmid=17248980}}</ref> This would, for example, cause organisms to eventually have a similar height. |
|||
A special case of natural selection is sexual selection, which is selection for any trait that increases mating success by increasing the attractiveness of an organism to potential mates.<ref>{{cite journal |last1=Andersson |first1=Malte |last2=Simmons |first2=Leigh W. |date=June 2006 |title=Sexual selection and mate choice |journal=Trends in Ecology & Evolution |volume=21 |issue=6 |pages=296–302 |pmid=16769428 |doi=10.1016/j.tree.2006.03.015 |issn=0169-5347 |url=http://academic.reed.edu/biology/professors/srenn/pages/teaching/2008_syllabus/2008_readings/2_anderson-simmons_2006.pdf |deadurl=no |archiveurl=https://web.archive.org/web/20130309112854/http://academic.reed.edu/biology/professors/srenn/pages/teaching/2008_syllabus/2008_readings/2_Anderson-Simmons_2006.pdf |archivedate=2013-03-09 |df= |citeseerx=10.1.1.595.4050 }}</ref> Traits that evolved through sexual selection are particularly prominent among males of several animal species. Although sexually favoured, traits such as cumbersome antlers, mating calls, large body size and bright colours often attract predation, which compromises the survival of individual males.<ref>{{cite journal |last1=Kokko |first1=Hanna |authorlink1=Hanna Kokko |last2=Brooks |first2=Robert |last3=McNamara |first3=John M. |last4=Houston |first4=Alasdair I. |date=July 7, 2002 |title=The sexual selection continuum |journal=[[Proceedings of the Royal Society#Proceedings of the Royal Society B|Proceedings of the Royal Society B]] |volume=269 |issue=1498 |pages=1331–1340 |doi=10.1098/rspb.2002.2020 |issn=0962-8452 |pmc=1691039 |pmid=12079655}}</ref><ref name="Balancing">{{cite journal |last1=Quinn |first1=Thomas P. |last2=Hendry |first2=Andrew P. |last3=Buck |first3=Gregory B. |year=2001 |title=Balancing natural and sexual selection in sockeye salmon: interactions between body size, reproductive opportunity and vulnerability to predation by bears |url=http://redpath-staff.mcgill.ca/hendry/QuinnetalEvolEcolRes2001.pdf |format=PDF |journal=Evolutionary Ecology Research |volume=3 |pages=917–937 |issn=1522-0613 |accessdate=2014-12-15 |deadurl=no |archiveurl=https://web.archive.org/web/20160305092304/http://redpath-staff.mcgill.ca/hendry/QuinnetalEvolEcolRes2001.pdf |archivedate=2016-03-05 |df= }}</ref> This survival disadvantage is balanced by higher reproductive success in males that show these [[Handicap principle|hard-to-fake]], sexually selected traits.<ref>{{cite journal |last1=Hunt |first1=John |last2=Brooks |first2=Robert |last3=Jennions |first3=Michael D. |last4=Smith |first4=Michael J. |last5=Bentsen |first5=Caroline L. |last6=Bussière |first6=Luc F. |date=December 23, 2004 |title=High-quality male field crickets invest heavily in sexual display but die young |journal=Nature |volume=432 |issue=7020 |pages=1024–1027 |bibcode=2004Natur.432.1024H |doi=10.1038/nature03084 |issn=0028-0836 |pmid=15616562 |display-authors=3}}</ref> |
|||
Natural selection most generally makes nature the measure against which individuals and individual traits, are more or less likely to survive. "Nature" in this sense refers to an [[ecosystem]], that is, a system in which organisms interact with every other element, [[Abiotic component|physical]] as well as [[Biotic component|biological]], in their local environment. [[Eugene Odum]], a founder of [[ecology]], defined an ecosystem as: "Any unit that includes all of the organisms...in a given area interacting with the physical environment so that a flow of energy leads to clearly defined trophic structure, biotic diversity, and material cycles (i.e., exchange of materials between living and nonliving parts) within the system...."<ref name="Odum1971">{{harvnb|Odum|1971|p=8}}</ref> Each population within an ecosystem occupies a distinct [[Ecological niche|niche]], or position, with distinct relationships to other parts of the system. These relationships involve the life history of the organism, its position in the [[food chain]] and its geographic range. This broad understanding of nature enables scientists to delineate specific forces which, together, comprise natural selection. |
|||
Natural selection can act at [[unit of selection|different levels of organisation]], such as genes, cells, individual organisms, groups of organisms and species.<ref name="Okasha07">{{harvnb|Okasha|2006}}</ref><ref name="Gould">{{cite journal |last=Gould |first=Stephen Jay |authorlink=Stephen Jay Gould |date=February 28, 1998 |title=Gulliver's further travels: the necessity and difficulty of a hierarchical theory of selection |journal=Philosophical Transactions of the Royal Society B: Biological Sciences |volume=353 |issue=1366 |pages=307–314 |doi=10.1098/rstb.1998.0211 |issn=0962-8436 |pmc=1692213 |pmid=9533127}}</ref><ref name=Mayr1997>{{cite journal |last=Mayr |first=Ernst |authorlink=Ernst Mayr |date=March 18, 1997 |title=The objects of selection |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=94 |issue=6 |pages=2091–2094 |bibcode=1997PNAS...94.2091M |doi=10.1073/pnas.94.6.2091 |issn=0027-8424 |pmc=33654 |pmid=9122151}}</ref> Selection can act at multiple levels simultaneously.<ref>{{harvnb|Maynard Smith|1998|pp=203–211; discussion 211–217}}</ref> An example of selection occurring below the level of the individual organism are genes called [[Transposable element|transposons]], which can replicate and spread throughout a genome.<ref>{{cite journal |last=Hickey |first=Donal A. |year=1992 |title=Evolutionary dynamics of transposable elements in prokaryotes and eukaryotes |journal=[[Genetica]] |volume=86 |issue=1–3 |pages=269–274 |doi=10.1007/BF00133725 |issn=0016-6707 |pmid=1334911}}</ref> Selection at a level above the individual, such as [[group selection]], may allow the evolution of cooperation, as discussed below.<ref>{{cite journal |last1=Gould |first1=Stephen Jay |last2=Lloyd |first2=Elisabeth A. |authorlink2=Elisabeth Lloyd |date=October 12, 1999 |title=Individuality and adaptation across levels of selection: how shall we name and generalise the unit of Darwinism? |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=96 |issue=21 |pages=11904–11909 |bibcode=1999PNAS...9611904G |doi=10.1073/pnas.96.21.11904 |issn=0027-8424 |pmc=18385 |pmid=10518549}}</ref> |
|||
=== Biased mutation === |
|||
In addition to being a major source of variation, mutation may also function as a mechanism of evolution when there are different probabilities at the molecular level for different mutations to occur, a process known as mutation bias.<ref>{{cite journal |last=Lynch |first=Michael |authorlink=Michael Lynch (geneticist) |date=May 15, 2007 |title=The frailty of adaptive hypotheses for the origins of organismal complexity |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=104 |issue=Suppl. 1 |pages=8597–8604 |bibcode=2007PNAS..104.8597L |doi=10.1073/pnas.0702207104 |issn=0027-8424 |pmid=17494740 |pmc=1876435}}</ref> If two genotypes, for example one with the nucleotide G and another with the nucleotide A in the same position, have the same fitness, but mutation from G to A happens more often than mutation from A to G, then genotypes with A will tend to evolve.<ref>{{cite journal |last1=Smith |first1=Nick G.C. |last2=Webster |first2=Matthew T. |last3=Ellegren |first3=Hans |date=September 2002 |title=Deterministic Mutation Rate Variation in the Human Genome |url=http://genome.cshlp.org/content/12/9/1350.abstract |journal=Genome Research |volume=12 |issue=9 |pages=1350–1356 |doi=10.1101/gr.220502 |issn=1088-9051 |pmc=186654 |pmid=12213772 |deadurl=no |archiveurl=https://web.archive.org/web/20140823062603/http://genome.cshlp.org/content/12/9/1350.abstract |archivedate=2014-08-23 |df= }}</ref> Different insertion vs. deletion mutation biases in different [[Taxon|taxa]] can lead to the evolution of different genome sizes.<ref>{{cite journal |last1=Petrov |first1=Dmitri A. |last2=Sangster |first2=Todd A. |last3=Johnston |first3=J. Spencer |last4=Hartl |first4=Daniel L. |last5=Shaw |first5=Kerry L. |date=February 11, 2000 |title=Evidence for DNA Loss as a Determinant of Genome Size |journal=Science |volume=287 |issue=5455 |pages=1060–1062 |bibcode=2000Sci...287.1060P |doi=10.1126/science.287.5455.1060 |issn=0036-8075 |pmid=10669421 |display-authors=3}}</ref><ref>{{cite journal |last=Petrov |first=Dmitri A. |date=May 2002 |title=DNA loss and evolution of genome size in ''Drosophila'' |journal=Genetica |volume=115 |issue=1 |pages=81–91 |doi=10.1023/A:1016076215168 |issn=0016-6707 |pmid=12188050}}</ref> Developmental or mutational biases have also been observed in morphological evolution.<ref>{{cite journal |last1=Kiontke |first1=Karin |last2=Barrière |first2=Antoine |last3=Kolotuev |first3=Irina |last4=Podbilewicz |first4=Benjamin |last5=Sommer |first5=Ralf |last6=Fitch |first6=David H.A. |last7=Félix |first7=Marie-Anne |date=November 2007 |title=Trends, Stasis, and Drift in the Evolution of Nematode Vulva Development |journal=Current Biology |volume=17 |issue=22 |pages=1925–1937 |doi=10.1016/j.cub.2007.10.061 |issn=0960-9822 |pmid=18024125 |display-authors=3|bibcode=1996CBio....6.1213A }}</ref><ref>{{cite journal |last1=Braendle |first1=Christian |last2=Baer |first2=Charles F. |last3=Félix |first3=Marie-Anne |date=March 12, 2010 |editor1-last=Barsh |editor1-first=Gregory S. |title=Bias and Evolution of the Mutationally Accessible Phenotypic Space in a Developmental System |journal=[[PLOS Genetics]] |volume=6 |issue=3 |page=e1000877 |doi=10.1371/journal.pgen.1000877 |issn=1553-7390 |pmid=20300655 |pmc=2837400}}</ref> For example, according to the [[Baldwin effect|phenotype-first theory of evolution]], mutations can eventually cause the [[genetic assimilation]] of traits that were previously [[phenotypic plasticity|induced by the environment]].<ref name="Palmer 2004">{{cite journal |last=Palmer |first=A. Richard |date=October 29, 2004 |title=Symmetry breaking and the evolution of development |journal=Science |pages=828–833 |volume=306 |issue=5697 |bibcode=2004Sci...306..828P |doi=10.1126/science.1103707 |issn=0036-8075 |pmid=15514148 |url=http://biology.duke.edu/nijhout/PDFs/Palmer04.pdf |deadurl=no |archiveurl=https://web.archive.org/web/20070612110233/http://www.biology.duke.edu/nijhout/PDFs/Palmer04.pdf |archivedate=2007-06-12 |df= |citeseerx=10.1.1.631.4256 }}</ref><ref>{{harvnb|West-Eberhard|2003|pages=140}}</ref><ref>{{harvnb|Pocheville|Danchin|2017}}</ref> |
|||
Mutation bias effects are superimposed on other processes. If selection would favour either one out of two mutations, but there is no extra advantage to having both, then the mutation that occurs the most frequently is the one that is most likely to become fixed in a population.<ref>{{cite journal |last1=Stoltzfus |first1=Arlin |last2=Yampolsky |first2=Lev Y. |date=September–October 2009 |title=Climbing Mount Probable: Mutation as a Cause of Nonrandomness in Evolution |journal=[[Journal of Heredity]] |volume=100 |issue=5 |pages=637–647 |doi=10.1093/jhered/esp048 |issn=0022-1503 |pmid=19625453}}</ref><ref>{{cite journal |last1=Yampolsky |first1=Lev Y. |last2=Stoltzfus |first2=Arlin |date=March 2001 |title=Bias in the introduction of variation as an orienting factor in evolution |journal=[[Evolution & Development]] |volume=3 |issue=2 |pages=73–83 |doi=10.1046/j.1525-142x.2001.003002073.x |issn=1520-541X |pmid=11341676}}</ref> Mutations leading to the loss of function of a gene are much more common than mutations that produce a new, fully functional gene. Most loss of function mutations are selected against. But when selection is weak, mutation bias towards loss of function can affect evolution.<ref>{{cite journal |last=Haldane |first=J.B.S. |date=January–February 1933 |title=The Part Played by Recurrent Mutation in Evolution |journal=The American Naturalist |volume=67 |issue=708 |pages=5–19 |doi=10.1086/280465 |issn=0003-0147 |jstor=2457127}}</ref> For example, [[Biological pigment|pigments]] are no longer useful when animals live in the darkness of caves, and tend to be lost.<ref>{{cite journal |last1=Protas |first1=Meredith |last2=Conrad |first2=Melissa |last3=Gross |first3=Joshua B. |last4=Tabin |first4=Clifford |authorlink4=Clifford Tabin |last5=Borowsky |first5=Richard |date=March 6, 2007 |title=Regressive Evolution in the Mexican Cave Tetra, ''Astyanax mexicanus'' |journal=Current Biology |volume=17 |issue=5 |pages=452–454 |doi=10.1016/j.cub.2007.01.051 |issn=0960-9822 |pmc=2570642 |pmid=17306543 |display-authors=3|bibcode=1996CBio....6.1213A }}</ref> This kind of loss of function can occur because of mutation bias, and/or because the function had a cost, and once the benefit of the function disappeared, natural selection leads to the loss. Loss of [[endospore|sporulation]] ability in ''[[Bacillus subtilis]]'' during laboratory evolution appears to have been caused by mutation bias, rather than natural selection against the cost of maintaining sporulation ability.<ref>{{Cite book |last1=Maughan |first1=Heather |last2=Masel |first2=Joanna |author2link=Joanna Masel |last3=Birky |first3=C. William, Jr. |last4=Nicholson |first4=Wayne L. |date=October 2007 |title=The Roles of Mutation Accumulation and Selection in Loss of Sporulation in Experimental Populations of ''Bacillus subtilis'' |journal=Genetics |volume=177 |issue=2 |pages=937–948 |doi=10.1534/genetics.107.075663 |issn=0016-6731 |pmc=2034656 |pmid=17720926|bibcode= }}</ref> When there is no selection for loss of function, the speed at which loss evolves depends more on the mutation rate than it does on the [[effective population size]],<ref>{{cite journal |last1=Masel |first1=Joanna |last2=King |first2=Oliver D. |last3=Maughan |first3=Heather |date=January 2007 |title=The Loss of Adaptive Plasticity during Long Periods of Environmental Stasis |journal=The American Naturalist |volume=169 |issue=1 |pages=38–46 |doi=10.1086/510212 |issn=0003-0147 |pmc=1766558 |pmid=17206583}}</ref> indicating that it is driven more by mutation bias than by genetic drift. In parasitic organisms, mutation bias leads to selection pressures as seen in ''[[Ehrlichia]]''. Mutations are biased towards [[antigen]]ic variants in outer-membrane proteins. |
|||
=== Genetic drift === |
|||
{{Further|Genetic drift|Effective population size}} |
|||
[[File:Allele-frequency.png|thumb|Simulation of genetic drift of 20 unlinked alleles in populations of 10 (top) and 100 (bottom). Drift to [[Fixation (population genetics)|fixation]] is more rapid in the smaller population.]] |
|||
Genetic drift is the random fluctuations of [[allele frequency|allele frequencies]] within a population from one generation to the next.<ref name="Futuyma2017b">{{cite book | last1=Futuyma | first1=Douglas J. | last2=Kirkpatrick | first2=Mark | year = 2017 | chapter = Natural selection and adaptation | title=Evolution | pages = 55–66 | edition = Fourth | publisher = Sunderland, Massachusetts: Sinauer Associates, Inc | isbn=978-1-60535-605-1}}</ref> When selective forces are absent or relatively weak, allele frequencies are equally likely to ''drift'' upward or downward at each successive generation because the alleles are subject to [[sampling error]].<ref name="Masel 2011">{{cite journal |last=Masel |first=Joanna |date=October 25, 2011 |title=Genetic drift |journal=Current Biology |volume=21 |issue=20 |pages=R837–R838 |doi=10.1016/j.cub.2011.08.007 |issn=0960-9822 |pmid=22032182|bibcode=1996CBio....6.1213A }}</ref> This drift halts when an allele eventually becomes fixed, either by disappearing from the population or replacing the other alleles entirely. Genetic drift may therefore eliminate some alleles from a population due to chance alone. Even in the absence of selective forces, genetic drift can cause two separate populations that began with the same genetic structure to drift apart into two divergent populations with different sets of alleles.<ref>{{cite journal |last=Lande |first=Russell |year=1989 |title=Fisherian and Wrightian theories of speciation |journal=[[Genome (journal)|Genome]] |volume=31 |issue=1 |pages=221–227 |doi=10.1139/g89-037 |issn=0831-2796 |pmid=2687093}}</ref> |
|||
The [[neutral theory of molecular evolution]] proposed that most evolutionary changes are the result of the fixation of [[neutral mutation]]s by genetic drift.<ref name="Kimura M 1991 367–86">{{cite journal |last=Kimura |first=Motoo |authorlink=Motoo Kimura |year=1991 |title=The neutral theory of molecular evolution: a review of recent evidence |url=https://www.jstage.jst.go.jp/article/jjg/66/4/66_4_367/_article |journal=[[Journal of Human Genetics|The Japanese Journal of Human Genetics]] |volume=66 |issue=4 |pages=367–386 |doi=10.1266/jjg.66.367 |issn=0021-504X |pmid=1954033 |deadurl=no |archiveurl=https://web.archive.org/web/20141216085551/https://www.jstage.jst.go.jp/article/jjg/66/4/66_4_367/_article |archivedate=2014-12-16 |df= }}</ref> Hence, in this model, most genetic changes in a population are the result of constant mutation pressure and genetic drift.<ref>{{cite journal |last=Kimura |first=Motoo |year=1989 |title=The neutral theory of molecular evolution and the world view of the neutralists |journal=Genome |volume=31 |issue=1 |pages=24–31 |doi=10.1139/g89-009 |issn=0831-2796 |pmid=2687096}}</ref> This form of the neutral theory is now largely abandoned, since it does not seem to fit the genetic variation seen in nature.<ref>{{cite journal |last=Kreitman |first=Martin |authorlink=Martin Kreitman |date=August 1996 |title=The neutral theory is dead. Long live the neutral theory |journal=BioEssays |volume=18 |issue=8 |pages=678–683; discussion 683 |doi=10.1002/bies.950180812 |issn=0265-9247 |pmid=8760341}}</ref><ref>{{cite journal |last=Leigh |first=E.G., Jr. |date=November 2007 |title=Neutral theory: a historical perspective |journal=[[Journal of Evolutionary Biology]] |volume=20 |issue=6 |pages=2075–2091 |doi=10.1111/j.1420-9101.2007.01410.x |issn=1010-061X |pmid=17956380}}</ref> However, a more recent and better-supported version of this model is the [[nearly neutral theory of molecular evolution|nearly neutral theory]], where a mutation that would be effectively neutral in a small population is not necessarily neutral in a large population.<ref name="Hurst" /> Other alternative theories propose that genetic drift is dwarfed by other stochastic forces in evolution, such as genetic hitchhiking, also known as genetic draft.<ref name="Masel 2011" /><ref name="gillespie 2001">{{cite journal |last=Gillespie |first=John H. |authorlink=John H. Gillespie |date=November 2001 |title=Is the population size of a species relevant to its evolution? |journal=Evolution |volume=55 |issue=11 |pages=2161–2169 |doi=10.1111/j.0014-3820.2001.tb00732.x |issn=0014-3820 |pmid=11794777}}</ref><ref>{{Cite book |last1=Neher |first1=Richard A. |last2=Shraiman |first2=Boris I. |date=August 2011 |title=Genetic Draft and Quasi-Neutrality in Large Facultatively Sexual Populations |journal=Genetics |volume=188 |issue=4 |pages=975–996 |doi=10.1534/genetics.111.128876 |issn=0016-6731 |pmc=3176096 |pmid=21625002|arxiv=1108.1635 |bibcode= }}</ref> |
|||
The time for a neutral allele to become fixed by genetic drift depends on population size, with fixation occurring more rapidly in smaller populations.<ref>{{cite journal |last1=Otto |first1=Sarah P. |last2=Whitlock |first2=Michael C. |date=June 1997 |title=The Probability of Fixation in Populations of Changing Size |url=http://www.genetics.org/content/146/2/723.full.pdf |format=PDF |journal=Genetics |volume=146 |issue=2 |pages=723–733 |issn=0016-6731 |pmc=1208011 |pmid=9178020 |accessdate=2014-12-18 |deadurl=no |archiveurl=https://web.archive.org/web/20150319042554/http://www.genetics.org/content/146/2/723.full.pdf |archivedate=2015-03-19 |df= }}</ref> The number of individuals in a population is not critical, but instead a measure known as the effective population size.<ref name="Charlesworth">{{cite journal |last=Charlesworth |first=Brian |authorlink=Brian Charlesworth |date=March 2009 |title=Fundamental concepts in genetics: effective population size and patterns of molecular evolution and variation |journal=Nature Reviews Genetics |volume=10 |issue=3 |pages=195–205 |doi=10.1038/nrg2526 |issn=1471-0056 |pmid=19204717}}</ref> The effective population is usually smaller than the total population since it takes into account factors such as the level of inbreeding and the stage of the lifecycle in which the population is the smallest.<ref name="Charlesworth" /> The effective population size may not be the same for every gene in the same population.<ref>{{cite journal |last1=Cutter |first1=Asher D. |last2=Choi |first2=Jae Young |date=August 2010 |title=Natural selection shapes nucleotide polymorphism across the genome of the nematode ''Caenorhabditis briggsae'' |journal=Genome Research |volume=20 |issue=8 |pages=1103–1111 |doi=10.1101/gr.104331.109 |issn=1088-9051 |pmc=2909573 |pmid=20508143 |ref=harv}}</ref> |
|||
It is usually difficult to measure the relative importance of selection and neutral processes, including drift.<ref>{{cite journal |last1=Mitchell-Olds |first1=Thomas |last2=Willis |first2=John H. |last3=Goldstein |first3=David B. |authorlink3=David B. Goldstein (geneticist) |date=November 2007 |title=Which evolutionary processes influence natural genetic variation for phenotypic traits? |journal=Nature Reviews Genetics |volume=8 |issue=11 |pages=845–856 |doi=10.1038/nrg2207 |issn=1471-0056 |pmid=17943192}}</ref> The comparative importance of adaptive and non-adaptive forces in driving evolutionary change is an area of [[Evolutionary biology|current research]].<ref>{{cite journal |last=Nei |first=Masatoshi |authorlink=Masatoshi Nei |date=December 2005 |title=Selectionism and Neutralism in Molecular Evolution |journal=[[Molecular Biology and Evolution]] |volume=22 |issue=12 |pages=2318–2342 |doi=10.1093/molbev/msi242 |issn=0737-4038 |pmc=1513187 |pmid=16120807}} |
|||
* {{cite journal |last=Nei |first=Masatoshi |date=May 2006 |title=Selectionism and Neutralism in Molecular Evolution |journal=Molecular Biology and Evolution |type=Erratum |volume=23 |issue=5 |page=1095 |doi=10.1093/molbev/msk009 |issn=0737-4038}}</ref> |
|||
=== Genetic hitchhiking === |
|||
{{Further|Genetic hitchhiking|Hill–Robertson effect|Selective sweep}} |
|||
Recombination allows alleles on the same strand of DNA to become separated. However, the rate of recombination is low (approximately two events per chromosome per generation). As a result, genes close together on a chromosome may not always be shuffled away from each other and genes that are close together tend to be inherited together, a phenomenon known as [[genetic linkage|linkage]].<ref>{{cite journal |last1=Lien |first1=Sigbjørn |last2=Szyda |first2=Joanna |last3=Schechinger |first3=Birgit |last4=Rappold |first4=Gudrun |last5=Arnheim |first5=Norm |date=February 2000 |title=Evidence for Heterogeneity in Recombination in the Human Pseudoautosomal Region: High Resolution Analysis by Sperm Typing and Radiation-Hybrid Mapping |journal=[[American Journal of Human Genetics]] |volume=66 |issue=2 |pages=557–566 |doi=10.1086/302754 |issn=0002-9297 |pmc=1288109 |pmid=10677316 |display-authors=3}}</ref> This tendency is measured by finding how often two alleles occur together on a single chromosome compared to [[independence (probability theory)|expectations]], which is called their [[linkage disequilibrium]]. A set of alleles that is usually inherited in a group is called a [[haplotype]]. This can be important when one allele in a particular haplotype is strongly beneficial: natural selection can drive a [[selective sweep]] that will also cause the other alleles in the haplotype to become more common in the population; this effect is called genetic hitchhiking or genetic draft.<ref>{{cite journal |last=Barton |first=Nicholas H. |authorlink=Nick Barton |date=November 29, 2000 |title=Genetic hitchhiking |journal=Philosophical Transactions of the Royal Society B: Biological Sciences |volume=355 |issue=1403 |pages=1553–1562 |doi=10.1098/rstb.2000.0716 |issn=0962-8436 |pmc=1692896 |pmid=11127900}}</ref> Genetic draft caused by the fact that some neutral genes are genetically linked to others that are under selection can be partially captured by an appropriate effective population size.<ref name="gillespie 2001" /> |
|||
=== Gene flow === |
|||
{{Further|Gene flow|Hybrid (biology)|Horizontal gene transfer}} |
|||
Gene flow involves the exchange of genes between populations and between species.<ref name="Morjan C, Rieseberg L 2004 1341–56" /> The presence or absence of gene flow fundamentally changes the course of evolution. Due to the complexity of organisms, any two completely isolated populations will eventually evolve genetic incompatibilities through neutral processes, as in the [[Bateson-Dobzhansky-Muller Model|Bateson-Dobzhansky-Muller model]], even if both populations remain essentially identical in terms of their adaptation to the environment. |
|||
If genetic differentiation between populations develops, gene flow between populations can introduce traits or alleles which are disadvantageous in the local population and this may lead to organisms within these populations evolving mechanisms that prevent mating with genetically distant populations, eventually resulting in the appearance of new species. Thus, exchange of genetic information between individuals is fundamentally important for the development of the [[Species problem#Mayr's Biological Species Concept|''Biological Species Concept'']] (BSC). |
|||
During the development of the modern synthesis, Sewall Wright developed his [[shifting balance theory]], which regarded gene flow between partially isolated populations as an important aspect of adaptive evolution.<ref>{{cite journal |last=Wright |first=Sewall |authorlink=Sewall Wright |year=1932 |title=The roles of mutation, inbreeding, crossbreeding and selection in evolution |url=http://www.blackwellpublishing.com/ridley/classictexts/wright.asp |journal=Proceedings of the VI International Congress of Genetrics |volume=1 |pages=356–366 |accessdate=2014-12-18 |deadurl=no |archiveurl=https://web.archive.org/web/20140823063103/http://www.blackwellpublishing.com/ridley/classictexts/wright.asp |archivedate=2014-08-23 |df= }}</ref> However, recently there has been substantial criticism of the importance of the shifting balance theory.<ref name="Coyne 1997">{{cite journal |last1=Coyne |first1=Jerry A. |authorlink1=Jerry Coyne |last2=Barton |first2=Nicholas H. |last3=Turelli |first3=Michael |date=June 1997 |title=Perspective: A Critique of Sewall Wright's Shifting Balance Theory of Evolution |journal=Evolution |volume=51 |issue=3 |pages=643–671 |doi=10.2307/2411143 |pmid=28568586 |issn=0014-3820|jstor=2411143 }}</ref> |
|||
== Outcomes == |
|||
[[File:Kishony lab-The Evolution of Bacteria on a Mega-Plate.webm|thumb|upright=1.5|thumbtime=106|A visual demonstration of rapid [[antibiotic resistance]] evolution by ''E. coli'' growing across a plate with increasing concentrations of [[trimethoprim]].<ref>{{Cite journal|last=Baym|first=Michael|last2=Lieberman|first2=Tami D.|last3=Kelsic|first3=Eric D.|last4=Chait|first4=Remy|last5=Gross|first5=Rotem|last6=Yelin|first6=Idan|last7=Kishony|first7=Roy|date=2016-09-09|title=Spatiotemporal microbial evolution on antibiotic landscapes|url=http://science.sciencemag.org/content/353/6304/1147|journal=Science|language=en|volume=353|issue=6304|pages=1147–1151|doi=10.1126/science.aag0822|issn=0036-8075|pmid=27609891|pmc=5534434|bibcode=2016Sci...353.1147B|deadurl=no|archiveurl=https://web.archive.org/web/20160923173125/http://science.sciencemag.org/content/353/6304/1147|archivedate=2016-09-23|df=}}</ref>]] |
|||
Evolution influences every aspect of the form and behaviour of organisms. Most prominent are the specific behavioural and physical adaptations that are the outcome of natural selection. These adaptations increase fitness by aiding activities such as finding food, avoiding [[Predation|predators]] or attracting mates. Organisms can also respond to selection by [[Co-operation (evolution)|cooperating]] with each other, usually by aiding their relatives or engaging in mutually beneficial [[symbiosis]]. In the longer term, evolution produces new species through splitting ancestral populations of organisms into new groups that cannot or will not interbreed. |
|||
These outcomes of evolution are distinguished based on time scale as [[macroevolution]] versus microevolution. Macroevolution refers to evolution that occurs at or above the level of species, in particular speciation and extinction; whereas microevolution refers to smaller evolutionary changes within a species or population, in particular shifts in [[allele frequency]] and adaptation.<ref name="ScottEC">{{cite journal |last1=Scott |first1=Eugenie C. |authorlink1=Eugenie Scott |last2=Matzke |first2=Nicholas J. |authorlink2=Nick Matzke |date=May 15, 2007 |title=Biological design in science classrooms |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=104 |issue=Suppl. 1 |pages=8669–8676 |bibcode=2007PNAS..104.8669S |doi=10.1073/pnas.0701505104 |issn=0027-8424 |pmid=17494747 |pmc=1876445}}</ref> In general, macroevolution is regarded as the outcome of long periods of microevolution.<ref>{{cite journal |last1=Hendry |first1=Andrew Paul |last2=Kinnison |first2=Michael T. |date=November 2001 |title=An introduction to microevolution: rate, pattern, process |journal=Genetica |volume='''112–113''' |issue=1 |pages=1–8 |doi=10.1023/A:1013368628607 |issn=0016-6707 |pmid=11838760}}</ref> Thus, the distinction between micro- and macroevolution is not a fundamental one—the difference is simply the time involved.<ref>{{cite journal |last=Leroi |first=Armand M. |authorlink=Armand Marie Leroi |date=March–April 2000 |title=The scale independence of evolution |journal=Evolution & Development |volume=2 |issue=2 |pages=67–77 |doi=10.1046/j.1525-142x.2000.00044.x |issn=1520-541X |pmid=11258392|citeseerx=10.1.1.120.1020 }}</ref> However, in macroevolution, the traits of the entire species may be important. For instance, a large amount of variation among individuals allows a species to rapidly adapt to new [[habitat]]s, lessening the chance of it going extinct, while a wide geographic range increases the chance of speciation, by making it more likely that part of the population will become isolated. In this sense, microevolution and macroevolution might involve selection at different levels—with microevolution acting on genes and organisms, versus macroevolutionary processes such as [[Unit of selection#Species selection and selection at higher taxonomic levels|species selection]] acting on entire species and affecting their rates of speciation and extinction.{{sfn|Gould|2002|pp=657–658}}<ref name="Gould_1994">{{cite journal |last=Gould |first=Stephen Jay |date=July 19, 1994 |title=Tempo and mode in the macroevolutionary reconstruction of Darwinism |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=91 |issue=15 |pages=6764–6771 |bibcode=1994PNAS...91.6764G |doi=10.1073/pnas.91.15.6764 |issn=0027-8424 |pmc=44281 |pmid=8041695}}</ref><ref name="Jablonski2000">{{cite journal |last=Jablonski |first=David |authorlink=David Jablonski |year=2000 |title=Micro- and macroevolution: scale and hierarchy in evolutionary biology and paleobiology |journal=[[Paleobiology (journal)|Paleobiology]] |volume=26 |issue=sp4 |pages=15–52 |doi=10.1666/0094-8373(2000)26[15:MAMSAH]2.0.CO;2 |issn=0094-8373}}</ref> |
|||
A common misconception is that evolution has goals, long-term plans, or an innate tendency for "progress", as expressed in beliefs such as orthogenesis and evolutionism; realistically however, evolution has no long-term goal and does not necessarily produce greater complexity.<ref name="sciam_1998">{{cite journal |last=Dougherty |first=Michael J. |date=July 20, 1998 |title=Is the human race evolving or devolving? |url=http://www.scientificamerican.com/article/is-the-human-race-evolvin/ |journal=Scientific American |issn=0036-8733 |accessdate=2015-09-11 |deadurl=no |archiveurl=https://wayback.archive-it.org/all/20140506224205/http://www.scientificamerican.com/article/is-the-human-race-evolvin/ |archivedate=2014-05-06 |df= }}</ref><ref>{{cite web |url=http://www.talkorigins.org/indexcc/CB/CB932.html |title=Claim CB932: Evolution of degenerate forms |date=July 22, 2003 |editor-last=Isaak |editor-first=Mark |website=[[TalkOrigins Archive]] |publisher=The TalkOrigins Foundation, Inc. |location=Houston, Texas |accessdate=2014-12-19 |deadurl=no |archiveurl=https://web.archive.org/web/20140823062949/http://www.talkorigins.org/indexcc/CB/CB932.html |archivedate=2014-08-23 |df= }}</ref><ref>{{harvnb|Lane|1996|p=61}}</ref> Although [[Evolution of biological complexity|complex species]] have evolved, they occur as a side effect of the overall number of organisms increasing and simple forms of life still remain more common in the biosphere.<ref name="Carroll_2001">{{cite journal |last=Carroll |first=Sean B. |authorlink=Sean B. Carroll |date=February 22, 2001 |title=Chance and necessity: the evolution of morphological complexity and diversity |journal=Nature |volume=409 |issue=6823 |pages=1102–1109 |bibcode=2001Natur.409.1102C |doi=10.1038/35059227 |issn=0028-0836 |pmid=11234024}}</ref> For example, the overwhelming majority of species are microscopic [[prokaryote]]s, which form about half the world's [[Biomass (ecology)|biomass]] despite their small size,<ref>{{cite journal |last1=Whitman |first1=William B. |last2=Coleman |first2=David C. |last3=Wiebe |first3=William J. |date=June 9, 1998 |title=Prokaryotes: The unseen majority |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=95 |issue=12 |pages=6578–6583 |bibcode=1998PNAS...95.6578W |doi=10.1073/pnas.95.12.6578 |issn=0027-8424 |pmc=33863 |pmid=9618454}}</ref> and constitute the vast majority of Earth's biodiversity.<ref name=Schloss>{{cite journal |last1=Schloss |first1=Patrick D. |last2=Handelsman |first2=Jo |authorlink2=Jo Handelsman |date=December 2004 |title=Status of the Microbial Census |journal=[[Microbiology and Molecular Biology Reviews]] |volume=68 |issue=4 |pages=686–691 |doi=10.1128/MMBR.68.4.686-691.2004 |issn=1092-2172 |pmc=539005 |pmid=15590780}}</ref> Simple organisms have therefore been the dominant form of life on Earth throughout its history and continue to be the main form of life up to the present day, with complex life only appearing more diverse because it is [[Sampling bias|more noticeable]].<ref>{{cite journal |last=Nealson |first=Kenneth H. |date=January 1999 |title=Post-Viking microbiology: new approaches, new data, new insights |journal=[[Origins of Life and Evolution of Biospheres]] |volume=29 |issue=1 |pages=73–93 |doi=10.1023/A:1006515817767 |issn=0169-6149 |pmid=11536899}}</ref> Indeed, the evolution of microorganisms is particularly important to [[Evolutionary biology|modern evolutionary research]], since their rapid reproduction allows the study of [[experimental evolution]] and the observation of evolution and adaptation in real time.<ref name="Buckling">{{cite journal |last1=Buckling |first1=Angus |last2=MacLean |first2=R. Craig |last3=Brockhurst |first3=Michael A. |last4=Colegrave |first4=Nick |date=February 12, 2009 |title=The Beagle in a bottle |journal=Nature |volume=457 |issue=7231 |pages=824–829 |bibcode=2009Natur.457..824B |doi=10.1038/nature07892 |issn=0028-0836 |pmid=19212400}}</ref><ref>{{cite journal |last1=Elena |first1=Santiago F. |last2=Lenski |first2=Richard E. |authorlink2=Richard Lenski |date=June 2003 |title=Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation |journal=Nature Reviews Genetics |volume=4 |issue=6 |pages=457–469 |doi=10.1038/nrg1088 |issn=1471-0056 |pmid=12776215}}</ref> |
|||
=== Adaptation === |
|||
{{details|Adaptation}} |
|||
[[File:Homology vertebrates-en.svg|thumb|upright=1.35|[[Homology (biology)|Homologous]] bones in the limbs of [[tetrapod]]s. The bones of these animals have the same basic structure, but have been [[adaptation|adapted]] for specific uses.]] |
|||
Adaptation is the process that makes organisms better suited to their habitat.<ref>{{harvnb|Mayr|1982|p=483}}: "Adaptation... could no longer be considered a static condition, a product of a creative past and became instead a continuing dynamic process."</ref><ref>The sixth edition of the ''Oxford Dictionary of Science'' (2010) defines ''adaptation'' as "Any change in the structure or functioning of successive generations of a population that makes it better suited to its environment."</ref> Also, the term adaptation may refer to a trait that is important for an organism's survival. For example, the adaptation of [[horse]]s' teeth to the grinding of grass. By using the term ''adaptation'' for the evolutionary process and ''adaptive trait'' for the product (the bodily part or function), the two senses of the word may be distinguished. Adaptations are produced by natural selection.<ref>{{cite journal |last=Orr |first=H. Allen |date=February 2005 |title=The genetic theory of adaptation: a brief history |journal=Nature Reviews Genetics |volume=6 |issue=2 |pages=119–127 |doi=10.1038/nrg1523 |issn=1471-0056 |pmid=15716908}}</ref> The following definitions are due to Theodosius Dobzhansky: |
|||
# ''Adaptation'' is the evolutionary process whereby an organism becomes better able to live in its habitat or habitats.<ref>{{harvnb|Dobzhansky|1968|pp=1–34}}</ref> |
|||
# ''Adaptedness'' is the state of being adapted: the degree to which an organism is able to live and reproduce in a given set of habitats.<ref>{{harvnb|Dobzhansky|1970|pp=4–6, 79–82, 84–87}}</ref> |
|||
# An ''adaptive trait'' is an aspect of the developmental pattern of the organism which enables or enhances the probability of that organism surviving and reproducing.<ref>{{cite journal |last=Dobzhansky |first=Theodosius |date=March 1956 |title=Genetics of Natural Populations. XXV. Genetic Changes in Populations of ''Drosophila pseudoobscura'' and ''Drosophila persimilis'' in Some Localities in California |journal=Evolution |volume=10 |issue=1 |pages=82–92 |doi=10.2307/2406099 |issn=0014-3820 |jstor=2406099}}</ref> |
|||
Adaptation may cause either the gain of a new feature, or the loss of an ancestral feature. An example that shows both types of change is bacterial adaptation to antibiotic selection, with genetic changes causing antibiotic resistance by both modifying the target of the drug, or increasing the activity of transporters that pump the drug out of the cell.<ref>{{cite journal |last1=Nakajima |first1=Akira |last2=Sugimoto |first2=Yohko |last3=Yoneyama |first3=Hiroshi |last4=Nakae |first4=Taiji |date=June 2002 |title=High-Level Fluoroquinolone Resistance in ''Pseudomonas aeruginosa'' Due to Interplay of the MexAB-OprM Efflux Pump and the DNA Gyrase Mutation |journal=Microbiology and Immunology |volume=46 |issue=6 |pages=391–395 |doi=10.1111/j.1348-0421.2002.tb02711.x |issn=1348-0421 |pmid=12153116}}</ref> Other striking examples are the bacteria ''[[Escherichia coli]]'' evolving the ability to use [[citric acid]] as a nutrient in a [[E. coli long-term evolution experiment|long-term laboratory experiment]],<ref>{{cite journal |last1=Blount |first1=Zachary D. |last2=Borland |first2=Christina Z. |last3=Lenski |first3=Richard E. |date=June 10, 2008 |title=Inaugural Article: Historical contingency and the evolution of a key innovation in an experimental population of ''Escherichia coli'' |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=105 |issue=23 |pages=7899–7906 |bibcode=2008PNAS..105.7899B |doi=10.1073/pnas.0803151105 |issn=0027-8424 |pmc=2430337 |pmid=18524956}}</ref> ''[[Flavobacterium]]'' evolving a novel enzyme that allows these bacteria to grow on the by-products of [[nylon]] manufacturing,<ref>{{cite journal |last1=Okada |first1=Hirosuke |last2=Negoro |first2=Seiji |last3=Kimura |first3=Hiroyuki |last4=Nakamura |first4=Shunichi |date=November 10, 1983 |title=Evolutionary adaptation of plasmid-encoded enzymes for degrading nylon oligomers |journal=Nature |volume=306 |issue=5939 |pages=203–206 |bibcode=1983Natur.306..203O |doi=10.1038/306203a0 |issn=0028-0836 |pmid=6646204}}</ref><ref>{{cite journal |last=Ohno |first=Susumu |authorlink=Susumu Ohno |date=April 1984 |title=Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=81 |issue=8 |pages=2421–2425 |bibcode=1984PNAS...81.2421O |doi=10.1073/pnas.81.8.2421 |issn=0027-8424 |pmc=345072 |pmid=6585807}}</ref> and the soil bacterium ''[[Sphingobium]]'' evolving an entirely new [[metabolic pathway]] that degrades the synthetic [[pesticide]] [[pentachlorophenol]].<ref>{{cite journal |last=Copley |first=Shelley D. |date=June 2000 |title=Evolution of a metabolic pathway for degradation of a toxic xenobiotic: the patchwork approach |journal=[[Trends (journals)|Trends in Biochemical Sciences]] |volume=25 |issue=6 |pages=261–265 |doi=10.1016/S0968-0004(00)01562-0 |issn=0968-0004 |pmid=10838562}}</ref><ref>{{cite journal |last1=Crawford |first1=Ronald L. |last2=Jung |first2=Carina M. |last3=Strap |first3=Janice L. |date=October 2007 |title=The recent evolution of pentachlorophenol (PCP)-4-monooxygenase (PcpB) and associated pathways for bacterial degradation of PCP |journal=[[Biodegradation (journal)|Biodegradation]] |volume=18 |issue=5 |pages=525–539 |doi=10.1007/s10532-006-9090-6 |issn=0923-9820 |pmid=17123025}}</ref> An interesting but still controversial idea is that some adaptations might increase the ability of organisms to generate genetic diversity and adapt by natural selection (increasing organisms' evolvability).<ref>{{cite journal |last=Eshel |first=Ilan |date=December 1973 |title=Clone-Selection and Optimal Rates of Mutation |journal=[[Applied Probability Trust|Journal of Applied Probability]] |volume=10 |issue=4 |pages=728–738 |doi=10.2307/3212376 |issn=1475-6072 |jstor=3212376}}</ref><ref>{{harvnb|Altenberg|1995|pp=205–259}}</ref><ref>{{cite journal |last1=Masel |first1=Joanna |last2=Bergman |first2=Aviv |date=July 2003 |title=The evolution of the evolvability properties of the yeast prion [PSI+] |journal=Evolution |volume=57 |issue=7 |pages=1498–1512 |doi=10.1111/j.0014-3820.2003.tb00358.x |issn=0014-3820 |pmid=12940355}}</ref><ref>{{Cite book |last1=Lancaster |first1=Alex K. |last2=Bardill |first2=J. Patrick |last3=True |first3=Heather L. |last4=Masel |first4=Joanna |date=February 2010 |title=The Spontaneous Appearance Rate of the Yeast Prion [''PSI''+] and Its Implications for the Evolution of the Evolvability Properties of the [''PSI''+] System |journal=Genetics |volume=184 |issue=2 |pages=393–400 |doi=10.1534/genetics.109.110213 |issn=0016-6731 |pmc=2828720 |pmid=19917766|bibcode= }}</ref><ref>{{cite journal |last1=Draghi |first1=Jeremy |last2=Wagner |first2=Günter P. |authorlink2=Günter P. Wagner |date=February 2008 |title=Evolution of evolvability in a developmental model |journal=Evolution |volume=62 |issue=2 |pages=301–315 |doi=10.1111/j.1558-5646.2007.00303.x |issn=0014-3820 |pmid=18031304}}</ref> |
|||
[[File:Whale skeleton.png|upright=1.35|thumb|left|A [[baleen whale]] skeleton, ''a'' and ''b'' label [[flipper (anatomy)|flipper]] bones, which were adapted from front [[leg]] bones: while ''c'' indicates [[Vestigiality|vestigial]] leg bones, suggesting an adaptation from land to sea.<ref name="transformation445">{{cite journal |last1=Bejder |first1=Lars |last2=Hall |first2=Brian K. |authorlink2=Brian K. Hall |date=November 2002 |title=Limbs in whales and limblessness in other vertebrates: mechanisms of evolutionary and developmental transformation and loss |journal=Evolution & Development |volume=4 |issue=6 |pages=445–458 |doi=10.1046/j.1525-142X.2002.02033.x |issn=1520-541X |pmid=12492145}}</ref>]] |
|||
Adaptation occurs through the gradual modification of existing structures. Consequently, structures with similar internal organisation may have different functions in related organisms. This is the result of a single ancestral structure being adapted to function in different ways. The bones within [[bat]] wings, for example, are very similar to those in [[mouse|mice]] feet and [[primate]] hands, due to the descent of all these structures from a common mammalian ancestor.<ref>{{cite journal |last1=Young |first1=Nathan M. |last2=HallgrÍmsson |first2=Benedikt |date=December 2005 |title=Serial homology and the evolution of mammalian limb covariation structure |journal=Evolution |volume=59 |issue=12 |pages=2691–2704 |doi=10.1554/05-233.1 |issn=0014-3820 |pmid=16526515}}</ref> However, since all living organisms are related to some extent,<ref name="Penny1999">{{cite journal |last1=Penny |first1=David |last2=Poole |first2=Anthony |date=December 1999 |title=The nature of the last universal common ancestor |journal=Current Opinion in Genetics & Development |volume=9 |issue=6 |pages=672–677 |doi=10.1016/S0959-437X(99)00020-9 |issn=0959-437X |pmid=10607605}}</ref> even organs that appear to have little or no structural similarity, such as [[arthropod]], [[squid]] and [[vertebrate]] eyes, or the limbs and wings of arthropods and vertebrates, can depend on a common set of homologous genes that control their assembly and function; this is called [[deep homology]].<ref>{{cite journal |last=Hall |first=Brian K. |date=August 2003 |title=Descent with modification: the unity underlying homology and homoplasy as seen through an analysis of development and evolution |journal=Biological Reviews |volume=78 |issue=3 |pages=409–433 |doi=10.1017/S1464793102006097 |issn=1464-7931 |pmid=14558591}}</ref><ref>{{cite journal |last1=Shubin |first1=Neil |authorlink1=Neil Shubin |last2=Tabin |first2=Clifford J. |authorlink2=Clifford Tabin |last3=Carroll |first3=Sean B. |date=February 12, 2009 |title=Deep homology and the origins of evolutionary novelty |journal=Nature |volume=457 |issue=7231 |pages=818–823 |bibcode=2009Natur.457..818S |doi=10.1038/nature07891 |issn=0028-0836 |pmid=19212399}}</ref> |
|||
During evolution, some structures may lose their original function and become [[Vestigiality|vestigial structures]].<ref name="Fong">{{cite journal |last1=Fong |first1=Daniel F. |last2=Kane |first2=Thomas C. |last3=Culver |first3=David C. |date=November 1995 |title=Vestigialization and Loss of Nonfunctional Characters |journal=Annual Review of Ecology and Systematics |volume=26 |pages=249–268 |doi=10.1146/annurev.es.26.110195.001341 |issn=1545-2069}}</ref> Such structures may have little or no function in a current species, yet have a clear function in ancestral species, or other closely related species. Examples include [[pseudogene]]s,<ref>{{cite journal |author1=ZhaoLei Zhang |last2=Gerstein |first2=Mark |date=August 2004 |title=Large-scale analysis of pseudogenes in the human genome |journal=Current Opinion in Genetics & Development |volume=14 |issue=4 |pages=328–335 |doi=10.1016/j.gde.2004.06.003 |issn=0959-437X |pmid=15261647}}</ref> the non-functional remains of eyes in blind cave-dwelling fish,<ref>{{cite journal |last=Jeffery |date=May–June 2005 |first1=William R. |title=Adaptive Evolution of Eye Degeneration in the Mexican Blind Cavefish |journal=Journal of Heredity |volume=96 |issue=3 |pages=185–196 |doi=10.1093/jhered/esi028 |issn=0022-1503 |pmid=15653557}}</ref> wings in flightless birds,<ref>{{cite journal |last1=Maxwell |first1=Erin E. |last2=Larsson |first2=Hans C.E. |date=May 2007 |title=Osteology and myology of the wing of the Emu (''Dromaius novaehollandiae'') and its bearing on the evolution of vestigial structures |journal=[[Journal of Morphology]] |volume=268 |issue=5 |pages=423–441 |doi=10.1002/jmor.10527 |issn=0362-2525 |pmid=17390336}}</ref> the presence of hip bones in whales and snakes,<ref name="transformation445" /> and sexual traits in organisms that reproduce via asexual reproduction.<ref>{{cite journal |last1=van der Kooi |first1=Casper J. |last2=Schwander |first2=Tanja |date=November 2014 |title=On the fate of sexual traits under asexuality |url=https://www.researchgate.net/publication/259824406 |format=PDF |journal=Biological Reviews |volume=89 |issue=4 |pages=805–819 |doi=10.1111/brv.12078 |issn=1464-7931 |pmid=24443922 |accessdate=2015-08-05 |deadurl=no |archiveurl=https://web.archive.org/web/20150723175840/http://www.researchgate.net/profile/Tanja_Schwander/publication/259824406_On_the_fate_of_sexual_traits_under_asexuality/links/53ff35a50cf283c3583c85f3.pdf |archivedate=2015-07-23 |df= }}</ref> Examples of [[Human vestigiality|vestigial structures in humans]] include [[Wisdom tooth|wisdom teeth]],<ref>{{cite journal |last1=Silvestri |first1=Anthony R., Jr. |last2=Singh |first2=Iqbal |date=April 2003 |title=The unresolved problem of the third molar: Would people be better off without it? |url=http://jada.ada.org/cgi/content/full/134/4/450 |journal=[[Journal of the American Dental Association]] |volume=134 |issue=4 |pages=450–455 |doi=10.14219/jada.archive.2003.0194 |issn=0002-8177 |pmid=12733778 |archiveurl=https://web.archive.org/web/20140823063158/http://jada.ada.org/content/134/4/450.full |archivedate=2014-08-23 |deadurl=yes |df= }}</ref> the [[coccyx]],<ref name="Fong" /> the [[vermiform appendix]],<ref name="Fong" /> and other behavioural vestiges such as [[goose bumps]]<ref>{{harvnb|Coyne|2009|p=62}}</ref><ref>{{harvnb|Darwin|1872|pp=101, 103}}</ref> and [[primitive reflexes]].<ref>{{harvnb|Gray|2007|p=66}}</ref><ref>{{harvnb|Coyne|2009|pp=85–86}}</ref><ref>{{harvnb|Stevens|1982|p=87}}</ref> |
|||
However, many traits that appear to be simple adaptations are in fact [[exaptation]]s: structures originally adapted for one function, but which coincidentally became somewhat useful for some other function in the process.{{sfn|Gould|2002|pp=1235–1236}} One example is the African lizard ''Holaspis guentheri'', which developed an extremely flat head for hiding in crevices, as can be seen by looking at its near relatives. However, in this species, the head has become so flattened that it assists in gliding from tree to tree—an exaptation.{{sfn|Gould|2002|pp=1235–1236}} Within cells, [[molecular machine]]s such as the bacterial [[Flagellum|flagella]]<ref>{{cite journal |last=Pallen |first=Mark J. |last2=Matzke |first2=Nicholas J. |date=October 2006 |title=From ''The Origin of Species'' to the origin of bacterial flagella |url=https://www.ocf.berkeley.edu/~matzke/matzke_cv/_pubs/Pallen_Matzke_2006_NRM_origin_flagella.pdf |type=PDF |journal=Nature Reviews Microbiology |volume=4 |issue=10 |pages=784–790 |doi=10.1038/nrmicro1493 |issn=1740-1526 |pmid=16953248 |accessdate=2014-12-25 |deadurl=yes |archiveurl=https://web.archive.org/web/20141226013207/https://www.ocf.berkeley.edu/~matzke/matzke_cv/_pubs/Pallen_Matzke_2006_NRM_origin_flagella.pdf |archivedate=2014-12-26 |df= }}</ref> and [[translocase of the inner membrane|protein sorting machinery]]<ref>{{cite journal |last1=Clements |first1=Abigail |last2=Bursac |first2=Dejan |last3=Gatsos |first3=Xenia |last4=Perry |first4=Andrew J. |last5=Civciristov |first5=Srgjan |last6=Celik |first6=Nermin |last7=Likic |first7=Vladimir A. |last8=Poggio |first8=Sebastian |last9=Jacobs-Wagner |first9=Christine |last10=Strugnell |first10=Richard A. |last11=Lithgow |first11=Trevor |date=September 15, 2009 |title=The reducible complexity of a mitochondrial molecular machine |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=106 |issue=37 |pages=15791–15795 |bibcode=2009PNAS..10615791C |doi=10.1073/pnas.0908264106 |issn=0027-8424 |pmid=19717453 |pmc=2747197 |display-authors=3}}</ref> evolved by the recruitment of several pre-existing proteins that previously had different functions.<ref name="ScottEC" /> Another example is the recruitment of enzymes from [[glycolysis]] and [[Drug metabolism|xenobiotic metabolism]] to serve as structural proteins called [[crystallin]]s within the lenses of organisms' eyes.<ref>{{harvnb|Piatigorsky|Kantorow|Gopal-Srivastava|Tomarev|1994|pp=241–250}}</ref><ref>{{cite journal |last=Wistow |first=Graeme |date=August 1993 |title=Lens crystallins: gene recruitment and evolutionary dynamism |journal=Trends in Biochemical Sciences |volume=18 |issue=8 |pages=301–306 |doi=10.1016/0968-0004(93)90041-K |issn=0968-0004 |pmid=8236445}}</ref> |
|||
An area of current investigation in evolutionary developmental biology is the developmental basis of adaptations and exaptations.<ref>{{cite journal |last1=Johnson |first1=Norman A. |last2=Porter |first2=Adam H. |date=November 2001 |title=Toward a new synthesis: population genetics and evolutionary developmental biology |journal=Genetica |volume='''112–113''' |issue=1 |pages=45–58 |doi=10.1023/A:1013371201773 |issn=0016-6707 |pmid=11838782}}</ref> This research addresses the origin and evolution of [[Embryogenesis|embryonic development]] and how modifications of development and developmental processes produce novel features.<ref>{{cite journal |last1=Baguñà |first1=Jaume |last2=Garcia-Fernàndez |first2=Jordi |year=2003 |title=Evo-Devo: the long and winding road |url=http://www.ijdb.ehu.es/web/paper.php?doi=14756346 |journal=[[The International Journal of Developmental Biology]] |volume=47 |issue=7–8 |pages=705–713 |issn=0214-6282 |pmid=14756346 |deadurl=no |archiveurl=https://web.archive.org/web/20141128011936/http://www.ijdb.ehu.es/web/paper.php?doi=14756346 |archivedate=2014-11-28 |df= }} |
|||
* {{cite journal |last=Love |first=Alan C. |date=March 2003 |title=Evolutionary Morphology, Innovation and the Synthesis of Evolutionary and Developmental Biology |journal=Biology and Philosophy |volume=18 |issue=2 |pages=309–345 |doi=10.1023/A:1023940220348 |issn=0169-3867}}</ref> These studies have shown that evolution can alter development to produce new structures, such as embryonic bone structures that develop into the jaw in other animals instead forming part of the [[Evolution of mammalian auditory ossicles|middle ear in mammals]].<ref>{{cite journal |last=Allin |first=Edgar F. |date=December 1975 |title=Evolution of the mammalian middle ear |journal=Journal of Morphology |volume=147 |issue=4 |pages=403–437 |doi=10.1002/jmor.1051470404 |issn=0362-2525 |pmid=1202224}}</ref> It is also possible for structures that have been lost in evolution to reappear due to changes in developmental genes, such as a mutation in chickens causing embryos to grow teeth similar to those of [[crocodile]]s.<ref>{{cite journal |last1=Harris |first1=Matthew P. |last2=Hasso |first2=Sean M. |last3=Ferguson |first3=Mark W.J. |last4=Fallon |first4=John F. |date=February 21, 2006 |title=The Development of Archosaurian First-Generation Teeth in a Chicken Mutant |journal=Current Biology |volume=16 |issue=4 |pages=371–377 |doi=10.1016/j.cub.2005.12.047 |issn=0960-9822 |pmid=16488870|bibcode=1996CBio....6.1213A }}</ref> It is now becoming clear that most alterations in the form of organisms are due to changes in a small set of conserved genes.<ref>{{cite journal |last=Carroll |first=Sean B. |date=July 11, 2008 |title=Evo-Devo and an Expanding Evolutionary Synthesis: A Genetic Theory of Morphological Evolution |journal=[[Cell (journal)|Cell]] |volume=134 |issue=1 |pages=25–36 |doi=10.1016/j.cell.2008.06.030 |issn=0092-8674 |pmid=18614008}}</ref> |
|||
=== Coevolution === |
|||
[[File:Thamnophis sirtalis sirtalis Wooster.jpg|thumb|[[Common Garter Snake|Common garter snake]] (''Thamnophis sirtalis sirtalis'') has evolved resistance to the [[anti-predator adaptation|defensive substance]] [[tetrodotoxin]] in its amphibian prey.]] |
|||
{{Further|Coevolution}} |
|||
Interactions between organisms can produce both conflict and cooperation. When the interaction is between pairs of species, such as a [[pathogen]] and a [[host (biology)|host]], or a predator and its prey, these species can develop matched sets of adaptations. Here, the evolution of one species causes adaptations in a second species. These changes in the second species then, in turn, cause new adaptations in the first species. This cycle of selection and response is called coevolution.<ref>{{cite journal |last=Wade |first=Michael J. |authorlink=Michael J. Wade |date=March 2007 |title=The co-evolutionary genetics of ecological communities |journal=Nature Reviews Genetics |volume=8 |issue=3 |pages=185–195 |doi=10.1038/nrg2031 |issn=1471-0056 |pmid=17279094}}</ref> An example is the production of [[tetrodotoxin]] in the [[rough-skinned newt]] and the evolution of tetrodotoxin resistance in its predator, the [[Common Garter Snake|common garter snake]]. In this predator-prey pair, an [[evolutionary arms race]] has produced high levels of toxin in the newt and correspondingly high levels of toxin resistance in the snake.<ref>{{cite journal |last1=Geffeney |first1=Shana |last2=Brodie |first2=Edmund D., Jr. |last3=Ruben |first3=Peter C. |last4=Brodie |first4=Edmund D., III |date=August 23, 2002 |title=Mechanisms of Adaptation in a Predator-Prey Arms Race: TTX-Resistant Sodium Channels |journal=Science |volume=297 |issue=5585 |pages=1336–1339 |bibcode=2002Sci...297.1336G |doi=10.1126/science.1074310 |issn=0036-8075 |pmid=12193784}} |
|||
* {{cite journal |last1=Brodie |first1=Edmund D., Jr. |last2=Ridenhour |first2=Benjamin J. |last3=Brodie |first3=Edmund D., III |date=October 2002 |title=The evolutionary response of predators to dangerous prey: hotspots and coldspots in the geographic mosaic of coevolution between garter snakes and newts |journal=Evolution |volume=56 |issue=10 |pages=2067–2082 |doi=10.1554/0014-3820(2002)056[2067:teropt]2.0.co;2 |issn=0014-3820 |pmid=12449493}} |
|||
* {{cite news |last=Carroll |first=Sean B. |date=December 21, 2009 |title=Whatever Doesn't Kill Some Animals Can Make Them Deadly |url=https://www.nytimes.com/2009/12/22/science/22creature.html |newspaper=The New York Times |location=New York |publisher=The New York Times Company |issn=0362-4331 |accessdate=2014-12-26 |deadurl=no |archiveurl=https://web.archive.org/web/20150423075609/http://www.nytimes.com/2009/12/22/science/22creature.html |archivedate=2015-04-23 |df= }}</ref> |
|||
=== Cooperation === |
|||
{{Further|Co-operation (evolution)}} |
|||
Not all co-evolved interactions between species involve conflict.<ref>{{cite journal |last=Sachs |first=Joel L. |date=September 2006 |title=Cooperation within and among species |journal=Journal of Evolutionary Biology |volume=19 |issue=5 |pages=1415–1418; discussion 1426–1436 |doi=10.1111/j.1420-9101.2006.01152.x |issn=1010-061X |pmid=16910971}} |
|||
* {{cite journal |last=Nowak |first=Martin A. |authorlink=Martin Nowak |date=December 8, 2006 |title=Five Rules for the Evolution of Cooperation |journal=Science |volume=314 |issue=5805 |pages=1560–1563 |bibcode=2006Sci...314.1560N |doi=10.1126/science.1133755 |issn=0036-8075 |pmc=3279745 |pmid=17158317}}</ref> Many cases of mutually beneficial interactions have evolved. For instance, an extreme cooperation exists between plants and the [[Mycorrhiza|mycorrhizal fungi]] that grow on their roots and aid the plant in absorbing nutrients from the soil.<ref>{{cite journal |last=Paszkowski |first=Uta |date=August 2006 |title=Mutualism and parasitism: the yin and yang of plant symbioses |journal=[[Current Opinion (Elsevier)|Current Opinion in Plant Biology]] |volume=9 |issue=4 |pages=364–370 |doi=10.1016/j.pbi.2006.05.008 |issn=1369-5266 |pmid=16713732}}</ref> This is a [[Reciprocity (evolution)|reciprocal]] relationship as the plants provide the fungi with sugars from [[photosynthesis]]. Here, the fungi actually grow inside plant cells, allowing them to exchange nutrients with their hosts, while sending [[signal transduction|signals]] that suppress the plant [[immune system]].<ref>{{cite journal |last1=Hause |first1=Bettina |last2=Fester |first2=Thomas |date=May 2005 |title=Molecular and cell biology of arbuscular mycorrhizal symbiosis |journal=[[Planta (journal)|Planta]] |volume=221 |issue=2 |pages=184–196 |doi=10.1007/s00425-004-1436-x |issn=0032-0935 |pmid=15871030}}</ref> |
|||
Coalitions between organisms of the same species have also evolved. An extreme case is the [[eusociality]] found in social insects, such as [[bee]]s, [[termite]]s and [[ant]]s, where sterile insects feed and guard the small number of organisms in a [[Colony (biology)|colony]] that are able to reproduce. On an even smaller scale, the somatic cells that make up the body of an animal limit their reproduction so they can maintain a stable organism, which then supports a small number of the animal's germ cells to produce offspring. Here, somatic cells respond to specific signals that instruct them whether to grow, remain as they are, or die. If cells ignore these signals and multiply inappropriately, their uncontrolled growth [[carcinogenesis|causes cancer]].<ref name="Bertram">{{cite journal |last=Bertram |first=John S. |date=December 2000 |title=The molecular biology of cancer |journal=[[Molecular Aspects of Medicine]] |volume=21 |issue=6 |pages=167–223 |doi=10.1016/S0098-2997(00)00007-8 |issn=0098-2997 |pmid=11173079}}</ref> |
|||
Such cooperation within species may have evolved through the process of [[kin selection]], which is where one organism acts to help raise a relative's offspring.<ref>{{cite journal |last1=Reeve |first1=H. Kern |last2=Hölldobler |first2=Bert |authorlink2=Bert Hölldobler |date=June 5, 2007 |title=The emergence of a superorganism through intergroup competition |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=104 |issue=23 |pages=9736–9740 |bibcode=2007PNAS..104.9736R |doi=10.1073/pnas.0703466104 |issn=0027-8424 |pmc=1887545 |pmid=17517608}}</ref> This activity is selected for because if the ''helping'' individual contains alleles which promote the helping activity, it is likely that its kin will ''also'' contain these alleles and thus those alleles will be passed on.<ref>{{cite journal |last1=Axelrod |first=Robert |last2=Hamilton |first2=W. D. |date=March 27, 1981 |title=The evolution of cooperation |journal=Science |volume=211 |issue=4489 |pages=1390–1396 |bibcode=1981Sci...211.1390A |doi=10.1126/science.7466396 |issn=0036-8075 |pmid=7466396}}</ref> Other processes that may promote cooperation include [[group selection]], where cooperation provides benefits to a group of organisms.<ref>{{cite journal |last1=Wilson |first1=Edward O. |last2=Hölldobler |first2=Bert |date=September 20, 2005 |title=Eusociality: Origin and consequences |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=102 |issue=38 |pages=13367–1371 |bibcode=2005PNAS..10213367W |doi=10.1073/pnas.0505858102 |issn=0027-8424 |pmc=1224642 |pmid=16157878}}</ref> |
|||
=== Speciation === |
|||
{{main|Speciation}} |
|||
{{Further|Assortative mating|Panmixia}} |
|||
[[File:Speciation modes edit.svg|left|thumb|upright=1.6|The four geographic modes of [[speciation]]]] |
|||
Speciation is the process where a species diverges into two or more descendant species.<ref name="Gavrilets">{{cite journal |last=Gavrilets |first=Sergey |date=October 2003 |title=Perspective: models of speciation: what have we learned in 40 years? |journal=Evolution |volume=57 |issue=10 |pages=2197–2215 |doi=10.1554/02-727 |issn=0014-3820 |pmid=14628909}}</ref> |
|||
There are multiple ways to define the concept of "species." The choice of definition is dependent on the particularities of the species concerned.<ref name="Queiroz">{{cite journal |last=de Queiroz |first=Kevin |date=May 3, 2005 |title=Ernst Mayr and the modern concept of species |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=102 |issue=Suppl. 1 |pages=6600–6607 |bibcode=2005PNAS..102.6600D |doi=10.1073/pnas.0502030102 |issn=0027-8424 |pmc=1131873 |pmid=15851674}}</ref> For example, some species concepts apply more readily toward sexually reproducing organisms while others lend themselves better toward asexual organisms. Despite the diversity of various species concepts, these various concepts can be placed into one of three broad philosophical approaches: interbreeding, ecological and phylogenetic.<ref name="Ereshefsky92">{{cite journal |last=Ereshefsky |first=Marc |authorlink=Marc Ereshefsky |date=December 1992 |title=Eliminative pluralism |journal=[[Philosophy of Science (journal)|Philosophy of Science]] |volume=59 |issue=4 |pages=671–690 |doi=10.1086/289701 |issn=0031-8248 |jstor=188136}}</ref> The ''Biological Species Concept'' (BSC) is a classic example of the interbreeding approach. Defined by evolutionary biologist [[Ernst Mayr]] in 1942, the BSC states that "species are groups of actually or potentially interbreeding natural populations, which are reproductively isolated from other such groups."<ref>{{harvnb|Mayr|1942|p=120}}</ref> Despite its wide and long-term use, the BSC like others is not without controversy, for example because these concepts cannot be applied to prokaryotes,<ref>{{cite journal |last1=Fraser |first1=Christophe |last2=Alm |first2=Eric J. |last3=Polz |first3=Martin F. |last4=Spratt |first4=Brian G. |last5=Hanage |first5=William P. |date=February 6, 2009 |title=The Bacterial Species Challenge: Making Sense of Genetic and Ecological Diversity |journal=Science |volume=323 |issue=5915 |pages=741–746 |bibcode=2009Sci...323..741F |doi=10.1126/science.1159388 |issn=0036-8075 |pmid=19197054 |display-authors=3}}</ref> and this is called the [[species problem]].<ref name="Queiroz" /> Some researchers have attempted a unifying monistic definition of species, while others adopt a pluralistic approach and suggest that there may be different ways to logically interpret the definition of a species.<ref name="Queiroz" /><ref name="Ereshefsky92" /> |
|||
[[Reproductive isolation|Barriers to reproduction]] between two diverging sexual populations are required for the populations to become new species. Gene flow may slow this process by spreading the new genetic variants also to the other populations. Depending on how far two species have diverged since their [[most recent common ancestor]], it may still be possible for them to produce offspring, as with horses and [[donkey]]s mating to produce [[mule]]s.<ref>{{cite journal |last=Short |first=Roger Valentine |date=October 1975 |title=The contribution of the mule to scientific thought |journal=Journal of Reproduction and Fertility. Supplement |issue=23 |pages=359–364 |issn=0449-3087 |oclc=1639439 |pmid=1107543}}</ref> Such hybrids are generally [[infertility|infertile]]. In this case, closely related species may regularly interbreed, but hybrids will be selected against and the species will remain distinct. However, viable hybrids are occasionally formed and these new species can either have properties intermediate between their parent species, or possess a totally new phenotype.<ref>{{cite journal |last1=Gross |first1=Briana L. |last2=Rieseberg |first2=Loren H. |date=May–June 2005 |title=The Ecological Genetics of Homoploid Hybrid Speciation |journal=Journal of Heredity |volume=96 |issue=3 |pages=241–252 |doi=10.1093/jhered/esi026 |issn=0022-1503 |pmc=2517139 |pmid=15618301}}</ref> The importance of hybridisation in producing [[hybrid speciation|new species]] of animals is unclear, although cases have been seen in many types of animals,<ref>{{cite journal |last1=Burke |first1=John M. |last2=Arnold |first2=Michael L. |date=December 2001 |title=Genetics and the fitness of hybrids |journal=Annual Review of Genetics |volume=35 |pages=31–52 |doi=10.1146/annurev.genet.35.102401.085719 |issn=0066-4197 |pmid=11700276}}</ref> with the [[gray tree frog]] being a particularly well-studied example.<ref>{{cite journal |last=Vrijenhoek |first=Robert C. |date=April 4, 2006 |title=Polyploid Hybrids: Multiple Origins of a Treefrog Species |journal=Current Biology |volume=16 |issue=7 |pages=R245–R247 |doi=10.1016/j.cub.2006.03.005 |issn=0960-9822 |pmid=16581499|bibcode=1996CBio....6.1213A }}</ref> |
|||
Speciation has been observed multiple times under both controlled laboratory conditions (see [[laboratory experiments of speciation]]) and in nature.<ref>{{cite journal |last1=Rice |first1=William R. |last2=Hostert |first2=Ellen E. |date=December 1993 |title=Laboratory Experiments on Speciation: What Have We Learned in 40 Years? |journal=Evolution |volume=47 |issue=6 |pages=1637–1653 |doi=10.2307/2410209 |pmid=28568007 |issn=0014-3820|jstor=2410209 }} |
|||
* {{cite journal |last1=Jiggins |first1=Chris D. |last2=Bridle |first2=Jon R. |date=March 2004 |title=Speciation in the apple maggot fly: a blend of vintages? |journal=Trends in Ecology & Evolution |volume=19 |issue=3 |pages=111–114 |doi=10.1016/j.tree.2003.12.008 |pmid=16701238 |issn=0169-5347}} |
|||
* {{cite web |url=http://www.talkorigins.org/faqs/faq-speciation.html |title=Observed Instances of Speciation |last=Boxhorn |first=Joseph |date=September 1, 1995 |website=TalkOrigins Archive |publisher=The TalkOrigins Foundation, Inc. |location=Houston, Texas |accessdate=2008-12-26 |deadurl=no |archiveurl=https://web.archive.org/web/20090122211743/http://talkorigins.org/faqs/faq-speciation.html |archivedate=2009-01-22 |df= }} |
|||
* {{cite journal |last1=Weinberg |first1=James R. |last2=Starczak |first2=Victoria R. |last3=Jörg |first3=Daniele |date=August 1992 |title=Evidence for Rapid Speciation Following a Founder Event in the Laboratory |journal=Evolution |volume=46 |issue=4 |pages=1214–1220 |doi=10.2307/2409766 |pmid=28564398 |issn=0014-3820 |jstor=2409766}}</ref> In sexually reproducing organisms, speciation results from reproductive isolation followed by genealogical divergence. There are four primary geographic modes of speciation. The most common in animals is [[allopatric speciation]], which occurs in populations initially isolated geographically, such as by [[habitat fragmentation]] or migration. Selection under these conditions can produce very rapid changes in the appearance and behaviour of organisms.<ref>{{cite journal |last1=Herrel |first1=Anthony |last2=Huyghe |first2=Katleen |last3=Vanhooydonck |first3=Bieke |last4=Backeljau |first4=Thierry |last5=Breugelmans |first5=Karin |last6=Grbac |first6=Irena |last7=Van Damme |first7=Raoul |last8=Irschick |first8=Duncan J. |date=March 25, 2008 |title=Rapid large-scale evolutionary divergence in morphology and performance associated with exploitation of a different dietary resource |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=105 |issue=12 |pages=4792–4795 |bibcode=2008PNAS..105.4792H |doi=10.1073/pnas.0711998105 |issn=0027-8424 |pmc=2290806 |pmid=18344323 |display-authors=3}}</ref><ref name="Losos1997">{{cite journal |last1=Losos |first1=Jonathan B. |last2=Warhelt |first2=Kenneth I. |last3=Schoener |first3=Thomas W. |date=May 1, 1997 |title=Adaptive differentiation following experimental island colonization in ''Anolis'' lizards |journal=Nature |volume=387 |issue=6628 |pages=70–73 |bibcode=1997Natur.387...70L |doi=10.1038/387070a0 |issn=0028-0836}}</ref> As selection and drift act independently on populations isolated from the rest of their species, separation may eventually produce organisms that cannot interbreed.<ref>{{cite journal |last1=Hoskin |first1=Conrad J. |last2=Higgle |first2=Megan |last3=McDonald |first3=Keith R. |last4=Moritz |first4=Craig |date=October 27, 2005 |title=Reinforcement drives rapid allopatric speciation |journal=Nature |pmid=16251964 |volume=437 |issue=7063 |pages=1353–1356 |bibcode=2005Natur.437.1353H |doi=10.1038/nature04004 |issn=0028-0836}}</ref> |
|||
The second mode of speciation is [[peripatric speciation]], which occurs when small populations of organisms become isolated in a new environment. This differs from allopatric speciation in that the isolated populations are numerically much smaller than the parental population. Here, the [[founder effect]] causes rapid speciation after an increase in [[inbreeding]] increases selection on homozygotes, leading to rapid genetic change.<ref>{{cite journal |last=Templeton |first=Alan R. |authorlink=Alan Templeton |date=April 1980 |title=The Theory of Speciation ''VIA'' the Founder Principle |url=http://www.genetics.org/content/94/4/1011.full.pdf+html |journal=Genetics |volume=94 |issue=4 |pages=1011–1038 |pmid=6777243 |issn=0016-6731 |pmc=1214177 |accessdate=2014-12-29 |deadurl=no |archiveurl=https://web.archive.org/web/20140823063455/http://www.genetics.org/content/94/4/1011.full.pdf+html |archivedate=2014-08-23 |df= }}</ref> |
|||
The third mode is [[parapatric speciation]]. This is similar to peripatric speciation in that a small population enters a new habitat, but differs in that there is no physical separation between these two populations. Instead, speciation results from the evolution of mechanisms that reduce gene flow between the two populations.<ref name="Gavrilets" /> Generally this occurs when there has been a drastic change in the environment within the parental species' habitat. One example is the grass ''[[Anthoxanthum|Anthoxanthum odoratum]]'', which can undergo parapatric speciation in response to localised metal pollution from mines.<ref>{{cite journal |last=Antonovics |first=Janis |authorlink=Janis Antonovics |date=July 2006 |title=Evolution in closely adjacent plant populations X: long-term persistence of prereproductive isolation at a mine boundary |url=http://www.nature.com/hdy/journal/v97/n1/full/6800835a.html |journal=[[Heredity (journal)|Heredity]] |volume=97 |issue=1 |pages=33–37 |doi=10.1038/sj.hdy.6800835 |issn=0018-067X |pmid=16639420 |accessdate=2014-12-29 |deadurl=no |archiveurl=https://web.archive.org/web/20140823063443/http://www.nature.com/hdy/journal/v97/n1/full/6800835a.html |archivedate=2014-08-23 |df= }}</ref> Here, plants evolve that have resistance to high levels of metals in the soil. Selection against interbreeding with the metal-sensitive parental population produced a gradual change in the flowering time of the metal-resistant plants, which eventually produced complete reproductive isolation. Selection against hybrids between the two populations may cause [[Reinforcement (speciation)|reinforcement]], which is the evolution of traits that promote mating within a species, as well as [[character displacement]], which is when two species become more distinct in appearance.<ref>{{cite journal |last1=Nosil |first1=Patrik |last2=Crespi |first2=Bernard J. |last3=Gries |first3=Regine |last4=Gries |first4=Gerhard |date=March 2007 |title=Natural selection and divergence in mate preference during speciation |journal=Genetica |volume=129 |issue=3 |pages=309–327 |doi=10.1007/s10709-006-0013-6 |pmid=16900317 |issn=0016-6707}}</ref> |
|||
[[File:Darwin's finches.jpeg|frame|[[Geographical isolation]] of [[Darwin's finches|finches]] on the [[Galápagos Islands]] produced over a dozen new species.]] |
|||
Finally, in [[sympatric speciation]] species diverge without geographic isolation or changes in habitat. This form is rare since even a small amount of gene flow may remove genetic differences between parts of a population.<ref>{{cite journal |last1=Savolainen |first1=Vincent |last2=Anstett |first2=Marie-Charlotte |last3=Lexer |first3=Christian |last4=Hutton |first4=Ian |last5=Clarkson |first5=James J. |last6=Norup |first6=Maria V. |last7=Powell |first7=Martyn P. |last8=Springate |first8=David |last9=Salamin |first9=Nicolas |last10=Baker |first10=William J. |date=May 11, 2006 |title=Sympatric speciation in palms on an oceanic island |journal=Nature |volume=441 |issue=7090 |pages=210–213 |bibcode=2006Natur.441..210S |doi=10.1038/nature04566 |issn=0028-0836 |pmid=16467788 |display-authors=3}} |
|||
* {{cite journal |last1=Barluenga |first1=Marta |last2=Stölting |first2=Kai N. |last3=Salzburger |first3=Walter |last4=Muschick |first4=Moritz |last5=Meyer |first5=Axel |authorlink5=Axel Meyer |date=February 9, 2006 |title=Sympatric speciation in Nicaraguan crater lake cichlid fish |journal=Nature |volume=439 |issue=7077 |pages=719–23 |bibcode=2006Natur.439..719B |doi=10.1038/nature04325 |issn=0028-0836 |pmid=16467837 |display-authors=3|url=http://nbn-resolving.de/urn:nbn:de:bsz:352-opus-34004 }}</ref> Generally, sympatric speciation in animals requires the evolution of both [[Polymorphism (biology)|genetic differences]] and [[Assortative mating|nonrandom mating]], to allow reproductive isolation to evolve.<ref>{{cite journal |last=Gavrilets |first=Sergey |date=March 21, 2006 |title=The Maynard Smith model of sympatric speciation |journal=Journal of Theoretical Biology |volume=239 |issue=2 |pages=172–182 |doi=10.1016/j.jtbi.2005.08.041 |issn=0022-5193 |pmid=16242727}}</ref> |
|||
One type of sympatric speciation involves [[crossbreed]]ing of two related species to produce a new hybrid species. This is not common in animals as animal hybrids are usually sterile. This is because during [[meiosis]] the [[homologous chromosome]]s from each parent are from different species and cannot successfully pair. However, it is more common in plants because plants often double their number of chromosomes, to form [[polyploidy|polyploids]].<ref>{{cite journal |last1=Wood |first1=Troy E. |last2=Takebayashi |first2=Naoki |last3=Barker |first3=Michael S. |last4=Mayrose |first4=Itay |last5=Greenspoon |first5=Philip B. |last6=Rieseberg |first6=Loren H. |date=August 18, 2009 |title=The frequency of polyploid speciation in vascular plants |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=106 |issue=33 |pages=13875–13879 |bibcode=2009PNAS..10613875W |doi=10.1073/pnas.0811575106 |issn=0027-8424 |pmc=2728988 |pmid=19667210 |display-authors=3}}</ref> This allows the chromosomes from each parental species to form matching pairs during meiosis, since each parent's chromosomes are represented by a pair already.<ref>{{cite journal |last1=Hegarty |first1=Matthew J. |last2=Hiscock |first2=Simon J. |date=May 20, 2008 |title=Genomic Clues to the Evolutionary Success of Polyploid Plants |journal=Current Biology |volume=18 |issue=10 |pages=R435–R444 |doi=10.1016/j.cub.2008.03.043 |issn=0960-9822 |pmid=18492478|bibcode=1996CBio....6.1213A }}</ref> An example of such a speciation event is when the plant species ''[[Arabidopsis thaliana]]'' and ''[[Arabidopsis arenosa]]'' crossbred to give the new species ''Arabidopsis suecica''.<ref>{{cite journal |last1=Jakobsson |first1=Mattias |last2=Hagenblad |first2=Jenny |last3=Tavaré |first3=Simon |authorlink3=Simon Tavaré |last4=Säll |first4=Torbjörn |last5=Halldén |first5=Christer |last6=Lind-Halldén |first6=Christina |last7=Nordborg |first7=Magnus |date=June 2006 |title=A Unique Recent Origin of the Allotetraploid Species ''Arabidopsis suecica'': Evidence from Nuclear DNA Markers |journal=Molecular Biology and Evolution |volume=23 |issue=6 |pages=1217–1231 |doi=10.1093/molbev/msk006 |issn=0737-4038 |pmid=16549398 |display-authors=3}}</ref> This happened about 20,000 years ago,<ref>{{cite journal |last=Säll |first1=Torbjörn |last2=Jakobsson |first2=Mattias |last3=Lind-Halldén |first3=Christina |last4=Halldén |first4=Christer |date=September 2003 |title=Chloroplast DNA indicates a single origin of the allotetraploid ''Arabidopsis suecica'' |journal=Journal of Evolutionary Biology |volume=16 |issue=5 |pages=1019–1029 |doi=10.1046/j.1420-9101.2003.00554.x |issn=1010-061X |pmid=14635917}}</ref> and the speciation process has been repeated in the laboratory, which allows the study of the genetic mechanisms involved in this process.<ref>{{cite journal |last1=Bomblies |first1=Kirsten |authorlink1=Kirsten Bomblies |last2=Weigel |first2=Detlef |authorlink2=Detlef Weigel |date=December 2007 |title=''Arabidopsis''—a model genus for speciation |journal=Current Opinion in Genetics & Development |volume=17 |issue=6 |pages=500–504 |doi=10.1016/j.gde.2007.09.006 |issn=0959-437X |pmid=18006296}}</ref> Indeed, chromosome doubling within a species may be a common cause of reproductive isolation, as half the doubled chromosomes will be unmatched when breeding with undoubled organisms.<ref name="Semon">{{cite journal |last1=Sémon |first1=Marie |last2=Wolfe |first2=Kenneth H. |date=December 2007 |title=Consequences of genome duplication |journal=Current Opinion in Genetics & Development |volume=17 |issue=6 |pages=505–512 |doi=10.1016/j.gde.2007.09.007 |issn=0959-437X |pmid=18006297}}</ref> |
|||
Speciation events are important in the theory of [[punctuated equilibrium]], which accounts for the pattern in the fossil record of short "bursts" of evolution interspersed with relatively long periods of stasis, where species remain relatively unchanged.<ref>{{harvnb|Eldredge|Gould|1972|pp=82–115}}</ref> In this theory, speciation and rapid evolution are linked, with natural selection and genetic drift acting most strongly on organisms undergoing speciation in novel habitats or small populations. As a result, the periods of stasis in the fossil record correspond to the parental population and the organisms undergoing speciation and rapid evolution are found in small populations or geographically restricted habitats and therefore rarely being preserved as fossils.<ref name="Gould_1994" /> |
|||
=== Extinction === |
|||
{{Further|Extinction}} |
|||
[[File:Palais de la Decouverte Tyrannosaurus rex p1050042.jpg|thumb|left|''[[Tyrannosaurus rex]]''. Non-[[bird|avian]] [[dinosaur]]s died out in the [[Cretaceous–Paleogene extinction event]] at the end of the [[Cretaceous]] period.]] |
|||
Extinction is the disappearance of an entire species. Extinction is not an unusual event, as species regularly appear through speciation and disappear through extinction.<ref>{{cite journal |last1=Benton |first1=Michael J. |authorlink=Michael Benton |date=April 7, 1995 |title=Diversification and extinction in the history of life |journal=Science |volume=268 |issue=5207 |pages=52–58 |bibcode=1995Sci...268...52B |doi=10.1126/science.7701342 |issn=0036-8075 |pmid=7701342}}</ref> Nearly all animal and plant species that have lived on Earth are now extinct,<ref>{{cite journal |last=Raup |first=David M. |authorlink=David M. Raup |date=March 28, 1986 |title=Biological extinction in Earth history |journal=Science |volume=231 |issue=4745 |pages=1528–1533 |bibcode=1986Sci...231.1528R |doi=10.1126/science.11542058 |issn=0036-8075 |pmid=11542058}}</ref> and extinction appears to be the ultimate fate of all species.<ref>{{cite journal |last1=Avise |first1=John C. |last2=Hubbell |first2=Stephen P. |authorlink2=Stephen P. Hubbell |last3=Ayala |first3=Francisco J. |date=August 12, 2008 |title=In the light of evolution II: Biodiversity and extinction |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=105 |issue=Suppl. 1 |pages=11453–11457 |bibcode=2008PNAS..10511453A |doi=10.1073/pnas.0802504105 |issn=0027-8424 |pmc=2556414 |pmid=18695213}}</ref> These extinctions have happened continuously throughout the history of life, although the rate of extinction spikes in occasional mass [[extinction event]]s.<ref name="Raup_1994">{{cite journal |last=Raup |first=David M. |date=July 19, 1994 |title=The role of extinction in evolution |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=91 |issue=15 |pages=6758–6763 |bibcode=1994PNAS...91.6758R |doi=10.1073/pnas.91.15.6758 |issn=0027-8424 |pmc=44280 |pmid=8041694}}</ref> The [[Cretaceous–Paleogene extinction event]], during which the non-avian dinosaurs became extinct, is the most well-known, but the earlier [[Permian–Triassic extinction event]] was even more severe, with approximately 96% of all marine species driven to extinction.<ref name="Raup_1994" /> The [[Holocene extinction|Holocene extinction event]] is an ongoing mass extinction associated with humanity's expansion across the globe over the past few thousand years. Present-day extinction rates are 100–1000 times greater than the background rate and up to 30% of current species may be extinct by the mid 21st century.<ref>{{cite journal |last1=Novacek |first1=Michael J. |last2=Cleland |first2=Elsa E. |date=May 8, 2001 |title=The current biodiversity extinction event: scenarios for mitigation and recovery |doi=10.1073/pnas.091093698 |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=98 |issue=10 |pages=5466–5470 |bibcode=2001PNAS...98.5466N |issn=0027-8424 |pmc=33235 |pmid=11344295}}</ref> Human activities are now the primary cause of the ongoing extinction event;<ref>{{cite journal |last1=Pimm |first1=Stuart |authorlink1=Stuart Pimm |last2=Raven |first2=Peter |authorlink2=Peter H. Raven |last3=Peterson |first3=Alan |last4=Şekercioğlu |first4=Çağan H. |last5=Ehrlich |first5=Paul R. |authorlink5=Paul R. Ehrlich |date=July 18, 2006 |title=Human impacts on the rates of recent, present and future bird extinctions |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=103 |issue=29 |pages=10941–10946 |bibcode=2006PNAS..10310941P |doi=10.1073/pnas.0604181103 |issn=0027-8424 |pmc=1544153 |pmid=16829570 |display-authors=3}} |
|||
* {{cite journal |last1=Barnosky |first1=Anthony D. |last2=Koch |first2=Paul L. |last3=Feranec |first3=Robert S. |last4=Wing |first4=Scott L. |last5=Shabel |first5=Alan B. |date=October 1, 2004 |title=Assessing the Causes of Late Pleistocene Extinctions on the Continents |journal=Science |volume=306 |issue=5693 |pages=70–75 |bibcode=2004Sci...306...70B |doi=10.1126/science.1101476 |issn=0036-8075 |pmid=15459379 |display-authors=3|citeseerx=10.1.1.574.332 }}</ref> [[global warming]] may further accelerate it in the future.<ref>{{cite journal |last1=Lewis |first1=Owen T. |date=January 29, 2006 |title=Climate change, species–area curves and the extinction crisis |journal=Philosophical Transactions of the Royal Society B: Biological Sciences |volume=361 |issue=1465 |pages=163–171 |doi=10.1098/rstb.2005.1712 |issn=0962-8436 |pmc=1831839 |pmid=16553315}}</ref> Despite the estimated extinction of more than 99 percent of all species that ever lived on Earth,<ref name="StearnsStearns1999">{{harvnb|Stearns|Stearns|1999|p=[https://books.google.com/books?id=0BHeC-tXIB4C&q=99%20percent X]}}</ref><ref name="NYT-20141108-MJN" /> about 1 trillion species are estimated to be on Earth currently with only one-thousandth of one percent described.<ref name="NSF-2016002">{{cite web |url=https://www.nsf.gov/news/news_summ.jsp?cntn_id=138446 |title=Researchers find that Earth may be home to 1 trillion species |author=<!--Not stated--> |date=May 2, 2016 |website=[[National Science Foundation]] |location=Arlington County, Virginia |accessdate=2016-05-06 |deadurl=no |archiveurl=https://web.archive.org/web/20160504111108/https://www.nsf.gov/news/news_summ.jsp?cntn_id=138446 |archivedate=2016-05-04 |df= }}</ref> |
|||
The role of extinction in evolution is not very well understood and may depend on which type of extinction is considered.<ref name="Raup_1994" /> The causes of the continuous "low-level" extinction events, which form the majority of extinctions, may be the result of competition between species for limited resources (the [[competitive exclusion principle]]).<ref name="Kutschera" /> If one species can out-compete another, this could produce species selection, with the fitter species surviving and the other species being driven to extinction.<ref name="Gould" /> The intermittent mass extinctions are also important, but instead of acting as a selective force, they drastically reduce diversity in a nonspecific manner and promote bursts of rapid evolution and speciation in survivors.<ref>{{cite journal |last=Jablonski |first=David |date=May 8, 2001 |title=Lessons from the past: Evolutionary impacts of mass extinctions |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=98 |issue=10 |pages=5393–5398 |bibcode=2001PNAS...98.5393J |doi=10.1073/pnas.101092598 |issn=0027-8424 |pmc=33224 |pmid=11344284}}</ref> |
|||
{{Clear}} |
|||
== Evolutionary history of life == |
|||
{{align|right|{{Life timeline}}}} |
|||
{{Main|Evolutionary history of life}} |
|||
{{See also|Timeline of evolutionary history of life}} |
|||
=== Origin of life === |
|||
{{Further|Abiogenesis|Earliest known life forms|Panspermia|RNA world hypothesis}} |
|||
The [[age of the Earth|Earth]] is about 4.54 billion years old.<ref name="USGS1997">{{cite web |url=http://pubs.usgs.gov/gip/geotime/age.html |title=Age of the Earth |date=July 9, 2007 |publisher=[[United States Geological Survey]] |accessdate=2015-05-31 |deadurl=no |archiveurl=https://web.archive.org/web/20051223072700/http://pubs.usgs.gov/gip/geotime/age.html |archivedate=2005-12-23 |df= }}</ref><ref name="Dalrymple 2001 205–221">{{harvnb|Dalrymple|2001|pp=205–221}}</ref><ref name="Elsevier">{{cite journal |last1=Manhesa |first1=Gérard |last2=Allègre |first2=Claude J. |authorlink2=Claude Allègre |last3=Dupréa |first3=Bernard |last4=Hamelin |first4=Bruno |date=May 1980 |title=Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics |journal=[[Earth and Planetary Science Letters]] |volume=47 |issue=3 |pages=370–382 |bibcode=1980E&PSL..47..370M |doi=10.1016/0012-821X(80)90024-2 |issn=0012-821X}}</ref> The earliest undisputed evidence of life on Earth dates from at least 3.5 billion years ago,<ref name="Origin1">{{cite journal |last1=Schopf |first1=J. William |authorlink1=J. William Schopf |last2=Kudryavtsev |first2=Anatoliy B. |last3=Czaja |first3=Andrew D. |last4=Tripathi |first4=Abhishek B. |date=October 5, 2007 |title=Evidence of Archean life: Stromatolites and microfossils |journal=[[Precambrian Research]] |volume=158 |pages=141–155 |issue=3–4 |doi=10.1016/j.precamres.2007.04.009 |issn=0301-9268|bibcode=2007PreR..158..141S }}</ref><ref name="RavenJohnson2002">{{harvnb|Raven|Johnson|2002|p=68}}</ref> during the [[Eoarchean]] Era after a geological [[Crust (geology)|crust]] started to solidify following the earlier molten [[Hadean]] Eon. Microbial mat fossils have been found in 3.48 billion-year-old sandstone in Western Australia.<ref name="AP-20131113">{{cite news |last=Borenstein |first=Seth |date=November 13, 2013 |title=Oldest fossil found: Meet your microbial mom |url=http://apnews.excite.com/article/20131113/DAA1VSC01.html |work=[[Excite]] |location=Yonkers, New York |publisher=[[Mindspark Interactive Network]] |agency=[[Associated Press]] |accessdate=2015-05-31 |deadurl=no |archiveurl=https://web.archive.org/web/20150629230719/http://apnews.excite.com/article/20131113/DAA1VSC01.html |archivedate=June 29, 2015 |df= }}</ref><ref name="TG-20131113-JP">{{cite news |last=Pearlman |first=Jonathan |date=November 13, 2013 |title='Oldest signs of life on Earth found' |url=https://www.telegraph.co.uk/news/science/science-news/10445788/Oldest-signs-of-life-on-Earth-found.html |newspaper=[[The Daily Telegraph]] |location=London |publisher=[[Telegraph Media Group]] |accessdate=2014-12-15 |deadurl=no |archiveurl=https://web.archive.org/web/20141216062531/http://www.telegraph.co.uk/news/science/science-news/10445788/Oldest-signs-of-life-on-Earth-found.html |archivedate=2014-12-16 |df= }}</ref><ref name="AST-20131108">{{cite journal |last1=Noffke |first1=Nora |last2=Christian |first2=Daniel |last3=Wacey |first3=David |last4=Hazen |first4=Robert M. |authorlink4=Robert Hazen |date=November 16, 2013 |title=Microbially Induced Sedimentary Structures Recording an Ancient Ecosystem in the ''ca.'' 3.48 Billion-Year-Old Dresser Formation, Pilbara, Western Australia |journal=[[Astrobiology (journal)|Astrobiology]] |volume=13 |issue=12 |pages=1103–1124 |bibcode=2013AsBio..13.1103N |doi=10.1089/ast.2013.1030 |issn=1531-1074 |pmc=3870916 |pmid=24205812}}</ref> Other early physical evidence of a biogenic substance is graphite in 3.7 billion-year-old [[Metasediment|metasedimentary rocks]] discovered in Western Greenland<ref name="NG-20131208">{{cite journal |last1=Ohtomo |first1=Yoko |last2=Kakegawa |first2=Takeshi |last3=Ishida |first3=Akizumi |last4=Nagase |first4=Toshiro |last5=Rosing |first5=Minik T. |display-authors=3 |date=January 2014 |title=Evidence for biogenic graphite in early Archaean Isua metasedimentary rocks |journal=[[Nature Geoscience]] |volume=7 |issue=1 |pages=25–28 |bibcode=2014NatGe...7...25O |doi=10.1038/ngeo2025 |issn=1752-0894}}</ref> as well as "remains of [[Biotic material|biotic life]]" found in 4.1 billion-year-old rocks in Western Australia.<ref name="AP-20151019">{{cite news |last=Borenstein |first=Seth |title=Hints of life on what was thought to be desolate early Earth |url=http://apnews.excite.com/article/20151019/us-sci--earliest_life-a400435d0d.html |date=19 October 2015 |work=[[Excite]] |location=Yonkers, NY |publisher=[[Mindspark Interactive Network]] |agency=[[Associated Press]] |deadurl=yes |archiveurl=https://web.archive.org/web/20151023200248/http://apnews.excite.com/article/20151019/us-sci--earliest_life-a400435d0d.html |archivedate=23 October 2015 |accessdate=8 October 2018}}</ref><ref name="PNAS-20151014-pdf">{{cite journal |last1=Bell |first1=Elizabeth A. |last2=Boehnike |first2=Patrick |last3=Harrison |first3=T. Mark |last4=Mao |first4=Wendy L. |date=November 24, 2015 |title=Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon |url=http://www.pnas.org/content/early/2015/10/14/1517557112.full.pdf |format=PDF |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=112 |issue=47 |pages=14518–14521 |doi=10.1073/pnas.1517557112 |issn=0027-8424 |accessdate=2015-12-30 |pmid=26483481 |pmc=4664351 |bibcode=2015PNAS..11214518B |deadurl=no |archiveurl=https://web.archive.org/web/20151106021508/http://www.pnas.org/content/early/2015/10/14/1517557112.full.pdf |archivedate=2015-11-06 |df= }}</ref> Commenting on the Australian findings, [[Stephen Blair Hedges]] wrote, "If life arose relatively quickly on Earth, then it could be common in the universe."<ref name="AP-20151019" /><ref>{{cite news |last=Schouten |first=Lucy |date=October 20, 2015 |title=When did life first emerge on Earth? Maybe a lot earlier than we thought |url=https://www.csmonitor.com/Science/2015/1020/When-did-life-first-emerge-on-Earth-Maybe-a-lot-earlier-than-we-thought |work=[[The Christian Science Monitor]] |location=Boston, Massachusetts |publisher=[[Christian Science Publishing Society]] |issn=0882-7729 |archive-url=https://web.archive.org/web/20160322214217/http://www.csmonitor.com/Science/2015/1020/When-did-life-first-emerge-on-Earth-Maybe-a-lot-earlier-than-we-thought |archive-date=2016-03-22 |dead-url=no |access-date=2018-07-11}}</ref> <!---Nevertheless, [[Late Heavy Bombardment#Geological consequences on Earth|several studies]] suggest that life on Earth may have started even earlier,<ref name="AB-20021014">{{cite web |last=Tenenbaum |first=David |title=When Did Life on Earth Begin? Ask a Rock |url=http://www.astrobio.net/exclusive/293/when-did-life-on-earth-begin-ask-a-rock |date=14 October 2002 |work=Astrobiology Magazine |accessdate=2014-04-13}}</ref> as early as 4.25 billion years ago according to one study,<ref name="NS-20080702">{{cite web |last=Courtland |first=Rachel |title=Did newborn Earth harbour life? |url=https://www.newscientist.com/article/dn14245-did-newborn-earth-harbour-life.html |date=2 July 2008 |work=[[New Scientist]] |accessdate=2014-04-13}}</ref> and 4.4 billion years ago according to another study.<ref name="RN-20090520">{{cite web |last=Steenhuysen |first=Julie |title=Study turns back clock on origins of life on Earth |url=https://www.reuters.com/article/2009/05/20/us-asteroids-idUSTRE54J5PX20090520 |date=20 May 2009 |work=[[Reuters]] |accessdate=2014-04-13}}</ref>---> In July 2016, scientists reported identifying a set of 355 [[gene]]s from the [[last universal common ancestor]] (LUCA) of all organisms living on Earth.<ref name="NYT-20160725">{{cite news |last=Wade |first=Nicholas |authorlink=Nicholas Wade |title=Meet Luca, the Ancestor of All Living Things |url=https://www.nytimes.com/2016/07/26/science/last-universal-ancestor.html |date=July 25, 2016 |newspaper=[[The New York Times]] |location=New York |publisher=[[The New York Times Company]] |issn=0362-4331 |accessdate=2016-07-25 |deadurl=no |archiveurl=https://web.archive.org/web/20160728053822/http://www.nytimes.com/2016/07/26/science/last-universal-ancestor.html |archivedate=July 28, 2016 |df= }}</ref> |
|||
More than 99 percent of all species, amounting to over five billion species,<ref name="Book-Biology">{{harvnb|McKinney|1997|p=[https://books.google.com/books?id=4LHnCAAAQBAJ&pg=PA110&lpg=PA110#v=onepage 110]}}</ref> that ever lived on Earth are estimated to be extinct.<ref name="StearnsStearns1999" /><ref name="NYT-20141108-MJN">{{cite news |last=Novacek |first=Michael J. |date=November 8, 2014 |title=Prehistory’s Brilliant Future |url=https://www.nytimes.com/2014/11/09/opinion/sunday/prehistorys-brilliant-future.html |newspaper=The New York Times |location=New York |publisher=The New York Times Company |issn=0362-4331 |accessdate=2014-12-25 |deadurl=no |archiveurl=https://web.archive.org/web/20141229225657/http://www.nytimes.com/2014/11/09/opinion/sunday/prehistorys-brilliant-future.html |archivedate=2014-12-29 |df= }}</ref> Estimates on the number of Earth's current species range from 10 million to 14 million,<ref name="PLoS-20110823">{{cite journal |last1=Mora |first1=Camilo |last2=Tittensor |first2=Derek P. |last3=Adl |first3=Sina |last4=Simpson |first4=Alastair G.B. |last5=Worm |first5=Boris |authorlink5=Boris Worm |display-authors=3 |date=August 23, 2011 |title=How Many Species Are There on Earth and in the Ocean? |journal=[[PLOS Biology]] |volume=9 |issue=8 |page=e1001127 |doi=10.1371/journal.pbio.1001127 |issn=1545-7885 |pmc=3160336 |pmid=21886479}}</ref><ref name="MillerSpoolman2012">{{harvnb|Miller|Spoolman|2012|p=[https://books.google.com/books?id=NYEJAAAAQBAJ&pg=PA62 62]}}</ref> of which about 1.9 million are estimated to have been named<ref name="Chapman2009">{{harvnb|Chapman|2009}}</ref> and 1.6 million documented in a central database to date,<ref name="col2016">{{cite web |url=http://www.catalogueoflife.org/annual-checklist/2016/info/ac |title=Species 2000 & ITIS Catalogue of Life, 2016 Annual Checklist |year=2016 |editor-last=Roskov |editor-first=Y. |editor2-last=Abucay |editor2-first=L. |editor3-last=Orrell |editor3-first=T. |editor4-last=Nicolson |editor4-first=D. |editor5-last=Flann |editor5-first=C. |editor6-last=Bailly |editor6-first=N. |editor7-last=Kirk |editor7-first=P. |editor8-last=Bourgoin |editor8-first=T. |editor9-last=DeWalt |editor9-first=R.E. |editor10-last=Decock |editor10-first=W. |editor11-last=De Wever |editor11-first=A. |display-editors=4 |website=Species 2000 |publisher=[[Naturalis Biodiversity Center]] |location=Leiden, Netherlands |issn=2405-884X |accessdate=2016-11-06 |deadurl=no |archiveurl=https://web.archive.org/web/20161112121623/http://www.catalogueoflife.org/annual-checklist/2016/info/ac |archivedate=2016-11-12 |df= }}</ref> leaving at least 80 percent not yet described. |
|||
Highly energetic chemistry is thought to have produced a self-replicating molecule around 4 billion years ago, and half a billion years later the [[Last universal common ancestor|last common ancestor of all life]] existed.<ref name="Doolittle_2000" /> The current scientific consensus is that the complex biochemistry that makes up life came from simpler chemical reactions.<ref>{{cite journal|last=Peretó |first=Juli |date=March 2005 |title=Controversies on the origin of life |url=http://www.im.microbios.org/0801/0801023.pdf |format=PDF |journal=International Microbiology |volume=8 |issue=1 |pages=23–31 |issn=1139-6709 |pmid=15906258 |deadurl=yes |archiveurl=https://web.archive.org/web/20150824074726/http://www.im.microbios.org/0801/0801023.pdf |archivedate=2015-08-24 |df= }}</ref> The beginning of life may have included self-replicating molecules such as [[RNA]]<ref>{{cite journal |last=Joyce |first=Gerald F. |authorlink=Gerald Joyce |date=July 11, 2002 |title=The antiquity of RNA-based evolution |journal=Nature |volume=418 |issue=6894 |pages=214–221 |bibcode=2002Natur.418..214J |doi=10.1038/418214a |issn=0028-0836 |pmid=12110897}}</ref> and the assembly of simple cells.<ref>{{cite journal |last1=Trevors |first1=Jack T. |last2=Psenner |first2=Roland |date=December 2001 |title=From self-assembly of life to present-day bacteria: a possible role for nanocells |journal=FEMS Microbiology Reviews |volume=25 |issue=5 |pages=573–582 |doi=10.1111/j.1574-6976.2001.tb00592.x |issn=1574-6976 |pmid=11742692}}</ref> |
|||
=== Common descent === |
|||
{{Further|Common descent|Evidence of common descent}} |
|||
All organisms on Earth are descended from a common ancestor or ancestral [[gene pool]].<ref name="Penny1999" /><ref>{{cite journal |last=Theobald |first=Douglas L. |date=May 13, 2010 |title=A formal test of the theory of universal common ancestry |journal=Nature |volume=465 |issue=7295 |pages=219–222 |bibcode=2010Natur.465..219T |doi=10.1038/nature09014 |issn=0028-0836 |pmid=20463738}}</ref> Current species are a stage in the process of evolution, with their diversity the product of a long series of speciation and extinction events.<ref>{{cite journal |last1=Bapteste |first1=Eric |last2=Walsh |first2=David A. |date=June 2005 |title=Does the 'Ring of Life' ring true? |journal=[[Trends (journals)|Trends in Microbiology]] |volume=13 |issue=6 |pages=256–261 |doi=10.1016/j.tim.2005.03.012 |issn=0966-842X |pmid=15936656}}</ref> The common descent of organisms was first deduced from four simple facts about organisms: First, they have geographic distributions that cannot be explained by local adaptation. Second, the diversity of life is not a set of completely unique organisms, but organisms that share morphological similarities. Third, vestigial traits with no clear purpose resemble functional ancestral traits and finally, that organisms can be classified using these similarities into a hierarchy of nested groups—similar to a family tree.<ref>{{harvnb|Darwin|1859|p=[http://darwin-online.org.uk/content/frameset?itemID=F373&viewtype=text&pageseq=16 1]}}</ref> However, modern research has suggested that, due to horizontal gene transfer, this "tree of life" may be more complicated than a simple branching tree since some genes have spread independently between distantly related species.<ref>{{cite journal |last1=Doolittle |first1=W. Ford |last2=Bapteste |first2=Eric |date=February 13, 2007 |title=Pattern pluralism and the Tree of Life hypothesis |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=104 |issue=7 |pages=2043–2049 |bibcode=2007PNAS..104.2043D |doi=10.1073/pnas.0610699104 |issn=0027-8424 |pmc=1892968 |pmid=17261804}}</ref><ref>{{cite journal |last1=Kunin |first1=Victor |last2=Goldovsky |first2=Leon |last3=Darzentas |first3=Nikos |last4=Ouzounis |first4=Christos A. |date=July 2005 |title=The net of life: Reconstructing the microbial phylogenetic network |journal=Genome Research |volume=15 |issue=7 |pages=954–959 |doi=10.1101/gr.3666505 |issn=1088-9051 |pmid=15965028 |pmc=1172039}}</ref> |
|||
[[File:Ape skeletons.png|upright=1.45|thumb|left|The [[Ape|hominoids]] are descendants of a [[common descent|common ancestor]].]] |
|||
Past species have also left records of their evolutionary history. Fossils, along with the comparative anatomy of present-day organisms, constitute the morphological, or anatomical, record.<ref name=Jablonski>{{cite journal |last=Jablonski |first=David |date=June 25, 1999 |title=The Future of the Fossil Record |journal=Science |volume=284 |issue=5423 |pages=2114–2116 |pmid=10381868 |doi=10.1126/science.284.5423.2114 |issn=0036-8075}}</ref> By comparing the anatomies of both modern and extinct species, paleontologists can infer the lineages of those species. However, this approach is most successful for organisms that had hard body parts, such as shells, bones or teeth. Further, as prokaryotes such as bacteria and archaea share a limited set of common morphologies, their fossils do not provide information on their ancestry. |
|||
More recently, evidence for common descent has come from the study of biochemical similarities between organisms. For example, all living cells use the same basic set of nucleotides and [[amino acid]]s.<ref>{{cite journal |last=Mason |first=Stephen F. |date=September 6, 1984 |title=Origins of biomolecular handedness |journal=Nature |volume=311 |issue=5981 |pages=19–23 |bibcode=1984Natur.311...19M |doi=10.1038/311019a0 |issn=0028-0836 |pmid=6472461}}</ref> The development of [[molecular genetics]] has revealed the record of evolution left in organisms' genomes: dating when species diverged through the [[molecular clock]] produced by mutations.<ref>{{cite journal |last1=Wolf |first1=Yuri I. |last2=Rogozin |first2=Igor B. |last3=Grishin |first3=Nick V. |last4=Koonin |first4=Eugene V. |authorlink4=Eugene Koonin |date=September 1, 2002 |title=Genome trees and the tree of life |journal=Trends in Genetics |volume=18 |issue=9 |pages=472–479 |doi=10.1016/S0168-9525(02)02744-0 |issn=0168-9525 |pmid=12175808}}</ref> For example, these DNA sequence comparisons have revealed that humans and chimpanzees share 98% of their genomes and analysing the few areas where they differ helps shed light on when the common ancestor of these species existed.<ref>{{cite journal |last1=Varki |first1=Ajit |authorlink1=Ajit Varki |last2=Altheide |first2=Tasha K. |date=December 2005 |title=Comparing the human and chimpanzee genomes: searching for needles in a haystack |journal=Genome Research |volume=15 |issue=12 |pages=1746–1758 |doi=10.1101/gr.3737405 |issn=1088-9051 |pmid=16339373}}</ref> |
|||
=== Evolution of life === |
|||
{{Main|Evolutionary history of life|Timeline of evolutionary history of life}} |
|||
{{PhylomapA|size=320px|align=right|caption=[[Phylogenetic tree|Evolutionary tree]] showing the divergence of modern species from their common ancestor in the centre.<ref name="Ciccarelli">{{cite journal |last1=Ciccarelli |first1=Francesca D. |last2=Doerks |first2=Tobias |last3=von Mering |first3=Christian |last4=Creevey |first4=Christopher J. |last5=Snel |first5=Berend |last6=Bork |first6=Peer |authorlink6=Peer Bork |date=March 3, 2006 |title=Toward Automatic Reconstruction of a Highly Resolved Tree of Life |journal=[[Science (journal)|Science]] |volume=311 |issue=5765 |pages=1283–1287 |bibcode=2006Sci...311.1283C |doi=10.1126/science.1123061 |issn=0036-8075 |pmid=16513982 |display-authors=3 |url=http://bioinformatics.bio.uu.nl/pdf/Ciccarelli.s06-311.pdf |deadurl=no |archiveurl=https://web.archive.org/web/20160304035346/http://bioinformatics.bio.uu.nl/pdf/Ciccarelli.s06-311.pdf |archivedate=2016-03-04 |df= |citeseerx=10.1.1.381.9514 }}</ref> The three [[Domain (biology)|domains]] are coloured, with [[bacteria]] blue, [[archaea]] green and [[eukaryote]]s red.}} |
|||
Prokaryotes inhabited the Earth from approximately 3–4 billion years ago.<ref name="Cavalier-Smith">{{cite journal |last=Cavalier-Smith |first=Thomas |authorlink=Thomas Cavalier-Smith |date=June 29, 2006 |title=Cell evolution and Earth history: stasis and revolution |journal=Philosophical Transactions of the Royal Society B: Biological Sciences |volume=361 |issue=1470 |pages=969–1006 |doi=10.1098/rstb.2006.1842 |issn=0962-8436 |pmc=1578732 |pmid=16754610}}</ref><ref>{{cite journal |last=Schopf |first=J. William |date=June 29, 2006 |title=Fossil evidence of Archaean life |journal=Philosophical Transactions of the Royal Society B: Biological Sciences |volume=361 |issue=1470 |pages=869–885 |doi=10.1098/rstb.2006.1834 |issn=0962-8436 |pmc=1578735 |pmid=16754604}} |
|||
* {{cite journal |last1=Altermann |first1=Wladyslaw |last2=Kazmierczak |first2=Józef |date=November 2003 |title=Archean microfossils: a reappraisal of early life on Earth |journal=Research in Microbiology |volume=154 |issue=9 |pages=611–617 |doi=10.1016/j.resmic.2003.08.006 |issn=0923-2508 |pmid=14596897}}</ref> No obvious changes in morphology or cellular organisation occurred in these organisms over the next few billion years.<ref>{{cite journal |last=Schopf |first=J. William |date=July 19, 1994 |title=Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic |journal=Proc. Natl. Acad. Sci. U.S.A. |volume=91 |issue=15 |pages=6735–6742 |bibcode=1994PNAS...91.6735S |doi=10.1073/pnas.91.15.6735 |issn=0027-8424 |pmc=44277 |pmid=8041691}}</ref> The eukaryotic cells emerged between 1.6–2.7 billion years ago. The next major change in cell structure came when bacteria were engulfed by eukaryotic cells, in a cooperative association called [[endosymbiont|endosymbiosis]].<ref name="rgruqh">{{cite journal |last1=Poole |first1=Anthony M. |last2=Penny |first2=David |date=January 2007 |title=Evaluating hypotheses for the origin of eukaryotes |journal=BioEssays |volume=29 |issue=1 |pages=74–84 |doi=10.1002/bies.20516 |issn=0265-9247 |pmid=17187354}}</ref><ref name="Dyall">{{cite journal |last1=Dyall |first1=Sabrina D. |last2=Brown |first2=Mark T. |last3=Johnson |first3=Patricia J. |authorlink3=Patricia J. Johnson |date=April 9, 2004 |title=Ancient Invasions: From Endosymbionts to Organelles |journal=Science |volume=304 |issue=5668 |pages=253–257 |bibcode=2004Sci...304..253D |doi=10.1126/science.1094884 |issn=0036-8075 |pmid=15073369}}</ref> The engulfed bacteria and the host cell then underwent coevolution, with the bacteria evolving into either mitochondria or [[hydrogenosome]]s.<ref>{{cite journal |last=Martin |first=William |date=October 2005 |title=The missing link between hydrogenosomes and mitochondria |journal=Trends in Microbiology |volume=13 |issue=10 |pages=457–459 |doi=10.1016/j.tim.2005.08.005 |issn=0966-842X |pmid=16109488}}</ref> Another engulfment of [[cyanobacteria]]l-like organisms led to the formation of chloroplasts in algae and plants.<ref>{{cite journal |last1=Lang |first1=B. Franz |last2=Gray |first2=Michael W. |last3=Burger |first3=Gertraud |date=December 1999 |title=Mitochondrial genome evolution and the origin of eukaryotes |journal=Annual Review of Genetics |volume=33 |pages=351–397 |doi=10.1146/annurev.genet.33.1.351 |issn=0066-4197 |pmid=10690412}} |
|||
* {{cite journal |last=McFadden |first=Geoffrey Ian |date=December 1, 1999 |title=Endosymbiosis and evolution of the plant cell |journal=Current Opinion in Plant Biology |volume=2 |issue=6 |pages=513–519 |doi=10.1016/S1369-5266(99)00025-4 |issn=1369-5266 |pmid=10607659}}</ref> |
|||
The history of life was that of the [[Unicellular organism|unicellular]] eukaryotes, prokaryotes and archaea until about 610 million years ago when multicellular organisms began to appear in the oceans in the [[Ediacara biota|Ediacaran]] period.<ref name="Cavalier-Smith" /><ref>{{cite journal |last1=DeLong |first1=Edward F. |authorlink1=Edward DeLong |last2=Pace |first2=Norman R. |authorlink2=Norman R. Pace |date=August 1, 2001 |title=Environmental Diversity of Bacteria and Archaea |journal=[[Systematic Biology]] |volume=50 |issue=4 |pages=470–478 |doi=10.1080/106351501750435040 |issn=1063-5157 |pmid=12116647 |url=http://sysbio.oxfordjournals.org/content/50/4/470.full.pdf |deadurl=no |archiveurl=https://web.archive.org/web/20160222022557/http://sysbio.oxfordjournals.org/content/50/4/470.full.pdf |archivedate=2016-02-22 |df= |citeseerx=10.1.1.321.8828 }}</ref> The [[Multicellular evolution|evolution of multicellularity]] occurred in multiple independent events, in organisms as diverse as [[sponge]]s, [[brown algae]], cyanobacteria, [[Slime mold|slime moulds]] and [[myxobacteria]].<ref>{{cite journal |last=Kaiser |first=Dale |authorlink=A. Dale Kaiser |date=December 2001 |title=Building a multicellular organism |journal=Annual Review of Genetics |volume=35 |pages=103–123 |doi=10.1146/annurev.genet.35.102401.090145 |issn=0066-4197 |pmid=11700279}}</ref> In January 2016, scientists reported that, about 800 million years ago, a minor genetic change in a single molecule called GK-PID may have allowed organisms to go from a single cell organism to one of many cells.<ref name="NYT-20160107">{{cite news |last=Zimmer |first=Carl |authorlink=Carl Zimmer |title=Genetic Flip Helped Organisms Go From One Cell to Many |url=https://www.nytimes.com/2016/01/12/science/genetic-flip-helped-organisms-go-from-one-cell-to-many.html |date=January 7, 2016 |newspaper=The New York Times |location=New York |publisher=The New York Times Company |issn=0362-4331 |accessdate=2016-01-07 |deadurl=no |archiveurl=https://web.archive.org/web/20160107204432/http://www.nytimes.com/2016/01/12/science/genetic-flip-helped-organisms-go-from-one-cell-to-many.html |archivedate=2016-01-07 |df= }}</ref> |
|||
Soon after the emergence of these first multicellular organisms, a remarkable amount of biological diversity appeared over approximately 10 million years, in an event called the [[Cambrian explosion]]. Here, the majority of [[Phylum|types]] of modern animals appeared in the fossil record, as well as unique lineages that subsequently became extinct.<ref name="Valentine_1999">{{cite journal |last1=Valentine |first1=James W. |authorlink1=James W. Valentine |last2=Jablonski |first2=David |last3=Erwin |first3=Douglas H. |authorlink3=Douglas Erwin |date=March 1, 1999 |title=Fossils, molecules and embryos: new perspectives on the Cambrian explosion |url=http://dev.biologists.org/content/126/5/851.full.pdf+html |journal=[[Development (journal)|Development]] |volume=126 |issue=5 |pages=851–859 |issn=0950-1991 |pmid=9927587 |accessdate=2014-12-30 |deadurl=no |archiveurl=https://web.archive.org/web/20150301063309/http://dev.biologists.org/content/126/5/851.full.pdf+html |archivedate=2015-03-01 |df= }}</ref> Various triggers for the Cambrian explosion have been proposed, including the accumulation of oxygen in the atmosphere from photosynthesis.<ref>{{cite journal |last=Ohno |first=Susumu |date=January 1997 |title=The reason for as well as the consequence of the Cambrian explosion in animal evolution |journal=Journal of Molecular Evolution |volume=44 |issue=Suppl. 1 |pages=S23–S27 |doi=10.1007/PL00000055 |issn=0022-2844 |pmid=9071008|bibcode=1997JMolE..44S..23O }} |
|||
* {{cite journal |last1=Valentine |first1=James W. |last2=Jablonski |first2=David |title=Morphological and developmental macroevolution: a paleontological perspective |url=http://www.ijdb.ehu.es/web/paper.php?doi=14756327 |year=2003 |journal=The International Journal of Developmental Biology |volume=47 |issue=7–8 |pages=517–522 |issn=0214-6282 |pmid=14756327 |accessdate=2014-12-30 |deadurl=no |archiveurl=https://web.archive.org/web/20141024234611/http://www.ijdb.ehu.es/web/paper.php?doi=14756327 |archivedate=2014-10-24 |df= }}</ref> |
|||
About 500 million years ago, plants and fungi colonised the land and were soon followed by arthropods and other animals.<ref>{{cite journal |last=Waters |first=Elizabeth R. |date=December 2003 |title=Molecular adaptation and the origin of land plants |journal=[[Molecular Phylogenetics and Evolution]] |volume=29 |issue=3 |pages=456–463 |doi=10.1016/j.ympev.2003.07.018 |issn=1055-7903 |pmid=14615186}}</ref> Insects were particularly successful and even today make up the majority of animal species.<ref>{{cite journal |last=Mayhew |first=Peter J. |authorlink=Peter Mayhew (biologist) |date=August 2007 |title=Why are there so many insect species? Perspectives from fossils and phylogenies |journal=Biological Reviews |volume=82 |issue=3 |pages=425–454 |doi=10.1111/j.1469-185X.2007.00018.x |issn=1464-7931 |pmid=17624962}}</ref> [[Amphibian]]s first appeared around 364 million years ago, followed by early [[amniote]]s and birds around 155 million years ago (both from "[[reptile]]"-like lineages), [[mammal]]s around 129 million years ago, [[homininae]] around 10 million years ago and [[Anatomically modern humans|modern humans]] around 250,000 years ago.<ref>{{cite journal |last=Carroll |first=Robert L. |authorlink=Robert L. Carroll |date=May 2007 |title=The Palaeozoic Ancestry of Salamanders, Frogs and Caecilians |journal=[[Zoological Journal of the Linnean Society]] |volume=150 |issue=Supplement s1 |pages=1–140 |doi=10.1111/j.1096-3642.2007.00246.x |issn=1096-3642 }}</ref><ref>{{cite journal |last1=Wible |first1=John R. |last2=Rougier |first2=Guillermo W. |last3=Novacek |first3=Michael J. |last4=Asher |first4=Robert J. |date=June 21, 2007 |title=Cretaceous eutherians and Laurasian origin for placental mammals near the K/T boundary |journal=Nature |volume=447 |issue=7147 |pages=1003–1006 |bibcode=2007Natur.447.1003W |doi=10.1038/nature05854 |issn=0028-0836 |pmid=17581585}}</ref><ref>{{cite journal |last=Witmer |first=Lawrence M. |authorlink=Lawrence Witmer |date=July 28, 2011 |title=Palaeontology: An icon knocked from its perch |journal=Nature |volume=475 |issue=7357 |pages=458–459 |doi=10.1038/475458a |issn=0028-0836 |pmid=21796198}}</ref> However, despite the evolution of these large animals, smaller organisms similar to the types that evolved early in this process continue to be highly successful and dominate the Earth, with the majority of both biomass and species being prokaryotes.<ref name=Schloss/> |
|||
== Applications == |
|||
{{Main|Applications of evolution|Selective breeding|Evolutionary computation}} |
|||
Concepts and models used in evolutionary biology, such as natural selection, have many applications.<ref name="Bull">{{cite journal |last1=Bull |first1=James J. |authorlink1=James J. Bull |last2=Wichman |first2=Holly A. |date=November 2001 |title=Applied evolution |journal=Annual Review of Ecology and Systematics |volume=32 |pages=183–217 |doi=10.1146/annurev.ecolsys.32.081501.114020 |issn=1545-2069}}</ref> |
|||
Artificial selection is the intentional selection of traits in a population of organisms. This has been used for thousands of years in the [[domestication]] of plants and animals.<ref>{{cite journal |last1=Doebley |first1=John F. |last2=Gaut |first2=Brandon S. |last3=Smith |first3=Bruce D. |authorlink3=Bruce D. Smith |date=December 29, 2006 |title=The Molecular Genetics of Crop Domestication |journal=Cell |volume=127 |issue=7 |pages=1309–1321 |doi=10.1016/j.cell.2006.12.006 |issn=0092-8674 |pmid=17190597}}</ref> More recently, such selection has become a vital part of [[genetic engineering]], with [[selectable marker]]s such as antibiotic resistance genes being used to manipulate DNA. Proteins with valuable properties have evolved by repeated rounds of mutation and selection (for example modified enzymes and new [[antibody|antibodies]]) in a process called [[directed evolution]].<ref>{{cite journal |last1=Jäckel |first1=Christian |last2=Kast |first2=Peter |last3=Hilvert |first3=Donald |date=June 2008 |title=Protein Design by Directed Evolution |journal=[[Annual Review of Biophysics]] |volume=37 |pages=153–173 |doi=10.1146/annurev.biophys.37.032807.125832 |issn=1936-122X |pmid=18573077}}</ref> |
|||
Understanding the changes that have occurred during an organism's evolution can reveal the genes needed to construct parts of the body, genes which may be involved in human [[genetic disorder]]s.<ref>{{cite journal |last=Maher |first=Brendan |date=April 8, 2009 |title=Evolution: Biology's next top model? |journal=Nature |volume=458 |issue=7239 |pages=695–698 |doi=10.1038/458695a |issn=0028-0836 |pmid=19360058}}</ref> For example, the [[Mexican tetra]] is an [[Albinism|albino]] cavefish that lost its eyesight during evolution. Breeding together different populations of this blind fish produced some offspring with functional eyes, since different mutations had occurred in the isolated populations that had evolved in different caves.<ref>{{cite journal |last=Borowsky |first=Richard |date=January 8, 2008 |title=Restoring sight in blind cavefish |journal=Current Biology |volume=18 |issue=1 |pages=R23–R24 |doi=10.1016/j.cub.2007.11.023 |issn=0960-9822 |pmid=18177707|bibcode=1996CBio....6.1213A }}</ref> This helped identify genes required for vision and pigmentation.<ref>{{cite journal |last1=Gross |first1=Joshua B. |last2=Borowsky |first2=Richard |last3=Tabin |first3=Clifford J. |date=January 2, 2009 |editor1-last=Barsh |editor1-first=Gregory S. |title=A novel role for ''Mc1r'' in the parallel evolution of depigmentation in independent populations of the cavefish ''Astyanax mexicanus'' |journal=PLOS Genetics |volume=5 |issue=1 |pages=e1000326 |doi=10.1371/journal.pgen.1000326 |issn=1553-7390 |pmc=2603666 |pmid=19119422}}</ref> |
|||
Evolutionary theory has many applications in medicine. Many human diseases are not static phenomena, but capable of evolution. Viruses, bacteria, fungi and [[cancer]]s evolve to be resistant to host [[immune system|immune defences]], as well as [[pharmaceutical drug]]s.<ref>{{cite journal|last1=Merlo|first1=Lauren M.F.|last2=Pepper|first2=John W.|last3=Reid|first3=Brian J.|last4=Maley|first4=Carlo C.|date=December 2006|title=Cancer as an evolutionary and ecological process.|url=|journal=[[Nat. Rev. Cancer|Nature Reviews Cancer]]|volume=6|issue=12|pages=924–935|doi=10.1038/nrc2013|issn=1474-175X|pmid=17109012|access-date=}}</ref><ref>{{cite journal|date=October 2012|title=Understanding the drug resistance mechanism of hepatitis C virus NS3/4A to ITMN-191 due to R155K, A156V, D168A/E mutations: a computational study.|url=|journal=[[Biochim. Biophys. Acta|Biochimica et Biophysica Acta (BBA) - General Subjects]]|volume=1820|issue=10|pages=1526–1534|doi=10.1016/j.bbagen.2012.06.001|issn=0304-4165|pmid=22698669|access-date=|last1=Pan|first1=Dabo|author2=Weiwei Xue|author3=Wenqi Zhang|author4=Huanxiang Liu|author5=Xiaojun Yao|display-authors=3}}</ref><ref>{{cite journal |last1=Woodford |first1=Neil |last2=Ellington |first2=Matthew J. |date=January 2007 |title=The emergence of antibiotic resistance by mutation. |journal=Clinical Microbiology and Infection |volume=13 |issue=1 |pages=5–18 |doi=10.1111/j.1469-0691.2006.01492.x |issn=1198-743X |pmid=17184282}}</ref> These same problems occur in agriculture with [[pesticide]]<ref>{{cite journal |last1=Labbé |first1=Pierrick |last2=Berticat |first2=Claire |last3=Berthomieu |first3=Arnaud |last4=Unal |first4=Sandra |last5=Bernard |first5=Clothilde |last6=Weill |first6=Mylène |last7=Lenormand |first7=Thomas |date=November 16, 2007 |title=Forty Years of Erratic Insecticide Resistance Evolution in the Mosquito ''Culex pipiens'' |journal=PLOS Genetics |volume=3 |issue=11 |pages=e205 |doi=10.1371/journal.pgen.0030205 |issn=1553-7390 |pmid=18020711 |display-authors=3 |pmc=2077897}}</ref> and [[herbicide]]<ref>{{cite journal |last=Neve |first=Paul |date=October 2007 |title=Challenges for herbicide resistance evolution and management: 50 years after Harper |journal=Weed Research |volume=47 |issue=5 |pages=365–369 |doi=10.1111/j.1365-3180.2007.00581.x |issn=0043-1737}}</ref> resistance. It is possible that we are facing the end of the effective life of most of available antibiotics<ref>{{cite journal |last1=Rodríguez-Rojas |first1=Alexandro |last2=Rodríguez-Beltrán |first2=Jerónimo |last3=Couce |first3=Alejandro |last4=Blázquez |first4=Jesús |date=August 2013 |title=Antibiotics and antibiotic resistance: A bitter fight against evolution |journal=[[International Journal of Medical Microbiology]] |volume=303 |issue=6–7 |pages=293–297 |doi=10.1016/j.ijmm.2013.02.004 |issn=1438-4221 |pmid=23517688}}</ref> and predicting the evolution and evolvability<ref>{{cite journal |last1=Schenk |first1=Martijn F. |last2=Szendro |first2=Ivan G. |last3=Krug |first3=Joachim |last4=de Visser |first4=J. Arjan G.M. |date=June 28, 2012 |title=Quantifying the Adaptive Potential of an Antibiotic Resistance Enzyme |journal=PLOS Genetics |volume=8 |issue=6 |pages=e1002783 |doi=10.1371/journal.pgen.1002783 |issn=1553-7390 |pmid=22761587 |pmc=3386231}}</ref> of our pathogens and devising strategies to slow or circumvent it is requiring deeper knowledge of the complex forces driving evolution at the molecular level.<ref>{{cite journal |last1=Read |first1=Andrew F. |last2=Lynch |first2=Penelope A. |last3=Thomas |first3=Matthew B. |date=April 7, 2009 |title=How to Make Evolution-Proof Insecticides for Malaria Control |journal=PLOS Biology |volume=7 |issue=4 |page=e1000058 |doi=10.1371/journal.pbio.1000058 |issn=1545-7885 |pmid=19355786 |pmc=3279047}}</ref> |
|||
In [[computer science]], simulations of evolution using [[evolutionary algorithm]]s and [[artificial life]] started in the 1960s and were extended with simulation of artificial selection.<ref>{{cite journal |last=Fraser |first=Alex S. |authorlink=Alex Fraser (scientist) |date=January 18, 1958 |title=Monte Carlo Analyses of Genetic Models |journal=Nature |volume=181 |issue=4603 |pages=208–209 |bibcode=1958Natur.181..208F |doi=10.1038/181208a0 |issn=0028-0836 |pmid=13504138}}</ref> Artificial evolution became a widely recognised optimisation method as a result of the work of [[Ingo Rechenberg]] in the 1960s. He used [[Evolution strategy|evolution strategies]] to solve complex engineering problems.<ref>{{harvnb|Rechenberg|1973}}</ref> [[Genetic algorithm]]s in particular became popular through the writing of [[John Henry Holland]].<ref>{{harvnb|Holland|1975}}</ref> Practical applications also include [[genetic programming|automatic evolution of computer programmes]].<ref>{{harvnb|Koza|1992}}</ref> Evolutionary algorithms are now used to solve multi-dimensional problems more efficiently than software produced by human designers and also to optimise the design of systems.<ref>{{cite journal |last=Jamshidi |first=Mo |date=August 15, 2003 |title=Tools for intelligent control: fuzzy controllers, neural networks and genetic algorithms |journal=[[Philosophical Transactions of the Royal Society A]] |volume=361 |issue=1809 |pages=1781–1808 |bibcode=2003RSPTA.361.1781J |doi=10.1098/rsta.2003.1225 |issn=1364-503X |pmid=12952685}}</ref> |
|||
== Social and cultural responses == |
|||
{{Further|Social effects of evolutionary theory|1860 Oxford evolution debate|Creation–evolution controversy|Objections to evolution|Evolution in fiction}} |
|||
[[File:Editorial cartoon depicting Charles Darwin as an ape (1871).jpg|upright|thumb|As evolution became widely accepted in the 1870s, [[caricature]]s of Charles Darwin with an [[ape]] or [[monkey]] body symbolised evolution.<ref>{{harvnb|Browne|2003|pp=376–379}}</ref>]] |
|||
In the 19th century, particularly after the publication of ''On the Origin of Species'' in 1859, the idea that life had evolved was an active source of academic debate centred on the philosophical, social and religious implications of evolution. Today, the modern evolutionary synthesis is accepted by a vast majority of scientists.<ref name="Kutschera" /> However, evolution remains a contentious concept for some [[Theism|theists]].<ref>For an overview of the philosophical, religious and cosmological controversies, see: |
|||
* {{harvnb|Dennett|1995}} |
|||
For the scientific and social reception of evolution in the 19th and early 20th centuries, see: |
|||
* {{cite book |last=Johnston |first=Ian C. |authorlink=Ian C. Johnston |year=1999 |chapter=Section Three: The Origins of Evolutionary Theory |chapter-url=https://malvma.viu.ca/~johnstoi/darwin/sect3.htm |title=... And Still We Evolve: A Handbook for the Early History of Modern Science |url=https://malvma.viu.ca/~johnstoi/darwin/title.htm |edition=3rd revised |location=Nanaimo, BC |publisher=Liberal Studies Department, [[Vancouver Island University|Malaspina University-College]] |accessdate=2015-01-01 |deadurl=no |archive-url=https://web.archive.org/web/20160416050826/http://records.viu.ca/~johnstoi/darwin/title.htm |archive-date=2016-04-16}} |
|||
* {{harvnb|Bowler|2003}} |
|||
* {{cite journal |last=Zuckerkandl |first=Emile |authorlink=Emile Zuckerkandl |date=December 30, 2006 |title=Intelligent design and biological complexity |journal=[[Gene (journal)|Gene]] |volume=385 |pages=2–18 |pmid=17011142 |doi=10.1016/j.gene.2006.03.025 |issn=0378-1119}}</ref> |
|||
While [[Level of support for evolution#Support for evolution by religious bodies|various religions and denominations]] have reconciled their beliefs with evolution through concepts such as [[theistic evolution]], there are [[creationism|creationists]] who believe that evolution is contradicted by the [[creation myth]]s found in their [[religion]]s and who raise various [[objections to evolution]].<ref name="ScottEC" /><ref name="Ross2005">{{cite journal |last=Ross |first=Marcus R. |authorlink=Marcus R. Ross |date=May 2005 |title=Who Believes What? Clearing up Confusion over Intelligent Design and Young-Earth Creationism |url=http://www.nagt.org/files/nagt/jge/abstracts/Ross_v53n3p319.pdf |format=PDF |journal=Journal of Geoscience Education |volume=53 |issue=3 |pages=319–323 |issn=1089-9995 |accessdate=2008-04-28 |bibcode=2005JGeEd..53..319R |doi=10.5408/1089-9995-53.3.319 |deadurl=no |archiveurl=https://web.archive.org/web/20080511204303/http://nagt.org/files/nagt/jge/abstracts/Ross_v53n3p319.pdf |archivedate=2008-05-11 |df= |citeseerx=10.1.1.404.1340 }}</ref><ref>{{cite journal|last=Hameed |first=Salman |date=December 12, 2008 |title=Bracing for Islamic Creationism |url=http://helios.hampshire.edu/~sahCS/Hameed-Science-Creationism.pdf |format=PDF |journal=Science |volume=322 |issue=5908 |pages=1637–1638 |doi=10.1126/science.1163672 |issn=0036-8075 |pmid=19074331 |deadurl=yes |archiveurl=https://web.archive.org/web/20141110031233/http://helios.hampshire.edu/~sahCS/Hameed-Science-Creationism.pdf |archivedate=2014-11-10 |df= }}</ref> As had been demonstrated by responses to the publication of ''[[Vestiges of the Natural History of Creation]]'' in 1844, the most controversial aspect of evolutionary biology is the implication of [[human evolution]] that humans share common ancestry with apes and that the mental and [[Evolution of morality|moral faculties]] of humanity have the same types of natural causes as other inherited traits in animals.<ref>{{harvnb|Bowler|2003}}</ref> In some countries, notably the United States, these tensions between science and religion have fuelled the current creation–evolution controversy, a religious conflict focusing on [[politics]] and [[creation and evolution in public education|public education]].<ref>{{cite journal |last=Miller |first=Jon D. |last2=Scott |first2=Eugenie C. |last3=Okamoto |first3=Shinji |date=August 11, 2006 |title=Public Acceptance of Evolution |journal=Science |volume=313 |issue=5788 |pages=765–766 |doi=10.1126/science.1126746 |issn=0036-8075 |pmid=16902112}}</ref> While other scientific fields such as [[physical cosmology|cosmology]]<ref name="wmap">{{cite journal |last1=Spergel |first1=David Nathaniel |authorlink1=David Spergel |last2=Verde |first2=Licia |last3=Peiris |first3=Hiranya V. |last4=Komatsu |first4=Eiichiro |last5=Nolta |first5=Michael R. |last6=Bennett |first6=Charles L. |authorlink6=Charles L. Bennett |last7=Halpern |first7=Mark |last8=Hinshaw |first8=Gary |last9=Jarosik |first9=Norman |year=2003 |title=First-Year Wilkinson Microwave Anisotropy Probe (WMAP) Observations: Determination of Cosmological Parameters |journal=[[The Astrophysical Journal|The Astrophysical Journal Supplement Series]] |volume=148 |issue=1 |pages=175–194 |arxiv=astro-ph/0302209 |bibcode=2003ApJS..148..175S |doi=10.1086/377226 |display-authors=3}}</ref> and [[Earth science]]<ref name="zircon">{{cite journal |last1=Wilde |first1=Simon A. |last2=Valley |first2=John W. |last3=Peck |first3=William H. |last4=Graham |first4=Colin M. |date=January 11, 2001 |title=Evidence from detrital zircons for the existence of continental crust and oceans on the Earth 4.4 Gyr ago |journal=Nature |volume=409 |issue=6817 |pages=175–178 |doi=10.1038/35051550 |issn=0028-0836 |pmid=11196637}}</ref> also conflict with literal interpretations of many [[religious text]]s, evolutionary biology experiences significantly more opposition from religious literalists. |
|||
The teaching of evolution in American secondary school biology classes was uncommon in most of the first half of the 20th century. The [[Scopes Trial]] decision of 1925 caused the subject to become very rare in American secondary biology textbooks for a generation, but it was gradually re-introduced later and became legally protected with the 1968 ''[[Epperson v. Arkansas]]'' decision. Since then, the competing religious belief of creationism was legally disallowed in secondary school curricula in various decisions in the 1970s and 1980s, but it returned in [[Pseudoscience|pseudoscientific]] form as [[intelligent design]] (ID), to be excluded once again in the 2005 ''[[Kitzmiller v. Dover Area School District]]'' case.<ref name="BioScience">{{cite journal |last=Branch |first=Glenn |authorlink=Glenn Branch |date=March 2007 |title=Understanding Creationism after ''Kitzmiller'' |journal=[[BioScience]] |volume=57 |issue=3 |pages=278–284 |doi=10.1641/B570313 |issn=0006-3568|bibcode=1985BioSc..35..499W }}</ref> The debate over Darwin's ideas did not generate significant controversy in China. <ref>https://www.cambridge.org/core/journals/british-journal-for-the-history-of-science/article/translation-and-transmutation-the-origin-of-species-in-china/3F5AED9F9D60EF2D7265927BB2B6AB3A</ref> |
|||
== See also == |
|||
{{Wikipedia books|Evolution}} |
|||
{{div col|colwidth=30em}} |
|||
* [[Argument from poor design]] |
|||
* [[Dual inheritance theory|Biocultural evolution]] |
|||
* [[Biological classification]] |
|||
* [[Evidence of common descent]] |
|||
* [[Evolution in Variable Environment]] |
|||
* [[Evolutionary anthropology]] |
|||
* [[Evolutionary ecology]] |
|||
* [[Evolutionary epistemology]] |
|||
* [[Evolutionary neuroscience]] |
|||
* [[Evolution of biological complexity]] |
|||
* [[Evolution of plants]] |
|||
* [[Project Steve]] |
|||
* [[Timeline of the evolutionary history of life]] |
|||
* [[Universal Darwinism]] |
|||
{{div col end}} |
|||
== References == |
|||
{{Reflist|30em}} |
|||
== Bibliography == |
|||
{{Refbegin|30em}} |
|||
* {{cite book |last=Altenberg |first=Lee |year=1995 |chapter=Genome growth and the evolution of the genotype-phenotype map |editor1-last=Banzhaf |editor1-first=Wolfgang |editor2-last=Eeckman |editor2-first=Frank H. |title=Evolution and Biocomputation: Computational Models of Evolution |series=Lecture Notes in Computer Science |volume=899 |pages=205–259 |location=Berlin; New York |publisher=[[Springer Science+Business Media|Springer-Verlag Berlin Heidelberg]] |doi=10.1007/3-540-59046-3_11 |issn=0302-9743 |isbn=978-3-540-59046-0 |lccn=95005970 |oclc=32049812 |ref=harv|citeseerx=10.1.1.493.6534 }} |
|||
* {{cite book |editor1-last=Ayala |editor1-first=Francisco J. |editor1-link=Francisco J. Ayala |editor2-last=Avise |editor2-first=John C. |editor2-link=John Avise |year=2014 |title=Essential Readings in Evolutionary Biology |location=Baltimore, Maryland |publisher=Johns Hopkins University Press |isbn=978-1-4214-1305-1 |lccn=2013027718 |oclc=854285705 |ref=harv}} |
|||
* {{cite book |last1=Birdsell |first1=John A. |last2=Wills |first2=Christopher |authorlink2=Christopher Wills |year=2003 |chapter=The Evolutionary Origin and Maintenance of Sexual Recombination: A Review of Contemporary Models |editor1-last=MacIntyre |editor1-first=Ross J. |editor2-last=Clegg |editor2-first=Michael T. |title=Evolutionary Biology |series=Evolutionary Biology |volume=33 |location=New York |publisher=[[Springer Science+Business Media]] |isbn=978-1-4419-3385-0 |issn=0071-3260 |oclc=751583918 |ref=harv}} |
|||
* {{cite book |last=Bowler |first=Peter J. |authorlink=Peter J. Bowler |year=1989 |title=The Mendelian Revolution: The Emergence of Hereditarian Concepts in Modern Science and Society |location=Baltimore, Maryland |publisher=Johns Hopkins University Press |isbn=978-0-8018-3888-0 |lccn=89030914 |oclc=19322402 |ref=harv}} |
|||
* {{cite book |last=Bowler |first=Peter J. |year=2003 |title=Evolution: The History of an Idea |edition=3rd completely rev. and expanded |location=Berkeley, California |publisher=[[University of California Press]] |isbn=978-0-520-23693-6 |lccn=2002007569 |oclc=49824702 |ref=harv}} |
|||
* {{cite book |last=Browne |first=Janet |authorlink=Janet Browne |year=2003 |title=Charles Darwin: The Power of Place |volume=2 |location=London |publisher=[[Random House|Pimlico]] |isbn=978-0-7126-6837-8 |lccn=94006598 |oclc=52327000 |ref=harv}} |
|||
* {{cite book |editor1-last=Burkhardt |editor1-first=Frederick |editor1-link=Frederick Burkhardt |editor2-last=Smith |editor2-first=Sydney |year=1991 |title=The Correspondence of Charles Darwin |series=The Correspondence of Charles Darwin |volume='''7''': 1858–1859 |location=Cambridge |publisher=[[Cambridge University Press]] |isbn=978-0-521-38564-0 |lccn=84045347 |oclc=185662993 |ref=harv}} |
|||
* {{cite book |last1=Carroll |first1=Sean B. |authorlink1=Sean B. Carroll |last2=Grenier |first2=Jennifer K. |last3=Weatherbee |first3=Scott D. |year=2005 |title=From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design |edition=2nd |location=Malden, Massachusetts |publisher=[[Wiley-Blackwell|Blackwell Publishing]] |isbn=978-1-4051-1950-4 |lccn=2003027991 |oclc=53972564 |ref=harv}} |
|||
* {{cite book |last=Chapman |first=Arthur D. |year=2009 |title=Numbers of Living Species in Australia and the World |edition=2nd |url=https://www.environment.gov.au/science/abrs/publications/other/numbers-living-species/ |accessdate=2016-11-06 |deadurl=no |archiveurl=https://web.archive.org/web/20161225064434/http://www.environment.gov.au/science/abrs/publications/other/numbers-living-species |archivedate=2016-12-25 |df= |location=Canberra |publisher=[[Department of the Environment, Water, Heritage and the Arts]]: [[Australian Biological Resources Study]] |isbn=978-0-642-56860-1 |oclc=780539206 |ref=harv}} |
|||
* {{cite book |last=Coyne |first=Jerry A. |authorlink=Jerry Coyne |year=2009 |title=Why Evolution is True |location=New York |publisher=[[Viking Press|Viking]] |isbn=978-0-670-02053-9 |lccn=2008033973 |oclc=233549529 |ref=harv}} |
|||
* {{cite book |editor1-last=Cracraft |editor1-first=Joel |editor2-last=Bybee |editor2-first=Rodger W. |year=2005 |title=Evolutionary Science and Society: Educating a New Generation |url=http://www.amnh.org/learn/resources/bscs_evolution.pdf |format=PDF |location=Colorado Springs, Colorado |publisher=[[Biological Sciences Curriculum Study]] |isbn=978-1-929614-23-3 |oclc=64228003 |accessdate=2014-12-06 |ref=harv}} "Revised Proceedings of the BSCS, AIBS Symposium November 2004, Chicago, Illinois" |
|||
* {{cite book |last=Dalrymple |first=G. Brent |authorlink=Brent Dalrymple |year=2001 |chapter=The age of the Earth in the twentieth century: a problem (mostly) solved |editor1-last=Lewis |editor1-first=C.L.E. |editor2-last=Knell |editor2-first=S.J. |title=The Age of the Earth: from 4004 BC to AD 2002 |journal=Geological Society, London, Special Publications |series=Geological Society Special Publication |volume=190 |issue=1 |pages=205–221 |bibcode=2001GSLSP.190..205D |doi=10.1144/gsl.sp.2001.190.01.14 |isbn=978-1-86239-093-5 |lccn=2003464816 |oclc=48570033 |ref=harv}} |
|||
* {{cite book |last=Darwin |first=Charles |authorlink=Charles Darwin |year=1859 |title=On the Origin of Species by Means of Natural Selection, or the Preservation of Favoured Races in the Struggle for Life |edition=1st |location=London |publisher=[[John Murray (publisher)|John Murray]] |lccn=06017473 |oclc=741260650 |ref=harv|title-link=On the Origin of Species }} The book is available from [http://darwin-online.org.uk/content/frameset?pageseq=1&itemID=F373&viewtype=side The Complete Work of Charles Darwin Online]. Retrieved 2014-11-21. |
|||
* {{cite book |last=Darwin |first=Charles |date=1872 |title=The Expression of the Emotions in Man and Animals |location=London |publisher=John Murray |lccn=04002793 |oclc=1102785 |ref=harv|title-link=The Expression of the Emotions in Man and Animals }} |
|||
* {{cite book |editor-last=Darwin |editor-first=Francis |editor-link=Francis Darwin |year=1909 |title=The foundations of The origin of species, a sketch written in 1842 |url=http://darwin-online.org.uk/converted/pdf/1909_Foundations_F1555.pdf |format=PDF |location=Cambridge |publisher=Printed at the University Press |lccn=61057537 |oclc=1184581 |accessdate=2014-11-27 |ref=harv}} |
|||
* {{cite book |last=Dawkins |first=Richard |authorlink=Richard Dawkins |year=1990 |title=The Blind Watchmaker |series=Penguin Science |location=London |publisher=[[Penguin Books]] |isbn=978-0-14-014481-9 |oclc=60143870 |ref=harv|title-link=The Blind Watchmaker }} |
|||
* {{cite book |last=Dennett |first=Daniel |authorlink=Daniel Dennett |year=1995 |title=Darwin's Dangerous Idea: Evolution and the Meanings of Life |location=New York |publisher=[[Simon & Schuster]] |isbn=978-0-684-80290-9 |lccn=94049158 |oclc=31867409 |ref=harv|title-link=Darwin's Dangerous Idea }} |
|||
* {{cite book |last=Dobzhansky |first=Theodosius |authorlink1=Theodosius Dobzhansky |year=1968 |chapter=On Some Fundamental Concepts of Darwinian Biology |editor1-last=Dobzhansky |editor1-first=Theodosius |editor2-last=Hecht |editor2-first=Max K. |editor3-last=Steere |editor3-first=William C. |title=Evolutionary Biology. Volume 2 |pages=1–34 |edition=1st |location=New York |publisher=[[Appleton-Century-Crofts]] |doi=10.1007/978-1-4684-8094-8_1 |oclc=24875357 |ref=harv|isbn=978-1-4684-8096-2 }} |
|||
* {{cite book |last=Dobzhansky |first=Theodosius |year=1970 |title=Genetics of the Evolutionary Process |location=New York |publisher=[[Columbia University Press]] |isbn=978-0-231-02837-0 |lccn=72127363 |oclc=97663 |ref=harv}} |
|||
* {{cite book |last1=Eldredge |first1=Niles |authorlink1=Niles Eldredge |last2=Gould |first2=Stephen Jay |authorlink2=Stephen Jay Gould |year=1972 |chapter=Punctuated equilibria: an alternative to phyletic gradualism |editor1-last=Schopf |editor1-first=Thomas J.M. |title=Models in Paleobiology |location=San Francisco, California |publisher=Freeman, Cooper |isbn=978-0-87735-325-6 |lccn=72078387 |oclc=572084 |ref=harv}} |
|||
** {{cite book |last=Eldredge |first=Niles |year=1985 |title=Time Frames: The Rethinking of Darwinian Evolution and the Theory of Punctuated Equilibria |location=New York |publisher=[[Simon & Schuster]] |isbn=978-0-671-49555-8 |lccn=84023632 |oclc=11443805}} |
|||
* {{cite book |author=Ewens |first=Warren J. |authorlink=Warren Ewens |year=2004 |title=Mathematical Population Genetics |series=Interdisciplinary Applied Mathematics |volume='''I'''. Theoretical Introduction |edition=2nd |location=New York |publisher=[[Springer Science+Business Media|Springer-Verlag New York]] |isbn=978-0-387-20191-7 |lccn=2003065728 |oclc=53231891 |ref=harv}} |
|||
* {{cite book |last=Futuyma |first=Douglas J. |authorlink=Douglas J. Futuyma |year=2004 |chapter=The Fruit of the Tree of Life: Insights into Evolution and Ecology |editor1-last=Cracraft |editor1-first=Joel |editor2-last=Donoghue |editor2-first=Michael J. |title=Assembling the Tree of Life |location=Oxford; New York |publisher=[[Oxford University Press]] |isbn=978-0-19-517234-8 |lccn=2003058012 |oclc=61342697 |ref=harv}} "Proceedings of a symposium held at the American Museum of Natural History in New York, 2002." |
|||
* {{cite book |last=Futuyma |first=Douglas J. |year=2005 |title=Evolution |location=Sunderland, Massachusetts |publisher=[[Sinauer Associates]] |isbn=978-0-87893-187-3 |lccn=2004029808 |oclc=57311264 |ref=harv}} |
|||
* {{cite book |last=Gould |first=Stephen Jay |year=2002 |title=The Structure of Evolutionary Theory |location=Cambridge, Massachusetts |publisher=[[Harvard University Press|Belknap Press of Harvard University Press]] |isbn=978-0-674-00613-3 |lccn=2001043556 |oclc=47869352 |ref=harv|title-link=The Structure of Evolutionary Theory }} |
|||
* {{cite book |last=Gray |first=Peter |authorlink=Peter Gray (psychologist) |year=2007 |title=Psychology |edition=5th |location=New York |publisher=[[Macmillan Publishers (United States)|Worth Publishers]] |isbn=978-0-7167-0617-5 |lccn=2006921149 |oclc=76872504 |ref=harv}} |
|||
* {{cite book |last1=Hall |first1=Brian K. |authorlink1=Brian K. Hall |last2=Hallgrímsson |first2=Benedikt |title=Strickberger's Evolution |year=2008 |edition=4th |location=Sudbury, Massachusetts |publisher=Jones and Bartlett Publishers |isbn=978-0-7637-0066-9 |lccn=2007008981 |oclc=85814089 |ref=harv}} |
|||
* {{cite book |last=Hennig |first=Willi |authorlink=Willi Hennig |year=1999 |origyear=Originally published 1966 (reprinted 1979); translated from the author's unpublished revision of ''Grundzüge einer Theorie der phylogenetischen Systematik'', published in 1950 |title=Phylogenetic Systematics |others=Translation by D. Dwight Davis and Rainer Zangerl; foreword by Donn E. Rosen, Gareth Nelson, and [[Colin Patterson (biologist)|Colin Patterson]] |edition=Reissue |location=Urbana, Illinois |publisher=[[University of Illinois Press]] |isbn=978-0-252-06814-0 |lccn=78031969 |oclc=722701473 |ref=harv}} |
|||
* {{cite book |last=Holland |first=John H. |authorlink=John Henry Holland |year=1975 |title=Adaptation in Natural and Artificial Systems: An Introductory Analysis with Applications to Biology, Control, and Artificial Intelligence |location=Ann Arbor, Michigan |publisher=[[University of Michigan Press]] |isbn=978-0-472-08460-9 |lccn=74078988 |oclc=1531617 |ref=harv}} |
|||
* {{cite book |last1=Jablonka |first1=Eva |authorlink1=Eva Jablonka |last2=Lamb |first2=Marion J. |authorlink2=Marion J. Lamb |year=2005 |title=Evolution in Four Dimensions: Genetic, Epigenetic, Behavioral, and Symbolic Variation in the History of Life |others=Illustrations by Anna Zeligowski |location=Cambridge, Massachusetts |publisher=[[MIT Press]] |isbn=978-0-262-10107-3 |lccn=2004058193 |oclc=61896061 |ref=harv}} |
|||
* {{cite book |last=Kampourakis |first=Kostas |year=2014 |title=Understanding Evolution |location=Cambridge; New York |publisher=Cambridge University Press |isbn=978-1-107-03491-4 |lccn=2013034917 |oclc=855585457 |ref=harv}} |
|||
* {{cite book |last1=Kirk |first1=Geoffrey |authorlink1=Geoffrey Kirk |last2=Raven |first2=John |authorlink2=John Raven |last3=Schofield |first3=Malcolm |year=1983 |title=The Presocratic Philosophers: A Critical History with a Selection of Texts |edition=2nd |location=Cambridge; New York |publisher=Cambridge University Press |isbn=978-0-521-27455-5 |lccn=82023505 |oclc=9081712 |ref=harv}} |
|||
* {{cite book |last=Koza |first=John R. |authorlink=John Koza |year=1992 |title=Genetic Programming: On the Programming of Computers by Means of Natural Selection |series=Complex Adaptive Systems |location=Cambridge, Massachusetts |publisher=MIT Press |isbn=978-0-262-11170-6 |lccn=92025785 |oclc=26263956 |ref=harv}} |
|||
* {{cite book |last=Lamarck |first=Jean-Baptiste |authorlink=Jean-Baptiste Lamarck |year=1809 |title=Philosophie Zoologique |location=Paris |publisher=Dentu et L'Auteur |oclc=2210044 |ref=harv|title-link=Philosophie Zoologique }} {{Internet Archive|id=philosophiezool06unkngoog|name=Philosophie zoologique (1809)}}. Retrieved 2014-11-29. |
|||
* {{cite book |last=Lane |first=David H. |year=1996 |title=The Phenomenon of Teilhard: Prophet for a New Age |edition=1st |location=Macon, Georgia |publisher=[[Mercer University Press]] |isbn=978-0-86554-498-7 |lccn=96008777 |oclc=34710780 |ref=harv}} |
|||
* {{cite book |author=Lucretius |authorlink=Lucretius |chapter=Book V, lines 855–877 |chapterurl=http://www.perseus.tufts.edu/hopper/text?doc=Perseus%3Atext%3A1999.02.0131%3Abook%3D5%3Acard%3D855 |title=De Rerum Natura |website=[[Perseus Project|Perseus Digital Library]] |others=Edited and translated by [[William Ellery Leonard]] (1916) |location=Medford/Somerville, Massachusetts |publisher=[[Tufts University]] |oclc=33233743 |accessdate=2014-11-25 |deadurl=no |archiveurl=https://web.archive.org/web/20140904053325/http://www.perseus.tufts.edu/hopper/text?doc=Perseus%3Atext%3A1999.02.0131%3Abook%3D5%3Acard%3D855 |archivedate=2014-09-04 |ref=harv|title-link=De rerum natura }} |
|||
* {{cite book |last=Magner |first=Lois N. |year=2002 |title=A History of the Life Sciences |edition=3rd rev. and expanded |location=New York |publisher=[[Marcel Dekker]] |isbn=978-0-8247-0824-5 |lccn=2002031313 |oclc=50410202 |ref=harv}} |
|||
* {{cite book |last=Mason |first=Stephen F. |year=1962 |title=A History of the Sciences |series=Collier Books. Science Library, CS9 |edition=New rev. |location=New York |publisher=[[Collier Books]] |lccn=62003378 |oclc=568032626 |ref=harv}} |
|||
* {{cite book |last=Maynard Smith |first=John |authorlink=John Maynard Smith |year=1978 |title=The Evolution of Sex |location=Cambridge; New York |publisher=Cambridge University Press |isbn=978-0-521-29302-0 |lccn=77085689 |oclc=3413793 |ref=harv}} |
|||
* {{cite book |last=Maynard Smith |first=John |year=1998 |chapter=The Units of Selection |editor1-last=Bock |editor1-first=Gregory R. |editor2-last=Goode |editor2-first=Jamie A. |title=The Limits of Reductionism in Biology |journal=Novartis Foundation Symposium |series=Novartis Foundation Symposia |volume=213 |pages=203–221 |location=Chichester, England |publisher=[[John Wiley & Sons]] |doi=10.1002/9780470515488.ch15 |isbn=978-0-471-97770-4 |lccn=98002779 |oclc=38311600 |pmid=9653725 |ref=harv}} "Papers from the Symposium on the Limits of Reductionism in Biology, held at the Novartis Foundation, London, May 13–15, 1997." |
|||
* {{cite book |last=Mayr |first=Ernst |authorlink=Ernst Mayr |year=1942 |title=Systematics and the Origin of Species from the Viewpoint of a Zoologist |series=Columbia Biological Series |volume=13 |location=New York |publisher=Columbia University Press |lccn=43001098 |oclc=766053 |ref=harv|title-link=Systematics and the Origin of Species }} |
|||
* {{cite book |last=Mayr |first=Ernst |year=1982 |title=The Growth of Biological Thought: Diversity, Evolution, and Inheritance |others=Translation of [[John Ray]] by E. Silk |location=Cambridge, Massachusetts |publisher=[[Harvard University Press|Belknap Press]] |isbn=978-0-674-36445-5 |lccn=81013204 |oclc=7875904 |ref=harv|title-link=The Growth of Biological Thought }} |
|||
* {{cite book |last=Mayr |first=Ernst |year=2002 |origyear=Originally published 2001; New York: [[Basic Books]] |title=What Evolution Is |series=Science Masters |location=London |publisher=[[Weidenfeld & Nicolson]] |isbn=978-0-297-60741-0 |lccn=2001036562 |oclc=248107061 |ref=harv}} |
|||
* {{cite book |last=McKinney |first=Michael L. |year=1997 |chapter=How do rare species avoid extinction? A paleontological view |editor1-last=Kunin |editor1-first=William E. |editor2-last=Gaston |editor2-first=Kevin J. |title=The Biology of Rarity: Causes and consequences of rare—common differences |edition=1st |location=London; New York |publisher=[[Chapman & Hall]] |isbn=978-0-412-63380-5 |lccn=96071014 |oclc=36442106 |ref=harv}} |
|||
* {{cite book |last1=Miller |first1=G. Tyler |last2=Spoolman |first2=Scott E. |year=2012 |title=Environmental Science |url=https://books.google.com/books?id=NYEJAAAAQBAJ&pg=PA62 |edition=14th |location=Belmont, California |publisher=[[Cengage Learning|Brooks/Cole]] |isbn=978-1-111-98893-7 |lccn=2011934330 |oclc=741539226 |accessdate=2014-12-27 |ref=harv}} |
|||
* {{cite book |last1=Moore |first1=Randy |last2=Decker |first2=Mark |last3=Cotner |first3=Sehoya |year=2010 |title=Chronology of the Evolution-Creationism Controversy |location=Santa Barbara, California |publisher=[[Greenwood Publishing Group|Greenwood Press]]/[[ABC-CLIO]] |isbn=978-0-313-36287-3 |lccn=2009039784 |oclc=422757410 |ref=harv}} |
|||
* {{cite book |last1=Nardon |first1=Paul |last2=Grenier |first2=Anne-Marie |year=1991 |chapter=Serial Endosymbiosis Theory and Weevil Evolution: The Role of Symbiosis |editor1-last=Margulis |editor1-first=Lynn |editor2-last=Fester |editor2-first=René |title=Symbiosis as a Source of Evolutionary Innovation: Speciation and Morphogenesis |location=Cambridge, Massachusetts |publisher=MIT Press |isbn=978-0-262-13269-5 |lccn=90020439 |oclc=22597587 |ref=harv}} "Based on a conference held in Bellagio, Italy, June 25–30, 1989" |
|||
* {{cite book |author1=National Academy of Sciences |authorlink1=National Academy of Sciences |author2=Institute of Medicine |authorlink2=Institute of Medicine |year=2008 |title=Science, Evolution, and Creationism |url=http://www.nap.edu/catalog.php?record_id=11876 |location=Washington, DC |publisher=National Academy Press |isbn=978-0-309-10586-6 |lccn=2007015904 |oclc=123539346 |accessdate=2014-11-22 |ref=NAS 2008}} |
|||
* {{cite book |last=Odum |first=Eugene P. |authorlink=Eugene Odum |year=1971 |title=Fundamentals of Ecology |edition=3rd |location=Philadelphia, Pennsylvania |publisher=[[Saunders (imprint)|Saunders]] |isbn=978-0-7216-6941-0 |lccn=76081826 |oclc=154846 |ref=harv}} |
|||
* {{cite book |last=Okasha |first=Samir |year=2006 |title=Evolution and the Levels of Selection |location=Oxford; New York |publisher=Oxford University Press |isbn=978-0-19-926797-2 |lccn=2006039679 |oclc=70985413 |ref=harv}} |
|||
* {{cite book |last=Panno |first=Joseph |title=The Cell: Evolution of the First Organism |year=2005 |series=Facts on File science library |location=New York |publisher=[[Infobase Publishing|Facts on File]] |isbn=978-0-8160-4946-2 |lccn=2003025841 |oclc=53901436 |ref=harv}} |
|||
* {{cite book |last1=Piatigorsky |first1=Joram |last2=Kantorow |first2=Marc |last3=Gopal-Srivastava |first3=Rashmi |last4=Tomarev |first4=Stanislav I. |year=1994 |chapter=Recruitment of enzymes and stress proteins as lens crystallins |editor1-last=Jansson |editor1-first=Bengt |editor2-last=Jörnvall |editor2-first=Hans |editor3-last=Rydberg |editor3-first=Ulf |editor4-last=Terenius |editor4-first=Lars |editor5-last=Vallee |editor5-first=Bert L. |display-editors=3 |title=Toward a Molecular Basis of Alcohol Use and Abuse |journal=Exs |series=Experientia |volume=71 |pages=241–50 |location=Basel; Boston |publisher=[[Birkhäuser|Birkhäuser Verlag]] |doi=10.1007/978-3-0348-7330-7_24 |isbn=978-3-7643-2940-2 |lccn=94010167 |oclc=30030941 |pmid=8032155 |ref=harv}} |
|||
* {{cite book |editor1-last=Pigliucci |editor1-first=Massimo |editor1-link=Massimo Pigliucci |editor2-last=Müller |editor2-first=Gerd B. |editor2-link=Gerd B. Müller |year=2010 |title=Evolution, the Extended Synthesis |url=http://muse.jhu.edu/books/9780262315142 |deadurl=no |archiveurl=https://web.archive.org/web/20150918231401/http://muse.jhu.edu/books/9780262315142 |archivedate=2015-09-18 |df= |location=Cambridge, Massachusetts |publisher=MIT Press |isbn=978-0-262-51367-8 |lccn=2009024587 |oclc=804875316 |ref=harv}} |
|||
* {{cite book |last1=Pocheville |first1=Arnaud |last2=Danchin |first2=Étienne |date=2017 |chapter=Chapter 3: Genetic assimilation and the paradox of blind variation |chapterurl=https://www.academia.edu/21713421 |editor-last1=Huneman |editor-first1=Philippe |editor-last2=Walsh |editor-first2=Denis M. |title=Challenging the Modern Synthesis |location=New York |publisher=Oxford University Press |isbn=978-0-19-937717-6 |lccn=2017448929 |oclc=1001337947 |ref=harv}} |
|||
* {{cite book |last=Provine |first=William B. |authorlink=Will Provine |year=1971 |title=The Origins of Theoretical Population Genetics |series=Chicago History of Science and Medicine |edition=2nd |location=Chicago, Illinois |publisher=[[University of Chicago Press]] |isbn=978-0-226-68464-2 |lccn=2001027561 |oclc=46660910 |ref=harv}} |
|||
* {{cite book |last=Provine |first=William B. |year=1988 |chapter=Progress in Evolution and Meaning in Life |editor-last=Nitecki |editor-first=Matthew H. |title=Evolutionary Progress |location=Chicago, Illinois |publisher=University of Chicago Press |isbn=978-0-226-58693-9 |lccn=88020835 |oclc=18380658 |ref=harv}} "This book is the result of the Spring Systematics Symposium held in May, 1987, at the Field Museum in Chicago" |
|||
* {{cite book |last=Quammen |first=David |authorlink=David Quammen |year=2006 |title=The Reluctant Mr. Darwin: An Intimate Portrait of Charles Darwin and the Making of His Theory of Evolution |series=Great Discoveries |edition=1st |location=New York |publisher=Atlas Books/[[W.W. Norton & Company]] |isbn=978-0-393-05981-6 |lccn=2006009864 |oclc=65400177 |ref=harv}} |
|||
* {{cite book |last1=Raven |first1=Peter H. |authorlink1=Peter H. Raven |last2=Johnson |first2=George B. |authorlink2=George B. Johnson |year=2002 |title=Biology |edition=6th |location=Boston, Massachusetts |publisher=[[McGraw-Hill Education|McGraw-Hill]] |isbn=978-0-07-112261-0 |lccn=2001030052 |oclc=45806501 |ref=harv}} |
|||
* {{cite book |last=Ray |first=John |authorlink=John Ray |year=1686 |title=Historia Plantarum |trans-title=History of Plants |volume=I |location=Londini |publisher=Typis Mariæ Clark |lccn=agr11000774 |oclc=2126030 |ref=harv}} |
|||
* {{cite book |last=Rechenberg |first=Ingo |authorlink=Ingo Rechenberg |year=1973 |title=Evolutionsstrategie; Optimierung technischer Systeme nach Prinzipien der biologischen Evolution |type=PhD thesis |series=Problemata |language=German |volume=15 |others=Afterword by [[Manfred Eigen]] |location=Stuttgart-Bad Cannstatt |publisher=Frommann-Holzboog |isbn=978-3-7728-0373-4 |lccn=74320689 |oclc=9020616 |ref=harv}} |
|||
* {{cite book |last=Ridley |first=Matt |authorlink=Matt Ridley |year=1993 |title=The Red Queen: Sex and the Evolution of Human Nature |location=New York |publisher=Viking |isbn=978-0-670-84357-2 |oclc=636657988 |ref=harv|title-link=The Red Queen: Sex and the Evolution of Human Nature }} |
|||
* {{cite book |last1=Stearns |first1=Beverly Peterson |last2=Stearns |first2=Stephen C. |author-link2=Stephen C. Stearns |year=1999 |title=Watching, from the Edge of Extinction |location=New Haven, Connecticut |publisher=[[Yale University Press]] |isbn=978-0-300-08469-6 |lccn=98034087 |oclc=803522914 |ref=harv}} |
|||
* {{cite book |last=Stevens |first=Anthony |authorlink=Anthony Stevens (Jungian analyst) |year=1982 |title=Archetype: A Natural History of the Self |location=London |publisher=[[Routledge|Routledge & Kegan Paul]] |isbn=978-0-7100-0980-7 |lccn=84672250 |oclc=10458367 |ref=harv}} |
|||
* {{cite book |last=West-Eberhard |first=Mary Jane |authorlink=Mary Jane West-Eberhard |year=2003 |title=Developmental Plasticity and Evolution |location=Oxford; New York |publisher=Oxford University Press |isbn=978-0-19-512235-0 |lccn=2001055164 |oclc=48398911 |ref=harv}} |
|||
* {{cite book |last1=Wiley |first1=E. O. |authorlink1=Edward O. Wiley |last2=Lieberman |first2=Bruce S. |year=2011 |title=Phylogenetics: Theory and Practice of Phylogenetic Systematics |edition=2nd |location=Hoboken, New Jersey |publisher=[[Wiley-Blackwell]] |isbn=978-0-470-90596-8 |lccn=2010044283 |oclc=741259265 |doi=10.1002/9781118017883 |ref=harv}} |
|||
* {{cite book |last=Wright |first=Sewall |authorlink=Sewall Wright |year=1984 |title=Genetic and Biometric Foundations |series=Evolution and the Genetics of Populations |volume=1 |location=Chicago, Illinois |publisher=University of Chicago Press |isbn=978-0-226-91038-3 |lccn=67025533 |oclc=246124737 |ref=harv}} |
|||
{{Refend}} |
|||
== Further reading == |
|||
{{further|Bibliography of biology}} |
|||
{{Library resources box |
|||
|onlinebooks=yes |
|||
|by=no |
|||
|lcheading=Evolution (Biology) |
|||
|label=Evolution |
|||
}} |
|||
{{refbegin}} |
|||
'''Introductory reading''' |
|||
* {{cite book |editor1-last=Barrett |editor1-first=Paul H. |editor2-last=Weinshank |editor2-first=Donald J. |editor3-last=Gottleber |editor3-first=Timothy T. |year=1981 |title=A Concordance to Darwin's Origin of Species, First Edition |location=Ithaca, New York |publisher=[[Cornell University Press]] |isbn=978-0-8014-1319-3 |lccn=80066893 |oclc=610057960}} |
|||
* {{cite book |last=Carroll |first=Sean B. |year=2005 |title=Endless Forms Most Beautiful: The New Science of Evo Devo and the Making of the Animal Kingdom |others=illustrations by Jamie W. Carroll, Josh P. Klaiss, Leanne M. Olds |edition=1st |location=New York |publisher=W.W. Norton & Company |isbn=978-0-393-06016-4 |lccn=2004029388 |oclc=57316841}} |
|||
* {{cite book |last1=Charlesworth |first1=Brian |authorlink1=Brian Charlesworth |last2=Charlesworth |first2=Deborah |authorlink2=Deborah Charlesworth |year=2003 |title=Evolution: A Very Short Introduction |series=Very Short Introductions |location=Oxford; New York |publisher=Oxford University Press |isbn=978-0-19-280251-4 |lccn=2003272247 |oclc=51668497}} |
|||
* {{cite book |last=Gould |first=Stephen Jay |year=1989 |title=Wonderful Life: The Burgess Shale and the Nature of History |edition=1st |location=New York |publisher=W.W. Norton & Company |isbn=978-0-393-02705-1 |lccn=88037469 |oclc=18983518|title-link=Wonderful Life (book) }} |
|||
* {{cite book |last=Jones |first=Steve |authorlink=Steve Jones (biologist) |year=1999 |title=Almost Like a Whale: The Origin of Species Updated |location=London; New York |publisher=[[Doubleday (publisher)|Doubleday]] |isbn=978-0-385-40985-8 |lccn=2002391059 |oclc=41420544|title-link=Almost Like a Whale }} |
|||
** {{cite book |last=Jones |first=Steve |year=2000 |title=Darwin's Ghost: The Origin of Species Updated |edition=1st |location=New York |publisher=[[Random House]] |isbn=978-0-375-50103-6 |lccn=99053246 |oclc=42690131 |author-mask=2}} American version. |
|||
* {{cite book |last=Mader |first=Sylvia S. |title=Biology |year=2007 |others=Significant contributions by Murray P. Pendarvis |edition=9th |location=Boston, Massachusetts |publisher=[[McGraw-Hill Education|McGraw-Hill Higher Education]] |isbn=978-0-07-246463-4 |lccn=2005027781 |oclc=61748307}} |
|||
* {{cite book |last=Maynard Smith |first=John |year=1993 |title=The Theory of Evolution |edition=Canto |location=Cambridge; New York |publisher=Cambridge University Press |isbn=978-0-521-45128-4 |lccn=93020358 |oclc=27676642|title-link=The Theory of Evolution }} |
|||
* {{cite book |last=Pallen |first=Mark J. |year=2009 |title=The Rough Guide to Evolution |series=Rough Guides Reference Guides |location=London; New York |publisher=[[Rough Guides]] |isbn=978-1-85828-946-5 |lccn=2009288090 |oclc=233547316}} |
|||
'''Advanced reading''' |
|||
* {{cite book |last1=Barton |first1=Nicholas H. |authorlink1=Nick Barton |last2=Briggs |first2=Derek E.G. |authorlink2=Derek Briggs |last3=Eisen |first3=Jonathan A. |authorlink3=Jonathan Eisen |last4=Goldstein |first4=David B. |last5=Patel |first5=Nipan H. |year=2007 |title=Evolution |location=Cold Spring Harbor, New York |publisher=Cold Spring Harbor Laboratory Press |isbn=978-0-87969-684-9 |lccn=2007010767 |oclc=86090399 |display-authors=3}} |
|||
* {{cite book |last1=Coyne |first1=Jerry A. |last2=Orr |first2=H. Allen |authorlink2=H. Allen Orr |year=2004 |title=Speciation |location=Sunderland, Massachusetts |publisher=Sinauer Associates |isbn=978-0-87893-089-0 |lccn=2004009505 |oclc=55078441}} |
|||
* {{cite book |last1=Bergstrom |first1=Carl T. |authorlink1=Carl Bergstrom |last2=Dugatkin |first2=Lee Alan |year=2012 |title=Evolution |edition=1st |location=New York |publisher=W.W. Norton & Company |isbn=978-0-393-91341-5 |lccn=2011036572 |oclc=729341924}} |
|||
* {{cite book |last=Gould |first=Stephen Jay |year=2002 |title=The Structure of Evolutionary Theory |location=Cambridge, Massachusetts |publisher=Belknap Press of Harvard University Press |isbn=978-0-674-00613-3 |lccn=2001043556 |oclc=47869352}} |
|||
* {{cite book |editor-last1=Hall |editor-first1=Brian K. |editor-last2=Olson |editor-first2=Wendy |year=2003 |title=Keywords and Concepts in Evolutionary Developmental Biology |location=Cambridge, Massachusetts |publisher=Harvard University Press |isbn=978-0-674-00904-2 |lccn=2002192201 |oclc=50761342}} |
|||
* {{cite book |last=Kauffman |first=Stuart A. |authorlink1=Stuart Kauffman |year=1993 |title=The Origins of Order: Self-organization and Selection in Evolution |url=https://books.google.com/?id=lZcSpRJz0dgC&printsec=frontcover |location=New York; Oxford |publisher=Oxford University Press |isbn=978-0-19-507951-7 |lccn=91011148 |oclc=895048122}} |
|||
* {{cite book |last1=Maynard Smith |first1=John |last2=Szathmáry |first2=Eörs |authorlink2=Eörs Szathmáry |year=1995 |title=The Major Transitions in Evolution |location=Oxford; New York |publisher=W.H. Freeman Spektrum |isbn=978-0-7167-4525-9 |lccn=94026965 |oclc=30894392|title-link=The Major Transitions in Evolution }} |
|||
* {{cite book |last=Mayr |first=Ernst |year=2001 |title=What Evolution Is |location=New York |publisher=Basic Books |isbn=978-0-465-04426-9 |lccn=2001036562 |oclc=47443814}} |
|||
* {{cite book |last=Minelli |first=Alessandro |authorlink=Alessandro Minelli |year=2009 |title=Forms of Becoming: The Evolutionary Biology of Development |others=Translation by Mark Epstein |location=Princeton, New Jersey; Oxford |publisher=[[Princeton University Press]] |isbn=978-0-691-13568-7 |lccn=2008028825 |oclc=233030259}} |
|||
{{refend}} |
|||
== External links == |
|||
<!-- IMPORTANT! Please do not add any links before discussing them on the talk page. --> |
|||
{{Spoken Wikipedia|Evolution.ogg|2005-04-18}} <!-- updated changed sections 2005-04-18 --> |
|||
{{Sister project links|evolution|voy=no}} |
|||
;General information |
|||
* {{In Our Time|"Evolution"|p00545gl}} |
|||
* {{cite journal |url=https://www.newscientist.com/topic/evolution |title=Evolution |journal=[[New Scientist]] |issn= 0262-4079 |accessdate=2011-05-30}} |
|||
* {{cite web |url=http://nationalacademies.org/evolution/ |title=Evolution Resources from the National Academies |publisher=[[National Academy of Sciences]] |location=Washington, DC |accessdate=2011-05-30}} |
|||
* {{cite web |url=http://evolution.berkeley.edu/ |title=Understanding Evolution: your one-stop resource for information on evolution |publisher=[[University of California, Berkeley]] |location=Berkeley, California |accessdate=2011-05-30}} |
|||
* {{cite web |url=https://www.nsf.gov/news/special_reports/darwin/textonly/index.jsp |title=Evolution of Evolution – 150 Years of Darwin's 'On the Origin of Species' |publisher=[[National Science Foundation]] |location=Arlington County, Virginia |accessdate=2011-05-30}} |
|||
* {{cite web |url=http://humanorigins.si.edu/evidence/human-evolution-timeline-interactive |title=Human Evolution Timeline Interactive |publisher=[[Smithsonian Institution]], [[National Museum of Natural History]] |accessdate=2018-07-14|date=2010-01-28 }} Adobe Flash required. |
|||
;Experiments concerning the process of biological evolution |
|||
* {{cite web |url=http://myxo.css.msu.edu/index.html |title=Experimental Evolution |last=Lenski |first=Richard E |authorlink=Richard Lenski |publisher=[[Michigan State University]] |location=East Lansing, Michigan |accessdate=2013-07-31}} |
|||
* {{cite journal |last1=Chastain |first1=Erick |last2=Livnat |first2=Adi |last3=Papadimitriou |first3=Christos |authorlink3=Christos Papadimitriou |last4=Vazirani |first4=Umesh |authorlink4=Umesh Vazirani |date=July 22, 2014 |title=Algorithms, games, and evolution |url=http://www.pnas.org/content/111/29/10620.full |journal=[[Proceedings of the National Academy of Sciences of the United States of America|Proc. Natl. Acad. Sci. U.S.A.]] |volume=111 |issue=29 |pages=10620–10623 |bibcode=2014PNAS..11110620C |doi=10.1073/pnas.1406556111 |pmid=24979793 |issn=0027-8424 |accessdate=2015-01-03|pmc=4115542}} |
|||
;Online lectures |
|||
* {{cite web |url=https://online-learning.harvard.edu/course/evolution-matters-lecture-series-0 |title=Evolution Matters Lecture Series |website=Harvard Online Learning Portal |publisher=[[Harvard University]] |location=Cambridge, Massachusetts |archive-url=https://web.archive.org/web/20171218132454/https://online-learning.harvard.edu/course/evolution-matters-lecture-series-0 |archive-date=2017-12-18 |dead-url=no |accessdate=2018-07-15}} |
|||
* {{cite web |url=https://oyc.yale.edu/ecology-and-evolutionary-biology/eeb-122 |title=EEB 122: Principles of Evolution, Ecology and Behavior |last=Stearns |first=Stephen C |authorlink=Stephen C. Stearns |website=[[Open Yale Courses]] |publisher=[[Yale University]] |location=New Haven, Connecticut |accessdate=2018-07-14 |archiveurl=https://web.archive.org/web/20171201233654/https://oyc.yale.edu/ecology-and-evolutionary-biology/eeb-122 |dead-url=no |archivedate=2017-12-01 |df= }} |
|||
{{Evolution|state=uncollapsed}} |
|||
{{Big History}} |
|||
{{Breakthrough of the Year}} |
|||
{{Featured article}} |
|||
{{Authority control}} |
|||
{{Portal bar|Evolutionary biology|Paleontology}} |
|||
[[Category:Biology theories]] |
|||
[[Category:Evolutionary biology|*]] |
|||
[[Category:Evolution| ]] |
|||
{{redirige aquí|Evolución}} |
{{redirige aquí|Evolución}} |
||
{{redirige aquí|Evolucionismo}} |
{{redirige aquí|Evolucionismo}} |
||
Revisión del 19:05 19 may 2019
Plantilla:About Plantilla:Pp-semi-protected Plantilla:Short description Plantilla:See introduction Plantilla:Evolutionary biology Plantilla:Use British English Evolution is change in the heritable characteristics of biological populations over successive generations.[1][2] These characteristics are the expressions of genes that are passed on from parent to offspring during reproduction. Different characteristics tend to exist within any given population as a result of mutation, genetic recombination and other sources of genetic variation.[3] Evolution occurs when evolutionary processes such as natural selection (including sexual selection) and genetic drift act on this variation, resulting in certain characteristics becoming more common or rare within a population.[4] It is this process of evolution that has given rise to biodiversity at every level of biological organisation, including the levels of species, individual organisms and molecules.[5][6]
The scientific theory of evolution by natural selection was proposed by Charles Darwin and Alfred Russel Wallace in the mid-19th century and was set out in detail in Darwin's book On the Origin of Species (1859).[7] Evolution by natural selection was first demonstrated by the observation that more offspring are often produced than can possibly survive. This is followed by three observable facts about living organisms: 1) traits vary among individuals with respect to their morphology, physiology and behaviour (phenotypic variation), 2) different traits confer different rates of survival and reproduction (differential fitness) and 3) traits can be passed from generation to generation (heritability of fitness).[8] Thus, in successive generations members of a population are more likely to be replaced by the progenies of parents with favourable characteristics that have enabled them to survive and reproduce in their respective environments. In the early 20th century, other competing ideas of evolution such as mutationism and orthogenesis were refuted as the modern synthesis reconciled Darwinian evolution with classical genetics, which established adaptive evolution as being caused by natural selection acting on Mendelian genetic variation.[9]
All life on Earth shares a last universal common ancestor (LUCA)[10][11][12] that lived approximately 3.5–3.8 billion years ago.[13] The fossil record includes a progression from early biogenic graphite,[14] to microbial mat fossils,[15][16][17] to fossilised multicellular organisms. Existing patterns of biodiversity have been shaped by repeated formations of new species (speciation), changes within species (anagenesis) and loss of species (extinction) throughout the evolutionary history of life on Earth.[18] Morphological and biochemical traits are more similar among species that share a more recent common ancestor, and can be used to reconstruct phylogenetic trees.[19][20]
Evolutionary biologists have continued to study various aspects of evolution by forming and testing hypotheses as well as constructing theories based on evidence from the field or laboratory and on data generated by the methods of mathematical and theoretical biology. Their discoveries have influenced not just the development of biology but numerous other scientific and industrial fields, including agriculture, medicine and computer science.[21]
History of evolutionary thought




Classical times
The proposal that one type of organism could descend from another type goes back to some of the first pre-Socratic Greek philosophers, such as Anaximander and Empedocles.[23] Such proposals survived into Roman times. The poet and philosopher Lucretius followed Empedocles in his masterwork De rerum natura (On the Nature of Things).[24][25]
Medieval
In contrast to these materialistic views, Aristotelianism considered all natural things as actualisations of fixed natural possibilities, known as forms.[26][27] This was part of a medieval teleological understanding of nature in which all things have an intended role to play in a divine cosmic order. Variations of this idea became the standard understanding of the Middle Ages and were integrated into Christian learning, but Aristotle did not demand that real types of organisms always correspond one-for-one with exact metaphysical forms and specifically gave examples of how new types of living things could come to be.[28]
Pre-Darwinian
In the 17th century, the new method of modern science rejected the Aristotelian approach. It sought explanations of natural phenomena in terms of physical laws that were the same for all visible things and that did not require the existence of any fixed natural categories or divine cosmic order. However, this new approach was slow to take root in the biological sciences, the last bastion of the concept of fixed natural types. John Ray applied one of the previously more general terms for fixed natural types, "species," to plant and animal types, but he strictly identified each type of living thing as a species and proposed that each species could be defined by the features that perpetuated themselves generation after generation.[29] The biological classification introduced by Carl Linnaeus in 1735 explicitly recognised the hierarchical nature of species relationships, but still viewed species as fixed according to a divine plan.[30]
Other naturalists of this time speculated on the evolutionary change of species over time according to natural laws. In 1751, Pierre Louis Maupertuis wrote of natural modifications occurring during reproduction and accumulating over many generations to produce new species.[31] Georges-Louis Leclerc, Comte de Buffon suggested that species could degenerate into different organisms, and Erasmus Darwin proposed that all warm-blooded animals could have descended from a single microorganism (or "filament").[32] The first full-fledged evolutionary scheme was Jean-Baptiste Lamarck's "transmutation" theory of 1809,[33] which envisaged spontaneous generation continually producing simple forms of life that developed greater complexity in parallel lineages with an inherent progressive tendency, and postulated that on a local level, these lineages adapted to the environment by inheriting changes caused by their use or disuse in parents.[34][35] (The latter process was later called Lamarckism.)[34][36][37][38] These ideas were condemned by established naturalists as speculation lacking empirical support. In particular, Georges Cuvier insisted that species were unrelated and fixed, their similarities reflecting divine design for functional needs. In the meantime, Ray's ideas of benevolent design had been developed by William Paley into the Natural Theology or Evidences of the Existence and Attributes of the Deity (1802), which proposed complex adaptations as evidence of divine design and which was admired by Charles Darwin.[39][40][41]
Darwinian revolution
The crucial break from the concept of constant typological classes or types in biology came with the theory of evolution through natural selection, which was formulated by Charles Darwin in terms of variable populations. Partly influenced by An Essay on the Principle of Population (1798) by Thomas Robert Malthus, Darwin noted that population growth would lead to a "struggle for existence" in which favourable variations prevailed as others perished. In each generation, many offspring fail to survive to an age of reproduction because of limited resources. This could explain the diversity of plants and animals from a common ancestry through the working of natural laws in the same way for all types of organism.[42][43][44][45] Darwin developed his theory of "natural selection" from 1838 onwards and was writing up his "big book" on the subject when Alfred Russel Wallace sent him a version of virtually the same theory in 1858. Their separate papers were presented together at an 1858 meeting of the Linnean Society of London.[46] At the end of 1859, Darwin's publication of his "abstract" as On the Origin of Species explained natural selection in detail and in a way that led to an increasingly wide acceptance of Darwin's concepts of evolution at the expense of alternative theories. Thomas Henry Huxley applied Darwin's ideas to humans, using paleontology and comparative anatomy to provide strong evidence that humans and apes shared a common ancestry. Some were disturbed by this since it implied that humans did not have a special place in the universe.[47]
Pangenesis and heredity
The mechanisms of reproductive heritability and the origin of new traits remained a mystery. Towards this end, Darwin developed his provisional theory of pangenesis.[48] In 1865, Gregor Mendel reported that traits were inherited in a predictable manner through the independent assortment and segregation of elements (later known as genes). Mendel's laws of inheritance eventually supplanted most of Darwin's pangenesis theory.[49] August Weismann made the important distinction between germ cells that give rise to gametes (such as sperm and egg cells) and the somatic cells of the body, demonstrating that heredity passes through the germ line only. Hugo de Vries connected Darwin's pangenesis theory to Weismann's germ/soma cell distinction and proposed that Darwin's pangenes were concentrated in the cell nucleus and when expressed they could move into the cytoplasm to change the cell's structure. De Vries was also one of the researchers who made Mendel's work well known, believing that Mendelian traits corresponded to the transfer of heritable variations along the germline.[50] To explain how new variants originate, de Vries developed a mutation theory that led to a temporary rift between those who accepted Darwinian evolution and biometricians who allied with de Vries.[35][51][52] In the 1930s, pioneers in the field of population genetics, such as Ronald Fisher, Sewall Wright and J. B. S. Haldane set the foundations of evolution onto a robust statistical philosophy. The false contradiction between Darwin's theory, genetic mutations, and Mendelian inheritance was thus reconciled.[53]
The 'modern synthesis'
In the 1920s and 1930s the so-called modern synthesis connected natural selection and population genetics, based on Mendelian inheritance, into a unified theory that applied generally to any branch of biology. The modern synthesis explained patterns observed across species in populations, through fossil transitions in palaeontology, and complex cellular mechanisms in developmental biology.[35][54] The publication of the structure of DNA by James Watson and Francis Crick with contribution of Rosalind Franklin in 1953 demonstrated a physical mechanism for inheritance.[55] Molecular biology improved understanding of the relationship between genotype and phenotype. Advancements were also made in phylogenetic systematics, mapping the transition of traits into a comparative and testable framework through the publication and use of evolutionary trees.[56][57] In 1973, evolutionary biologist Theodosius Dobzhansky penned that "nothing in biology makes sense except in the light of evolution," because it has brought to light the relations of what first seemed disjointed facts in natural history into a coherent explanatory body of knowledge that describes and predicts many observable facts about life on this planet.[58]
Further syntheses
Since then, the modern synthesis has been further extended to explain biological phenomena across the full and integrative scale of the biological hierarchy, from genes to species. One extension, known as evolutionary developmental biology and informally called "evo-devo," emphasises how changes between generations (evolution) acts on patterns of change within individual organisms (development).[59][60][61] Since the beginning of the 21st century and in light of discoveries made in recent decades, some biologists have argued for an extended evolutionary synthesis, which would account for the effects of non-genetic inheritance modes, such as epigenetics, parental effects, ecological inheritance and cultural inheritance, and evolvability.[62][63]
Heredity

Evolution in organisms occurs through changes in heritable traits—the inherited characteristics of an organism. In humans, for example, eye colour is an inherited characteristic and an individual might inherit the "brown-eye trait" from one of their parents.[64] Inherited traits are controlled by genes and the complete set of genes within an organism's genome (genetic material) is called its genotype.[65]
The complete set of observable traits that make up the structure and behaviour of an organism is called its phenotype. These traits come from the interaction of its genotype with the environment.[66] As a result, many aspects of an organism's phenotype are not inherited. For example, suntanned skin comes from the interaction between a person's genotype and sunlight; thus, suntans are not passed on to people's children. However, some people tan more easily than others, due to differences in genotypic variation; a striking example are people with the inherited trait of albinism, who do not tan at all and are very sensitive to sunburn.[67]
Heritable traits are passed from one generation to the next via DNA, a molecule that encodes genetic information.[65] DNA is a long biopolymer composed of four types of bases. The sequence of bases along a particular DNA molecule specify the genetic information, in a manner similar to a sequence of letters spelling out a sentence. Before a cell divides, the DNA is copied, so that each of the resulting two cells will inherit the DNA sequence. Portions of a DNA molecule that specify a single functional unit are called genes; different genes have different sequences of bases. Within cells, the long strands of DNA form condensed structures called chromosomes. The specific location of a DNA sequence within a chromosome is known as a locus. If the DNA sequence at a locus varies between individuals, the different forms of this sequence are called alleles. DNA sequences can change through mutations, producing new alleles. If a mutation occurs within a gene, the new allele may affect the trait that the gene controls, altering the phenotype of the organism.[68] However, while this simple correspondence between an allele and a trait works in some cases, most traits are more complex and are controlled by quantitative trait loci (multiple interacting genes).[69][70]
Recent findings have confirmed important examples of heritable changes that cannot be explained by changes to the sequence of nucleotides in the DNA. These phenomena are classed as epigenetic inheritance systems.[71] DNA methylation marking chromatin, self-sustaining metabolic loops, gene silencing by RNA interference and the three-dimensional conformation of proteins (such as prions) are areas where epigenetic inheritance systems have been discovered at the organismic level.[72][73] Developmental biologists suggest that complex interactions in genetic networks and communication among cells can lead to heritable variations that may underlay some of the mechanics in developmental plasticity and canalisation.[74] Heritability may also occur at even larger scales. For example, ecological inheritance through the process of niche construction is defined by the regular and repeated activities of organisms in their environment. This generates a legacy of effects that modify and feed back into the selection regime of subsequent generations. Descendants inherit genes plus environmental characteristics generated by the ecological actions of ancestors.[75] Other examples of heritability in evolution that are not under the direct control of genes include the inheritance of cultural traits and symbiogenesis.[76][77]
Variation
An individual organism's phenotype results from both its genotype and the influence from the environment it has lived in. A substantial part of the phenotypic variation in a population is caused by genotypic variation.[70] The modern evolutionary synthesis defines evolution as the change over time in this genetic variation. The frequency of one particular allele will become more or less prevalent relative to other forms of that gene. Variation disappears when a new allele reaches the point of fixation—when it either disappears from the population or replaces the ancestral allele entirely.[78]
Natural selection will only cause evolution if there is enough genetic variation in a population. Before the discovery of Mendelian genetics, one common hypothesis was blending inheritance. But with blending inheritance, genetic variance would be rapidly lost, making evolution by natural selection implausible. The Hardy–Weinberg principle provides the solution to how variation is maintained in a population with Mendelian inheritance. The frequencies of alleles (variations in a gene) will remain constant in the absence of selection, mutation, migration and genetic drift.[79]
Variation comes from mutations in the genome, reshuffling of genes through sexual reproduction and migration between populations (gene flow). Despite the constant introduction of new variation through mutation and gene flow, most of the genome of a species is identical in all individuals of that species.[80] However, even relatively small differences in genotype can lead to dramatic differences in phenotype: for example, chimpanzees and humans differ in only about 5% of their genomes.[81]
Mutation

Mutations are changes in the DNA sequence of a cell's genome. When mutations occur, they may alter the product of a gene, or prevent the gene from functioning, or have no effect. Based on studies in the fly Drosophila melanogaster, it has been suggested that if a mutation changes a protein produced by a gene, this will probably be harmful, with about 70% of these mutations having damaging effects, and the remainder being either neutral or weakly beneficial.[82]
Mutations can involve large sections of a chromosome becoming duplicated (usually by genetic recombination), which can introduce extra copies of a gene into a genome.[83] Extra copies of genes are a major source of the raw material needed for new genes to evolve.[84] This is important because most new genes evolve within gene families from pre-existing genes that share common ancestors.[85] For example, the human eye uses four genes to make structures that sense light: three for colour vision and one for night vision; all four are descended from a single ancestral gene.[86]
New genes can be generated from an ancestral gene when a duplicate copy mutates and acquires a new function. This process is easier once a gene has been duplicated because it increases the redundancy of the system; one gene in the pair can acquire a new function while the other copy continues to perform its original function.[87][88] Other types of mutations can even generate entirely new genes from previously noncoding DNA.[89][90]
The generation of new genes can also involve small parts of several genes being duplicated, with these fragments then recombining to form new combinations with new functions.[91][92] When new genes are assembled from shuffling pre-existing parts, domains act as modules with simple independent functions, which can be mixed together to produce new combinations with new and complex functions.[93] For example, polyketide synthases are large enzymes that make antibiotics; they contain up to one hundred independent domains that each catalyse one step in the overall process, like a step in an assembly line.[94]
Sex and recombination
In asexual organisms, genes are inherited together, or linked, as they cannot mix with genes of other organisms during reproduction. In contrast, the offspring of sexual organisms contain random mixtures of their parents' chromosomes that are produced through independent assortment. In a related process called homologous recombination, sexual organisms exchange DNA between two matching chromosomes.[95] Recombination and reassortment do not alter allele frequencies, but instead change which alleles are associated with each other, producing offspring with new combinations of alleles.[96] Sex usually increases genetic variation and may increase the rate of evolution.[97][98]
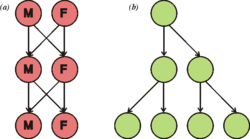
The two-fold cost of sex was first described by John Maynard Smith.[99] The first cost is that in sexually dimorphic species only one of the two sexes can bear young. (This cost does not apply to hermaphroditic species, like most plants and many invertebrates.) The second cost is that any individual who reproduces sexually can only pass on 50% of its genes to any individual offspring, with even less passed on as each new generation passes.[100] Yet sexual reproduction is the more common means of reproduction among eukaryotes and multicellular organisms. The Red Queen hypothesis has been used to explain the significance of sexual reproduction as a means to enable continual evolution and adaptation in response to coevolution with other species in an ever-changing environment.[100][101][102][103]
Gene flow
Gene flow is the exchange of genes between populations and between species.[104] It can therefore be a source of variation that is new to a population or to a species. Gene flow can be caused by the movement of individuals between separate populations of organisms, as might be caused by the movement of mice between inland and coastal populations, or the movement of pollen between heavy-metal-tolerant and heavy-metal-sensitive populations of grasses.
Gene transfer between species includes the formation of hybrid organisms and horizontal gene transfer. Horizontal gene transfer is the transfer of genetic material from one organism to another organism that is not its offspring; this is most common among bacteria.[105] In medicine, this contributes to the spread of antibiotic resistance, as when one bacteria acquires resistance genes it can rapidly transfer them to other species.[106] Horizontal transfer of genes from bacteria to eukaryotes such as the yeast Saccharomyces cerevisiae and the adzuki bean weevil Callosobruchus chinensis has occurred.[107][108] An example of larger-scale transfers are the eukaryotic bdelloid rotifers, which have received a range of genes from bacteria, fungi and plants.[109] Viruses can also carry DNA between organisms, allowing transfer of genes even across biological domains.[110]
Large-scale gene transfer has also occurred between the ancestors of eukaryotic cells and bacteria, during the acquisition of chloroplasts and mitochondria. It is possible that eukaryotes themselves originated from horizontal gene transfers between bacteria and archaea.[111]
Mechanisms

From a neo-Darwinian perspective, evolution occurs when there are changes in the frequencies of alleles within a population of interbreeding organisms,[79] for example, the allele for black colour in a population of moths becoming more common. Mechanisms that can lead to changes in allele frequencies include natural selection, genetic drift, genetic hitchhiking, mutation and gene flow.
Natural selection
Evolution by means of natural selection is the process by which traits that enhance survival and reproduction become more common in successive generations of a population. It has often been called a "self-evident" mechanism because it necessarily follows from three simple facts:[8]
- Variation exists within populations of organisms with respect to morphology, physiology, and behaviour (phenotypic variation).
- Different traits confer different rates of survival and reproduction (differential fitness).
- These traits can be passed from generation to generation (heritability of fitness).
More offspring are produced than can possibly survive, and these conditions produce competition between organisms for survival and reproduction. Consequently, organisms with traits that give them an advantage over their competitors are more likely to pass on their traits to the next generation than those with traits that do not confer an advantage.[112] This teleonomy is the quality whereby the process of natural selection creates and preserves traits that are seemingly fitted for the functional roles they perform.[113] Consequences of selection include nonrandom mating[114] and genetic hitchhiking.
The central concept of natural selection is the evolutionary fitness of an organism.[115] Fitness is measured by an organism's ability to survive and reproduce, which determines the size of its genetic contribution to the next generation.[115] However, fitness is not the same as the total number of offspring: instead fitness is indicated by the proportion of subsequent generations that carry an organism's genes.[116] For example, if an organism could survive well and reproduce rapidly, but its offspring were all too small and weak to survive, this organism would make little genetic contribution to future generations and would thus have low fitness.[115]
If an allele increases fitness more than the other alleles of that gene, then with each generation this allele will become more common within the population. These traits are said to be "selected for." Examples of traits that can increase fitness are enhanced survival and increased fecundity. Conversely, the lower fitness caused by having a less beneficial or deleterious allele results in this allele becoming rarer—they are "selected against."[117] Importantly, the fitness of an allele is not a fixed characteristic; if the environment changes, previously neutral or harmful traits may become beneficial and previously beneficial traits become harmful.[68] However, even if the direction of selection does reverse in this way, traits that were lost in the past may not re-evolve in an identical form (see Dollo's law).[118][119] However, a re-activation of dormant genes, as long as they have not been eliminated from the genome and were only suppressed perhaps for hundreds of generations, can lead to the re-occurrence of traits thought to be lost like hindlegs in dolphins, teeth in chickens, wings in wingless stick insects, tails and additional nipples in humans etc.[120] "Throwbacks" such as these are known as atavisms.

· Graph 1 shows directional selection, in which a single extreme phenotype is favoured.
· Graph 2 depicts stabilizing selection, where the intermediate phenotype is favoured over the extreme traits.
· Graph 3 shows disruptive selection, in which the extreme phenotypes are favoured over the intermediate.
Natural selection within a population for a trait that can vary across a range of values, such as height, can be categorised into three different types. The first is directional selection, which is a shift in the average value of a trait over time—for example, organisms slowly getting taller.[121] Secondly, disruptive selection is selection for extreme trait values and often results in two different values becoming most common, with selection against the average value. This would be when either short or tall organisms had an advantage, but not those of medium height. Finally, in stabilising selection there is selection against extreme trait values on both ends, which causes a decrease in variance around the average value and less diversity.[112][122] This would, for example, cause organisms to eventually have a similar height.
A special case of natural selection is sexual selection, which is selection for any trait that increases mating success by increasing the attractiveness of an organism to potential mates.[123] Traits that evolved through sexual selection are particularly prominent among males of several animal species. Although sexually favoured, traits such as cumbersome antlers, mating calls, large body size and bright colours often attract predation, which compromises the survival of individual males.[124][125] This survival disadvantage is balanced by higher reproductive success in males that show these hard-to-fake, sexually selected traits.[126]
Natural selection most generally makes nature the measure against which individuals and individual traits, are more or less likely to survive. "Nature" in this sense refers to an ecosystem, that is, a system in which organisms interact with every other element, physical as well as biological, in their local environment. Eugene Odum, a founder of ecology, defined an ecosystem as: "Any unit that includes all of the organisms...in a given area interacting with the physical environment so that a flow of energy leads to clearly defined trophic structure, biotic diversity, and material cycles (i.e., exchange of materials between living and nonliving parts) within the system...."[127] Each population within an ecosystem occupies a distinct niche, or position, with distinct relationships to other parts of the system. These relationships involve the life history of the organism, its position in the food chain and its geographic range. This broad understanding of nature enables scientists to delineate specific forces which, together, comprise natural selection.
Natural selection can act at different levels of organisation, such as genes, cells, individual organisms, groups of organisms and species.[128][129][130] Selection can act at multiple levels simultaneously.[131] An example of selection occurring below the level of the individual organism are genes called transposons, which can replicate and spread throughout a genome.[132] Selection at a level above the individual, such as group selection, may allow the evolution of cooperation, as discussed below.[133]
Biased mutation
In addition to being a major source of variation, mutation may also function as a mechanism of evolution when there are different probabilities at the molecular level for different mutations to occur, a process known as mutation bias.[134] If two genotypes, for example one with the nucleotide G and another with the nucleotide A in the same position, have the same fitness, but mutation from G to A happens more often than mutation from A to G, then genotypes with A will tend to evolve.[135] Different insertion vs. deletion mutation biases in different taxa can lead to the evolution of different genome sizes.[136][137] Developmental or mutational biases have also been observed in morphological evolution.[138][139] For example, according to the phenotype-first theory of evolution, mutations can eventually cause the genetic assimilation of traits that were previously induced by the environment.[140][141][142]
Mutation bias effects are superimposed on other processes. If selection would favour either one out of two mutations, but there is no extra advantage to having both, then the mutation that occurs the most frequently is the one that is most likely to become fixed in a population.[143][144] Mutations leading to the loss of function of a gene are much more common than mutations that produce a new, fully functional gene. Most loss of function mutations are selected against. But when selection is weak, mutation bias towards loss of function can affect evolution.[145] For example, pigments are no longer useful when animals live in the darkness of caves, and tend to be lost.[146] This kind of loss of function can occur because of mutation bias, and/or because the function had a cost, and once the benefit of the function disappeared, natural selection leads to the loss. Loss of sporulation ability in Bacillus subtilis during laboratory evolution appears to have been caused by mutation bias, rather than natural selection against the cost of maintaining sporulation ability.[147] When there is no selection for loss of function, the speed at which loss evolves depends more on the mutation rate than it does on the effective population size,[148] indicating that it is driven more by mutation bias than by genetic drift. In parasitic organisms, mutation bias leads to selection pressures as seen in Ehrlichia. Mutations are biased towards antigenic variants in outer-membrane proteins.
Genetic drift
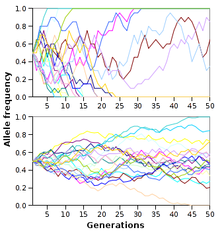
Genetic drift is the random fluctuations of allele frequencies within a population from one generation to the next.[149] When selective forces are absent or relatively weak, allele frequencies are equally likely to drift upward or downward at each successive generation because the alleles are subject to sampling error.[150] This drift halts when an allele eventually becomes fixed, either by disappearing from the population or replacing the other alleles entirely. Genetic drift may therefore eliminate some alleles from a population due to chance alone. Even in the absence of selective forces, genetic drift can cause two separate populations that began with the same genetic structure to drift apart into two divergent populations with different sets of alleles.[151]
The neutral theory of molecular evolution proposed that most evolutionary changes are the result of the fixation of neutral mutations by genetic drift.[152] Hence, in this model, most genetic changes in a population are the result of constant mutation pressure and genetic drift.[153] This form of the neutral theory is now largely abandoned, since it does not seem to fit the genetic variation seen in nature.[154][155] However, a more recent and better-supported version of this model is the nearly neutral theory, where a mutation that would be effectively neutral in a small population is not necessarily neutral in a large population.[112] Other alternative theories propose that genetic drift is dwarfed by other stochastic forces in evolution, such as genetic hitchhiking, also known as genetic draft.[150][156][157]
The time for a neutral allele to become fixed by genetic drift depends on population size, with fixation occurring more rapidly in smaller populations.[158] The number of individuals in a population is not critical, but instead a measure known as the effective population size.[159] The effective population is usually smaller than the total population since it takes into account factors such as the level of inbreeding and the stage of the lifecycle in which the population is the smallest.[159] The effective population size may not be the same for every gene in the same population.[160]
It is usually difficult to measure the relative importance of selection and neutral processes, including drift.[161] The comparative importance of adaptive and non-adaptive forces in driving evolutionary change is an area of current research.[162]
Genetic hitchhiking
Recombination allows alleles on the same strand of DNA to become separated. However, the rate of recombination is low (approximately two events per chromosome per generation). As a result, genes close together on a chromosome may not always be shuffled away from each other and genes that are close together tend to be inherited together, a phenomenon known as linkage.[163] This tendency is measured by finding how often two alleles occur together on a single chromosome compared to expectations, which is called their linkage disequilibrium. A set of alleles that is usually inherited in a group is called a haplotype. This can be important when one allele in a particular haplotype is strongly beneficial: natural selection can drive a selective sweep that will also cause the other alleles in the haplotype to become more common in the population; this effect is called genetic hitchhiking or genetic draft.[164] Genetic draft caused by the fact that some neutral genes are genetically linked to others that are under selection can be partially captured by an appropriate effective population size.[156]
Gene flow
Gene flow involves the exchange of genes between populations and between species.[104] The presence or absence of gene flow fundamentally changes the course of evolution. Due to the complexity of organisms, any two completely isolated populations will eventually evolve genetic incompatibilities through neutral processes, as in the Bateson-Dobzhansky-Muller model, even if both populations remain essentially identical in terms of their adaptation to the environment.
If genetic differentiation between populations develops, gene flow between populations can introduce traits or alleles which are disadvantageous in the local population and this may lead to organisms within these populations evolving mechanisms that prevent mating with genetically distant populations, eventually resulting in the appearance of new species. Thus, exchange of genetic information between individuals is fundamentally important for the development of the Biological Species Concept (BSC).
During the development of the modern synthesis, Sewall Wright developed his shifting balance theory, which regarded gene flow between partially isolated populations as an important aspect of adaptive evolution.[165] However, recently there has been substantial criticism of the importance of the shifting balance theory.[166]
Outcomes
Evolution influences every aspect of the form and behaviour of organisms. Most prominent are the specific behavioural and physical adaptations that are the outcome of natural selection. These adaptations increase fitness by aiding activities such as finding food, avoiding predators or attracting mates. Organisms can also respond to selection by cooperating with each other, usually by aiding their relatives or engaging in mutually beneficial symbiosis. In the longer term, evolution produces new species through splitting ancestral populations of organisms into new groups that cannot or will not interbreed.
These outcomes of evolution are distinguished based on time scale as macroevolution versus microevolution. Macroevolution refers to evolution that occurs at or above the level of species, in particular speciation and extinction; whereas microevolution refers to smaller evolutionary changes within a species or population, in particular shifts in allele frequency and adaptation.[168] In general, macroevolution is regarded as the outcome of long periods of microevolution.[169] Thus, the distinction between micro- and macroevolution is not a fundamental one—the difference is simply the time involved.[170] However, in macroevolution, the traits of the entire species may be important. For instance, a large amount of variation among individuals allows a species to rapidly adapt to new habitats, lessening the chance of it going extinct, while a wide geographic range increases the chance of speciation, by making it more likely that part of the population will become isolated. In this sense, microevolution and macroevolution might involve selection at different levels—with microevolution acting on genes and organisms, versus macroevolutionary processes such as species selection acting on entire species and affecting their rates of speciation and extinction.[171][172][173]
A common misconception is that evolution has goals, long-term plans, or an innate tendency for "progress", as expressed in beliefs such as orthogenesis and evolutionism; realistically however, evolution has no long-term goal and does not necessarily produce greater complexity.[174][175][176] Although complex species have evolved, they occur as a side effect of the overall number of organisms increasing and simple forms of life still remain more common in the biosphere.[177] For example, the overwhelming majority of species are microscopic prokaryotes, which form about half the world's biomass despite their small size,[178] and constitute the vast majority of Earth's biodiversity.[179] Simple organisms have therefore been the dominant form of life on Earth throughout its history and continue to be the main form of life up to the present day, with complex life only appearing more diverse because it is more noticeable.[180] Indeed, the evolution of microorganisms is particularly important to modern evolutionary research, since their rapid reproduction allows the study of experimental evolution and the observation of evolution and adaptation in real time.[181][182]
Adaptation
Adaptation is the process that makes organisms better suited to their habitat.[183][184] Also, the term adaptation may refer to a trait that is important for an organism's survival. For example, the adaptation of horses' teeth to the grinding of grass. By using the term adaptation for the evolutionary process and adaptive trait for the product (the bodily part or function), the two senses of the word may be distinguished. Adaptations are produced by natural selection.[185] The following definitions are due to Theodosius Dobzhansky:
- Adaptation is the evolutionary process whereby an organism becomes better able to live in its habitat or habitats.[186]
- Adaptedness is the state of being adapted: the degree to which an organism is able to live and reproduce in a given set of habitats.[187]
- An adaptive trait is an aspect of the developmental pattern of the organism which enables or enhances the probability of that organism surviving and reproducing.[188]
Adaptation may cause either the gain of a new feature, or the loss of an ancestral feature. An example that shows both types of change is bacterial adaptation to antibiotic selection, with genetic changes causing antibiotic resistance by both modifying the target of the drug, or increasing the activity of transporters that pump the drug out of the cell.[189] Other striking examples are the bacteria Escherichia coli evolving the ability to use citric acid as a nutrient in a long-term laboratory experiment,[190] Flavobacterium evolving a novel enzyme that allows these bacteria to grow on the by-products of nylon manufacturing,[191][192] and the soil bacterium Sphingobium evolving an entirely new metabolic pathway that degrades the synthetic pesticide pentachlorophenol.[193][194] An interesting but still controversial idea is that some adaptations might increase the ability of organisms to generate genetic diversity and adapt by natural selection (increasing organisms' evolvability).[195][196][197][198][199]
Adaptation occurs through the gradual modification of existing structures. Consequently, structures with similar internal organisation may have different functions in related organisms. This is the result of a single ancestral structure being adapted to function in different ways. The bones within bat wings, for example, are very similar to those in mice feet and primate hands, due to the descent of all these structures from a common mammalian ancestor.[201] However, since all living organisms are related to some extent,[202] even organs that appear to have little or no structural similarity, such as arthropod, squid and vertebrate eyes, or the limbs and wings of arthropods and vertebrates, can depend on a common set of homologous genes that control their assembly and function; this is called deep homology.[203][204]
During evolution, some structures may lose their original function and become vestigial structures.[205] Such structures may have little or no function in a current species, yet have a clear function in ancestral species, or other closely related species. Examples include pseudogenes,[206] the non-functional remains of eyes in blind cave-dwelling fish,[207] wings in flightless birds,[208] the presence of hip bones in whales and snakes,[200] and sexual traits in organisms that reproduce via asexual reproduction.[209] Examples of vestigial structures in humans include wisdom teeth,[210] the coccyx,[205] the vermiform appendix,[205] and other behavioural vestiges such as goose bumps[211][212] and primitive reflexes.[213][214][215]
However, many traits that appear to be simple adaptations are in fact exaptations: structures originally adapted for one function, but which coincidentally became somewhat useful for some other function in the process.[216] One example is the African lizard Holaspis guentheri, which developed an extremely flat head for hiding in crevices, as can be seen by looking at its near relatives. However, in this species, the head has become so flattened that it assists in gliding from tree to tree—an exaptation.[216] Within cells, molecular machines such as the bacterial flagella[217] and protein sorting machinery[218] evolved by the recruitment of several pre-existing proteins that previously had different functions.[168] Another example is the recruitment of enzymes from glycolysis and xenobiotic metabolism to serve as structural proteins called crystallins within the lenses of organisms' eyes.[219][220]
An area of current investigation in evolutionary developmental biology is the developmental basis of adaptations and exaptations.[221] This research addresses the origin and evolution of embryonic development and how modifications of development and developmental processes produce novel features.[222] These studies have shown that evolution can alter development to produce new structures, such as embryonic bone structures that develop into the jaw in other animals instead forming part of the middle ear in mammals.[223] It is also possible for structures that have been lost in evolution to reappear due to changes in developmental genes, such as a mutation in chickens causing embryos to grow teeth similar to those of crocodiles.[224] It is now becoming clear that most alterations in the form of organisms are due to changes in a small set of conserved genes.[225]
Coevolution

Interactions between organisms can produce both conflict and cooperation. When the interaction is between pairs of species, such as a pathogen and a host, or a predator and its prey, these species can develop matched sets of adaptations. Here, the evolution of one species causes adaptations in a second species. These changes in the second species then, in turn, cause new adaptations in the first species. This cycle of selection and response is called coevolution.[226] An example is the production of tetrodotoxin in the rough-skinned newt and the evolution of tetrodotoxin resistance in its predator, the common garter snake. In this predator-prey pair, an evolutionary arms race has produced high levels of toxin in the newt and correspondingly high levels of toxin resistance in the snake.[227]
Cooperation
Not all co-evolved interactions between species involve conflict.[228] Many cases of mutually beneficial interactions have evolved. For instance, an extreme cooperation exists between plants and the mycorrhizal fungi that grow on their roots and aid the plant in absorbing nutrients from the soil.[229] This is a reciprocal relationship as the plants provide the fungi with sugars from photosynthesis. Here, the fungi actually grow inside plant cells, allowing them to exchange nutrients with their hosts, while sending signals that suppress the plant immune system.[230]
Coalitions between organisms of the same species have also evolved. An extreme case is the eusociality found in social insects, such as bees, termites and ants, where sterile insects feed and guard the small number of organisms in a colony that are able to reproduce. On an even smaller scale, the somatic cells that make up the body of an animal limit their reproduction so they can maintain a stable organism, which then supports a small number of the animal's germ cells to produce offspring. Here, somatic cells respond to specific signals that instruct them whether to grow, remain as they are, or die. If cells ignore these signals and multiply inappropriately, their uncontrolled growth causes cancer.[231]
Such cooperation within species may have evolved through the process of kin selection, which is where one organism acts to help raise a relative's offspring.[232] This activity is selected for because if the helping individual contains alleles which promote the helping activity, it is likely that its kin will also contain these alleles and thus those alleles will be passed on.[233] Other processes that may promote cooperation include group selection, where cooperation provides benefits to a group of organisms.[234]
Speciation

Speciation is the process where a species diverges into two or more descendant species.[235]
There are multiple ways to define the concept of "species." The choice of definition is dependent on the particularities of the species concerned.[236] For example, some species concepts apply more readily toward sexually reproducing organisms while others lend themselves better toward asexual organisms. Despite the diversity of various species concepts, these various concepts can be placed into one of three broad philosophical approaches: interbreeding, ecological and phylogenetic.[237] The Biological Species Concept (BSC) is a classic example of the interbreeding approach. Defined by evolutionary biologist Ernst Mayr in 1942, the BSC states that "species are groups of actually or potentially interbreeding natural populations, which are reproductively isolated from other such groups."[238] Despite its wide and long-term use, the BSC like others is not without controversy, for example because these concepts cannot be applied to prokaryotes,[239] and this is called the species problem.[236] Some researchers have attempted a unifying monistic definition of species, while others adopt a pluralistic approach and suggest that there may be different ways to logically interpret the definition of a species.[236][237]
Barriers to reproduction between two diverging sexual populations are required for the populations to become new species. Gene flow may slow this process by spreading the new genetic variants also to the other populations. Depending on how far two species have diverged since their most recent common ancestor, it may still be possible for them to produce offspring, as with horses and donkeys mating to produce mules.[240] Such hybrids are generally infertile. In this case, closely related species may regularly interbreed, but hybrids will be selected against and the species will remain distinct. However, viable hybrids are occasionally formed and these new species can either have properties intermediate between their parent species, or possess a totally new phenotype.[241] The importance of hybridisation in producing new species of animals is unclear, although cases have been seen in many types of animals,[242] with the gray tree frog being a particularly well-studied example.[243]
Speciation has been observed multiple times under both controlled laboratory conditions (see laboratory experiments of speciation) and in nature.[244] In sexually reproducing organisms, speciation results from reproductive isolation followed by genealogical divergence. There are four primary geographic modes of speciation. The most common in animals is allopatric speciation, which occurs in populations initially isolated geographically, such as by habitat fragmentation or migration. Selection under these conditions can produce very rapid changes in the appearance and behaviour of organisms.[245][246] As selection and drift act independently on populations isolated from the rest of their species, separation may eventually produce organisms that cannot interbreed.[247]
The second mode of speciation is peripatric speciation, which occurs when small populations of organisms become isolated in a new environment. This differs from allopatric speciation in that the isolated populations are numerically much smaller than the parental population. Here, the founder effect causes rapid speciation after an increase in inbreeding increases selection on homozygotes, leading to rapid genetic change.[248]
The third mode is parapatric speciation. This is similar to peripatric speciation in that a small population enters a new habitat, but differs in that there is no physical separation between these two populations. Instead, speciation results from the evolution of mechanisms that reduce gene flow between the two populations.[235] Generally this occurs when there has been a drastic change in the environment within the parental species' habitat. One example is the grass Anthoxanthum odoratum, which can undergo parapatric speciation in response to localised metal pollution from mines.[249] Here, plants evolve that have resistance to high levels of metals in the soil. Selection against interbreeding with the metal-sensitive parental population produced a gradual change in the flowering time of the metal-resistant plants, which eventually produced complete reproductive isolation. Selection against hybrids between the two populations may cause reinforcement, which is the evolution of traits that promote mating within a species, as well as character displacement, which is when two species become more distinct in appearance.[250]
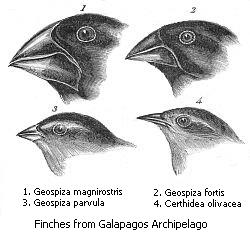
Finally, in sympatric speciation species diverge without geographic isolation or changes in habitat. This form is rare since even a small amount of gene flow may remove genetic differences between parts of a population.[251] Generally, sympatric speciation in animals requires the evolution of both genetic differences and nonrandom mating, to allow reproductive isolation to evolve.[252]
One type of sympatric speciation involves crossbreeding of two related species to produce a new hybrid species. This is not common in animals as animal hybrids are usually sterile. This is because during meiosis the homologous chromosomes from each parent are from different species and cannot successfully pair. However, it is more common in plants because plants often double their number of chromosomes, to form polyploids.[253] This allows the chromosomes from each parental species to form matching pairs during meiosis, since each parent's chromosomes are represented by a pair already.[254] An example of such a speciation event is when the plant species Arabidopsis thaliana and Arabidopsis arenosa crossbred to give the new species Arabidopsis suecica.[255] This happened about 20,000 years ago,[256] and the speciation process has been repeated in the laboratory, which allows the study of the genetic mechanisms involved in this process.[257] Indeed, chromosome doubling within a species may be a common cause of reproductive isolation, as half the doubled chromosomes will be unmatched when breeding with undoubled organisms.[258]
Speciation events are important in the theory of punctuated equilibrium, which accounts for the pattern in the fossil record of short "bursts" of evolution interspersed with relatively long periods of stasis, where species remain relatively unchanged.[259] In this theory, speciation and rapid evolution are linked, with natural selection and genetic drift acting most strongly on organisms undergoing speciation in novel habitats or small populations. As a result, the periods of stasis in the fossil record correspond to the parental population and the organisms undergoing speciation and rapid evolution are found in small populations or geographically restricted habitats and therefore rarely being preserved as fossils.[172]
Extinction

Extinction is the disappearance of an entire species. Extinction is not an unusual event, as species regularly appear through speciation and disappear through extinction.[260] Nearly all animal and plant species that have lived on Earth are now extinct,[261] and extinction appears to be the ultimate fate of all species.[262] These extinctions have happened continuously throughout the history of life, although the rate of extinction spikes in occasional mass extinction events.[263] The Cretaceous–Paleogene extinction event, during which the non-avian dinosaurs became extinct, is the most well-known, but the earlier Permian–Triassic extinction event was even more severe, with approximately 96% of all marine species driven to extinction.[263] The Holocene extinction event is an ongoing mass extinction associated with humanity's expansion across the globe over the past few thousand years. Present-day extinction rates are 100–1000 times greater than the background rate and up to 30% of current species may be extinct by the mid 21st century.[264] Human activities are now the primary cause of the ongoing extinction event;[265] global warming may further accelerate it in the future.[266] Despite the estimated extinction of more than 99 percent of all species that ever lived on Earth,[267][268] about 1 trillion species are estimated to be on Earth currently with only one-thousandth of one percent described.[269]
The role of extinction in evolution is not very well understood and may depend on which type of extinction is considered.[263] The causes of the continuous "low-level" extinction events, which form the majority of extinctions, may be the result of competition between species for limited resources (the competitive exclusion principle).[59] If one species can out-compete another, this could produce species selection, with the fitter species surviving and the other species being driven to extinction.[129] The intermittent mass extinctions are also important, but instead of acting as a selective force, they drastically reduce diversity in a nonspecific manner and promote bursts of rapid evolution and speciation in survivors.[270]
Evolutionary history of life
Origin of life
The Earth is about 4.54 billion years old.[271][272][273] The earliest undisputed evidence of life on Earth dates from at least 3.5 billion years ago,[13][274] during the Eoarchean Era after a geological crust started to solidify following the earlier molten Hadean Eon. Microbial mat fossils have been found in 3.48 billion-year-old sandstone in Western Australia.[15][16][17] Other early physical evidence of a biogenic substance is graphite in 3.7 billion-year-old metasedimentary rocks discovered in Western Greenland[14] as well as "remains of biotic life" found in 4.1 billion-year-old rocks in Western Australia.[275][276] Commenting on the Australian findings, Stephen Blair Hedges wrote, "If life arose relatively quickly on Earth, then it could be common in the universe."[275][277] In July 2016, scientists reported identifying a set of 355 genes from the last universal common ancestor (LUCA) of all organisms living on Earth.[278]
More than 99 percent of all species, amounting to over five billion species,[279] that ever lived on Earth are estimated to be extinct.[267][268] Estimates on the number of Earth's current species range from 10 million to 14 million,[280][281] of which about 1.9 million are estimated to have been named[282] and 1.6 million documented in a central database to date,[283] leaving at least 80 percent not yet described.
Highly energetic chemistry is thought to have produced a self-replicating molecule around 4 billion years ago, and half a billion years later the last common ancestor of all life existed.[11] The current scientific consensus is that the complex biochemistry that makes up life came from simpler chemical reactions.[284] The beginning of life may have included self-replicating molecules such as RNA[285] and the assembly of simple cells.[286]
Common descent
All organisms on Earth are descended from a common ancestor or ancestral gene pool.[202][287] Current species are a stage in the process of evolution, with their diversity the product of a long series of speciation and extinction events.[288] The common descent of organisms was first deduced from four simple facts about organisms: First, they have geographic distributions that cannot be explained by local adaptation. Second, the diversity of life is not a set of completely unique organisms, but organisms that share morphological similarities. Third, vestigial traits with no clear purpose resemble functional ancestral traits and finally, that organisms can be classified using these similarities into a hierarchy of nested groups—similar to a family tree.[289] However, modern research has suggested that, due to horizontal gene transfer, this "tree of life" may be more complicated than a simple branching tree since some genes have spread independently between distantly related species.[290][291]
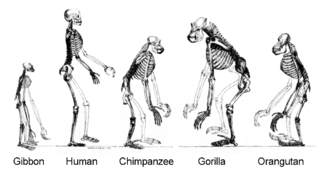
Past species have also left records of their evolutionary history. Fossils, along with the comparative anatomy of present-day organisms, constitute the morphological, or anatomical, record.[292] By comparing the anatomies of both modern and extinct species, paleontologists can infer the lineages of those species. However, this approach is most successful for organisms that had hard body parts, such as shells, bones or teeth. Further, as prokaryotes such as bacteria and archaea share a limited set of common morphologies, their fossils do not provide information on their ancestry.
More recently, evidence for common descent has come from the study of biochemical similarities between organisms. For example, all living cells use the same basic set of nucleotides and amino acids.[293] The development of molecular genetics has revealed the record of evolution left in organisms' genomes: dating when species diverged through the molecular clock produced by mutations.[294] For example, these DNA sequence comparisons have revealed that humans and chimpanzees share 98% of their genomes and analysing the few areas where they differ helps shed light on when the common ancestor of these species existed.[295]
Evolution of life
Plantilla:PhylomapA Prokaryotes inhabited the Earth from approximately 3–4 billion years ago.[296][297] No obvious changes in morphology or cellular organisation occurred in these organisms over the next few billion years.[298] The eukaryotic cells emerged between 1.6–2.7 billion years ago. The next major change in cell structure came when bacteria were engulfed by eukaryotic cells, in a cooperative association called endosymbiosis.[299][300] The engulfed bacteria and the host cell then underwent coevolution, with the bacteria evolving into either mitochondria or hydrogenosomes.[301] Another engulfment of cyanobacterial-like organisms led to the formation of chloroplasts in algae and plants.[302]
The history of life was that of the unicellular eukaryotes, prokaryotes and archaea until about 610 million years ago when multicellular organisms began to appear in the oceans in the Ediacaran period.[296][303] The evolution of multicellularity occurred in multiple independent events, in organisms as diverse as sponges, brown algae, cyanobacteria, slime moulds and myxobacteria.[304] In January 2016, scientists reported that, about 800 million years ago, a minor genetic change in a single molecule called GK-PID may have allowed organisms to go from a single cell organism to one of many cells.[305]
Soon after the emergence of these first multicellular organisms, a remarkable amount of biological diversity appeared over approximately 10 million years, in an event called the Cambrian explosion. Here, the majority of types of modern animals appeared in the fossil record, as well as unique lineages that subsequently became extinct.[306] Various triggers for the Cambrian explosion have been proposed, including the accumulation of oxygen in the atmosphere from photosynthesis.[307]
About 500 million years ago, plants and fungi colonised the land and were soon followed by arthropods and other animals.[308] Insects were particularly successful and even today make up the majority of animal species.[309] Amphibians first appeared around 364 million years ago, followed by early amniotes and birds around 155 million years ago (both from "reptile"-like lineages), mammals around 129 million years ago, homininae around 10 million years ago and modern humans around 250,000 years ago.[310][311][312] However, despite the evolution of these large animals, smaller organisms similar to the types that evolved early in this process continue to be highly successful and dominate the Earth, with the majority of both biomass and species being prokaryotes.[179]
Applications
Concepts and models used in evolutionary biology, such as natural selection, have many applications.[313]
Artificial selection is the intentional selection of traits in a population of organisms. This has been used for thousands of years in the domestication of plants and animals.[314] More recently, such selection has become a vital part of genetic engineering, with selectable markers such as antibiotic resistance genes being used to manipulate DNA. Proteins with valuable properties have evolved by repeated rounds of mutation and selection (for example modified enzymes and new antibodies) in a process called directed evolution.[315]
Understanding the changes that have occurred during an organism's evolution can reveal the genes needed to construct parts of the body, genes which may be involved in human genetic disorders.[316] For example, the Mexican tetra is an albino cavefish that lost its eyesight during evolution. Breeding together different populations of this blind fish produced some offspring with functional eyes, since different mutations had occurred in the isolated populations that had evolved in different caves.[317] This helped identify genes required for vision and pigmentation.[318]
Evolutionary theory has many applications in medicine. Many human diseases are not static phenomena, but capable of evolution. Viruses, bacteria, fungi and cancers evolve to be resistant to host immune defences, as well as pharmaceutical drugs.[319][320][321] These same problems occur in agriculture with pesticide[322] and herbicide[323] resistance. It is possible that we are facing the end of the effective life of most of available antibiotics[324] and predicting the evolution and evolvability[325] of our pathogens and devising strategies to slow or circumvent it is requiring deeper knowledge of the complex forces driving evolution at the molecular level.[326]
In computer science, simulations of evolution using evolutionary algorithms and artificial life started in the 1960s and were extended with simulation of artificial selection.[327] Artificial evolution became a widely recognised optimisation method as a result of the work of Ingo Rechenberg in the 1960s. He used evolution strategies to solve complex engineering problems.[328] Genetic algorithms in particular became popular through the writing of John Henry Holland.[329] Practical applications also include automatic evolution of computer programmes.[330] Evolutionary algorithms are now used to solve multi-dimensional problems more efficiently than software produced by human designers and also to optimise the design of systems.[331]
Social and cultural responses

In the 19th century, particularly after the publication of On the Origin of Species in 1859, the idea that life had evolved was an active source of academic debate centred on the philosophical, social and religious implications of evolution. Today, the modern evolutionary synthesis is accepted by a vast majority of scientists.[59] However, evolution remains a contentious concept for some theists.[333]
While various religions and denominations have reconciled their beliefs with evolution through concepts such as theistic evolution, there are creationists who believe that evolution is contradicted by the creation myths found in their religions and who raise various objections to evolution.[168][334][335] As had been demonstrated by responses to the publication of Vestiges of the Natural History of Creation in 1844, the most controversial aspect of evolutionary biology is the implication of human evolution that humans share common ancestry with apes and that the mental and moral faculties of humanity have the same types of natural causes as other inherited traits in animals.[336] In some countries, notably the United States, these tensions between science and religion have fuelled the current creation–evolution controversy, a religious conflict focusing on politics and public education.[337] While other scientific fields such as cosmology[338] and Earth science[339] also conflict with literal interpretations of many religious texts, evolutionary biology experiences significantly more opposition from religious literalists.
The teaching of evolution in American secondary school biology classes was uncommon in most of the first half of the 20th century. The Scopes Trial decision of 1925 caused the subject to become very rare in American secondary biology textbooks for a generation, but it was gradually re-introduced later and became legally protected with the 1968 Epperson v. Arkansas decision. Since then, the competing religious belief of creationism was legally disallowed in secondary school curricula in various decisions in the 1970s and 1980s, but it returned in pseudoscientific form as intelligent design (ID), to be excluded once again in the 2005 Kitzmiller v. Dover Area School District case.[340] The debate over Darwin's ideas did not generate significant controversy in China. [341]
See also
- Argument from poor design
- Biocultural evolution
- Biological classification
- Evidence of common descent
- Evolution in Variable Environment
- Evolutionary anthropology
- Evolutionary ecology
- Evolutionary epistemology
- Evolutionary neuroscience
- Evolution of biological complexity
- Evolution of plants
- Project Steve
- Timeline of the evolutionary history of life
- Universal Darwinism
|}
References
- ↑ Hall y Hallgrímsson, 2008, pp. 4–6
- ↑ «Evolution Resources». Washington, DC: National Academies of Sciences, Engineering, and Medicine. 2016. Archivado desde el original el 3 de junio de 2016.
- ↑ Futuyma, Douglas J.; Kirkpatrick, Mark (2017). «Mutation and variation». Evolution (Fourth edición). Sunderland, Massachusetts: Sinauer Associates, Inc. pp. 79-102. ISBN 978-1-60535-605-1.
- ↑ Scott-Phillips, Thomas C.; Laland, Kevin N.; Shuker, David M.; Dickins, Thomas E.; West, Stuart A. (May 2014). «The Niche Construction Perspective: A Critical Appraisal». Evolution 68 (5): 1231-1243. ISSN 0014-3820. PMC 4261998. PMID 24325256. doi:10.1111/evo.12332. «Evolutionary processes are generally thought of as processes by which these changes occur. Four such processes are widely recognized: natural selection (in the broad sense, to include sexual selection), genetic drift, mutation, and migration (Fisher 1930; Haldane 1932). The latter two generate variation; the first two sort it.»
- ↑ Hall y Hallgrímsson, 2008, pp. 3–5
- ↑ Voet, Donald; Voet, Judith G.; Pratt, Charlotte W. (2016). «Introduction to the chemistry of life». Fundamentals of Biochemistry: Life at the molecular level (Fifth edición). Hoboken, New Jersey: Wiley. pp. 1-22. ISBN 1-11-891840-1.
- ↑ Darwin, Charles (1860). On the Origin of Species (2nd edición). London: John Murray. p. 490.
- ↑ a b Lewontin, Richard C. (November 1970). «The Units of Selection». Annual Review of Ecology and Systematics 1: 1-18. ISSN 1545-2069. JSTOR 2096764. doi:10.1146/annurev.es.01.110170.000245. Archivado desde el original el 6 de febrero de 2015.
- ↑ Futuyma, Douglas J.; Kirkpatrick, Mark (2017). «Evolutionary Biology». Evolution (Fourth edición). Sunderland, Massachusetts: Sinauer Associates, Inc. pp. 3-26. ISBN 978-1-60535-605-1.
- ↑ Kampourakis, 2014, pp. 127–129
- ↑ a b Doolittle, W. Ford (February 2000). «Uprooting the Tree of Life» (PDF). Scientific American 282 (2): 90-95. Bibcode:2000SciAm.282b..90D. ISSN 0036-8733. PMID 10710791. doi:10.1038/scientificamerican0200-90. Archivado desde el original el 7 de septiembre de 2006. Consultado el 5 de abril de 2015.
- ↑ Glansdorff, Nicolas; Ying Xu; Labedan, Bernard (July 9, 2008). «The Last Universal Common Ancestor: emergence, constitution and genetic legacy of an elusive forerunner». Biology Direct 3: 29. ISSN 1745-6150. PMC 2478661. PMID 18613974. doi:10.1186/1745-6150-3-29.
- ↑ a b Schopf, J. William; Kudryavtsev, Anatoliy B.; Czaja, Andrew D.; Tripathi, Abhishek B. (October 5, 2007). «Evidence of Archean life: Stromatolites and microfossils». Precambrian Research 158 (3–4): 141-155. Bibcode:2007PreR..158..141S. ISSN 0301-9268. doi:10.1016/j.precamres.2007.04.009.
- ↑ a b Ohtomo, Yoko; Kakegawa, Takeshi; Ishida, Akizumi et al. (January 2014). «Evidence for biogenic graphite in early Archaean Isua metasedimentary rocks». Nature Geoscience 7 (1): 25-28. Bibcode:2014NatGe...7...25O. ISSN 1752-0894. doi:10.1038/ngeo2025.
- ↑ a b Borenstein, Seth (November 13, 2013). «Oldest fossil found: Meet your microbial mom». Excite (Yonkers, New York: Mindspark Interactive Network). Associated Press. Archivado desde el original el June 29, 2015. Consultado el 31 de mayo de 2015.
- ↑ a b Pearlman, Jonathan (November 13, 2013). «'Oldest signs of life on Earth found'». The Daily Telegraph (London: Telegraph Media Group). Archivado desde el original el 16 de diciembre de 2014. Consultado el 15 de diciembre de 2014.
- ↑ a b Noffke, Nora; Christian, Daniel; Wacey, David; Hazen, Robert M. (November 16, 2013). «Microbially Induced Sedimentary Structures Recording an Ancient Ecosystem in the ca. 3.48 Billion-Year-Old Dresser Formation, Pilbara, Western Australia». Astrobiology 13 (12): 1103-1124. Bibcode:2013AsBio..13.1103N. ISSN 1531-1074. PMC 3870916. PMID 24205812. doi:10.1089/ast.2013.1030.
- ↑ Futuyma, 2004, p. 33
- ↑ Panno, 2005, pp. xv-16
- ↑ NAS 2008, p. 17 (enlace roto disponible en este archivo).
- ↑ Futuyma, Douglas J., ed. (1999). «Evolution, Science, and Society: Evolutionary Biology and the National Research Agenda» (PDF) (Executive summary). New Brunswick, New Jersey: Office of University Publications, Rutgers, The State University of New Jersey. OCLC 43422991. Archivado desde el original el 31 de enero de 2012. Consultado el 24 de noviembre de 2014.
- ↑ Darwin, 1909, p. 53
- ↑ Kirk, Raven y Schofield, 1983, pp. 100–142, 280–321
- ↑ Lucretius,
- ↑ Sedley, David (2003). «Lucretius and the New Empedocles» (PDF). Leeds International Classical Studies 2 (4). ISSN 1477-3643. Archivado desde el original el 23 de agosto de 2014. Consultado el 25 de noviembre de 2014.
- ↑ Torrey, Harry Beal; Felin, Frances (March 1937). «Was Aristotle an Evolutionist?». The Quarterly Review of Biology 12 (1): 1-18. ISSN 0033-5770. JSTOR 2808399. doi:10.1086/394520.
- ↑ Hull, David L. (December 1967). «The Metaphysics of Evolution». The British Journal for the History of Science 3 (4): 309-337. JSTOR 4024958. doi:10.1017/S0007087400002892.
- ↑ Mason, 1962, pp. 43–44
- ↑ Mayr, 1982, pp. 256–257
- ↑ Waggoner, Ben (July 7, 2000). «Carl Linnaeus (1707-1778)». Evolution (Online exhibit). Berkeley, California: University of California Museum of Paleontology. Archivado desde el original el 30 de abril de 2011. Consultado el 11 de febrero de 2012.
- ↑ Bowler, 2003, pp. 73–75
- ↑ «Erasmus Darwin (1731-1802)». Evolution (Online exhibit). Berkeley, California: University of California Museum of Paleontology. October 4, 1995. Archivado desde el original el 19 de enero de 2012. Consultado el 11 de febrero de 2012.
- ↑ Lamarck, 1809
- ↑ a b Nardon y Grenier, 1991, p. 162
- ↑ a b c Gould, 2002[página requerida]
- ↑ Ghiselin, Michael T. (September–October 1994). «The Imaginary Lamarck: A Look at Bogus 'History' in Schoolbooks». The Textbook Letter. OCLC 23228649. Archivado desde el original el 12 de febrero de 2008. Consultado el 23 de enero de 2008.
- ↑ Magner, 2002[página requerida]
- ↑ Jablonka, Eva; Lamb, Marion J. (August 2007). «Précis of Evolution in Four Dimensions». Behavioral and Brain Sciences 30 (4): 353-365. ISSN 0140-525X. PMID 18081952. doi:10.1017/S0140525X07002221.
- ↑ Burkhardt y Smith, 1991
- «Darwin, C. R. to Lubbock, John». Darwin Correspondence Project. Cambridge, UK: University of Cambridge. Archivado desde el original el 15 de diciembre de 2014. Consultado el 1 de diciembre de 2014. Letter 2532, November 22, 1859.
- ↑ Sulloway, Frank J. (June 2009). «Why Darwin rejected intelligent design». Journal of Biosciences 34 (2): 173-183. ISSN 0250-5991. PMID 19550032. doi:10.1007/s12038-009-0020-8.
- ↑ Dawkins, 1990[página requerida]
- ↑ Sober, Elliott (June 16, 2009). «Did Darwin write the Origin backwards?». Proc. Natl. Acad. Sci. U.S.A. 106 (Suppl. 1): 10048-10055. Bibcode:2009PNAS..10610048S. ISSN 0027-8424. PMC 2702806. PMID 19528655. doi:10.1073/pnas.0901109106.
- ↑ Mayr, 2002, p. 165
- ↑ Bowler, 2003, pp. 145–146
- ↑ Sokal, Robert R.; Crovello, Theodore J. (March–April 1970). «The Biological Species Concept: A Critical Evaluation». The American Naturalist 104 (936): 127-153. ISSN 0003-0147. JSTOR 2459191. doi:10.1086/282646.
- ↑ Darwin, Charles; Wallace, Alfred (August 20, 1858). «On the Tendency of Species to form Varieties; and on the Perpetuation of Varieties and Species by Natural Means of Selection». Journal of the Proceedings of the Linnean Society of London. Zoology 3 (9): 45-62. ISSN 1096-3642. doi:10.1111/j.1096-3642.1858.tb02500.x. Archivado desde el original el 14 de julio de 2007. Consultado el 13 de mayo de 2007.
- ↑ Desmond, Adrian J. (July 17, 2014). «Thomas Henry Huxley». Encyclopædia Britannica Online. Chicago, Illinois: Encyclopædia Britannica, Inc. Archivado desde el original el January 19, 2015. Consultado el 2 de diciembre de 2014.
- ↑ Liu, Y. S.; Zhou, X. M.; Zhi, M. X.; Li, X. J.; Wang, Q. L. (September 2009). «Darwin's contributions to genetics». Journal of Applied Genetics 50 (3): 177-184. ISSN 1234-1983. PMID 19638672. doi:10.1007/BF03195671.
- ↑ Weiling, Franz (July 1991). «Historical study: Johann Gregor Mendel 1822–1884». American Journal of Medical Genetics 40 (1): 1-25; discussion 26. PMID 1887835. doi:10.1002/ajmg.1320400103.
- ↑ Wright, 1984, p. 480
- ↑ Provine, 1971
- ↑ Stamhuis, Ida H.; Meijer, Onno G.; Zevenhuizen, Erik J. A. (June 1999). «Hugo de Vries on Heredity, 1889-1903: Statistics, Mendelian Laws, Pangenes, Mutations». Isis 90 (2): 238-267. JSTOR 237050. PMID 10439561. doi:10.1086/384323.
- ↑ Quammen, 2006[página requerida]
- ↑ Bowler, 1989[página requerida]
- ↑ Watson, J. D.; Crick, F. H. C. (April 25, 1953). «Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid» (PDF). Nature 171 (4356): 737-738. Bibcode:1953Natur.171..737W. ISSN 0028-0836. PMID 13054692. doi:10.1038/171737a0. Archivado desde el original el 23 de agosto de 2014. Consultado el 4 de diciembre de 2014. «It has not escaped our notice that the specific pairing we have postulated immediately suggests a possible copying mechanism for the genetic material.»
- ↑ Hennig, 1999, p. 280
- ↑ Wiley y Lieberman, 2011[página requerida]
- ↑ Dobzhansky, Theodosius (March 1973). «Nothing in Biology Makes Sense Except in the Light of Evolution». The American Biology Teacher 35 (3): 125-129. JSTOR 4444260. doi:10.2307/4444260. Archivado desde el original el 23 de octubre de 2015. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ a b c Kutschera, Ulrich; Niklas, Karl J. (June 2004). «The modern theory of biological evolution: an expanded synthesis». Naturwissenschaften 91 (6): 255-276. Bibcode:2004NW.....91..255K. ISSN 1432-1904. PMID 15241603. doi:10.1007/s00114-004-0515-y.
- ↑ Cracraft y Bybee, 2005[página requerida]
- ↑ Avise, John C.; Ayala, Francisco J. (11 de mayo de 2010). «In the light of evolution IV: The human condition» (PDF). Proc. Natl. Acad. Sci. U.S.A. 107 (Suppl. 2): 8897-8901. ISSN 0027-8424. PMC 3024015. PMID 20460311. doi:10.1073/pnas.1003214107. Archivado desde el original el 23 de agosto de 2014. Consultado el 29 de diciembre de 2014.
- ↑ Danchin, Étienne; Charmantier, Anne; Champagne, Frances A.; Mesoudi, Alex; Pujol, Benoit; Blanchet, Simon (June 2011). «Beyond DNA: integrating inclusive inheritance into an extended theory of evolution». Nature Reviews Genetics 12 (7): 475-486. ISSN 1471-0056. PMID 21681209. doi:10.1038/nrg3028.
- ↑ Pigliucci y Müller, 2010
- ↑ Sturm, Richard A.; Frudakis, Tony N. (August 2004). «Eye colour: portals into pigmentation genes and ancestry». Trends in Genetics 20 (8): 327-332. ISSN 0168-9525. PMID 15262401. doi:10.1016/j.tig.2004.06.010.
- ↑ a b Pearson, Helen (25 de mayo de 2006). «Genetics: What is a gene?». Nature 441 (7092): 398-401. Bibcode:2006Natur.441..398P. ISSN 0028-0836. PMID 16724031. doi:10.1038/441398a.
- ↑ Visscher, Peter M.; Hill, William G.; Wray, Naomi R. (April 2008). «Heritability in the genomics era — concepts and misconceptions». Nature Reviews Genetics 9 (4): 255-266. ISSN 1471-0056. PMID 18319743. doi:10.1038/nrg2322.
- ↑ Oetting, William S.; Brilliant, Murray H.; King, Richard A. (August 1996). «The clinical spectrum of albinism in humans». Molecular Medicine Today 2 (8): 330-335. ISSN 1357-4310. PMID 8796918. doi:10.1016/1357-4310(96)81798-9.
- ↑ a b Futuyma, 2005[página requerida]
- ↑ Phillips, Patrick C. (November 2008). «Epistasis—the essential role of gene interactions in the structure and evolution of genetic systems». Nature Reviews Genetics 9 (11): 855-867. ISSN 1471-0056. PMC 2689140. PMID 18852697. doi:10.1038/nrg2452.
- ↑ a b Rongling Wu; Min Lin (March 2006). «Functional mapping — how to map and study the genetic architecture of dynamic complex traits». Nature Reviews Genetics 7 (3): 229-237. ISSN 1471-0056. PMID 16485021. doi:10.1038/nrg1804.
- ↑ Jablonka, Eva; Raz, Gal (June 2009). «Transgenerational Epigenetic Inheritance: Prevalence, Mechanisms, and Implications for the Study of Heredity and Evolution». The Quarterly Review of Biology 84 (2): 131-176. ISSN 0033-5770. PMID 19606595. doi:10.1086/598822. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Bossdorf, Oliver; Arcuri, Davide; Richards, Christina L.; Pigliucci, Massimo (May 2010). «Experimental alteration of DNA methylation affects the phenotypic plasticity of ecologically relevant traits in Arabidopsis thaliana». Evolutionary Ecology (Submitted manuscript) 24 (3): 541-553. ISSN 0269-7653. doi:10.1007/s10682-010-9372-7.
- ↑ Jablonka y Lamb, 2005[página requerida]
- ↑ Jablonka, Eva; Lamb, Marion J. (December 2002). «The Changing Concept of Epigenetics». Annals of the New York Academy of Sciences 981 (1): 82-96. Bibcode:2002NYASA.981...82J. ISSN 0077-8923. PMID 12547675. doi:10.1111/j.1749-6632.2002.tb04913.x.
- ↑ Laland, Kevin N.; Sterelny, Kim (September 2006). «Perspective: Seven Reasons (Not) to Neglect Niche Construction». Evolution 60 (9): 1751-1762. ISSN 0014-3820. doi:10.1111/j.0014-3820.2006.tb00520.x.
- ↑ Chapman, Michael J.; Margulis, Lynn (December 1998). «Morphogenesis by symbiogenesis» (PDF). International Microbiology 1 (4): 319-326. ISSN 1139-6709. PMID 10943381. Archivado desde el original el 23 de agosto de 2014. Consultado el 9 de diciembre de 2014.
- ↑ Wilson, David Sloan; Wilson, Edward O. (December 2007). «Rethinking the Theoretical Foundation of Sociobiology» (PDF). The Quarterly Review of Biology 82 (4): 327-348. ISSN 0033-5770. PMID 18217526. doi:10.1086/522809. Archivado desde el original el 11 de mayo de 2011.
- ↑ Amos, William; Harwood, John (February 28, 1998). «Factors affecting levels of genetic diversity in natural populations». Philosophical Transactions of the Royal Society B: Biological Sciences 353 (1366): 177-186. ISSN 0962-8436. PMC 1692205. PMID 9533122. doi:10.1098/rstb.1998.0200.
- ↑ a b Ewens, 2004[página requerida]
- ↑ Butlin, Roger K.; Tregenza, Tom (February 28, 1998). «Levels of genetic polymorphism: marker loci versus quantitative traits». Philosophical Transactions of the Royal Society B: Biological Sciences 353 (1366): 187-198. ISSN 0962-8436. PMC 1692210. PMID 9533123. doi:10.1098/rstb.1998.0201.
- Butlin, Roger K.; Tregenza, Tom (December 29, 2000). «Correction for Butlin and Tregenza, Levels of genetic polymorphism: marker loci versus quantitative traits». Philosophical Transactions of the Royal Society B: Biological Sciences 355 (1404): 1865. ISSN 0962-8436. doi:10.1098/rstb.2000.2000. «Some of the values in table 1 on p. 193 were given incorrectly. The errors do not affect the conclusions drawn in the paper. The corrected table is reproduced below.»
- ↑ Wetterbom, Anna; Sevov, Marie; Cavelier, Lucia; Bergström, Tomas F. (November 2006). «Comparative Genomic Analysis of Human and Chimpanzee Indicates a Key Role for Indels in Primate Evolution». Journal of Molecular Evolution 63 (5): 682-690. Bibcode:2006JMolE..63..682W. ISSN 0022-2844. PMID 17075697. doi:10.1007/s00239-006-0045-7.
- ↑ Sawyer, Stanley A.; Parsch, John; Zhang Zhi; Hartl, Daniel L. (April 17, 2007). «Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila». Proc. Natl. Acad. Sci. U.S.A. 104 (16): 6504-6510. Bibcode:2007PNAS..104.6504S. ISSN 0027-8424. PMC 1871816. PMID 17409186. doi:10.1073/pnas.0701572104.
- ↑ Hastings, P. J.; Lupski, James R.; Rosenberg, Susan M.; Ira, Grzegorz (August 2009). «Mechanisms of change in gene copy number». Nature Reviews Genetics 10 (8): 551-564. ISSN 1471-0056. PMC 2864001. PMID 19597530. doi:10.1038/nrg2593.
- ↑ Carroll, Grenier y Weatherbee, 2005[página requerida]
- ↑ Harrison, Paul M.; Gerstein, Mark (17 de mayo de 2002). «Studying Genomes Through the Aeons: Protein Families, Pseudogenes and Proteome Evolution». Journal of Molecular Biology 318 (5): 1155-1174. ISSN 0022-2836. PMID 12083509. doi:10.1016/S0022-2836(02)00109-2.
- ↑ Bowmaker, James K. (May 1998). «Evolution of colour vision in vertebrates». Eye 12 (3b): 541-547. ISSN 0950-222X. PMID 9775215. doi:10.1038/eye.1998.143.
- ↑ Gregory, T. Ryan; Hebert, Paul D. N. (April 1999). «The Modulation of DNA Content: Proximate Causes and Ultimate Consequences». Genome Research 9 (4): 317-324. ISSN 1088-9051. PMID 10207154. doi:10.1101/gr.9.4.317 (inactivo 2018-11-14). Archivado desde el original el 23 de agosto de 2014. Consultado el 11 de diciembre de 2014. Parámetro desconocido
|doi-broken-date=ignorado (ayuda) - ↑ Hurles, Matthew (July 13, 2004). «Gene Duplication: The Genomic Trade in Spare Parts». PLOS Biology 2 (7): e206. ISSN 1545-7885. PMC 449868. PMID 15252449. doi:10.1371/journal.pbio.0020206.
- ↑ Liu, Na; Okamura, Katsutomo; Tyler, David M. et al. (October 2008). «The evolution and functional diversification of animal microRNA genes». Cell Research 18 (10): 985-996. ISSN 1001-0602. PMC 2712117. PMID 18711447. doi:10.1038/cr.2008.278. Archivado desde el original el 2 de febrero de 2015. Consultado el 11 de diciembre de 2014.
- ↑ Siepel, Adam (October 2009). «Darwinian alchemy: Human genes from noncoding DNA». Genome Research 19 (10): 1693-1695. ISSN 1088-9051. PMC 2765273. PMID 19797681. doi:10.1101/gr.098376.109. Archivado desde el original el 23 de agosto de 2014. Consultado el 11 de diciembre de 2014.
- ↑ Orengo, Christine A.; Thornton, Janet M. (July 2005). «Protein families and their evolution—a structural perspective». Annual Review of Biochemistry 74: 867-900. ISSN 0066-4154. PMID 15954844. doi:10.1146/annurev.biochem.74.082803.133029.
- ↑ Long, Manyuan; Betrán, Esther; Thornton, Kevin; Wang, Wen (November 2003). «The origin of new genes: glimpses from the young and old». Nature Reviews Genetics 4 (11): 865-875. ISSN 1471-0056. PMID 14634634. doi:10.1038/nrg1204.
- ↑ Wang, Minglei; Caetano-Anollés, Gustavo (January 14, 2009). «The Evolutionary Mechanics of Domain Organization in Proteomes and the Rise of Modularity in the Protein World». Structure 17 (1): 66-78. ISSN 1357-4310. PMID 19141283. doi:10.1016/j.str.2008.11.008.
- ↑ Weissman, Kira J.; Müller, Rolf (April 14, 2008). «Protein–Protein Interactions in Multienzyme Megasynthetases». ChemBioChem 9 (6): 826-848. ISSN 1439-4227. PMID 18357594. doi:10.1002/cbic.200700751.
- ↑ Radding, Charles M. (December 1982). «Homologous Pairing and Strand Exchange in Genetic Recombination». Annual Review of Genetics 16: 405-437. ISSN 0066-4197. PMID 6297377. doi:10.1146/annurev.ge.16.120182.002201.
- ↑ Agrawal, Aneil F. (September 5, 2006). «Evolution of Sex: Why Do Organisms Shuffle Their Genotypes?». Current Biology 16 (17): R696-R704. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMID 16950096. doi:10.1016/j.cub.2006.07.063.
- ↑ Peters, Andrew D.; Otto, Sarah P. (June 2003). «Liberating genetic variance through sex». BioEssays 25 (6): 533-537. ISSN 0265-9247. PMID 12766942. doi:10.1002/bies.10291.
- ↑ Goddard, Matthew R.; Godfray, H. Charles J.; Burt, Austin (March 31, 2005). «Sex increases the efficacy of natural selection in experimental yeast populations». Nature 434 (7033): 636-640. Bibcode:2005Natur.434..636G. ISSN 0028-0836. PMID 15800622. doi:10.1038/nature03405.
- ↑ Maynard Smith, 1978[página requerida]
- ↑ a b Ridley, 1993[página requerida]
- ↑ Van Valen, Leigh (1973). «A New Evolutionary Law» (PDF). Evolutionary Theory 1: 1-30. ISSN 0093-4755. Archivado desde el original el 22 de diciembre de 2014. Consultado el 24 de diciembre de 2014.
- ↑ Hamilton, W. D.; Axelrod, Robert; Tanese, Reiko (1 de mayo de 1990). «Sexual reproduction as an adaptation to resist parasites (a review)». Proc. Natl. Acad. Sci. U.S.A. 87 (9): 3566-3573. Bibcode:1990PNAS...87.3566H. ISSN 0027-8424. PMC 53943. PMID 2185476. doi:10.1073/pnas.87.9.3566.
- ↑ Birdsell y Wills, 2003, pp. 113–117
- ↑ a b Morjan, Carrie L.; Rieseberg, Loren H. (June 2004). «How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles». Molecular Ecology 13 (6): 1341-1356. ISSN 0962-1083. PMC 2600545. PMID 15140081. doi:10.1111/j.1365-294X.2004.02164.x.
- ↑ Boucher, Yan; Douady, Christophe J.; Papke, R. Thane et al. (December 2003). «Lateral gene transfer and the origins of prokaryotic groups». Annual Review of Genetics 37: 283-328. ISSN 0066-4197. PMID 14616063. doi:10.1146/annurev.genet.37.050503.084247.
- ↑ Walsh, Timothy R. (October 2006). «Combinatorial genetic evolution of multiresistance». Current Opinion in Microbiology 9 (5): 476-482. ISSN 1369-5274. PMID 16942901. doi:10.1016/j.mib.2006.08.009.
- ↑ Kondo, Natsuko; Nikoh, Naruo; Ijichi, Nobuyuki et al. (October 29, 2002). «Genome fragment of Wolbachia endosymbiont transferred to X chromosome of host insect». Proc. Natl. Acad. Sci. U.S.A. 99 (22): 14280-14285. Bibcode:2002PNAS...9914280K. ISSN 0027-8424. PMC 137875. PMID 12386340. doi:10.1073/pnas.222228199.
- ↑ Sprague, George F., Jr. (December 1991). «Genetic exchange between kingdoms». Current Opinion in Genetics & Development 1 (4): 530-533. ISSN 0959-437X. PMID 1822285. doi:10.1016/S0959-437X(05)80203-5.
- ↑ Gladyshev, Eugene A.; Meselson, Matthew; Arkhipova, Irina R. (30 de mayo de 2008). «Massive Horizontal Gene Transfer in Bdelloid Rotifers». Science (Submitted manuscript) 320 (5880): 1210-1213. Bibcode:2008Sci...320.1210G. ISSN 0036-8075. PMID 18511688. doi:10.1126/science.1156407.
- ↑ Baldo, Angela M.; McClure, Marcella A. (September 1999). «Evolution and Horizontal Transfer of dUTPase-Encoding Genes in Viruses and Their Hosts». Journal of Virology 73 (9): 7710-7721. ISSN 0022-538X. PMC 104298. PMID 10438861.
- ↑ Rivera, Maria C.; Lake, James A. (September 9, 2004). «The ring of life provides evidence for a genome fusion origin of eukaryotes». Nature 431 (7005): 152-155. Bibcode:2004Natur.431..152R. ISSN 0028-0836. PMID 15356622. doi:10.1038/nature02848.
- ↑ a b c Hurst, Laurence D. (February 2009). «Fundamental concepts in genetics: genetics and the understanding of selection». Nature Reviews Genetics 10 (2): 83-93. ISSN 1471-0056. PMID 19119264. doi:10.1038/nrg2506.
- ↑ Darwin, 1859, Chapter XIV
- ↑ Otto, Sarah P.; Servedio, Maria R.; Nuismer, Scott L. (August 2008). «Frequency-Dependent Selection and the Evolution of Assortative Mating». Genetics 179 (4). pp. 2091-2112. ISSN 0016-6731. PMC 2516082. PMID 18660541. doi:10.1534/genetics.107.084418.
- ↑ a b c Orr, H. Allen (August 2009). «Fitness and its role in evolutionary genetics». Nature Reviews Genetics 10 (8): 531-539. ISSN 1471-0056. PMC 2753274. PMID 19546856. doi:10.1038/nrg2603.
- ↑ Haldane, J.B.S. (March 14, 1959). «The Theory of Natural Selection To-Day». Nature 183 (4663): 710-713. Bibcode:1959Natur.183..710H. ISSN 0028-0836. PMID 13644170. doi:10.1038/183710a0.
- ↑ Lande, Russell; Arnold, Stevan J. (November 1983). «The Measurement of Selection on Correlated Characters». Evolution 37 (6): 1210-1226. ISSN 0014-3820. JSTOR 2408842. PMID 28556011. doi:10.2307/2408842.
- ↑ Goldberg, Emma E.; Igić, Boris (November 2008). «On phylogenetic tests of irreversible evolution». Evolution 62 (11): 2727-2741. ISSN 0014-3820. PMID 18764918. doi:10.1111/j.1558-5646.2008.00505.x.
- ↑ Collin, Rachel; Miglietta, Maria Pia (November 2008). «Reversing opinions on Dollo's Law». Trends in Ecology & Evolution 23 (11): 602-609. ISSN 0169-5347. PMID 18814933. doi:10.1016/j.tree.2008.06.013.
- ↑ Tomić, Nenad; Meyer-Rochow, Victor Benno (2011). «Atavisms: Medical, Genetic, and Evolutionary Implications». Perspectives in Biology and Medicine 54 (3): 332-353. ISSN 0031-5982. PMID 21857125. doi:10.1353/pbm.2011.0034.
- ↑ Hoekstra, Hopi E.; Hoekstra, Jonathan M.; Berrigan, David et al. (July 31, 2001). «Strength and tempo of directional selection in the wild». Proc. Natl. Acad. Sci. U.S.A. 98 (16): 9157-9160. Bibcode:2001PNAS...98.9157H. ISSN 0027-8424. PMC 55389. PMID 11470913. doi:10.1073/pnas.161281098.
- ↑ Felsenstein, Joseph (November 1979). «Excursions along the Interface between Disruptive and Stabilizing Selection». Genetics 93 (3): 773-795. ISSN 0016-6731. PMC 1214112. PMID 17248980.
- ↑ Andersson, Malte; Simmons, Leigh W. (June 2006). «Sexual selection and mate choice». Trends in Ecology & Evolution 21 (6): 296-302. ISSN 0169-5347. PMID 16769428. doi:10.1016/j.tree.2006.03.015. Archivado desde el original el 9 de marzo de 2013. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Kokko, Hanna; Brooks, Robert; McNamara, John M.; Houston, Alasdair I. (July 7, 2002). «The sexual selection continuum». Proceedings of the Royal Society B 269 (1498): 1331-1340. ISSN 0962-8452. PMC 1691039. PMID 12079655. doi:10.1098/rspb.2002.2020.
- ↑ Quinn, Thomas P.; Hendry, Andrew P.; Buck, Gregory B. (2001). «Balancing natural and sexual selection in sockeye salmon: interactions between body size, reproductive opportunity and vulnerability to predation by bears» (PDF). Evolutionary Ecology Research 3: 917-937. ISSN 1522-0613. Archivado desde el original el 5 de marzo de 2016. Consultado el 15 de diciembre de 2014.
- ↑ Hunt, John; Brooks, Robert; Jennions, Michael D. et al. (December 23, 2004). «High-quality male field crickets invest heavily in sexual display but die young». Nature 432 (7020): 1024-1027. Bibcode:2004Natur.432.1024H. ISSN 0028-0836. PMID 15616562. doi:10.1038/nature03084.
- ↑ Odum, 1971, p. 8
- ↑ Okasha, 2006
- ↑ a b Gould, Stephen Jay (February 28, 1998). «Gulliver's further travels: the necessity and difficulty of a hierarchical theory of selection». Philosophical Transactions of the Royal Society B: Biological Sciences 353 (1366): 307-314. ISSN 0962-8436. PMC 1692213. PMID 9533127. doi:10.1098/rstb.1998.0211.
- ↑ Mayr, Ernst (March 18, 1997). «The objects of selection». Proc. Natl. Acad. Sci. U.S.A. 94 (6): 2091-2094. Bibcode:1997PNAS...94.2091M. ISSN 0027-8424. PMC 33654. PMID 9122151. doi:10.1073/pnas.94.6.2091.
- ↑ Maynard Smith, 1998, pp. 203–211; discussion 211–217
- ↑ Hickey, Donal A. (1992). «Evolutionary dynamics of transposable elements in prokaryotes and eukaryotes». Genetica 86 (1–3): 269-274. ISSN 0016-6707. PMID 1334911. doi:10.1007/BF00133725.
- ↑ Gould, Stephen Jay; Lloyd, Elisabeth A. (October 12, 1999). «Individuality and adaptation across levels of selection: how shall we name and generalise the unit of Darwinism?». Proc. Natl. Acad. Sci. U.S.A. 96 (21): 11904-11909. Bibcode:1999PNAS...9611904G. ISSN 0027-8424. PMC 18385. PMID 10518549. doi:10.1073/pnas.96.21.11904.
- ↑ Lynch, Michael (15 de mayo de 2007). «The frailty of adaptive hypotheses for the origins of organismal complexity». Proc. Natl. Acad. Sci. U.S.A. 104 (Suppl. 1): 8597-8604. Bibcode:2007PNAS..104.8597L. ISSN 0027-8424. PMC 1876435. PMID 17494740. doi:10.1073/pnas.0702207104.
- ↑ Smith, Nick G.C.; Webster, Matthew T.; Ellegren, Hans (September 2002). «Deterministic Mutation Rate Variation in the Human Genome». Genome Research 12 (9): 1350-1356. ISSN 1088-9051. PMC 186654. PMID 12213772. doi:10.1101/gr.220502. Archivado desde el original el 23 de agosto de 2014.
- ↑ Petrov, Dmitri A.; Sangster, Todd A.; Johnston, J. Spencer et al. (February 11, 2000). «Evidence for DNA Loss as a Determinant of Genome Size». Science 287 (5455): 1060-1062. Bibcode:2000Sci...287.1060P. ISSN 0036-8075. PMID 10669421. doi:10.1126/science.287.5455.1060.
- ↑ Petrov, Dmitri A. (May 2002). «DNA loss and evolution of genome size in Drosophila». Genetica 115 (1): 81-91. ISSN 0016-6707. PMID 12188050. doi:10.1023/A:1016076215168.
- ↑ Kiontke, Karin; Barrière, Antoine; Kolotuev, Irina et al. (November 2007). «Trends, Stasis, and Drift in the Evolution of Nematode Vulva Development». Current Biology 17 (22): 1925-1937. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMID 18024125. doi:10.1016/j.cub.2007.10.061.
- ↑ Braendle, Christian; Baer, Charles F.; Félix, Marie-Anne (March 12, 2010). «Bias and Evolution of the Mutationally Accessible Phenotypic Space in a Developmental System». En Barsh, Gregory S., ed. PLOS Genetics 6 (3): e1000877. ISSN 1553-7390. PMC 2837400. PMID 20300655. doi:10.1371/journal.pgen.1000877.
- ↑ Palmer, A. Richard (October 29, 2004). «Symmetry breaking and the evolution of development». Science 306 (5697): 828-833. Bibcode:2004Sci...306..828P. ISSN 0036-8075. PMID 15514148. doi:10.1126/science.1103707. Archivado desde el original el 12 de junio de 2007. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ West-Eberhard, 2003
- ↑ Pocheville y Danchin, 2017
- ↑ Stoltzfus, Arlin; Yampolsky, Lev Y. (September–October 2009). «Climbing Mount Probable: Mutation as a Cause of Nonrandomness in Evolution». Journal of Heredity 100 (5): 637-647. ISSN 0022-1503. PMID 19625453. doi:10.1093/jhered/esp048.
- ↑ Yampolsky, Lev Y.; Stoltzfus, Arlin (March 2001). «Bias in the introduction of variation as an orienting factor in evolution». Evolution & Development 3 (2): 73-83. ISSN 1520-541X. PMID 11341676. doi:10.1046/j.1525-142x.2001.003002073.x.
- ↑ Haldane, J.B.S. (January–February 1933). «The Part Played by Recurrent Mutation in Evolution». The American Naturalist 67 (708): 5-19. ISSN 0003-0147. JSTOR 2457127. doi:10.1086/280465.
- ↑ Protas, Meredith; Conrad, Melissa; Gross, Joshua B. et al. (March 6, 2007). «Regressive Evolution in the Mexican Cave Tetra, Astyanax mexicanus». Current Biology 17 (5): 452-454. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMC 2570642. PMID 17306543. doi:10.1016/j.cub.2007.01.051.
- ↑ Maughan, Heather; Masel, Joanna; Birky, C. William, Jr.; Nicholson, Wayne L. (October 2007). «The Roles of Mutation Accumulation and Selection in Loss of Sporulation in Experimental Populations of Bacillus subtilis». Genetics 177 (2). pp. 937-948. ISSN 0016-6731. PMC 2034656. PMID 17720926. doi:10.1534/genetics.107.075663.
- ↑ Masel, Joanna; King, Oliver D.; Maughan, Heather (January 2007). «The Loss of Adaptive Plasticity during Long Periods of Environmental Stasis». The American Naturalist 169 (1): 38-46. ISSN 0003-0147. PMC 1766558. PMID 17206583. doi:10.1086/510212.
- ↑ Futuyma, Douglas J.; Kirkpatrick, Mark (2017). «Natural selection and adaptation». Evolution (Fourth edición). Sunderland, Massachusetts: Sinauer Associates, Inc. pp. 55-66. ISBN 978-1-60535-605-1.
- ↑ a b Masel, Joanna (October 25, 2011). «Genetic drift». Current Biology 21 (20): R837-R838. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMID 22032182. doi:10.1016/j.cub.2011.08.007.
- ↑ Lande, Russell (1989). «Fisherian and Wrightian theories of speciation». Genome 31 (1): 221-227. ISSN 0831-2796. PMID 2687093. doi:10.1139/g89-037.
- ↑ Kimura, Motoo (1991). «The neutral theory of molecular evolution: a review of recent evidence». The Japanese Journal of Human Genetics 66 (4): 367-386. ISSN 0021-504X. PMID 1954033. doi:10.1266/jjg.66.367. Archivado desde el original el 16 de diciembre de 2014.
- ↑ Kimura, Motoo (1989). «The neutral theory of molecular evolution and the world view of the neutralists». Genome 31 (1): 24-31. ISSN 0831-2796. PMID 2687096. doi:10.1139/g89-009.
- ↑ Kreitman, Martin (August 1996). «The neutral theory is dead. Long live the neutral theory». BioEssays 18 (8): 678-683; discussion 683. ISSN 0265-9247. PMID 8760341. doi:10.1002/bies.950180812.
- ↑ Leigh, E.G., Jr. (November 2007). «Neutral theory: a historical perspective». Journal of Evolutionary Biology 20 (6): 2075-2091. ISSN 1010-061X. PMID 17956380. doi:10.1111/j.1420-9101.2007.01410.x.
- ↑ a b Gillespie, John H. (November 2001). «Is the population size of a species relevant to its evolution?». Evolution 55 (11): 2161-2169. ISSN 0014-3820. PMID 11794777. doi:10.1111/j.0014-3820.2001.tb00732.x.
- ↑ Neher, Richard A.; Shraiman, Boris I. (August 2011). «Genetic Draft and Quasi-Neutrality in Large Facultatively Sexual Populations». Genetics 188 (4). pp. 975-996. ISSN 0016-6731. PMC 3176096. PMID 21625002. arXiv:1108.1635. doi:10.1534/genetics.111.128876.
- ↑ Otto, Sarah P.; Whitlock, Michael C. (June 1997). «The Probability of Fixation in Populations of Changing Size» (PDF). Genetics 146 (2): 723-733. ISSN 0016-6731. PMC 1208011. PMID 9178020. Archivado desde el original el 19 de marzo de 2015. Consultado el 18 de diciembre de 2014.
- ↑ a b Charlesworth, Brian (March 2009). «Fundamental concepts in genetics: effective population size and patterns of molecular evolution and variation». Nature Reviews Genetics 10 (3): 195-205. ISSN 1471-0056. PMID 19204717. doi:10.1038/nrg2526.
- ↑ Cutter, Asher D.; Choi, Jae Young (August 2010). «Natural selection shapes nucleotide polymorphism across the genome of the nematode Caenorhabditis briggsae». Genome Research 20 (8): 1103-1111. ISSN 1088-9051. PMC 2909573. PMID 20508143. doi:10.1101/gr.104331.109.
- ↑ Mitchell-Olds, Thomas; Willis, John H.; Goldstein, David B. (November 2007). «Which evolutionary processes influence natural genetic variation for phenotypic traits?». Nature Reviews Genetics 8 (11): 845-856. ISSN 1471-0056. PMID 17943192. doi:10.1038/nrg2207.
- ↑ Nei, Masatoshi (December 2005). «Selectionism and Neutralism in Molecular Evolution». Molecular Biology and Evolution 22 (12): 2318-2342. ISSN 0737-4038. PMC 1513187. PMID 16120807. doi:10.1093/molbev/msi242.
- Nei, Masatoshi (May 2006). «Selectionism and Neutralism in Molecular Evolution». Molecular Biology and Evolution (Erratum) 23 (5): 1095. ISSN 0737-4038. doi:10.1093/molbev/msk009.
- ↑ Lien, Sigbjørn; Szyda, Joanna; Schechinger, Birgit et al. (February 2000). «Evidence for Heterogeneity in Recombination in the Human Pseudoautosomal Region: High Resolution Analysis by Sperm Typing and Radiation-Hybrid Mapping». American Journal of Human Genetics 66 (2): 557-566. ISSN 0002-9297. PMC 1288109. PMID 10677316. doi:10.1086/302754.
- ↑ Barton, Nicholas H. (November 29, 2000). «Genetic hitchhiking». Philosophical Transactions of the Royal Society B: Biological Sciences 355 (1403): 1553-1562. ISSN 0962-8436. PMC 1692896. PMID 11127900. doi:10.1098/rstb.2000.0716.
- ↑ Wright, Sewall (1932). «The roles of mutation, inbreeding, crossbreeding and selection in evolution». Proceedings of the VI International Congress of Genetrics 1: 356-366. Archivado desde el original el 23 de agosto de 2014. Consultado el 18 de diciembre de 2014.
- ↑ Coyne, Jerry A.; Barton, Nicholas H.; Turelli, Michael (June 1997). «Perspective: A Critique of Sewall Wright's Shifting Balance Theory of Evolution». Evolution 51 (3): 643-671. ISSN 0014-3820. JSTOR 2411143. PMID 28568586. doi:10.2307/2411143.
- ↑ Baym, Michael; Lieberman, Tami D.; Kelsic, Eric D.; Chait, Remy; Gross, Rotem; Yelin, Idan; Kishony, Roy (9 de septiembre de 2016). «Spatiotemporal microbial evolution on antibiotic landscapes». Science (en inglés) 353 (6304): 1147-1151. Bibcode:2016Sci...353.1147B. ISSN 0036-8075. PMC 5534434. PMID 27609891. doi:10.1126/science.aag0822. Archivado desde el original el 23 de septiembre de 2016.
- ↑ a b c Scott, Eugenie C.; Matzke, Nicholas J. (15 de mayo de 2007). «Biological design in science classrooms». Proc. Natl. Acad. Sci. U.S.A. 104 (Suppl. 1): 8669-8676. Bibcode:2007PNAS..104.8669S. ISSN 0027-8424. PMC 1876445. PMID 17494747. doi:10.1073/pnas.0701505104.
- ↑ Hendry, Andrew Paul; Kinnison, Michael T. (November 2001). «An introduction to microevolution: rate, pattern, process». Genetica. 112–113 (1): 1-8. ISSN 0016-6707. PMID 11838760. doi:10.1023/A:1013368628607.
- ↑ Leroi, Armand M. (March–April 2000). «The scale independence of evolution». Evolution & Development 2 (2): 67-77. ISSN 1520-541X. PMID 11258392. doi:10.1046/j.1525-142x.2000.00044.x. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Gould, 2002, pp. 657–658.
- ↑ a b Gould, Stephen Jay (July 19, 1994). «Tempo and mode in the macroevolutionary reconstruction of Darwinism». Proc. Natl. Acad. Sci. U.S.A. 91 (15): 6764-6771. Bibcode:1994PNAS...91.6764G. ISSN 0027-8424. PMC 44281. PMID 8041695. doi:10.1073/pnas.91.15.6764.
- ↑ Jablonski, David (2000). «Micro- and macroevolution: scale and hierarchy in evolutionary biology and paleobiology». Paleobiology 26 (sp4): 15-52. ISSN 0094-8373. doi:10.1666/0094-8373(2000)26[15:MAMSAH]2.0.CO;2.
- ↑ Dougherty, Michael J. (July 20, 1998). «Is the human race evolving or devolving?». Scientific American. ISSN 0036-8733. Archivado desde el original el 6 de mayo de 2014. Consultado el 11 de septiembre de 2015.
- ↑ Isaak, Mark, ed. (July 22, 2003). «Claim CB932: Evolution of degenerate forms». TalkOrigins Archive. Houston, Texas: The TalkOrigins Foundation, Inc. Archivado desde el original el 23 de agosto de 2014. Consultado el 19 de diciembre de 2014.
- ↑ Lane, 1996, p. 61
- ↑ Carroll, Sean B. (February 22, 2001). «Chance and necessity: the evolution of morphological complexity and diversity». Nature 409 (6823): 1102-1109. Bibcode:2001Natur.409.1102C. ISSN 0028-0836. PMID 11234024. doi:10.1038/35059227.
- ↑ Whitman, William B.; Coleman, David C.; Wiebe, William J. (June 9, 1998). «Prokaryotes: The unseen majority». Proc. Natl. Acad. Sci. U.S.A. 95 (12): 6578-6583. Bibcode:1998PNAS...95.6578W. ISSN 0027-8424. PMC 33863. PMID 9618454. doi:10.1073/pnas.95.12.6578.
- ↑ a b Schloss, Patrick D.; Handelsman, Jo (December 2004). «Status of the Microbial Census». Microbiology and Molecular Biology Reviews 68 (4): 686-691. ISSN 1092-2172. PMC 539005. PMID 15590780. doi:10.1128/MMBR.68.4.686-691.2004.
- ↑ Nealson, Kenneth H. (January 1999). «Post-Viking microbiology: new approaches, new data, new insights». Origins of Life and Evolution of Biospheres 29 (1): 73-93. ISSN 0169-6149. PMID 11536899. doi:10.1023/A:1006515817767.
- ↑ Buckling, Angus; MacLean, R. Craig; Brockhurst, Michael A.; Colegrave, Nick (February 12, 2009). «The Beagle in a bottle». Nature 457 (7231): 824-829. Bibcode:2009Natur.457..824B. ISSN 0028-0836. PMID 19212400. doi:10.1038/nature07892.
- ↑ Elena, Santiago F.; Lenski, Richard E. (June 2003). «Evolution experiments with microorganisms: the dynamics and genetic bases of adaptation». Nature Reviews Genetics 4 (6): 457-469. ISSN 1471-0056. PMID 12776215. doi:10.1038/nrg1088.
- ↑ Mayr, 1982, p. 483: "Adaptation... could no longer be considered a static condition, a product of a creative past and became instead a continuing dynamic process."
- ↑ The sixth edition of the Oxford Dictionary of Science (2010) defines adaptation as "Any change in the structure or functioning of successive generations of a population that makes it better suited to its environment."
- ↑ Orr, H. Allen (February 2005). «The genetic theory of adaptation: a brief history». Nature Reviews Genetics 6 (2): 119-127. ISSN 1471-0056. PMID 15716908. doi:10.1038/nrg1523.
- ↑ Dobzhansky, 1968, pp. 1–34
- ↑ Dobzhansky, 1970, pp. 4–6, 79–82, 84–87
- ↑ Dobzhansky, Theodosius (March 1956). «Genetics of Natural Populations. XXV. Genetic Changes in Populations of Drosophila pseudoobscura and Drosophila persimilis in Some Localities in California». Evolution 10 (1): 82-92. ISSN 0014-3820. JSTOR 2406099. doi:10.2307/2406099.
- ↑ Nakajima, Akira; Sugimoto, Yohko; Yoneyama, Hiroshi; Nakae, Taiji (June 2002). «High-Level Fluoroquinolone Resistance in Pseudomonas aeruginosa Due to Interplay of the MexAB-OprM Efflux Pump and the DNA Gyrase Mutation». Microbiology and Immunology 46 (6): 391-395. ISSN 1348-0421. PMID 12153116. doi:10.1111/j.1348-0421.2002.tb02711.x.
- ↑ Blount, Zachary D.; Borland, Christina Z.; Lenski, Richard E. (June 10, 2008). «Inaugural Article: Historical contingency and the evolution of a key innovation in an experimental population of Escherichia coli». Proc. Natl. Acad. Sci. U.S.A. 105 (23): 7899-7906. Bibcode:2008PNAS..105.7899B. ISSN 0027-8424. PMC 2430337. PMID 18524956. doi:10.1073/pnas.0803151105.
- ↑ Okada, Hirosuke; Negoro, Seiji; Kimura, Hiroyuki; Nakamura, Shunichi (November 10, 1983). «Evolutionary adaptation of plasmid-encoded enzymes for degrading nylon oligomers». Nature 306 (5939): 203-206. Bibcode:1983Natur.306..203O. ISSN 0028-0836. PMID 6646204. doi:10.1038/306203a0.
- ↑ Ohno, Susumu (April 1984). «Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence». Proc. Natl. Acad. Sci. U.S.A. 81 (8): 2421-2425. Bibcode:1984PNAS...81.2421O. ISSN 0027-8424. PMC 345072. PMID 6585807. doi:10.1073/pnas.81.8.2421.
- ↑ Copley, Shelley D. (June 2000). «Evolution of a metabolic pathway for degradation of a toxic xenobiotic: the patchwork approach». Trends in Biochemical Sciences 25 (6): 261-265. ISSN 0968-0004. PMID 10838562. doi:10.1016/S0968-0004(00)01562-0.
- ↑ Crawford, Ronald L.; Jung, Carina M.; Strap, Janice L. (October 2007). «The recent evolution of pentachlorophenol (PCP)-4-monooxygenase (PcpB) and associated pathways for bacterial degradation of PCP». Biodegradation 18 (5): 525-539. ISSN 0923-9820. PMID 17123025. doi:10.1007/s10532-006-9090-6.
- ↑ Eshel, Ilan (December 1973). «Clone-Selection and Optimal Rates of Mutation». Journal of Applied Probability 10 (4): 728-738. ISSN 1475-6072. JSTOR 3212376. doi:10.2307/3212376.
- ↑ Altenberg, 1995, pp. 205–259
- ↑ Masel, Joanna; Bergman, Aviv (July 2003). «The evolution of the evolvability properties of the yeast prion [PSI+]». Evolution 57 (7): 1498-1512. ISSN 0014-3820. PMID 12940355. doi:10.1111/j.0014-3820.2003.tb00358.x.
- ↑ Lancaster, Alex K.; Bardill, J. Patrick; True, Heather L.; Masel, Joanna (February 2010). «The Spontaneous Appearance Rate of the Yeast Prion [PSI+] and Its Implications for the Evolution of the Evolvability Properties of the [PSI+] System». Genetics 184 (2). pp. 393-400. ISSN 0016-6731. PMC 2828720. PMID 19917766. doi:10.1534/genetics.109.110213.
- ↑ Draghi, Jeremy; Wagner, Günter P. (February 2008). «Evolution of evolvability in a developmental model». Evolution 62 (2): 301-315. ISSN 0014-3820. PMID 18031304. doi:10.1111/j.1558-5646.2007.00303.x.
- ↑ a b Bejder, Lars; Hall, Brian K. (November 2002). «Limbs in whales and limblessness in other vertebrates: mechanisms of evolutionary and developmental transformation and loss». Evolution & Development 4 (6): 445-458. ISSN 1520-541X. PMID 12492145. doi:10.1046/j.1525-142X.2002.02033.x.
- ↑ Young, Nathan M.; HallgrÍmsson, Benedikt (December 2005). «Serial homology and the evolution of mammalian limb covariation structure». Evolution 59 (12): 2691-2704. ISSN 0014-3820. PMID 16526515. doi:10.1554/05-233.1.
- ↑ a b Penny, David; Poole, Anthony (December 1999). «The nature of the last universal common ancestor». Current Opinion in Genetics & Development 9 (6): 672-677. ISSN 0959-437X. PMID 10607605. doi:10.1016/S0959-437X(99)00020-9.
- ↑ Hall, Brian K. (August 2003). «Descent with modification: the unity underlying homology and homoplasy as seen through an analysis of development and evolution». Biological Reviews 78 (3): 409-433. ISSN 1464-7931. PMID 14558591. doi:10.1017/S1464793102006097.
- ↑ Shubin, Neil; Tabin, Clifford J.; Carroll, Sean B. (February 12, 2009). «Deep homology and the origins of evolutionary novelty». Nature 457 (7231): 818-823. Bibcode:2009Natur.457..818S. ISSN 0028-0836. PMID 19212399. doi:10.1038/nature07891.
- ↑ a b c Fong, Daniel F.; Kane, Thomas C.; Culver, David C. (November 1995). «Vestigialization and Loss of Nonfunctional Characters». Annual Review of Ecology and Systematics 26: 249-268. ISSN 1545-2069. doi:10.1146/annurev.es.26.110195.001341.
- ↑ ZhaoLei Zhang; Gerstein, Mark (August 2004). «Large-scale analysis of pseudogenes in the human genome». Current Opinion in Genetics & Development 14 (4): 328-335. ISSN 0959-437X. PMID 15261647. doi:10.1016/j.gde.2004.06.003.
- ↑ Jeffery, William R. (May–June 2005). «Adaptive Evolution of Eye Degeneration in the Mexican Blind Cavefish». Journal of Heredity 96 (3): 185-196. ISSN 0022-1503. PMID 15653557. doi:10.1093/jhered/esi028.
- ↑ Maxwell, Erin E.; Larsson, Hans C.E. (May 2007). «Osteology and myology of the wing of the Emu (Dromaius novaehollandiae) and its bearing on the evolution of vestigial structures». Journal of Morphology 268 (5): 423-441. ISSN 0362-2525. PMID 17390336. doi:10.1002/jmor.10527.
- ↑ van der Kooi, Casper J.; Schwander, Tanja (November 2014). «On the fate of sexual traits under asexuality» (PDF). Biological Reviews 89 (4): 805-819. ISSN 1464-7931. PMID 24443922. doi:10.1111/brv.12078. Archivado desde el original el 23 de julio de 2015. Consultado el 5 de agosto de 2015.
- ↑ Silvestri, Anthony R., Jr.; Singh, Iqbal (April 2003). «The unresolved problem of the third molar: Would people be better off without it?». Journal of the American Dental Association 134 (4): 450-455. ISSN 0002-8177. PMID 12733778. doi:10.14219/jada.archive.2003.0194. Archivado desde el original el 23 de agosto de 2014.
- ↑ Coyne, 2009, p. 62
- ↑ Darwin, 1872, pp. 101, 103
- ↑ Gray, 2007, p. 66
- ↑ Coyne, 2009, pp. 85–86
- ↑ Stevens, 1982, p. 87
- ↑ a b Gould, 2002, pp. 1235–1236.
- ↑ Pallen, Mark J.; Matzke, Nicholas J. (October 2006). «From The Origin of Species to the origin of bacterial flagella». Nature Reviews Microbiology (PDF) 4 (10): 784-790. ISSN 1740-1526. PMID 16953248. doi:10.1038/nrmicro1493. Archivado desde el original el 26 de diciembre de 2014. Consultado el 25 de diciembre de 2014.
- ↑ Clements, Abigail; Bursac, Dejan; Gatsos, Xenia et al. (September 15, 2009). «The reducible complexity of a mitochondrial molecular machine». Proc. Natl. Acad. Sci. U.S.A. 106 (37): 15791-15795. Bibcode:2009PNAS..10615791C. ISSN 0027-8424. PMC 2747197. PMID 19717453. doi:10.1073/pnas.0908264106.
- ↑ Piatigorsky et al., 1994, pp. 241–250
- ↑ Wistow, Graeme (August 1993). «Lens crystallins: gene recruitment and evolutionary dynamism». Trends in Biochemical Sciences 18 (8): 301-306. ISSN 0968-0004. PMID 8236445. doi:10.1016/0968-0004(93)90041-K.
- ↑ Johnson, Norman A.; Porter, Adam H. (November 2001). «Toward a new synthesis: population genetics and evolutionary developmental biology». Genetica. 112–113 (1): 45-58. ISSN 0016-6707. PMID 11838782. doi:10.1023/A:1013371201773.
- ↑ Baguñà, Jaume; Garcia-Fernàndez, Jordi (2003). «Evo-Devo: the long and winding road». The International Journal of Developmental Biology 47 (7–8): 705-713. ISSN 0214-6282. PMID 14756346. Archivado desde el original el 28 de noviembre de 2014.
- Love, Alan C. (March 2003). «Evolutionary Morphology, Innovation and the Synthesis of Evolutionary and Developmental Biology». Biology and Philosophy 18 (2): 309-345. ISSN 0169-3867. doi:10.1023/A:1023940220348.
- ↑ Allin, Edgar F. (December 1975). «Evolution of the mammalian middle ear». Journal of Morphology 147 (4): 403-437. ISSN 0362-2525. PMID 1202224. doi:10.1002/jmor.1051470404.
- ↑ Harris, Matthew P.; Hasso, Sean M.; Ferguson, Mark W.J.; Fallon, John F. (February 21, 2006). «The Development of Archosaurian First-Generation Teeth in a Chicken Mutant». Current Biology 16 (4): 371-377. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMID 16488870. doi:10.1016/j.cub.2005.12.047.
- ↑ Carroll, Sean B. (July 11, 2008). «Evo-Devo and an Expanding Evolutionary Synthesis: A Genetic Theory of Morphological Evolution». Cell 134 (1): 25-36. ISSN 0092-8674. PMID 18614008. doi:10.1016/j.cell.2008.06.030.
- ↑ Wade, Michael J. (March 2007). «The co-evolutionary genetics of ecological communities». Nature Reviews Genetics 8 (3): 185-195. ISSN 1471-0056. PMID 17279094. doi:10.1038/nrg2031.
- ↑ Geffeney, Shana; Brodie, Edmund D., Jr.; Ruben, Peter C.; Brodie, Edmund D., III (August 23, 2002). «Mechanisms of Adaptation in a Predator-Prey Arms Race: TTX-Resistant Sodium Channels». Science 297 (5585): 1336-1339. Bibcode:2002Sci...297.1336G. ISSN 0036-8075. PMID 12193784. doi:10.1126/science.1074310.
- Brodie, Edmund D., Jr.; Ridenhour, Benjamin J.; Brodie, Edmund D., III (October 2002). «The evolutionary response of predators to dangerous prey: hotspots and coldspots in the geographic mosaic of coevolution between garter snakes and newts». Evolution 56 (10): 2067-2082. ISSN 0014-3820. PMID 12449493. doi:10.1554/0014-3820(2002)056[2067:teropt]2.0.co;2.
- Carroll, Sean B. (December 21, 2009). «Whatever Doesn't Kill Some Animals Can Make Them Deadly». The New York Times (New York: The New York Times Company). ISSN 0362-4331. Archivado desde el original el 23 de abril de 2015. Consultado el 26 de diciembre de 2014.
- ↑ Sachs, Joel L. (September 2006). «Cooperation within and among species». Journal of Evolutionary Biology 19 (5): 1415-1418; discussion 1426-1436. ISSN 1010-061X. PMID 16910971. doi:10.1111/j.1420-9101.2006.01152.x.
- Nowak, Martin A. (December 8, 2006). «Five Rules for the Evolution of Cooperation». Science 314 (5805): 1560-1563. Bibcode:2006Sci...314.1560N. ISSN 0036-8075. PMC 3279745. PMID 17158317. doi:10.1126/science.1133755.
- ↑ Paszkowski, Uta (August 2006). «Mutualism and parasitism: the yin and yang of plant symbioses». Current Opinion in Plant Biology 9 (4): 364-370. ISSN 1369-5266. PMID 16713732. doi:10.1016/j.pbi.2006.05.008.
- ↑ Hause, Bettina; Fester, Thomas (May 2005). «Molecular and cell biology of arbuscular mycorrhizal symbiosis». Planta 221 (2): 184-196. ISSN 0032-0935. PMID 15871030. doi:10.1007/s00425-004-1436-x.
- ↑ Bertram, John S. (December 2000). «The molecular biology of cancer». Molecular Aspects of Medicine 21 (6): 167-223. ISSN 0098-2997. PMID 11173079. doi:10.1016/S0098-2997(00)00007-8.
- ↑ Reeve, H. Kern; Hölldobler, Bert (June 5, 2007). «The emergence of a superorganism through intergroup competition». Proc. Natl. Acad. Sci. U.S.A. 104 (23): 9736-9740. Bibcode:2007PNAS..104.9736R. ISSN 0027-8424. PMC 1887545. PMID 17517608. doi:10.1073/pnas.0703466104.
- ↑ Axelrod, Robert; Hamilton, W. D. (March 27, 1981). «The evolution of cooperation». Science 211 (4489): 1390-1396. Bibcode:1981Sci...211.1390A. ISSN 0036-8075. PMID 7466396. doi:10.1126/science.7466396.
- ↑ Wilson, Edward O.; Hölldobler, Bert (September 20, 2005). «Eusociality: Origin and consequences». Proc. Natl. Acad. Sci. U.S.A. 102 (38): 13367-1371. Bibcode:2005PNAS..10213367W. ISSN 0027-8424. PMC 1224642. PMID 16157878. doi:10.1073/pnas.0505858102.
- ↑ a b Gavrilets, Sergey (October 2003). «Perspective: models of speciation: what have we learned in 40 years?». Evolution 57 (10): 2197-2215. ISSN 0014-3820. PMID 14628909. doi:10.1554/02-727.
- ↑ a b c de Queiroz, Kevin (3 de mayo de 2005). «Ernst Mayr and the modern concept of species». Proc. Natl. Acad. Sci. U.S.A. 102 (Suppl. 1): 6600-6607. Bibcode:2005PNAS..102.6600D. ISSN 0027-8424. PMC 1131873. PMID 15851674. doi:10.1073/pnas.0502030102.
- ↑ a b Ereshefsky, Marc (December 1992). «Eliminative pluralism». Philosophy of Science 59 (4): 671-690. ISSN 0031-8248. JSTOR 188136. doi:10.1086/289701.
- ↑ Mayr, 1942, p. 120
- ↑ Fraser, Christophe; Alm, Eric J.; Polz, Martin F. et al. (February 6, 2009). «The Bacterial Species Challenge: Making Sense of Genetic and Ecological Diversity». Science 323 (5915): 741-746. Bibcode:2009Sci...323..741F. ISSN 0036-8075. PMID 19197054. doi:10.1126/science.1159388.
- ↑ Short, Roger Valentine (October 1975). «The contribution of the mule to scientific thought». Journal of Reproduction and Fertility. Supplement (23): 359-364. ISSN 0449-3087. OCLC 1639439. PMID 1107543.
- ↑ Gross, Briana L.; Rieseberg, Loren H. (May–June 2005). «The Ecological Genetics of Homoploid Hybrid Speciation». Journal of Heredity 96 (3): 241-252. ISSN 0022-1503. PMC 2517139. PMID 15618301. doi:10.1093/jhered/esi026.
- ↑ Burke, John M.; Arnold, Michael L. (December 2001). «Genetics and the fitness of hybrids». Annual Review of Genetics 35: 31-52. ISSN 0066-4197. PMID 11700276. doi:10.1146/annurev.genet.35.102401.085719.
- ↑ Vrijenhoek, Robert C. (April 4, 2006). «Polyploid Hybrids: Multiple Origins of a Treefrog Species». Current Biology 16 (7): R245-R247. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMID 16581499. doi:10.1016/j.cub.2006.03.005.
- ↑ Rice, William R.; Hostert, Ellen E. (December 1993). «Laboratory Experiments on Speciation: What Have We Learned in 40 Years?». Evolution 47 (6): 1637-1653. ISSN 0014-3820. JSTOR 2410209. PMID 28568007. doi:10.2307/2410209.
- Jiggins, Chris D.; Bridle, Jon R. (March 2004). «Speciation in the apple maggot fly: a blend of vintages?». Trends in Ecology & Evolution 19 (3): 111-114. ISSN 0169-5347. PMID 16701238. doi:10.1016/j.tree.2003.12.008.
- Boxhorn, Joseph (September 1, 1995). «Observed Instances of Speciation». TalkOrigins Archive. Houston, Texas: The TalkOrigins Foundation, Inc. Archivado desde el original el 22 de enero de 2009. Consultado el 26 de diciembre de 2008.
- Weinberg, James R.; Starczak, Victoria R.; Jörg, Daniele (August 1992). «Evidence for Rapid Speciation Following a Founder Event in the Laboratory». Evolution 46 (4): 1214-1220. ISSN 0014-3820. JSTOR 2409766. PMID 28564398. doi:10.2307/2409766.
- ↑ Herrel, Anthony; Huyghe, Katleen; Vanhooydonck, Bieke et al. (March 25, 2008). «Rapid large-scale evolutionary divergence in morphology and performance associated with exploitation of a different dietary resource». Proc. Natl. Acad. Sci. U.S.A. 105 (12): 4792-4795. Bibcode:2008PNAS..105.4792H. ISSN 0027-8424. PMC 2290806. PMID 18344323. doi:10.1073/pnas.0711998105.
- ↑ Losos, Jonathan B.; Warhelt, Kenneth I.; Schoener, Thomas W. (1 de mayo de 1997). «Adaptive differentiation following experimental island colonization in Anolis lizards». Nature 387 (6628): 70-73. Bibcode:1997Natur.387...70L. ISSN 0028-0836. doi:10.1038/387070a0.
- ↑ Hoskin, Conrad J.; Higgle, Megan; McDonald, Keith R.; Moritz, Craig (October 27, 2005). «Reinforcement drives rapid allopatric speciation». Nature 437 (7063): 1353-1356. Bibcode:2005Natur.437.1353H. ISSN 0028-0836. PMID 16251964. doi:10.1038/nature04004.
- ↑ Templeton, Alan R. (April 1980). «The Theory of Speciation VIA the Founder Principle». Genetics 94 (4): 1011-1038. ISSN 0016-6731. PMC 1214177. PMID 6777243. Archivado desde el original el 23 de agosto de 2014. Consultado el 29 de diciembre de 2014.
- ↑ Antonovics, Janis (July 2006). «Evolution in closely adjacent plant populations X: long-term persistence of prereproductive isolation at a mine boundary». Heredity 97 (1): 33-37. ISSN 0018-067X. PMID 16639420. doi:10.1038/sj.hdy.6800835. Archivado desde el original el 23 de agosto de 2014. Consultado el 29 de diciembre de 2014.
- ↑ Nosil, Patrik; Crespi, Bernard J.; Gries, Regine; Gries, Gerhard (March 2007). «Natural selection and divergence in mate preference during speciation». Genetica 129 (3): 309-327. ISSN 0016-6707. PMID 16900317. doi:10.1007/s10709-006-0013-6.
- ↑ Savolainen, Vincent; Anstett, Marie-Charlotte; Lexer, Christian et al. (11 de mayo de 2006). «Sympatric speciation in palms on an oceanic island». Nature 441 (7090): 210-213. Bibcode:2006Natur.441..210S. ISSN 0028-0836. PMID 16467788. doi:10.1038/nature04566.
- Barluenga, Marta; Stölting, Kai N.; Salzburger, Walter et al. (February 9, 2006). «Sympatric speciation in Nicaraguan crater lake cichlid fish». Nature 439 (7077): 719-23. Bibcode:2006Natur.439..719B. ISSN 0028-0836. PMID 16467837. doi:10.1038/nature04325.
- ↑ Gavrilets, Sergey (March 21, 2006). «The Maynard Smith model of sympatric speciation». Journal of Theoretical Biology 239 (2): 172-182. ISSN 0022-5193. PMID 16242727. doi:10.1016/j.jtbi.2005.08.041.
- ↑ Wood, Troy E.; Takebayashi, Naoki; Barker, Michael S. et al. (August 18, 2009). «The frequency of polyploid speciation in vascular plants». Proc. Natl. Acad. Sci. U.S.A. 106 (33): 13875-13879. Bibcode:2009PNAS..10613875W. ISSN 0027-8424. PMC 2728988. PMID 19667210. doi:10.1073/pnas.0811575106.
- ↑ Hegarty, Matthew J.; Hiscock, Simon J. (20 de mayo de 2008). «Genomic Clues to the Evolutionary Success of Polyploid Plants». Current Biology 18 (10): R435-R444. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMID 18492478. doi:10.1016/j.cub.2008.03.043.
- ↑ Jakobsson, Mattias; Hagenblad, Jenny; Tavaré, Simon et al. (June 2006). «A Unique Recent Origin of the Allotetraploid Species Arabidopsis suecica: Evidence from Nuclear DNA Markers». Molecular Biology and Evolution 23 (6): 1217-1231. ISSN 0737-4038. PMID 16549398. doi:10.1093/molbev/msk006.
- ↑ Säll, Torbjörn; Jakobsson, Mattias; Lind-Halldén, Christina; Halldén, Christer (September 2003). «Chloroplast DNA indicates a single origin of the allotetraploid Arabidopsis suecica». Journal of Evolutionary Biology 16 (5): 1019-1029. ISSN 1010-061X. PMID 14635917. doi:10.1046/j.1420-9101.2003.00554.x.
- ↑ Bomblies, Kirsten; Weigel, Detlef (December 2007). «Arabidopsis—a model genus for speciation». Current Opinion in Genetics & Development 17 (6): 500-504. ISSN 0959-437X. PMID 18006296. doi:10.1016/j.gde.2007.09.006.
- ↑ Sémon, Marie; Wolfe, Kenneth H. (December 2007). «Consequences of genome duplication». Current Opinion in Genetics & Development 17 (6): 505-512. ISSN 0959-437X. PMID 18006297. doi:10.1016/j.gde.2007.09.007.
- ↑ Eldredge y Gould, 1972, pp. 82–115
- ↑ Benton, Michael J. (April 7, 1995). «Diversification and extinction in the history of life». Science 268 (5207): 52-58. Bibcode:1995Sci...268...52B. ISSN 0036-8075. PMID 7701342. doi:10.1126/science.7701342.
- ↑ Raup, David M. (March 28, 1986). «Biological extinction in Earth history». Science 231 (4745): 1528-1533. Bibcode:1986Sci...231.1528R. ISSN 0036-8075. PMID 11542058. doi:10.1126/science.11542058.
- ↑ Avise, John C.; Hubbell, Stephen P.; Ayala, Francisco J. (August 12, 2008). «In the light of evolution II: Biodiversity and extinction». Proc. Natl. Acad. Sci. U.S.A. 105 (Suppl. 1): 11453-11457. Bibcode:2008PNAS..10511453A. ISSN 0027-8424. PMC 2556414. PMID 18695213. doi:10.1073/pnas.0802504105.
- ↑ a b c Raup, David M. (July 19, 1994). «The role of extinction in evolution». Proc. Natl. Acad. Sci. U.S.A. 91 (15): 6758-6763. Bibcode:1994PNAS...91.6758R. ISSN 0027-8424. PMC 44280. PMID 8041694. doi:10.1073/pnas.91.15.6758.
- ↑ Novacek, Michael J.; Cleland, Elsa E. (8 de mayo de 2001). «The current biodiversity extinction event: scenarios for mitigation and recovery». Proc. Natl. Acad. Sci. U.S.A. 98 (10): 5466-5470. Bibcode:2001PNAS...98.5466N. ISSN 0027-8424. PMC 33235. PMID 11344295. doi:10.1073/pnas.091093698.
- ↑ Pimm, Stuart; Raven, Peter; Peterson, Alan et al. (July 18, 2006). «Human impacts on the rates of recent, present and future bird extinctions». Proc. Natl. Acad. Sci. U.S.A. 103 (29): 10941-10946. Bibcode:2006PNAS..10310941P. ISSN 0027-8424. PMC 1544153. PMID 16829570. doi:10.1073/pnas.0604181103.
- Barnosky, Anthony D.; Koch, Paul L.; Feranec, Robert S. et al. (October 1, 2004). «Assessing the Causes of Late Pleistocene Extinctions on the Continents». Science 306 (5693): 70-75. Bibcode:2004Sci...306...70B. ISSN 0036-8075. PMID 15459379. doi:10.1126/science.1101476. Parámetro desconocido
|citeseerx=ignorado (ayuda)
- Barnosky, Anthony D.; Koch, Paul L.; Feranec, Robert S. et al. (October 1, 2004). «Assessing the Causes of Late Pleistocene Extinctions on the Continents». Science 306 (5693): 70-75. Bibcode:2004Sci...306...70B. ISSN 0036-8075. PMID 15459379. doi:10.1126/science.1101476. Parámetro desconocido
- ↑ Lewis, Owen T. (January 29, 2006). «Climate change, species–area curves and the extinction crisis». Philosophical Transactions of the Royal Society B: Biological Sciences 361 (1465): 163-171. ISSN 0962-8436. PMC 1831839. PMID 16553315. doi:10.1098/rstb.2005.1712.
- ↑ a b Stearns y Stearns, 1999, p. X
- ↑ a b Novacek, Michael J. (November 8, 2014). «Prehistory’s Brilliant Future». The New York Times (New York: The New York Times Company). ISSN 0362-4331. Archivado desde el original el 29 de diciembre de 2014. Consultado el 25 de diciembre de 2014.
- ↑ «Researchers find that Earth may be home to 1 trillion species». National Science Foundation. Arlington County, Virginia. 2 de mayo de 2016. Archivado desde el original el 4 de mayo de 2016. Consultado el 6 de mayo de 2016.
- ↑ Jablonski, David (8 de mayo de 2001). «Lessons from the past: Evolutionary impacts of mass extinctions». Proc. Natl. Acad. Sci. U.S.A. 98 (10): 5393-5398. Bibcode:2001PNAS...98.5393J. ISSN 0027-8424. PMC 33224. PMID 11344284. doi:10.1073/pnas.101092598.
- ↑ «Age of the Earth». United States Geological Survey. July 9, 2007. Archivado desde el original el 23 de diciembre de 2005. Consultado el 31 de mayo de 2015.
- ↑ Dalrymple, 2001, pp. 205–221
- ↑ Manhesa, Gérard; Allègre, Claude J.; Dupréa, Bernard; Hamelin, Bruno (May 1980). «Lead isotope study of basic-ultrabasic layered complexes: Speculations about the age of the earth and primitive mantle characteristics». Earth and Planetary Science Letters 47 (3): 370-382. Bibcode:1980E&PSL..47..370M. ISSN 0012-821X. doi:10.1016/0012-821X(80)90024-2.
- ↑ Raven y Johnson, 2002, p. 68
- ↑ a b Borenstein, Seth (19 October 2015). «Hints of life on what was thought to be desolate early Earth». Excite (Yonkers, NY: Mindspark Interactive Network). Associated Press. Archivado desde el original el 23 October 2015. Consultado el 8 October 2018.
- ↑ Bell, Elizabeth A.; Boehnike, Patrick; Harrison, T. Mark; Mao, Wendy L. (November 24, 2015). «Potentially biogenic carbon preserved in a 4.1 billion-year-old zircon» (PDF). Proc. Natl. Acad. Sci. U.S.A. 112 (47): 14518-14521. Bibcode:2015PNAS..11214518B. ISSN 0027-8424. PMC 4664351. PMID 26483481. doi:10.1073/pnas.1517557112. Archivado desde el original el 6 de noviembre de 2015. Consultado el 30 de diciembre de 2015.
- ↑ Schouten, Lucy (October 20, 2015). «When did life first emerge on Earth? Maybe a lot earlier than we thought». The Christian Science Monitor (Boston, Massachusetts: Christian Science Publishing Society). ISSN 0882-7729. Archivado desde el original el 22 de marzo de 2016. Consultado el 11 de julio de 2018.
- ↑ Wade, Nicholas (July 25, 2016). «Meet Luca, the Ancestor of All Living Things». The New York Times (New York: The New York Times Company). ISSN 0362-4331. Archivado desde el original el July 28, 2016. Consultado el 25 de julio de 2016.
- ↑ McKinney, 1997, p. 110
- ↑ Mora, Camilo; Tittensor, Derek P.; Adl, Sina et al. (August 23, 2011). «How Many Species Are There on Earth and in the Ocean?». PLOS Biology 9 (8): e1001127. ISSN 1545-7885. PMC 3160336. PMID 21886479. doi:10.1371/journal.pbio.1001127.
- ↑ Miller y Spoolman, 2012, p. 62
- ↑ Chapman, 2009
- ↑ Roskov, Y.; Abucay, L.; Orrell, T.; Nicolson, D. et al., eds. (2016). «Species 2000 & ITIS Catalogue of Life, 2016 Annual Checklist». Species 2000. Leiden, Netherlands: Naturalis Biodiversity Center. ISSN 2405-884X. Archivado desde el original el 12 de noviembre de 2016. Consultado el 6 de noviembre de 2016.
- ↑ Peretó, Juli (March 2005). «Controversies on the origin of life» (PDF). International Microbiology 8 (1): 23-31. ISSN 1139-6709. PMID 15906258. Archivado desde el original el 24 de agosto de 2015.
- ↑ Joyce, Gerald F. (July 11, 2002). «The antiquity of RNA-based evolution». Nature 418 (6894): 214-221. Bibcode:2002Natur.418..214J. ISSN 0028-0836. PMID 12110897. doi:10.1038/418214a.
- ↑ Trevors, Jack T.; Psenner, Roland (December 2001). «From self-assembly of life to present-day bacteria: a possible role for nanocells». FEMS Microbiology Reviews 25 (5): 573-582. ISSN 1574-6976. PMID 11742692. doi:10.1111/j.1574-6976.2001.tb00592.x.
- ↑ Theobald, Douglas L. (13 de mayo de 2010). «A formal test of the theory of universal common ancestry». Nature 465 (7295): 219-222. Bibcode:2010Natur.465..219T. ISSN 0028-0836. PMID 20463738. doi:10.1038/nature09014.
- ↑ Bapteste, Eric; Walsh, David A. (June 2005). «Does the 'Ring of Life' ring true?». Trends in Microbiology 13 (6): 256-261. ISSN 0966-842X. PMID 15936656. doi:10.1016/j.tim.2005.03.012.
- ↑ Darwin, 1859, p. 1
- ↑ Doolittle, W. Ford; Bapteste, Eric (February 13, 2007). «Pattern pluralism and the Tree of Life hypothesis». Proc. Natl. Acad. Sci. U.S.A. 104 (7): 2043-2049. Bibcode:2007PNAS..104.2043D. ISSN 0027-8424. PMC 1892968. PMID 17261804. doi:10.1073/pnas.0610699104.
- ↑ Kunin, Victor; Goldovsky, Leon; Darzentas, Nikos; Ouzounis, Christos A. (July 2005). «The net of life: Reconstructing the microbial phylogenetic network». Genome Research 15 (7): 954-959. ISSN 1088-9051. PMC 1172039. PMID 15965028. doi:10.1101/gr.3666505.
- ↑ Jablonski, David (June 25, 1999). «The Future of the Fossil Record». Science 284 (5423): 2114-2116. ISSN 0036-8075. PMID 10381868. doi:10.1126/science.284.5423.2114.
- ↑ Mason, Stephen F. (September 6, 1984). «Origins of biomolecular handedness». Nature 311 (5981): 19-23. Bibcode:1984Natur.311...19M. ISSN 0028-0836. PMID 6472461. doi:10.1038/311019a0.
- ↑ Wolf, Yuri I.; Rogozin, Igor B.; Grishin, Nick V.; Koonin, Eugene V. (September 1, 2002). «Genome trees and the tree of life». Trends in Genetics 18 (9): 472-479. ISSN 0168-9525. PMID 12175808. doi:10.1016/S0168-9525(02)02744-0.
- ↑ Varki, Ajit; Altheide, Tasha K. (December 2005). «Comparing the human and chimpanzee genomes: searching for needles in a haystack». Genome Research 15 (12): 1746-1758. ISSN 1088-9051. PMID 16339373. doi:10.1101/gr.3737405.
- ↑ a b Cavalier-Smith, Thomas (June 29, 2006). «Cell evolution and Earth history: stasis and revolution». Philosophical Transactions of the Royal Society B: Biological Sciences 361 (1470): 969-1006. ISSN 0962-8436. PMC 1578732. PMID 16754610. doi:10.1098/rstb.2006.1842.
- ↑ Schopf, J. William (June 29, 2006). «Fossil evidence of Archaean life». Philosophical Transactions of the Royal Society B: Biological Sciences 361 (1470): 869-885. ISSN 0962-8436. PMC 1578735. PMID 16754604. doi:10.1098/rstb.2006.1834.
- ↑ Schopf, J. William (July 19, 1994). «Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic». Proc. Natl. Acad. Sci. U.S.A. 91 (15): 6735-6742. Bibcode:1994PNAS...91.6735S. ISSN 0027-8424. PMC 44277. PMID 8041691. doi:10.1073/pnas.91.15.6735.
- ↑ Poole, Anthony M.; Penny, David (January 2007). «Evaluating hypotheses for the origin of eukaryotes». BioEssays 29 (1): 74-84. ISSN 0265-9247. PMID 17187354. doi:10.1002/bies.20516.
- ↑ Dyall, Sabrina D.; Brown, Mark T.; Johnson, Patricia J. (April 9, 2004). «Ancient Invasions: From Endosymbionts to Organelles». Science 304 (5668): 253-257. Bibcode:2004Sci...304..253D. ISSN 0036-8075. PMID 15073369. doi:10.1126/science.1094884.
- ↑ Martin, William (October 2005). «The missing link between hydrogenosomes and mitochondria». Trends in Microbiology 13 (10): 457-459. ISSN 0966-842X. PMID 16109488. doi:10.1016/j.tim.2005.08.005.
- ↑ Lang, B. Franz; Gray, Michael W.; Burger, Gertraud (December 1999). «Mitochondrial genome evolution and the origin of eukaryotes». Annual Review of Genetics 33: 351-397. ISSN 0066-4197. PMID 10690412. doi:10.1146/annurev.genet.33.1.351.
- ↑ DeLong, Edward F.; Pace, Norman R. (August 1, 2001). «Environmental Diversity of Bacteria and Archaea». Systematic Biology 50 (4): 470-478. ISSN 1063-5157. PMID 12116647. doi:10.1080/106351501750435040. Archivado desde el original el 22 de febrero de 2016. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Kaiser, Dale (December 2001). «Building a multicellular organism». Annual Review of Genetics 35: 103-123. ISSN 0066-4197. PMID 11700279. doi:10.1146/annurev.genet.35.102401.090145.
- ↑ Zimmer, Carl (January 7, 2016). «Genetic Flip Helped Organisms Go From One Cell to Many». The New York Times (New York: The New York Times Company). ISSN 0362-4331. Archivado desde el original el 7 de enero de 2016. Consultado el 7 de enero de 2016.
- ↑ Valentine, James W.; Jablonski, David; Erwin, Douglas H. (March 1, 1999). «Fossils, molecules and embryos: new perspectives on the Cambrian explosion». Development 126 (5): 851-859. ISSN 0950-1991. PMID 9927587. Archivado desde el original el 1 de marzo de 2015. Consultado el 30 de diciembre de 2014.
- ↑ Ohno, Susumu (January 1997). «The reason for as well as the consequence of the Cambrian explosion in animal evolution». Journal of Molecular Evolution 44 (Suppl. 1): S23-S27. Bibcode:1997JMolE..44S..23O. ISSN 0022-2844. PMID 9071008. doi:10.1007/PL00000055.
- Valentine, James W.; Jablonski, David (2003). «Morphological and developmental macroevolution: a paleontological perspective». The International Journal of Developmental Biology 47 (7–8): 517-522. ISSN 0214-6282. PMID 14756327. Archivado desde el original el 24 de octubre de 2014. Consultado el 30 de diciembre de 2014.
- ↑ Waters, Elizabeth R. (December 2003). «Molecular adaptation and the origin of land plants». Molecular Phylogenetics and Evolution 29 (3): 456-463. ISSN 1055-7903. PMID 14615186. doi:10.1016/j.ympev.2003.07.018.
- ↑ Mayhew, Peter J. (August 2007). «Why are there so many insect species? Perspectives from fossils and phylogenies». Biological Reviews 82 (3): 425-454. ISSN 1464-7931. PMID 17624962. doi:10.1111/j.1469-185X.2007.00018.x.
- ↑ Carroll, Robert L. (May 2007). «The Palaeozoic Ancestry of Salamanders, Frogs and Caecilians». Zoological Journal of the Linnean Society 150 (Supplement s1): 1-140. ISSN 1096-3642. doi:10.1111/j.1096-3642.2007.00246.x.
- ↑ Wible, John R.; Rougier, Guillermo W.; Novacek, Michael J.; Asher, Robert J. (June 21, 2007). «Cretaceous eutherians and Laurasian origin for placental mammals near the K/T boundary». Nature 447 (7147): 1003-1006. Bibcode:2007Natur.447.1003W. ISSN 0028-0836. PMID 17581585. doi:10.1038/nature05854.
- ↑ Witmer, Lawrence M. (July 28, 2011). «Palaeontology: An icon knocked from its perch». Nature 475 (7357): 458-459. ISSN 0028-0836. PMID 21796198. doi:10.1038/475458a.
- ↑ Bull, James J.; Wichman, Holly A. (November 2001). «Applied evolution». Annual Review of Ecology and Systematics 32: 183-217. ISSN 1545-2069. doi:10.1146/annurev.ecolsys.32.081501.114020.
- ↑ Doebley, John F.; Gaut, Brandon S.; Smith, Bruce D. (December 29, 2006). «The Molecular Genetics of Crop Domestication». Cell 127 (7): 1309-1321. ISSN 0092-8674. PMID 17190597. doi:10.1016/j.cell.2006.12.006.
- ↑ Jäckel, Christian; Kast, Peter; Hilvert, Donald (June 2008). «Protein Design by Directed Evolution». Annual Review of Biophysics 37: 153-173. ISSN 1936-122X. PMID 18573077. doi:10.1146/annurev.biophys.37.032807.125832.
- ↑ Maher, Brendan (April 8, 2009). «Evolution: Biology's next top model?». Nature 458 (7239): 695-698. ISSN 0028-0836. PMID 19360058. doi:10.1038/458695a.
- ↑ Borowsky, Richard (January 8, 2008). «Restoring sight in blind cavefish». Current Biology 18 (1): R23-R24. Bibcode:1996CBio....6.1213A. ISSN 0960-9822. PMID 18177707. doi:10.1016/j.cub.2007.11.023.
- ↑ Gross, Joshua B.; Borowsky, Richard; Tabin, Clifford J. (January 2, 2009). «A novel role for Mc1r in the parallel evolution of depigmentation in independent populations of the cavefish Astyanax mexicanus». En Barsh, Gregory S., ed. PLOS Genetics 5 (1): e1000326. ISSN 1553-7390. PMC 2603666. PMID 19119422. doi:10.1371/journal.pgen.1000326.
- ↑ Merlo, Lauren M.F.; Pepper, John W.; Reid, Brian J.; Maley, Carlo C. (December 2006). «Cancer as an evolutionary and ecological process.». Nature Reviews Cancer 6 (12): 924-935. ISSN 1474-175X. PMID 17109012. doi:10.1038/nrc2013.
- ↑ Pan, Dabo; Weiwei Xue; Wenqi Zhang et al. (October 2012). «Understanding the drug resistance mechanism of hepatitis C virus NS3/4A to ITMN-191 due to R155K, A156V, D168A/E mutations: a computational study.». Biochimica et Biophysica Acta (BBA) - General Subjects 1820 (10): 1526-1534. ISSN 0304-4165. PMID 22698669. doi:10.1016/j.bbagen.2012.06.001.
- ↑ Woodford, Neil; Ellington, Matthew J. (January 2007). «The emergence of antibiotic resistance by mutation.». Clinical Microbiology and Infection 13 (1): 5-18. ISSN 1198-743X. PMID 17184282. doi:10.1111/j.1469-0691.2006.01492.x.
- ↑ Labbé, Pierrick; Berticat, Claire; Berthomieu, Arnaud et al. (November 16, 2007). «Forty Years of Erratic Insecticide Resistance Evolution in the Mosquito Culex pipiens». PLOS Genetics 3 (11): e205. ISSN 1553-7390. PMC 2077897. PMID 18020711. doi:10.1371/journal.pgen.0030205.
- ↑ Neve, Paul (October 2007). «Challenges for herbicide resistance evolution and management: 50 years after Harper». Weed Research 47 (5): 365-369. ISSN 0043-1737. doi:10.1111/j.1365-3180.2007.00581.x.
- ↑ Rodríguez-Rojas, Alexandro; Rodríguez-Beltrán, Jerónimo; Couce, Alejandro; Blázquez, Jesús (August 2013). «Antibiotics and antibiotic resistance: A bitter fight against evolution». International Journal of Medical Microbiology 303 (6–7): 293-297. ISSN 1438-4221. PMID 23517688. doi:10.1016/j.ijmm.2013.02.004.
- ↑ Schenk, Martijn F.; Szendro, Ivan G.; Krug, Joachim; de Visser, J. Arjan G.M. (June 28, 2012). «Quantifying the Adaptive Potential of an Antibiotic Resistance Enzyme». PLOS Genetics 8 (6): e1002783. ISSN 1553-7390. PMC 3386231. PMID 22761587. doi:10.1371/journal.pgen.1002783.
- ↑ Read, Andrew F.; Lynch, Penelope A.; Thomas, Matthew B. (April 7, 2009). «How to Make Evolution-Proof Insecticides for Malaria Control». PLOS Biology 7 (4): e1000058. ISSN 1545-7885. PMC 3279047. PMID 19355786. doi:10.1371/journal.pbio.1000058.
- ↑ Fraser, Alex S. (January 18, 1958). «Monte Carlo Analyses of Genetic Models». Nature 181 (4603): 208-209. Bibcode:1958Natur.181..208F. ISSN 0028-0836. PMID 13504138. doi:10.1038/181208a0.
- ↑ Rechenberg, 1973
- ↑ Holland, 1975
- ↑ Koza, 1992
- ↑ Jamshidi, Mo (August 15, 2003). «Tools for intelligent control: fuzzy controllers, neural networks and genetic algorithms». Philosophical Transactions of the Royal Society A 361 (1809): 1781-1808. Bibcode:2003RSPTA.361.1781J. ISSN 1364-503X. PMID 12952685. doi:10.1098/rsta.2003.1225.
- ↑ Browne, 2003, pp. 376–379
- ↑ For an overview of the philosophical, religious and cosmological controversies, see:
For the scientific and social reception of evolution in the 19th and early 20th centuries, see:
- Johnston, Ian C. (1999). «Section Three: The Origins of Evolutionary Theory». ... And Still We Evolve: A Handbook for the Early History of Modern Science (3rd revised edición). Nanaimo, BC: Liberal Studies Department, Malaspina University-College. Archivado desde el original el 16 de abril de 2016. Consultado el 1 de enero de 2015.
- Bowler, 2003
- Zuckerkandl, Emile (December 30, 2006). «Intelligent design and biological complexity». Gene 385: 2-18. ISSN 0378-1119. PMID 17011142. doi:10.1016/j.gene.2006.03.025.
- ↑ Ross, Marcus R. (May 2005). «Who Believes What? Clearing up Confusion over Intelligent Design and Young-Earth Creationism» (PDF). Journal of Geoscience Education 53 (3): 319-323. Bibcode:2005JGeEd..53..319R. ISSN 1089-9995. doi:10.5408/1089-9995-53.3.319. Archivado desde el original el 11 de mayo de 2008. Consultado el 28 de abril de 2008. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Hameed, Salman (December 12, 2008). «Bracing for Islamic Creationism» (PDF). Science 322 (5908): 1637-1638. ISSN 0036-8075. PMID 19074331. doi:10.1126/science.1163672. Archivado desde el original el 10 de noviembre de 2014.
- ↑ Bowler, 2003
- ↑ Miller, Jon D.; Scott, Eugenie C.; Okamoto, Shinji (August 11, 2006). «Public Acceptance of Evolution». Science 313 (5788): 765-766. ISSN 0036-8075. PMID 16902112. doi:10.1126/science.1126746.
- ↑ Spergel, David Nathaniel; Verde, Licia; Peiris, Hiranya V. et al. (2003). «First-Year Wilkinson Microwave Anisotropy Probe (WMAP) Observations: Determination of Cosmological Parameters». The Astrophysical Journal Supplement Series 148 (1): 175-194. Bibcode:2003ApJS..148..175S. arXiv:astro-ph/0302209. doi:10.1086/377226.
- ↑ Wilde, Simon A.; Valley, John W.; Peck, William H.; Graham, Colin M. (January 11, 2001). «Evidence from detrital zircons for the existence of continental crust and oceans on the Earth 4.4 Gyr ago». Nature 409 (6817): 175-178. ISSN 0028-0836. PMID 11196637. doi:10.1038/35051550.
- ↑ Branch, Glenn (March 2007). «Understanding Creationism after Kitzmiller». BioScience 57 (3): 278-284. Bibcode:1985BioSc..35..499W. ISSN 0006-3568. doi:10.1641/B570313.
- ↑ https://www.cambridge.org/core/journals/british-journal-for-the-history-of-science/article/translation-and-transmutation-the-origin-of-species-in-china/3F5AED9F9D60EF2D7265927BB2B6AB3A
Bibliography
- Altenberg, Lee (1995). «Genome growth and the evolution of the genotype-phenotype map». En Banzhaf, Wolfgang; Eeckman, Frank H., eds. Evolution and Biocomputation: Computational Models of Evolution. Lecture Notes in Computer Science 899. Berlin; New York: Springer-Verlag Berlin Heidelberg. pp. 205-259. ISBN 978-3-540-59046-0. ISSN 0302-9743. LCCN 95005970. OCLC 32049812. doi:10.1007/3-540-59046-3_11. Parámetro desconocido
|citeseerx=ignorado (ayuda) - Ayala, Francisco J.; Avise, John C., eds. (2014). Essential Readings in Evolutionary Biology. Baltimore, Maryland: Johns Hopkins University Press. ISBN 978-1-4214-1305-1. LCCN 2013027718. OCLC 854285705.
- Birdsell, John A.; Wills, Christopher (2003). «The Evolutionary Origin and Maintenance of Sexual Recombination: A Review of Contemporary Models». En MacIntyre, Ross J.; Clegg, Michael T., eds. Evolutionary Biology. Evolutionary Biology 33. New York: Springer Science+Business Media. ISBN 978-1-4419-3385-0. ISSN 0071-3260. OCLC 751583918.
- Bowler, Peter J. (1989). The Mendelian Revolution: The Emergence of Hereditarian Concepts in Modern Science and Society. Baltimore, Maryland: Johns Hopkins University Press. ISBN 978-0-8018-3888-0. LCCN 89030914. OCLC 19322402.
- Bowler, Peter J. (2003). Evolution: The History of an Idea (3rd completely rev. and expanded edición). Berkeley, California: University of California Press. ISBN 978-0-520-23693-6. LCCN 2002007569. OCLC 49824702.
- Browne, Janet (2003). Charles Darwin: The Power of Place 2. London: Pimlico. ISBN 978-0-7126-6837-8. LCCN 94006598. OCLC 52327000.
- Burkhardt, Frederick; Smith, Sydney, eds. (1991). The Correspondence of Charles Darwin. The Correspondence of Charles Darwin. 7: 1858–1859. Cambridge: Cambridge University Press. ISBN 978-0-521-38564-0. LCCN 84045347. OCLC 185662993.
- Carroll, Sean B.; Grenier, Jennifer K.; Weatherbee, Scott D. (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design (2nd edición). Malden, Massachusetts: Blackwell Publishing. ISBN 978-1-4051-1950-4. LCCN 2003027991. OCLC 53972564.
- Chapman, Arthur D. (2009). Numbers of Living Species in Australia and the World (2nd edición). Canberra: Department of the Environment, Water, Heritage and the Arts: Australian Biological Resources Study. ISBN 978-0-642-56860-1. OCLC 780539206. Archivado desde el original el 25 de diciembre de 2016. Consultado el 6 de noviembre de 2016.
- Coyne, Jerry A. (2009). Why Evolution is True. New York: Viking. ISBN 978-0-670-02053-9. LCCN 2008033973. OCLC 233549529.
- Cracraft, Joel; Bybee, Rodger W., eds. (2005). Evolutionary Science and Society: Educating a New Generation (PDF). Colorado Springs, Colorado: Biological Sciences Curriculum Study. ISBN 978-1-929614-23-3. OCLC 64228003. Consultado el 6 de diciembre de 2014. "Revised Proceedings of the BSCS, AIBS Symposium November 2004, Chicago, Illinois"
- Dalrymple, G. Brent (2001). The age of the Earth in the twentieth century: a problem (mostly) solved. En Lewis, C.L.E.; Knell, S.J., eds. «The Age of the Earth: from 4004 BC to AD 2002». Geological Society, London, Special Publications. Geological Society Special Publication 190 (1). pp. 205-221. Bibcode:2001GSLSP.190..205D. ISBN 978-1-86239-093-5. LCCN 2003464816. OCLC 48570033. doi:10.1144/gsl.sp.2001.190.01.14.
- Darwin, Charles (1859). On the Origin of Species by Means of Natural Selection, or the Preservation of Favoured Races in the Struggle for Life (1st edición). London: John Murray. LCCN 06017473. OCLC 741260650. Parámetro desconocido
|title-link=ignorado (ayuda) The book is available from The Complete Work of Charles Darwin Online. Retrieved 2014-11-21. - Darwin, Charles (1872). The Expression of the Emotions in Man and Animals. London: John Murray. LCCN 04002793. OCLC 1102785. Parámetro desconocido
|title-link=ignorado (ayuda) - Darwin, Francis, ed. (1909). The foundations of The origin of species, a sketch written in 1842 (PDF). Cambridge: Printed at the University Press. LCCN 61057537. OCLC 1184581. Consultado el 27 de noviembre de 2014.
- Dawkins, Richard (1990). The Blind Watchmaker. Penguin Science. London: Penguin Books. ISBN 978-0-14-014481-9. OCLC 60143870. Parámetro desconocido
|title-link=ignorado (ayuda) - Dennett, Daniel (1995). Darwin's Dangerous Idea: Evolution and the Meanings of Life. New York: Simon & Schuster. ISBN 978-0-684-80290-9. LCCN 94049158. OCLC 31867409. Parámetro desconocido
|title-link=ignorado (ayuda) - Dobzhansky, Theodosius (1968). «On Some Fundamental Concepts of Darwinian Biology». En Dobzhansky, Theodosius; Hecht, Max K.; Steere, William C., eds. Evolutionary Biology. Volume 2 (1st edición). New York: Appleton-Century-Crofts. pp. 1-34. ISBN 978-1-4684-8096-2. OCLC 24875357. doi:10.1007/978-1-4684-8094-8_1.
- Dobzhansky, Theodosius (1970). Genetics of the Evolutionary Process. New York: Columbia University Press. ISBN 978-0-231-02837-0. LCCN 72127363. OCLC 97663.
- Eldredge, Niles; Gould, Stephen Jay (1972). «Punctuated equilibria: an alternative to phyletic gradualism». En Schopf, Thomas J.M., ed. Models in Paleobiology. San Francisco, California: Freeman, Cooper. ISBN 978-0-87735-325-6. LCCN 72078387. OCLC 572084.
- Eldredge, Niles (1985). Time Frames: The Rethinking of Darwinian Evolution and the Theory of Punctuated Equilibria. New York: Simon & Schuster. ISBN 978-0-671-49555-8. LCCN 84023632. OCLC 11443805.
- Ewens, Warren J. (2004). Mathematical Population Genetics. Interdisciplinary Applied Mathematics. I. Theoretical Introduction (2nd edición). New York: Springer-Verlag New York. ISBN 978-0-387-20191-7. LCCN 2003065728. OCLC 53231891.
- Futuyma, Douglas J. (2004). «The Fruit of the Tree of Life: Insights into Evolution and Ecology». En Cracraft, Joel; Donoghue, Michael J., eds. Assembling the Tree of Life. Oxford; New York: Oxford University Press. ISBN 978-0-19-517234-8. LCCN 2003058012. OCLC 61342697. "Proceedings of a symposium held at the American Museum of Natural History in New York, 2002."
- Futuyma, Douglas J. (2005). Evolution. Sunderland, Massachusetts: Sinauer Associates. ISBN 978-0-87893-187-3. LCCN 2004029808. OCLC 57311264.
- Gould, Stephen Jay (2002). The Structure of Evolutionary Theory. Cambridge, Massachusetts: Belknap Press of Harvard University Press. ISBN 978-0-674-00613-3. LCCN 2001043556. OCLC 47869352. Parámetro desconocido
|title-link=ignorado (ayuda) - Gray, Peter (2007). Psychology (5th edición). New York: Worth Publishers. ISBN 978-0-7167-0617-5. LCCN 2006921149. OCLC 76872504.
- Hall, Brian K.; Hallgrímsson, Benedikt (2008). Strickberger's Evolution (4th edición). Sudbury, Massachusetts: Jones and Bartlett Publishers. ISBN 978-0-7637-0066-9. LCCN 2007008981. OCLC 85814089.
- Hennig, Willi (1999) [Originally published 1966 (reprinted 1979); translated from the author's unpublished revision of Grundzüge einer Theorie der phylogenetischen Systematik, published in 1950]. Phylogenetic Systematics. Translation by D. Dwight Davis and Rainer Zangerl; foreword by Donn E. Rosen, Gareth Nelson, and Colin Patterson (Reissue edición). Urbana, Illinois: University of Illinois Press. ISBN 978-0-252-06814-0. LCCN 78031969. OCLC 722701473.
- Holland, John H. (1975). Adaptation in Natural and Artificial Systems: An Introductory Analysis with Applications to Biology, Control, and Artificial Intelligence. Ann Arbor, Michigan: University of Michigan Press. ISBN 978-0-472-08460-9. LCCN 74078988. OCLC 1531617.
- Jablonka, Eva; Lamb, Marion J. (2005). Evolution in Four Dimensions: Genetic, Epigenetic, Behavioral, and Symbolic Variation in the History of Life. Illustrations by Anna Zeligowski. Cambridge, Massachusetts: MIT Press. ISBN 978-0-262-10107-3. LCCN 2004058193. OCLC 61896061.
- Kampourakis, Kostas (2014). Understanding Evolution. Cambridge; New York: Cambridge University Press. ISBN 978-1-107-03491-4. LCCN 2013034917. OCLC 855585457.
- Kirk, Geoffrey; Raven, John; Schofield, Malcolm (1983). The Presocratic Philosophers: A Critical History with a Selection of Texts (2nd edición). Cambridge; New York: Cambridge University Press. ISBN 978-0-521-27455-5. LCCN 82023505. OCLC 9081712.
- Koza, John R. (1992). Genetic Programming: On the Programming of Computers by Means of Natural Selection. Complex Adaptive Systems. Cambridge, Massachusetts: MIT Press. ISBN 978-0-262-11170-6. LCCN 92025785. OCLC 26263956.
- Lamarck, Jean-Baptiste (1809). Philosophie Zoologique. Paris: Dentu et L'Auteur. OCLC 2210044. Parámetro desconocido
|title-link=ignorado (ayuda) Evolución biológica en Internet Archive. Retrieved 2014-11-29. - Lane, David H. (1996). The Phenomenon of Teilhard: Prophet for a New Age (1st edición). Macon, Georgia: Mercer University Press. ISBN 978-0-86554-498-7. LCCN 96008777. OCLC 34710780.
- Lucretius. Book V, lines 855–877. «De Rerum Natura». Perseus Digital Library. Edited and translated by William Ellery Leonard (1916) (Medford/Somerville, Massachusetts: Tufts University). OCLC 33233743. Archivado desde el original el 4 de septiembre de 2014
|urlarchivo=requiere|url=(ayuda). Consultado el 25 de noviembre de 2014. Parámetro desconocido|title-link=ignorado (ayuda) - Magner, Lois N. (2002). A History of the Life Sciences (3rd rev. and expanded edición). New York: Marcel Dekker. ISBN 978-0-8247-0824-5. LCCN 2002031313. OCLC 50410202.
- Mason, Stephen F. (1962). A History of the Sciences. Collier Books. Science Library, CS9 (New rev. edición). New York: Collier Books. LCCN 62003378. OCLC 568032626.
- Maynard Smith, John (1978). The Evolution of Sex. Cambridge; New York: Cambridge University Press. ISBN 978-0-521-29302-0. LCCN 77085689. OCLC 3413793.
- Maynard Smith, John (1998). The Units of Selection. En Bock, Gregory R.; Goode, Jamie A., eds. «The Limits of Reductionism in Biology». Novartis Foundation Symposium. Novartis Foundation Symposia 213 (Chichester, England: John Wiley & Sons). pp. 203-221. ISBN 978-0-471-97770-4. LCCN 98002779. OCLC 38311600. PMID 9653725. doi:10.1002/9780470515488.ch15. "Papers from the Symposium on the Limits of Reductionism in Biology, held at the Novartis Foundation, London, May 13–15, 1997."
- Mayr, Ernst (1942). Systematics and the Origin of Species from the Viewpoint of a Zoologist. Columbia Biological Series 13. New York: Columbia University Press. LCCN 43001098. OCLC 766053. Parámetro desconocido
|title-link=ignorado (ayuda) - Mayr, Ernst (1982). The Growth of Biological Thought: Diversity, Evolution, and Inheritance. Translation of John Ray by E. Silk. Cambridge, Massachusetts: Belknap Press. ISBN 978-0-674-36445-5. LCCN 81013204. OCLC 7875904. Parámetro desconocido
|title-link=ignorado (ayuda) - Mayr, Ernst (2002) [Originally published 2001; New York: Basic Books]. What Evolution Is. Science Masters. London: Weidenfeld & Nicolson. ISBN 978-0-297-60741-0. LCCN 2001036562. OCLC 248107061.
- McKinney, Michael L. (1997). «How do rare species avoid extinction? A paleontological view». En Kunin, William E.; Gaston, Kevin J., eds. The Biology of Rarity: Causes and consequences of rare—common differences (1st edición). London; New York: Chapman & Hall. ISBN 978-0-412-63380-5. LCCN 96071014. OCLC 36442106.
- Miller, G. Tyler; Spoolman, Scott E. (2012). Environmental Science (14th edición). Belmont, California: Brooks/Cole. ISBN 978-1-111-98893-7. LCCN 2011934330. OCLC 741539226. Consultado el 27 de diciembre de 2014.
- Moore, Randy; Decker, Mark; Cotner, Sehoya (2010). Chronology of the Evolution-Creationism Controversy. Santa Barbara, California: Greenwood Press/ABC-CLIO. ISBN 978-0-313-36287-3. LCCN 2009039784. OCLC 422757410.
- Nardon, Paul; Grenier, Anne-Marie (1991). «Serial Endosymbiosis Theory and Weevil Evolution: The Role of Symbiosis». En Margulis, Lynn; Fester, René, eds. Symbiosis as a Source of Evolutionary Innovation: Speciation and Morphogenesis. Cambridge, Massachusetts: MIT Press. ISBN 978-0-262-13269-5. LCCN 90020439. OCLC 22597587. "Based on a conference held in Bellagio, Italy, June 25–30, 1989"
- National Academy of Sciences; Institute of Medicine (2008). Science, Evolution, and Creationism. Washington, DC: National Academy Press. ISBN 978-0-309-10586-6. LCCN 2007015904. OCLC 123539346. Consultado el 22 de noviembre de 2014.
- Odum, Eugene P. (1971). Fundamentals of Ecology (3rd edición). Philadelphia, Pennsylvania: Saunders. ISBN 978-0-7216-6941-0. LCCN 76081826. OCLC 154846.
- Okasha, Samir (2006). Evolution and the Levels of Selection. Oxford; New York: Oxford University Press. ISBN 978-0-19-926797-2. LCCN 2006039679. OCLC 70985413.
- Panno, Joseph (2005). The Cell: Evolution of the First Organism. Facts on File science library. New York: Facts on File. ISBN 978-0-8160-4946-2. LCCN 2003025841. OCLC 53901436.
- Piatigorsky, Joram; Kantorow, Marc; Gopal-Srivastava, Rashmi; Tomarev, Stanislav I. (1994). Recruitment of enzymes and stress proteins as lens crystallins. En Jansson, Bengt; Jörnvall, Hans; Rydberg, Ulf et al., eds. «Toward a Molecular Basis of Alcohol Use and Abuse». Exs. Experientia 71 (Basel; Boston: Birkhäuser Verlag). pp. 241-50. ISBN 978-3-7643-2940-2. LCCN 94010167. OCLC 30030941. PMID 8032155. doi:10.1007/978-3-0348-7330-7_24.
- Pigliucci, Massimo; Müller, Gerd B., eds. (2010). Evolution, the Extended Synthesis. Cambridge, Massachusetts: MIT Press. ISBN 978-0-262-51367-8. LCCN 2009024587. OCLC 804875316. Archivado desde el original el 18 de septiembre de 2015.
- Pocheville, Arnaud; Danchin, Étienne (2017). «Chapter 3: Genetic assimilation and the paradox of blind variation». En Huneman, Philippe; Walsh, Denis M., eds. Challenging the Modern Synthesis. New York: Oxford University Press. ISBN 978-0-19-937717-6. LCCN 2017448929. OCLC 1001337947.
- Provine, William B. (1971). The Origins of Theoretical Population Genetics. Chicago History of Science and Medicine (2nd edición). Chicago, Illinois: University of Chicago Press. ISBN 978-0-226-68464-2. LCCN 2001027561. OCLC 46660910.
- Provine, William B. (1988). «Progress in Evolution and Meaning in Life». En Nitecki, Matthew H., ed. Evolutionary Progress. Chicago, Illinois: University of Chicago Press. ISBN 978-0-226-58693-9. LCCN 88020835. OCLC 18380658. "This book is the result of the Spring Systematics Symposium held in May, 1987, at the Field Museum in Chicago"
- Quammen, David (2006). The Reluctant Mr. Darwin: An Intimate Portrait of Charles Darwin and the Making of His Theory of Evolution. Great Discoveries (1st edición). New York: Atlas Books/W.W. Norton & Company. ISBN 978-0-393-05981-6. LCCN 2006009864. OCLC 65400177.
- Raven, Peter H.; Johnson, George B. (2002). Biology (6th edición). Boston, Massachusetts: McGraw-Hill. ISBN 978-0-07-112261-0. LCCN 2001030052. OCLC 45806501.
- Ray, John (1686). Historia Plantarum [History of Plants] I. Londini: Typis Mariæ Clark. LCCN agr11000774. OCLC 2126030.
- Rechenberg, Ingo (1973). Evolutionsstrategie; Optimierung technischer Systeme nach Prinzipien der biologischen Evolution (PhD thesis). Problemata (en german) 15. Afterword by Manfred Eigen. Stuttgart-Bad Cannstatt: Frommann-Holzboog. ISBN 978-3-7728-0373-4. LCCN 74320689. OCLC 9020616.
- Ridley, Matt (1993). The Red Queen: Sex and the Evolution of Human Nature. New York: Viking. ISBN 978-0-670-84357-2. OCLC 636657988. Parámetro desconocido
|title-link=ignorado (ayuda) - Stearns, Beverly Peterson; Stearns, Stephen C. (1999). Watching, from the Edge of Extinction. New Haven, Connecticut: Yale University Press. ISBN 978-0-300-08469-6. LCCN 98034087. OCLC 803522914.
- Stevens, Anthony (1982). Archetype: A Natural History of the Self. London: Routledge & Kegan Paul. ISBN 978-0-7100-0980-7. LCCN 84672250. OCLC 10458367.
- West-Eberhard, Mary Jane (2003). Developmental Plasticity and Evolution. Oxford; New York: Oxford University Press. ISBN 978-0-19-512235-0. LCCN 2001055164. OCLC 48398911.
- Wiley, E. O.; Lieberman, Bruce S. (2011). Phylogenetics: Theory and Practice of Phylogenetic Systematics (2nd edición). Hoboken, New Jersey: Wiley-Blackwell. ISBN 978-0-470-90596-8. LCCN 2010044283. OCLC 741259265. doi:10.1002/9781118017883.
- Wright, Sewall (1984). Genetic and Biometric Foundations. Evolution and the Genetics of Populations 1. Chicago, Illinois: University of Chicago Press. ISBN 978-0-226-91038-3. LCCN 67025533. OCLC 246124737.
Further reading
Plantilla:Library resources box
Introductory reading
- Barrett, Paul H.; Weinshank, Donald J.; Gottleber, Timothy T., eds. (1981). A Concordance to Darwin's Origin of Species, First Edition. Ithaca, New York: Cornell University Press. ISBN 978-0-8014-1319-3. LCCN 80066893. OCLC 610057960.
- Carroll, Sean B. (2005). Endless Forms Most Beautiful: The New Science of Evo Devo and the Making of the Animal Kingdom. illustrations by Jamie W. Carroll, Josh P. Klaiss, Leanne M. Olds (1st edición). New York: W.W. Norton & Company. ISBN 978-0-393-06016-4. LCCN 2004029388. OCLC 57316841.
- Charlesworth, Brian; Charlesworth, Deborah (2003). Evolution: A Very Short Introduction. Very Short Introductions. Oxford; New York: Oxford University Press. ISBN 978-0-19-280251-4. LCCN 2003272247. OCLC 51668497.
- Gould, Stephen Jay (1989). Wonderful Life: The Burgess Shale and the Nature of History (1st edición). New York: W.W. Norton & Company. ISBN 978-0-393-02705-1. LCCN 88037469. OCLC 18983518. Parámetro desconocido
|title-link=ignorado (ayuda) - Jones, Steve (1999). Almost Like a Whale: The Origin of Species Updated. London; New York: Doubleday. ISBN 978-0-385-40985-8. LCCN 2002391059. OCLC 41420544. Parámetro desconocido
|title-link=ignorado (ayuda)- —— (2000). Darwin's Ghost: The Origin of Species Updated (1st edición). New York: Random House. ISBN 978-0-375-50103-6. LCCN 99053246. OCLC 42690131. American version.
- Mader, Sylvia S. (2007). Biology. Significant contributions by Murray P. Pendarvis (9th edición). Boston, Massachusetts: McGraw-Hill Higher Education. ISBN 978-0-07-246463-4. LCCN 2005027781. OCLC 61748307.
- Maynard Smith, John (1993). The Theory of Evolution (Canto edición). Cambridge; New York: Cambridge University Press. ISBN 978-0-521-45128-4. LCCN 93020358. OCLC 27676642. Parámetro desconocido
|title-link=ignorado (ayuda) - Pallen, Mark J. (2009). The Rough Guide to Evolution. Rough Guides Reference Guides. London; New York: Rough Guides. ISBN 978-1-85828-946-5. LCCN 2009288090. OCLC 233547316.
Advanced reading
- Barton, Nicholas H.; Briggs, Derek E.G.; Eisen, Jonathan A. et al. (2007). Evolution. Cold Spring Harbor, New York: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-684-9. LCCN 2007010767. OCLC 86090399.
- Coyne, Jerry A.; Orr, H. Allen (2004). Speciation. Sunderland, Massachusetts: Sinauer Associates. ISBN 978-0-87893-089-0. LCCN 2004009505. OCLC 55078441.
- Bergstrom, Carl T.; Dugatkin, Lee Alan (2012). Evolution (1st edición). New York: W.W. Norton & Company. ISBN 978-0-393-91341-5. LCCN 2011036572. OCLC 729341924.
- Gould, Stephen Jay (2002). The Structure of Evolutionary Theory. Cambridge, Massachusetts: Belknap Press of Harvard University Press. ISBN 978-0-674-00613-3. LCCN 2001043556. OCLC 47869352.
- Hall, Brian K.; Olson, Wendy, eds. (2003). Keywords and Concepts in Evolutionary Developmental Biology. Cambridge, Massachusetts: Harvard University Press. ISBN 978-0-674-00904-2. LCCN 2002192201. OCLC 50761342.
- Kauffman, Stuart A. (1993). The Origins of Order: Self-organization and Selection in Evolution. New York; Oxford: Oxford University Press. ISBN 978-0-19-507951-7. LCCN 91011148. OCLC 895048122.
- Maynard Smith, John; Szathmáry, Eörs (1995). The Major Transitions in Evolution. Oxford; New York: W.H. Freeman Spektrum. ISBN 978-0-7167-4525-9. LCCN 94026965. OCLC 30894392. Parámetro desconocido
|title-link=ignorado (ayuda) - Mayr, Ernst (2001). What Evolution Is. New York: Basic Books. ISBN 978-0-465-04426-9. LCCN 2001036562. OCLC 47443814.
- Minelli, Alessandro (2009). Forms of Becoming: The Evolutionary Biology of Development. Translation by Mark Epstein. Princeton, New Jersey; Oxford: Princeton University Press. ISBN 978-0-691-13568-7. LCCN 2008028825. OCLC 233030259.
External links
- General information
- Plantilla:In Our Time
- «Evolution». New Scientist. ISSN 0262-4079. Consultado el 30 de mayo de 2011.
- «Evolution Resources from the National Academies». Washington, DC: National Academy of Sciences. Consultado el 30 de mayo de 2011.
- «Understanding Evolution: your one-stop resource for information on evolution». Berkeley, California: University of California, Berkeley. Consultado el 30 de mayo de 2011.
- «Evolution of Evolution – 150 Years of Darwin's 'On the Origin of Species'». Arlington County, Virginia: National Science Foundation. Consultado el 30 de mayo de 2011.
- «Human Evolution Timeline Interactive». Smithsonian Institution, National Museum of Natural History. 28 de enero de 2010. Consultado el 14 de julio de 2018. Adobe Flash required.
- Experiments concerning the process of biological evolution
- Lenski, Richard E. «Experimental Evolution». East Lansing, Michigan: Michigan State University. Consultado el 31 de julio de 2013.
- Chastain, Erick; Livnat, Adi; Papadimitriou, Christos; Vazirani, Umesh (July 22, 2014). «Algorithms, games, and evolution». Proc. Natl. Acad. Sci. U.S.A. 111 (29): 10620-10623. Bibcode:2014PNAS..11110620C. ISSN 0027-8424. PMC 4115542. PMID 24979793. doi:10.1073/pnas.1406556111. Consultado el 3 de enero de 2015.
- Online lectures
- «Evolution Matters Lecture Series». Harvard Online Learning Portal. Cambridge, Massachusetts: Harvard University. Archivado desde el original el 18 de diciembre de 2017. Consultado el 15 de julio de 2018.
- Stearns, Stephen C. «EEB 122: Principles of Evolution, Ecology and Behavior». Open Yale Courses. New Haven, Connecticut: Yale University. Archivado desde el original el 1 de diciembre de 2017. Consultado el 14 de julio de 2018.
Plantilla:Evolution Plantilla:Big History Plantilla:Breakthrough of the Year Plantilla:Featured article
 Portal:Evolutionary biology. Contenido relacionado con Paleontology.
Portal:Evolutionary biology. Contenido relacionado con Paleontology.
Plantilla:Evolución biológica La evolución biológica es el conjunto de cambios en caracteres fenotípicos y genéticos de poblaciones biológicas a través de generaciones. Dicho proceso ha originado la diversidad de formas de vida que existen sobre la Tierra a partir de un antepasado común.[1][2] Los procesos evolutivos han producido la biodiversidad en cada nivel de la organización biológica, incluyendo los de especie, población, organismos individuales y molecular (evolución molecular).[3] Toda la vida en la Tierra procede de un último antepasado común universal que existió entre hace 3800 y 3500 millones de años.[4][5]
La palabra evolución se utiliza para describir los cambios y fue aplicada por primera vez en el siglo XVIII por un biólogo suizo, Charles Bonnet, en su obra Consideration sur les corps organisés.[6][7] No obstante, el concepto de que la vida en la Tierra evolucionó a partir de un ancestro común ya había sido formulado por varios filósofos griegos,[8] y la hipótesis de que las especies se transforman continuamente fue postulada por numerosos científicos de los siglos XVIII y XIX, a los cuales Charles Darwin citó en el primer capítulo de su libro El origen de las especies.[9] Sin embargo, fue el propio Darwin en 1859,[10] quien sintetizó un cuerpo coherente de observaciones y profundizó el mecanismo de cambio llamado selección natural, lo que consolidó el concepto de la evolución biológica hasta convertirlo en una verdadera teoría científica.[2] Anteriormente, el concepto de selección natural ya había sido aportado por Al-Jahiz en el año 77, en su Libro de los animales, con postulados claves sobre la lucha por la sobrevivencia de las especies, y la herencia de características exitosas mediante reproducción.[11]
La evolución como propiedad inherente a los seres vivos no es materia de debate en la comunidad científica dedicada a su estudio;[2] sin embargo, los mecanismos que explican la transformación y diversificación de las especies se hallan bajo intensa y continua investigación científica.
Dos naturalistas, Charles Darwin y Alfred Russel Wallace, propusieron en forma independiente en 1858 que la selección natural era el mecanismo básico responsable del origen de nuevas variantes genotípicas y en última instancia, de nuevas especies.[12][13]
Actualmente, la teoría de la evolución combina las propuestas de Darwin y Wallace con las leyes de Mendel y otros avances posteriores en la genética; por eso se la denomina síntesis moderna o «teoría sintética».[2] Según esta teoría, la evolución se define como un cambio en la frecuencia de los alelos de una población a lo largo de las generaciones. Este cambio puede ser causado por diferentes mecanismos, tales como la selección natural, la deriva genética, la mutación y la migración o flujo genético. La teoría sintética recibe en la actualidad una aceptación general de la comunidad científica, aunque también algunas críticas. Los avances de otras disciplinas relacionadas, como la biología molecular, la genética del desarrollo o la paleontología han enriquecido la teoría sintética desde su formulación, en torno a 1940.[14] Actualmente siguen surgiendo hipótesis sobre los mecanismos del cambio evolutivo basadas en datos empíricos tomados de organismos vivos.[15][16]
La evolución como un hecho probado
Evidencia del proceso evolutivo
La evidencia del proceso evolutivo surge del conjunto de pruebas que los científicos han reunido para demostrar que la evolución es un proceso característico de la materia viva y que todos los organismos que viven en la Tierra descienden de un último antepasado común universal.[17] Las especies actuales son un estado en el proceso evolutivo y su riqueza relativa y niveles de complejidad biológica son el producto de una larga serie de eventos de especiación y de extinción.[18]
La existencia de un ancestro común puede deducirse a partir de unas características simples de los organismos. Primero, existe evidencia proveniente de la biogeografía: tanto Charles Darwin como Alfred Russell Wallace se percataron de que la distribución geográfica de especies diferentes depende de la distancia y el aislamiento de las áreas que ocupan, y no de condiciones ecológicas y climatológicas similares, como sería de esperar si las especies hubieran aparecido al mismo tiempo ya adaptadas a su medio ambiente. Posteriormente, el descubrimiento de la tectónica de placas fue muy importante para la teoría de la evolución, al proporcionar una explicación para las similitudes entre muchos grupos de especies en continentes que se encontraban unidos en el pasado.[19] Segundo, la diversidad de la vida sobre la Tierra no se resuelve en un conjunto de organismos completamente únicos, sino que los mismos comparten una gran cantidad de similitudes morfológicas. Así, cuando se comparan los órganos de los distintos seres vivos, se encuentran semejanzas en su constitución que señalan el parentesco que existe entre especies diferentes. Estas semejanzas y su origen permiten clasificar a los órganos en homólogos, si tienen un mismo origen embrionario y evolutivo, y análogos, si tienen diferente origen embrionario y evolutivo pero la misma función. Los estudios anatómicos han encontrado homología en muchas estructuras superficialmente tan diferentes como las espinas de los cactos y las trampas de varias plantas insectívoras que indican que son simplemente hojas que han experimentado modificaciones adaptativas.[20] Los procesos evolutivos explican asimismo la presencia de órganos vestigiales, que están reducidos y no tienen función aparente, pero que muestran claramente que derivan de órganos funcionales presentes en otras especies, tales como los huesos rudimentarios de las patas posteriores presentes en algunas serpientes.[2]
La embriología, a través de los estudios comparativos de las etapas embrionarias de distintas clases de animales, ofrece otro conjunto de indicios del proceso evolutivo. Se ha encontrado que en estas primeras etapas del desarrollo, muchos organismos muestran características comunes que sugieren la existencia de un patrón de desarrollo compartido entre ellas, lo que, a su vez, sugiere la existencia de un antepasado común. El hecho de que los embriones tempranos de vertebrados como los mamíferos y aves posean hendiduras branquiales, que luego desaparecen conforme avanza el desarrollo, puede explicarse si se se hallan emparentados con los peces.[21]
Otro grupo de pistas proviene del campo de la sistemática. Los organismos pueden ser clasificados usando las similitudes mencionadas en grupos anidados jerárquicamente, muy similares a un árbol genealógico.[22][10] Si bien las investigaciones modernas sugieren que, debido a la transferencia horizontal de genes, este árbol de la vida puede ser más complicado que lo que se pensaba, ya que muchos genes se han distribuido independientemente entre especies distantemente relacionadas.[23][24]
Las especies que han vivido en épocas remotas han dejado registros de su historia evolutiva. Los fósiles, conjuntamente con la anatomía comparada de los organismos actuales, constituyen la evidencia paleontológica del proceso evolutivo. Mediante la comparación de las anatomías de las especies modernas con las ya extintas, los paleontólogos pueden inferir los linajes a los que unas y otras pertenecen. Sin embargo, la investigación paleontológica para buscar conexiones evolutivas tiene ciertas limitaciones. De hecho, es útil solo en aquellos organismos que presentan partes del cuerpo duras, tales como caparazones, dientes o huesos. Más aún, ciertos otros organismos, como los procariotas ―las bacterias y arqueas― presentan una cantidad limitada de características comunes, por lo que sus fósiles no proveen información sobre sus ancestros.[25]
Un método más reciente para probar el proceso evolutivo es el estudio de las similitudes bioquímicas entre los organismos. Por ejemplo, todas las células utilizan el mismo conjunto básico de nucleótidos y aminoácidos.[26] El desarrollo de la genética molecular ha revelado que el registro evolutivo reside en el genoma de cada organismo y que es posible datar el momento de la divergencia de las especies a través del reloj molecular basado en las mutaciones acumuladas en el proceso de evolución molecular.[27] Por ejemplo, la comparación entre las secuencias del ADN del humano y del chimpancé ha confirmado la estrecha similitud entre las dos especies y ha ayudado a elucidar cuándo existió el ancestro común de ambas.[28]
El origen de la vida
El origen de la vida, aunque atañe al estudio de los seres vivos, es un tema que no es abordado por la teoría de la evolución; pues esta última solo se ocupa del cambio en los seres vivos, y no del origen, cambios e interacciones de las moléculas orgánicas de las que estos proceden.[29]
No se sabe mucho sobre las etapas más tempranas y previas al desarrollo de la vida, y los intentos realizados para tratar de desvelar la historia más temprana del origen de la vida generalmente se enfocan en el comportamiento de las macromoléculas, debido a que el consenso científico actual es que la compleja bioquímica que constituye la vida provino de reacciones químicas simples, si bien persisten las controversias acerca de cómo ocurrieron las mismas.[30] Sin embargo, los científicos están de acuerdo en que todos los organismos existentes comparten ciertas características ―incluyendo la presencia de estructura celular y de código genético― que estarían relacionadas con el origen de la vida.[31]
Tampoco está claro cuáles fueron los primeros desarrollos de la vida (protobiontes), la estructura de los primeros seres vivos o la identidad y la naturaleza del último antepasado común universal.[32][33] Las bacterias, los primeros organismos que dejaron una huella en el registro fósil, son demasiado complejas para haber surgido directamente de los materiales no vivos.[34] La falta de indicios geoquímicos o fósiles de organismos anteriores ha dejado un amplio campo libre para las hipótesis. Aunque no hay consenso científico sobre cómo comenzó la vida, se acepta la existencia del último antepasado común universal porque sería prácticamente imposible que dos o más linajes separados pudieran haber desarrollado de manera independiente los muchos complejos mecanismos bioquímicos comunes a todos los organismos vivos.[26][35]
Se ha propuesto que el inicio de la vida pueden haber sido moléculas autorreplicantes como el ARN,[36] o ensamblajes de células simples denominadas nanocélulas.[37] Una hipótesis alternativa es la del comienzo de la vida en otras partes del Universo, desde donde habría llegado a la Tierra en cometas o meteoritos, en el proceso denominado panspermia.[38]
La evolución de la vida en la Tierra

Detallados estudios químicos basados en isótopos de carbono de rocas del eón Arcaico sugieren que las primeras formas de vida emergieron en la Tierra probablemente hace más de 3800 millones de años, en la era Eoarcaica, y hay claros indicios geoquímicos ―tales como la presencia en rocas antiguas de isótopos de azufre producidos por la reducción microbiana de sulfatos― que indican su presencia en la era Paleoarcaica, hace 3470 millones de años.[40] Los estromatolitos ―capas de roca producidas por comunidades de microorganismos― más antiguos se reconocen en estratos de 3450 millones de años, mientras que los microfósiles filiformes más antiguos, morfológicamente similares a cianobacterias, se encuentran en estratos de sílex de 3450 millones de años hallados en Australia.[41][42][43]
Asimismo, los fósiles moleculares derivados de los lípidos de la membrana plasmática y del resto de la célula ―denominados «biomarcadores»― confirman que ciertos organismos similares a cianobacterias habitaron los océanos arcaicos hace más de 2700 millones de años. Estos microbios fotoautótrofos liberaban oxígeno, que comenzó a acumularse en la atmósfera hace aproximadamente 2200 millones de años y transformó definitivamente la composición de esta.[44][43] La aparición de una atmósfera rica en oxígeno tras el surgimiento de organismos fotosintéticos puede también rastrearse por los depósitos laminares de hierro y las bandas rojas de los óxidos de hierro posteriores. La abundancia de oxígeno posibilitó el desarrollo de la respiración celular aeróbica, que emergió hace aproximadamente 2000 millones de años.[45]
Desde la formación de estas primeras formas de vida compleja, los procariotas, hace 3000 a 4000 millones de años,[46][47] pasaron miles de millones de años sin ningún cambio significativo en la morfología u organización celular en estos organismos,[48] hasta que surgieron los eucariotas a partir de la integración de bacterias en otras células procariotas, formando una asociación cooperativa entredenominada endosimbiosis.[49] Las bacterias incorporadas a sus células hospedantes y estas iniciaron un proceso de coevolución, por el cual las bacterias originaron las mitocondrias o hidrogenosomas.[50]
Un segundo evento independiente de endosimbiosis llevó a la formación de los cloroplastos a partir de cianobacteria alojadas en las antepasadas de las algas y plantas. La evidencia tanto bioquímica como paleontológica indica que las primeras células eucarióticas surgieron entre hace unos 2000 a 1500 millones de años, aunque probablemente los atributos clave de su fisiología evolucionaron antes.[51][43][52]
La historia de la vida sobre la Tierra fue la de los eucariotas unicelulares, procariotas y arqueas hasta hace aproximadamente 610 millones de años, momento en el que los primeros organismos multicelulares aparecieron en los océanos en el período denominado Ediacárico.[46][53][54]
Es posible que algunos organismos ediacáricos estuvieran estrechamente relacionados con grupos que predominaron más adelante, como los poríferos o los cnidarios.[55] No obstante, debido a la dificultad a la hora de deducir las relaciones evolutivas en estos organismos, algunos paleontólogos han sugerido que la biota de Ediacara representa una rama completamente extinta, un «experimento fallido» de la vida multicelular, y que la vida multicelular posterior volvió a evolucionar más tarde a partir de organismos unicelulares no relacionados.[54] En cualquier caso, la evolución de los organismos pluricelulares ocurrió en múltiples eventos independientes, en organismos tan diversos como las esponjas, algas pardas, cianobacterias, hongos mucosos y mixobacterias.[56]
Poco después de la aparición de los primeros organismos multicelulares, una gran diversidad de formas de vida apareció en un período de diez millones de años, en un evento denominado explosión cámbrica, un lapso breve en términos geológicos pero que implicó una diversificación animal sin paralelo documentada en los fósiles encontrados en los sedimentos de Burgess Shale, Canadá. Durante este período, la mayoría de los filos animales actuales aparecieron en los registros fósiles, como así también una gran cantidad de linajes únicos que ulteriormente se extinguieron. La mayoría de los planes corporales de los animales modernos se originaron durante este período.[57]
Entre los posibles desencadenantes de la explosión cámbrica se incluye la acumulación de oxígeno en la atmósfera debido a la fotosíntesis.[58][59]
Aproximadamente hace 500 millones de años, las plantas y los hongos colonizaron la tierra y les siguieron rápidamente los artrópodos y otros animales.[60]
Los anfibios aparecieron en la historia de la Tierra hace alrededor de 300 millones de años, seguidos por los primeros amniotas, y luego por los mamíferos, hace unos 200 millones de años, y las aves, hace 100 millones de años. Sin embargo, los organismos microscópicos, similares a aquellos que evolucionaron tempranamente, continúan siendo la forma de vida predominante en la Tierra, ya que la mayor parte de las especies y la biomasa terrestre está constituida por procariotas.[42]
Teorías científicas acerca de la evolución
Historia del pensamiento evolucionista

Varios filósofos griegos de la antigüedad contemplaron la posibilidad de cambios en los organismos vivos a través del tiempo. Anaximandro (ca. 610-546 a. C.) sugirió que los primeros animales vivían en el agua y que dieron origen a los animales terrestres.[62] Empédocles (ca. 490-430 a. C.) escribió que los primeros seres vivos provenían de la tierra y las especies surgieron mediante procesos naturales sin un organizador o una causa final.[63] Las obras de Aristóteles (384-322 a. C.), el primer naturalista cuyo trabajo se ha conservado con detalle, contienen observaciones e interpretaciones muy sagaces, si bien mezcladas con mitos y errores diversos que reflejan el estado del conocimiento en su época;[64] es notable su esfuerzo en exponer las relaciones existentes entre los seres vivos como una scala naturae ―tal como se describe en Historia animalium― en la que los organismos se clasifican de acuerdo con una estructura jerárquica, «escalera de la vida» o «cadena del Ser», ordenados según la complejidad de sus estructuras y funciones, con los organismos que muestran una mayor vitalidad y capacidad de movimiento descritos como «organismos superiores».[65][66]
Algunos antiguos pensadores chinos expresaron igualmente la idea de que las especies biológicas cambian. Zhuangzi, un filósofo taoísta que vivió alrededor del siglo IV a. C., mencionó que las formas de vida tienen una habilidad innata o el poder (hua 化) para transformarse y adaptarse a su entorno.[67]
Según Joseph Needham, el taoísmo niega explícitamente la inmutabilidad de las especies biológicas y los filósofos taoístas especularon que las mismas desarrollaron diferentes atributos en respuesta a distintos entornos. De hecho, el taoísmo se refiere a los seres humanos, la naturaleza y el cielo como existentes en un estado de «constante transformación», en contraste con la visión más estática de la naturaleza típica del pensamiento occidental.[68]


Si bien la idea de la evolución biológica ha existido desde épocas remotas y en diferentes culturas —por ejemplo, en la sociedad musulmana la esbozaron en el siglo IX Al-Jahiz y en el siglo XIII Nasir al-Din al-Tusi respectivamente—,[69] la teoría moderna no se estableció hasta llegados los siglos XVIII y XIX, con la contribución de científicos como Christian Pander, Jean-Baptiste Lamarck y Charles Darwin.[70]
En el siglo XVIII, la oposición entre fijismo y transformismo fue ambigua. Algunos autores, por ejemplo, admitieron la transformación de las especies a nivel de géneros, pero negaban la posibilidad de que cambiaran de un género a otro. Otros naturalistas hablaban de «progresión» en la naturaleza orgánica, pero es muy difícil determinar si con ello hacían referencia a una transformación real de las especies o se trataba, simplemente, de una modulación de la clásica idea de la scala naturae.[71]
Jean-Baptiste Lamarck (1744-1829) formuló la primera teoría de la evolución y propuso que los organismos, es toda su variedad, habían evolucionado desde formas simples creadas por Dios y postuló que los responsables de esa evolución habían sido los propios organismos por su capacidad de adaptarse al ambiente: los cambios en ese ambiente generaban nuevas necesidades en los organismos y esas nuevas necesidades conllevarían una modificación de los mismos que sería heredable. Se apoyó para la formulación de su teoría en la existencia de restos de formas intermedias extintas.[8] Con esta teoría Lamarck se enfrentó a la creencia general por la que todas las especies habían sido creadas y permanecían inmutables desde su creación y también se opuso al influyente Georges Cuvier (1769-1832) que justificaba la desaparición de las especies no porque fueran formas intermedias entre las primigenias y las actuales, sino porque se trataba de formas de vida diferentes, extinguidas en los diferentes cataclismos geológicos sufridos por la Tierra.[72][73]

No fue sino hasta la publicación de El origen de las especies de Charles Darwin cuando el hecho de la evolución comenzó a ser ampliamente aceptado. Una carta de Alfred Russel Wallace, en la cual revelaba su propio descubrimiento de la selección natural, impulsó a Darwin a publicar su trabajo en evolución. Por lo tanto, a veces se les concede a ambos el crédito por la teoría de la evolución, llamándola también teoría de Darwin-Wallace.[66]
Un debate particularmente interesante en el campo evolutivo fue el que sostuvieron los naturalistas franceses Georges Cuvier y Étienne Geoffroy Saint-Hilaire en el año 1830. Ambos discrepaban en los criterios fundamentales para describir las relaciones entre los seres vivos; mientras Cuvier se basaba en características anatómicas funcionales, Geoffroy daba más importancia a la morfología. La distinción entre función y forma trajo consigo el desarrollo de dos campos de investigación, conocidos respectivamente como anatomía funcional y anatomía trascendental. Gracias al trabajo del anatomista británico Richard Owen, los dos puntos de vista empezaron a reconciliarse, proceso completado en la teoría de la evolución de Darwin.[74][75]
A pesar de que la teoría de Darwin sacudió profundamente la opinión científica con respecto al desarrollo de la vida, llegando incluso tener influencias sociales, no pudo explicar la fuente de variación existente entre las especies, y además la propuesta de Darwin de la existencia de un mecanismo hereditario (pangénesis) no satisfizo a la mayoría de los biólogos. No fue recién hasta fines del siglo XIX y comienzos del XX, que estos mecanismos pudieron establecerse.[76]
Cuando alrededor del 1900 se «redescubrió» el trabajo que Gregor Mendel llevó a cabo a fines del siglo XIX sobre la naturaleza de la herencia, se estableció una discusión entre los mendelianos (Charles Benedict Davenport) y los biométricos (Walter Frank Raphael Weldon y Karl Pearson), quienes insistían en que la mayoría de los caminos importantes para la evolución debían mostrar una variación continua que no era explicable a través del análisis mendeliano. Finalmente, los dos modelos fueron conciliados y fusionados, principalmente a través del trabajo del biólogo y estadístico Ronald Fisher.[72] Este enfoque combinado, que aplica un modelo estadístico riguroso a las teorías de Mendel de la herencia vía genes, se dio a conocer en los años 1930 y 1940 y se conoce como la teoría sintética de la evolución.[77]
En los años de la década de 1940, siguiendo el experimento de Griffith, Avery, MacLeod y McCarty lograron identificar de forma definitiva al ácido desoxirribonucléico (ADN) como el «principio transformador» responsable de la transmisión de la información genética.[78] En 1953, Francis Crick y James Watson publicaron su famoso trabajo sobre la estructura del ADN, basado en la investigación de Rosalind Franklin y Maurice Wilkins. Estos avances iniciaron la era de la biología molecular y condujeron a la interpretación de la evolución como un proceso molecular.
A mediados de la década de 1970, Motoo Kimura formuló la teoría neutralista de la evolución molecular, estableciendo de manera firme la importancia de la deriva génica como el principal mecanismo de la evolución. Hasta la fecha continúan los debates en esta área de investigación. Uno de los más importantes es acerca de la teoría del equilibrio puntuado, una teoría propuesta por Niles Eldredge y Stephen Jay Gould para explicar la escasez de formas transicionales entre especies.[79]
Darwinismo

Fotografía de Julia Margaret Cameron.
Esta etapa del pensamiento evolutivo se inicia con la publicación en agosto de 1858 de un trabajo conjunto de Darwin y Wallace,[12] al que siguió en 1859 el libro de Darwin El origen de las especies, en el que que designa el principio de la selección natural como el principal motor del proceso evolutivo y acepta la tesis lamarckiana de la herencia de los caracteres adquiridos como una fuente de variabilidad biológica; por este motivo, aunque Wallace rechazaba el lamarckismo, se acepta la denominación de «Lamarck-Darwin-Wallace» para referirse a este estadio.[2]
El trabajo de 1858 contenía «una muy ingeniosa teoría para explicar la aparición y perpetuación de las variedades y de las formas específicas en nuestro planeta» según palabras del prólogo escrito por Charles Lyell (1797-1895) y William Jackson Hooker (1785-1865). De hecho, este trabajo presentó por primera vez la hipótesis de la selección natural. Esta hipótesis contenía cinco afirmaciones fundamentales: (1) todos los organismos producen más descendencia de la que el ambiente puede sostener; (2) existe una abundante variabilidad intraespecífica para la mayoría de los caracteres; (3) la competencia por los recursos limitados lleva a la lucha «por la vida» (según Darwin) o «por la existencia» (según Wallace); (4) se produce descendencia con modificaciones heredables; y (5) como resultado, se originan nuevas especies.[80] Lyell y Hooker reconocieron a Darwin como el primero en formular las ideas presentadas en el trabajo conjunto, adjuntando como prueba un ensayo de Darwin de 1944 y una carta que envió a Asa Gray en 1857, ambos publicados junto con un artículo de Wallace. Un análisis comparativo detallado de las publicaciones de Darwin y Wallace revela que las contribuciones de este último fueron más importantes de lo que usualmente se suele reconocer, [81][82]
Treinta años más tarde, el codescubridor de la selección natural publicó una serie de conferencias bajo el título de «Darwinism» que tratan los mismos temas que ya había tratado Darwin, pero a la luz de los hechos y de los datos que eran desconocidos en tiempos de Darwin, quien falleció en 1882.[83] Sin embargo, en su Origen de las especies', Darwin fue el primero en resumir un conjunto coherente de observaciones que solidificó el concepto de la evolución de la vida en una verdadera teoría científica ―es decir, en un sistema de hipótesis―.[84]
La lista de las propuestas de Darwin presentadas en esta obra se expone a continuación:[2]
1. Los actos sobrenaturales del Creador son incompatibles con los hechos empíricos de la naturaleza.2. Toda la vida evolucionó a partir de una o de pocas formas simples de organismos.
3. Las especies evolucionan a partir de variedades preexistentes por medio de la selección natural.
4. El nacimiento de una especie es gradual y de larga duración.
5. Los taxones superiores (géneros, familias, etc.) evolucionan a través de los mismos mecanismos que los responsables del origen de las especies.
6. Cuanto mayor es la similitud entre los taxones, más estrechamente relacionados se hallan entre sí y más corto es el tiempo de su divergencia desde el último ancestro común.
7. La extinción es principalmente el resultado de la competencia interespecífica.
8. El registro geológico es incompleto: la ausencia de formas de transición entre las especies y taxones de mayor rango se debe a las lagunas en el conocimiento actual.
Neodarwinismo
Neodarwinismo es un término acuñado en 1895 por el naturalista y psicólogo inglés George John Romanes (1848-1894) en su obra Darwin and after Darwin.[85] El término describe un estado en el desarrollo de la teoría evolutiva que se remonta al citólogo y zoólogo germano August Weismann (1834-1914), quien en 1892 aportó evidencia experimental en contra de la herencia lamarckiana y postuló que el desarrollo del organismo no influye en el material hereditario y que la reproducción sexual en cada generación introduce nuevas variaciones en la población de individuos. La selección natural, entonces, puede actuar sobre la variabilidad de la población y determina el curso del cambio evolutivo.[86] El neodarwinismo enriqueció el concepto original de Darwin, al destacar el origen de las variaciones entre individuos y excluir la herencia lamarckiana como una explicación viable del mecanismo de herencia. Wallace, quien popularizó el término «darwinismo» en 1889,[83] incorporó plenamente las nuevas conclusiones de Weismann y fue, por consiguiente, uno de los primeros proponentes del neodarwinismo.[2]
Síntesis evolutiva moderna
Este sistema de hipótesis del proceso evolutivo se originó entre 1937 y 1950.[87] En contraste con el neodarwinismo de Weismann y Wallace, que daba primacía a la selección natural y postulaba la genética mendeliana como el mecanismo de transmisión de caracteres entre generaciones, la teoría sintética incorporó datos de campos diversos de la biología, como la genética molecular, la sistemática y la paleontología e introdujo nuevos mecanismos para la evolución. Por estas razones, se trata de diferentes teorías aunque a veces se usen los términos indistintamente.[88][89][90]
De acuerdo a la gran mayoría de los historiadores de la Biología, los conceptos básicos de la teoría sintética están basados esencialmente en el contenido de seis libros, cuyos autores fueron: el naturalista y genetista ruso americano Theodosius Dobzhansky (1900-1975); el naturalista y taxónomo alemán americano Ernst Mayr (1904-2005); el zoólogo británico Julian Huxley (1887-1975); el paleontólogo americano George G. Simpson (1902-1984); el zoólogo germano Bernhard Rensch (1900-1990) y el botánico estadounidense George Ledyard Stebbins (1906-2000).[89]
Los términos «síntesis evolutiva» y «teoría sintética» fueron acuñados por Julian Huxley en su libro Evolución: la síntesis moderna (1942), en el que también introdujo el término Biología evolutiva en vez de la frase «estudio de la evolución».[91][92] De hecho Huxley fue el primero en señalar que la evolución «debía ser considerada el problema más central y el más importante de la biología y cuya explicación debía ser abordada mediante hechos y métodos de cada rama de la ciencia, desde la ecología, la genética, la paleontología, la embriología, la sistemática hasta la anatomía comparada y la distribución geográfica, sin olvidar los de otras disciplinas como la geología, la geografía y las matemáticas».[93]
La llamada «síntesis evolutiva moderna» es una robusta teoría que actualmente proporciona explicaciones y modelos matemáticos de los mecanismos generales de la evolución o los fenómenos evolutivos, como la adaptación o la especiación. Como cualquier teoría científica, sus hipótesis están sujetas a constante crítica y comprobación experimental.
- Los entes dónde actúa la evolución son las poblaciones de organismos y no los individuos. Theodosius Dobzhansky, uno de los fundadores de la síntesis moderna, lo expresó la evolución del siguiente modo: «La evolución es un cambio en la composición genética de las poblaciones. El estudio de los mecanismos evolutivos corresponde a la genética poblacional».[94] Esta idea llevó al «concepto biológico de especie» desarrollado por Mayr en 1942: una comunidad de poblaciones que se entrecruzan y que está reproductivamente aislada de otras comunidades.[95][96]
- La variabilidad fenotípica y genética en las poblaciones de plantas y de animales se produce por la recombinación genética ―reorganización de segmentos de cromosomas durante la reproducción sexual— y por las mutaciones aleatorias. La cantidad de variación genética que una población de organismos con reproducción sexual puede producir es enorme. Considérese la posibilidad de un solo individuo con un número «N» de genes, cada uno con solo dos alelos. Este individuo puede producir 2N espermatozoides u óvulos genéticamente diferentes. Debido a que la reproducción sexual implica dos progenitores, cada descendiente puede, por tanto, poseer una de las 4N combinaciones diferentes de genotipos. Así, si cada progenitor tiene 150 genes con dos alelos cada uno ―una subestimación del genoma humano―, cada uno de los padres puede dar lugar a más de 1045 gametos genéticamente diferentes y más de 1090 descendientes genéticamente diferentes.
- La selección natural es la fuerza más importante que modela el curso de la evolución fenotípica. En ambientes cambiantes, la selección direccional es de especial importancia, porque produce un cambio en la media de la población hacia un fenotipo nuevo que se adapta mejor a las condiciones ambientales alteradas. Además, en las poblaciones pequeñas, la deriva génica aleatoria ―la pérdida de genes del acervo genético― puede ser significativa.
- La especiación puede definirse como «un paso en el proceso evolutivo (en el que) las formas... se vuelven incapaces de hibridarse».[97] Se han descubierto y estudiado en profundidad diversos mecanismos de aislamiento reproductivo. Se cree que el aislamiento geográfico de la población fundadora es responsable del origen de las nuevas especies en las islas y otros hábitats aislados y es probable que la especiación alopátrica ―evolución divergente de poblaciones que están geográficamente aisladas unas de otras― sea el mecanismo de especiación predominante en el origen de muchas especies de animales.[98] Sin embargo, la especiación simpátrica ―la aparición de nuevos especies sin aislamiento geográfico― también está documentada en muchos taxones, sobre todo en las plantas vasculares, los insectos, los peces y las aves.[99]
- Las transiciones evolutivas en estas poblaciones suelen ser graduales, es decir, las nuevas especies evolucionan a partir de las variedades preexistentes por medio de procesos lentos y en cada etapa se mantiene su adaptación específica.
- La macroevolución ―la evolución filogenética por encima del nivel de especie o la aparición de taxones superiores― es un proceso gradual, paso a paso, que no es más que la extrapolación de la microevolución ―el origen de las razas y variedades, y de las especies―.
Síntesis evolutiva moderna
En la época de Darwin los científicos no conocían cómo se heredaban las características. Posteriormente se descubrió la relación de la mayoría de las características hereditarias con entidades persistentes llamadas genes, fragmentos de las moléculas lineales de ácido desoxirribonucleico (ADN) del núcleo de las células. El ADN varía entre los miembros de una misma especie y también sufre cambios, mutaciones, o reorganizaciones por recombinación genética.
Variabilidad
El fenotipo de un organismo individual es el resultado de su genotipo y la influencia del ambiente en el que vive y ha vivido. Una parte sustancial de la variación entre fenotipos dentro de una población está causada por las diferencias entre sus genotipos.[100] La síntesis evolutiva moderna define la evolución como el cambio de esa variación genética a través del tiempo. La frecuencia de cada alelo fluctúa, siendo más o menos prevalente en relación con otras formas alternativas del mismo gen. Las fuerzas evolutivas actúan mediante la dirección de esos cambios en las frecuencias alélicas en uno u otro sentido. La variación de una población para un gen dado desaparece cuando se produce la fijación de un alelo que ha reemplazado enteramente a todas las otras formas alternativas de ese mismo gen.[101]
La variabilidad surge en las poblaciones naturales por mutaciones en el material genético, migraciones entre poblaciones (flujo genético) y por la reorganización de los genes a través de la reproducción sexual. La variabilidad también puede provenir del intercambio de genes entre diferentes especies, por ejemplo a través de la transferencia horizontal de genes en las bacterias o la hibridación interespecífica en las plantas.[102] A pesar de la constante introducción de variantes nuevas a través de estos procesos, la mayor parte del genoma de una especie es idéntica en todos los individuos que pertenecen a la misma.[103] Sin embargo, aun pequeños cambios en el genotipo pueden llevar a modificaciones sustanciales del fenotipo. Así, los chimpancés y los seres humanos, por ejemplo, solo difieren en aproximadamente el 5 % de sus genomas.[104]
Mutación
Darwin no conocía la fuente de las variaciones en los organismos individuales, pero observó que parecían ocurrir aleatoriamente. En trabajos posteriores se atribuyó la mayor parte de estas variaciones a las mutaciones. La mutación es un cambio permanente y transmisible en el material genético ―usualmente el ADN o el ARN― de una célula, producido por «errores de copia» en el material genético durante la división celular o por la exposición a radiación, sustancias químicas o la acción de virus. Las mutaciones aleatorias ocurren constantemente en el genoma de todos los organismos, creando nueva variabilidad genética.[105][106][107] Las mutaciones pueden no tener efecto alguno sobre el fenotipo del organismo, o ser perjudiciales o beneficiosas. A modo de ejemplo, los estudios realizados sobre la mosca de la fruta (Drosophila melanogaster), sugieren que, si una mutación determina un cambio en la proteína producida por un gen, ese cambio será perjudicial en el 70 % de los casos y neutro o levemente beneficioso en los restantes.[108]
La frecuencia de nuevas mutaciones en un gen o secuencia de ADN en cada generación se denomina tasa de mutación. En escenarios de rápido cambio ambiental, una tasa de mutación alta aumenta la probabilidad de que algunos individuos tengan una variante genética adecuada para adaptarse y sobrevivir; por otro lado, también aumenta el número de mutaciones perjudiciales o deletéreas que disminuyen la adaptación de los individuos y eleva la probabilidad de extinción de la especie.[109] Debido a los efectos contrapuestos que las mutaciones pueden tener sobre los organismos,[105] la tasa de mutación óptima para una población es una compensación entre costos y beneficios,[110] que depende de la especie y refleja la historia evolutiva como respuesta a los retos impuestos por el ambiente.[109] Los virus, por ejemplo, presentan una alta tasa de mutación,[111] lo que supone una ventaja adaptativa ya que deben evolucionar rápida y constantemente para sortear a los sistemas inmunes de los organismos que afectan.[112]
La duplicación génica introduce en el genoma copias extras de un gen y, de ese modo, proporciona el material de base para que las nuevas copias inicien su propio camino evolutivo.[113][114][115] Si el gen inicial sigue funcionando normalmente, sus copias pueden adquirir nuevas mutaciones sin perjuicio para el organismo que los alberga y llegar con el tiempo a adoptar nuevas funciones.[116][117] Por ejemplo, en los seres humanos son necesarios cuatro genes para construir las estructuras necesarias para detectar la luz: tres para la visión de los colores y uno para la visión nocturna. Los cuatro genes han evolucionado a partir de un solo gen ancestral por duplicación y posterior divergencia.[118] Otros tipos de mutación pueden ocasionalmente crear nuevos genes a partir del denominado ADN no codificante.[119][120] También pueden surgir nuevos genes con diferentes funciones a partir de fragmentos de genes duplicados que se recombinan para formar nuevas secuencias de ADN.[121][122][123]
Las mutaciones cromosómicas ―también denominadas, aberraciones cromosómicas― son una fuente adicional de variabilidad hereditaria. Así, las translocaciones, inversiones, deleciones, translocaciones robertsonianas y duplicaciones, usualmente ocasionan variantes fenotípicas que se transmiten a la descendencia. Por ejemplo, en el género Homo tuvo lugar una fusión cromosómica que dio lugar al cromosoma 2 de los seres humanos, mientras que otros simios conservan 24 pares de cromosomas.[124] No obstante las consecuencias fenotípicas que pueden tener tales mutaciones cromosómicas, su mayor importancia evolutiva reside en acelerar la divergencia de las poblaciones que presentan diferentes configuraciones cromosómica: el flujo genético entre ellas se reduce severamente debido a la esterilidad o semiesterilidad de los individuos heterocigóticos. De este modo, las mutaciones cromosómicas actúan como mecanismos de aislamiento reproductivo que conducen a que las diferentes poblaciones mantengan su identidad como especies a través del tiempo.[125]
Los fragmentos de ADN que pueden cambiar de posición en los cromosomas, tales como los transposones, constituyen una importante fracción del material genético de plantas y animales y pueden haber desempeñado un papel destacado en su evolución.[126] Al insertarse en o escindirse de otras partes del genoma estas secuencias pueden activar, inhibir, eliminar o mutar otros genes y, por ende, crear nueva variabilidad genética.[106] Asimismo, ciertas de estas secuencias se repiten miles o millones de veces en el genoma y muchas de ellas han adoptado funciones importantes, como por ejemplo, la regulación de la expresión genética.[127]
Recombinación genética
La recombinación genética es el proceso mediante el cual la información genética se redistribuye por transposición de fragmentos de ADN entre dos cromosomas durante la meiosis ―y más raramente en la mitosis―. Los efectos son similares a los de las mutaciones, es decir, si los cambios no son deletéreos se transmiten a la descendencia y contribuyen a incrementar la diversidad dentro de cada especie.
En los organismos asexuales, los genes se heredan en conjunto, o ligados, ya que no se mezclan con los de otros organismos durante los ciclos de recombinación que usualmente se producen durante la reproducción sexual. En contraste, los descendientes de los organismos que se reproducen sexualmente contienen una mezcla aleatoria de los cromosomas de sus progenitores, la cual se produce durante la recombinación meiótica y la posterior fecundación.[128] La recombinación no altera las frecuencias alélicas sino que modifica la asociación existente entre alelos pertenecientes a genes diferentes, produciendo descendientes con combinaciones únicas de genes.[129] La recombinación generalmente incrementa la variabilidad genética y puede aumentar también las tasas de evolución.[130][131][132] No obstante, la existencia de la reproducción asexual, tal como ocurre en las plantas apomícticas o en los animales partenogenéticos, indica que este modo de reproducción puede también ser ventajoso en ciertos ambientes.[133] Jens Christian Clausen fue uno de los primeros en reconocer formalmente que la apomixis, particularmente la apomixis facultativa, no necesariamente conduce a una pérdida de variabilidad genética y de potencial evolutivo. Utilizando una analogía entre el proceso adaptativo y la producción a gran escala de automóviles, Clausen arguyó que la combinación de sexualidad (que permite la producción de nuevos genotipos) y de apomixis (que permite la producción ilimitada de los genotipos más adaptados) potencia la capacidad de una especie para el cambio adaptativo.[134]
Aunque el proceso de recombinación posibilita que los genes agrupados en un cromosoma puedan heredarse independientemente, la tasa de recombinación es baja ―aproximadamente dos eventos por cromosoma y por generación―. Como resultado, los genes adyacentes tienden a heredarse conjuntamente, en un fenómeno que se denomina ligamiento.[135] Un grupo de alelos que usualmente se heredan conjuntamente por hallarse ligados se denomina haplotipo. Cuando uno de los alelos en haplotipo es altamente beneficioso, la selección natural puede conducir a un barrido selectivo que aumenta la proporción dentro de la población del resto de los alelos en el haplotipo; este efecto se denomina arrastre por ligamiento o «efecto autostop» (en inglés, genetic hitchhiking).[136]
Cuando los alelos no se recombinan, como es el caso en el cromosoma Y de los mamíferos o en los organismos asexuales, los genes con mutaciones deletéreas se acumulan, lo que se denomina trinquete de Muller (Muller ratchet en inglés).[137][138] De este modo, al romper los conjuntos de genes ligados, la reproducción sexual facilita la eliminación de las mutaciones perjudiciales y la retención de las beneficiosas,[139] además de la aparición de individuos con combinaciones genéticas nuevas y favorables. Estos beneficios deben contrarrestar otros efectos perjudiciales de la reproducción sexual, como la menor tasa reproductiva de las poblaciones de organismos sexuales y la separación de combinaciones favorables de genes. En todas las especies sexuales, y con la excepción de los organismos hermafroditas, cada población está constituida por individuos de dos sexos, de los cuales solo uno es capaz de engendrar la prole. En una especie asexual, en cambio, todos los miembros de la población tienen esa capacidad, lo que implica un crecimiento más rápido de la población asexual en cada generación. Otro costo del sexo es que los machos y las hembras deben buscarse entre ellos para aparearse y la selección sexual suele favorecer caracteres que reducen la aptitud de los individuos. Este costo del sexo fue expresado por primera vez en términos matemáticos por John Maynard Smith.[140][139] Las razones de la evolución de la reproducción sexual son todavía poco claras y es un interrogante que constituye un área activa de investigación en Biología evolutiva,[141][142] que ha inspirado ideas tales como la hipótesis de la Reina Roja.[143] El escritor científico Matt Ridley, que popularizó el término en su libro The Red Queen: Sex and the Evolution of Human Nature, sostiene que existe una carrera armamentista cíclica entre los organismos y sus parásitos y especula que el sexo sirve para preservar los genes circunstancialmente desfavorables pero potencialmente beneficiosos ante futuros cambios en las poblaciones de parásitos.
Genética de poblaciones
Como se ha descrito previamente, desde un punto de vista genético la evolución es un cambio intergeneracional en la frecuencia de los alelos dentro de una población que comparte un mismo patrimonio genético.[144] Una población es un grupo de individuos de la misma especie que comparten un ámbito geográfico. Por ejemplo, todas las polillas de una misma especie que viven en un bosque aislado forman una población. Un gen determinado dentro de la población puede presentar diversas formas alternativas, que son las responsables de la variación entre los diferentes fenotipos de los organismos. Un ejemplo puede ser un gen de la coloración en las polillas que tenga dos alelos: uno para color blanco y otro para color negro. El patrimonio o acervo genético es el conjunto completo de los alelos de una población, de forma que cada alelo aparece un número determinado de veces en un acervo génico. La fracción de genes del patrimonio genético que están representadas por un alelo determinado recibe el nombre de frecuencia alélica, por ejemplo, la fracción de polillas en la población que presentan el alelo para color negro. La evolución tiene lugar cuando hay cambios en la frecuencia alélica en una población de organismos que se reproducen entre ellos, por ejemplo, si el alelo para color negro se hace más común en una población de polillas.[145]
Para comprender los mecanismos que hacen que evolucione una población, es útil conocer las condiciones necesarias para que la población no evolucione. El principio de Hardy-Weinberg determina que la frecuencia de los alelos de una población suficientemente grande permanecerá constante solo si la única fuerza que actúa es la recombinación aleatoria de alelos durante la formación de los gametos y la posterior combinación de los mismos durante la fertilización.[146] En ese caso, la población se encuentra en equilibrio de Hardy-Weinberg y, por lo tanto, no evoluciona.[145]
Flujo genético

El flujo genético es el intercambio de genes entre poblaciones, usualmente de la misma especie. Como ejemplos de flujo génico se pueden mencionar el cruzamiento de individuos tras la inmigración de una población en el territorio de otra, o, en el caso de las plantas, el intercambio de polen entre poblaciones diferentes. La transferencia de genes entre especies conlleva la formación de híbridos o la transferencia horizontal de genes.[148]
La inmigración y la emigración de individuos en las poblaciones naturales pueden causar cambios en las frecuencias alélicas, como así también la introducción ―o desaparición― de variantes alélicas dentro de un acervo genético ya establecido. Las separaciones físicas en el tiempo, espacio o nichos ecológicos específicos que puede existir entre las poblaciones naturales restringen o imposibilitan el flujo génico. Además de estas restricciones al intercambio de genes entre poblaciones existen otros mecanismos de aislamiento reproductivo conformados por características, comportamientos y procesos fisiológicos que impiden que los miembros de dos especies diferentes puedan cruzarse o aparearse entre sí, producir descendencia o que esta sea viable o fértil. Estas barreras constituyen una fase indispensable en la formación de nuevas especies ya que mantienen las características propias de las mismas a través del tiempo al restringir o eliminar el flujo genético entre los individuos de diferentes poblaciones.[149][150][151][152]
Las especies distintas pueden ser interfértiles, dependiendo de cuánto han divergido desde su ancestro común; por ejemplo, la yegua y el asno pueden aparearse y producir la mula.[153] Tales híbridos son generalmente estériles debido a las diferencias cromosómicas entre las especies parentales, que impiden el emparejamiento correcto de los cromosomas durante la meiosis. En este caso, las especies estrechamente relacionadas pueden cruzarse con regularidad, pero la selección natural actúa contra los híbridos. Sin embargo, de vez en cuando se forman híbridos viables y fértiles que pueden presentar propiedades intermedias entre sus especies paternales o poseer un fenotipo totalmente nuevo.[154]
La importancia de la hibridación en la creación de nuevas especies de animales no está clara, aunque existen ejemplos bien documentados como el de la rana Hyla versicolor.[155][156] La hibridación es, sin embargo, un mecanismo importante de formación de nuevas especies en las plantas, ya que estas toleran la poliploidía ―la duplicación de todos los cromosomas de un organismo― más fácilmente que los animales;[157][158] la poliploidía restaura la fertilidad en los híbridos interespecíficos debido a que cada cromosoma es capaz de aparearse con un compañero idéntico durante la meiosis.[159][160]
Los mecanismos de la evolución
Hay dos mecanismos básicos de cambio evolutivo: la selección natural y la deriva genética. La selección natural favorece a los genes que mejoran la capacidad de supervivencia y reproducción del organismo. La deriva genética es el cambio en la frecuencia de los alelos, provocado por transmisión aleatoria de los genes de una generación a la siguiente. La importancia relativa de la selección natural y de la deriva genética en una población varía dependiendo de la fuerza de la selección y del tamaño poblacional efectivo, que es el número de ejemplares de esa población capaces de reproducirse.[161] La selección natural suele predominar en las poblaciones grandes, mientras que la deriva genética predomina en las pequeñas. El predominio de la deriva genética en poblaciones pequeñas puede llevar incluso a la fijación de mutaciones ligeramente deletéreas.[162] Como resultado de ello, los cambios en el tamaño de una población pueden influir significativamente en el curso de la evolución. Los denominados «cuellos de botella», o descensos drásticos temporarios del tamaño efectivo de la población, suponen una pérdida o erosión de la variabilidad genética y conllevan la formación de poblaciones genéticamente más uniformes. Los cuellos de botella pueden ser el resultado de catástrofes, variaciones en el medio ambiente o alteraciones en el flujo genético causadas por una migración reducida, la expansión a nuevos hábitats, o una subdivisión de la población.[161]
Selección natural




La selección natural es el proceso por el cual las mutaciones genéticas que mejoran la capacidad reproductiva se vuelven, y permanecen, cada vez más frecuentes en las sucesivas generaciones de una población. Se la califica a menudo de «mecanismo autoevidente», pues es la consecuencia necesaria de tres hechos simples: (a) dentro de las poblaciones de organismos hay variación heredable (b) los organismos producen más descendientes de los que pueden sobrevivir, y (c) tales descendientes tienen diferentes capacidades para sobrevivir y reproducirse.[163]
El concepto central de la selección natural es la aptitud biológica de un organismo.[164] La aptitud, ajuste o adecuación influye en la medida de la contribución genética de un organismo a la generación siguiente.[164] Sin embargo, la aptitud no es simplemente igual al número total de descendientes de un determinado organismo, ya que también cuantifica la proporción de generaciones posteriores que llevan los genes de ese organismo.[165] Por ejemplo, si un organismo puede sobrevivir y reproducirse pero sus descendientes son demasiado pequeños o enfermizos como para llegar a la edad reproductiva, la contribución genética de ese organismo a las futuras generaciones será muy baja y, por ende, su aptitud también lo es.[164]
Por consiguiente, si un alelo aumenta la aptitud más que otros, con cada generación el alelo será más común dentro de la población. Se dice que tales rasgos son «seleccionados favorablemente». Una mejora de la supervivencia o una mayor fecundidad son ejemplos de rasgos que pueden aumentar la aptitud. En cambio, la menor aptitud causada por un alelo menos beneficioso o deletéreo hace que este sea cada vez más raro en la población y sufra una «selección negativa».[166] Hay que subrayar que la aptitud de un alelo no es una característica fija: si el ambiente cambia, los rasgos que antes eran neutros o nocivos pueden ser beneficiosos y viceversa.[167] Por ejemplo, la polilla Biston betularia presenta dos colores, uno claro denominado forma typica y otro oscuro llamado forma carbonaria. La forma typica, como su nombre indica, es la más frecuente en esta especie. No obstante, durante la revolución industrial en el Reino Unido los troncos de muchos árboles sobre los que se posaban las polillas se ennegrecieron por el hollín, lo que les daba las polillas de color oscuro una mayor oportunidad de sobrevivir y producir más descendientes al pasar más fácilmente desapercibidas para los depredadores. Sólo cincuenta años después de que se descubriera la primera polilla melánica, casi la totalidad de las polillas del área industrial de Manchester eran oscuras. Este proceso se revirtió a causa de la «Ley del aire limpio» (Clean Air Act) de 1956, que redujo la polución industrial. Al aclararse el color de los troncos, las polillas oscuras volvieron a ser más fácilmente visibles por los depredadores y su número disminuyó.[168] Sin embargo, aunque la dirección de la selección cambie, los rasgos que se hubiesen perdido en el pasado no pueden volver a obtenerse de forma idéntica ―situación que describe la Ley de Dollo o «Ley de la irreversibilidad evolutiva»―.[169] De acuerdo con esta hipótesis, una estructura u órgano que se ha perdido o descartado durante el transcurso de la evolución no volverá a aparecer en ese mismo linaje de organismos.[170][171]
Según Richard Dawkins, esta hipótesis es «una declaración sobre la improbabilidad estadística de seguir exactamente la misma trayectoria evolutiva dos veces o, de hecho, una misma trayectoria particular en ambas direcciones».[172]
Dentro de una población, la selección natural para un determinado rasgo que varía en forma continua, como la altura, se puede categorizar en tres tipos diferentes. El primero es la «selección direccional», que es un cambio en el valor medio de un rasgo a lo largo del tiempo; por ejemplo, cuando los organismos cada vez son más altos.[173] En segundo lugar se halla la «selección disruptiva» que es la selección de los valores extremos de un determinado rasgo, lo que a menudo determina que los valores extremos sean más comunes y que la selección actúe en contra del valor medio; esto implica, en el ejemplo anterior, que los organismos bajos y altos tienen una ventaja, pero los de altura media no. Finalmente, en la «selección estabilizadora», la selección actúa en contra de los valores extremos, lo que determina una disminución de la varianza alrededor del promedio y una menor variabilidad de la población para ese carácter en particular;[163][174] si se diera este tipo de selección, todos los organismos de una población adquirirían paulatinamente una altura similar.
Un tipo especial de selección natural es la selección sexual, que actúa a favor de cualquier rasgo que aumente el éxito reproductivo por aumentar el atractivo de un organismo para sus parejas potenciales.[175] Ciertos rasgos adquiridos por los machos por selección sexual ―tales como los cuernos voluminosos, cantos de apareamiento o colores brillantes― pueden reducir las posibilidades de supervivencia, por ejemplo, por atraer a los depredadores.[176] No obstante, Esta desventaja reproductiva se compensa por un mayor éxito reproductivo de los machos que presentan estos rasgos.[177]
Un área de estudio activo es la denominada «unidad de selección»; se ha dicho que la selección natural actúa a nivel de genes, células, organismos individuales, grupos de organismos e incluso especies.[178][179] Ninguno de estos modelos es mutuamente exclusivo, y la selección puede actuar en múltiples niveles a la vez.[180] Por ejemplo, debajo del nivel del individuo, hay genes denominados transposones que intentan replicarse en todo el genoma.[181] La selección por encima del nivel del individuo, como la selección de grupo, puede permitir la evolución de la cooperación.[182]
Deriva genética

La deriva genética es el cambio en la frecuencia de los alelos entre una generación y la siguiente, y tiene lugar porque los alelos de la descendencia son una muestra aleatoria de los padres, y por el papel que juega el azar en la hora de determinar si un ejemplar determinado sobrevivirá y se reproducirá.[101] En términos matemáticos, los alelos están sujetos a errores de muestreo. Como resultado de ello, cuando las fuerzas selectivas están ausentes o son relativamente débiles, la frecuencia de los alelos tiende a «derivar» hacia arriba o hacia abajo aleatoriamente (en un paseo aleatorio). Esta deriva se detiene cuando un alelo se convierte finalmente fijado, es decir, o bien desaparece de la población, o bien sustituye totalmente el resto de genes. Así pues, la deriva genética puede eliminar algunos alelos de una población simplemente debido al azar. Incluso en la ausencia de fuerzas selectivas, la deriva genética puede hacer que dos poblaciones separadas que empiezan con la misma estructura genética se separen en dos poblaciones divergentes con un conjunto de alelos diferentes.[183]
El tiempo necesario para que un alelo quede fijado por la deriva genética depende del tamaño de la población; la fijación tiene lugar más rápido en poblaciones más pequeñas.[184] La medida precisa de las poblaciones que es importante en este caso recibe el nombre de tamaño poblacional efectivo, que fue definida por Sewall Wright como el número teórico de ejemplares reproductivos que presenten el mismo grado observado de consanguinidad.
Aunque la selección natural es responsable de la adaptación, la importancia relativa de las dos fuerzas, selección natural y deriva genética, como impulsoras del cambio evolutivo en general es actualmente un campo de investigación en la biología evolutiva.[185] Estas investigaciones fueron inspiradas por la teoría neutralista de la evolución molecular, que postula que la mayoría de cambios evolutivos son el resultado de la fijación de mutaciones neutras, que no tienen ningún efecto inmediato sobre la aptitud de un organismo.[186] Por tanto, en este modelo, la mayoría de los cambios genéticos en una población son el resultado de una presión de mutación constante y de deriva genética.[187]
Las consecuencias de la evolución
Adaptación

La adaptación es el proceso mediante el cual una población se adecua mejor a su hábitat y también el cambio en la estructura o en el funcionamiento de un organismo que lo hace más adecuado a su entorno.[188][189] Este proceso se produce por selección natural[190] durante muchas generaciones[191] y es uno de los fenómenos básicos de la biología.[192]
La importancia de una adaptación sólo puede entenderse en relación con el total de la biología de la especie. Julian Huxley[193]
Las especies tienden a adaptarse a diferentes nichos ecológicos para reducir al mínimo la competencia entre ellas.[194] Esto se conoce como principio de exclusión competitiva en ecología: dos especies no pueden ocupar el mismo nicho en el mismo ambiente por un largo tiempo.[195]
La adaptación no implica necesariamente cambios importantes en parte física de un cuerpo. Como ejemplo puede mencionarse a los trematodos ―parásitos internos con estructuras corporales muy simples pero con un ciclo de vida muy complejo― en los que sus adaptaciones a un medio ambiente tan inusual no son el producto de caracteres observables a simple vista sino en aspectos críticos de su ciclo vital.[196] En general, el concepto de adaptación incluye, además del proceso adaptativo mismo, todos los aspectos de los organismos, de las poblaciones o de las especies que son su resultado. Mediante la utilización del término «adaptación» para el proceso evolutivo y «rasgo o carácter adaptativo» para el producto del mismo, los dos sentidos del concepto se distinguen perfectamente. Según Theodosius Dobzhansky la «adaptación» es el proceso evolutivo por el cual un organismo se vuelve más capaz de vivir en su hábitat o hábitats,[197] mientras que la «adaptabilidad» es el estado de estar adaptado, o sea, el grado en que un organismo es capaz de vivir y reproducirse en un determinado conjunto de hábitats.[198] Finalmente, un «carácter adaptativo» es uno de los aspectos del desarrollo de un organismo que aumenta su probabilidad de sobrevivir y reproducirse.[199]
La adaptación a veces implica la ganancia de una nueva característica; ejemplos notables son la evolución en laboratorio de las bacterias Escherichia coli para que puedan ser capaces de utilizar el ácido cítrico como un nutriente, cuando las bacterias de tipo silvestre no lo pueden hacer;[200] la evolución de una nueva enzima en Flavobacterium que permite que estas bacterias puedan crecer en los subproductos de la fabricación del nylon;[201][202] y la evolución de una vía metabólica completamente nueva en la bacteria del suelo Sphingobium que le permite degradar el pesticida sintético pentaclorofenol.[203][204] En ocasiones, también puede darse la pérdida de una función ancestral. Un ejemplo que muestra los dos tipos de cambio es la adaptación de la bacteria Pseudomonas aeruginosa a la fluoroquinolona con cambios genéticos que modifican la molécula sobre la que actúa y por el aumento de la actividad de los transportadores que bombean el antibiótico fuera de la célula.[205] Una idea todavía controvertida, es que algunas adaptaciones pueden aumentar la capacidad de los organismos para generar diversidad genética y para adaptarse por selección natural ―o sea, aumentarían la capacidad de evolución―.[206][207]

Una consecuencia de la adaptación es la existencia de estructuras con organización interna similar y diferentes funciones en organismos relacionados. Este es el resultado de la modificación de una estructura ancestral para adaptarla a diferentes ambientes y nichos ecológicos. Los huesos de las alas de los murciélagos, por ejemplo, son muy similares a los de los pies del ratón y los de las manos de los primates, debido a que todas estas estructuras estaban presentes en un ancestro común de los mamíferos.[209] Dado que todos los organismos vivos están relacionados en cierta medida, incluso las estructuras que presentan diferencias profundas, como los ojos de los artrópodos, del calamar y de los vertebrados, o las extremidades y las alas de artrópodos y vertebrados, dependen de un conjunto común de genes homólogos que controlan su desarrollo y funcionamiento, lo que se denomina homología profunda.[210][211]
Durante la adaptación pueden aparecer estructuras vestigiales,[212] carentes de funcionalidad en una especie; sin embargo, la misma estructura es funcional en la especie ancestral o en otras especies relacionadas. Los ejemplos incluyen los pseudogenes,[213] los ojos de los peces cavernícolas ciegos,[214] las alas en las especies de aves que no vuelan[215] y los huesos de la cadera presentes en las ballenas y en las serpientes.[208] En los seres humanos también existen ejemplos de estructuras vestigiales, como las muelas de juicio,[216] el coxis,[212] el apéndice vermiforme,[212] y reacciones involuntarias como la piel de gallina[217] y otros reflejos primitivos.[218][219][220][221]
Algunos rasgos que parecen ser simples adaptaciones son, de hecho, exaptaciones: estructuras originalmente adaptadas para una función, pero que coincidentalmente se hicieron útiles para otro propósito.[222] Un ejemplo es el lagarto africano Holaspis guentheri que desarrolló una cabeza y tronco muy aplastados para esconderse en las grietas, como puede observarse en otros lagartos del mismo género. Sin embargo, en esta especie, el cuerpo achatado le permite planear de árbol en árbol.[222] Los pulmones de los peces pulmonados ancestrales son una exaptación de las vejigas natatorias de los peces teleósteos empleadas como regulador de la flotación.[223]
Una rama de la biología evolutiva estudia el desarrollo de las adaptaciones y de las exaptaciones.[224] Esta área de investigación aborda el origen y la evolución del desarrollo embrionario y cómo surgen nuevas características a partir de modificaciones del desarrollo.[225] Estos estudios han demostrado, por ejemplo, que las mismas estructuras óseas de los embriones que forman parte de la mandíbula en algunos animales, en cambio forman parte del oído medio en los mamíferos.[226] Cambios en los genes que controlan el desarrollo también pueden causar que reaparezcan estructuras perdidas durante la evolución, como por ejemplo, los dientes de embriones de pollos mutantes, similares a los de los cocodrilos.[227] De hecho, cada vez es más claro que la mayoría de las alteraciones en la forma de los organismos se deben a cambios en un pequeño conjunto de genes conservados.[228]
Coevolución
La interacción entre organismos puede producir conflicto o cooperación. Cuando interactúan dos especies diferentes, como un patógeno y un hospedador, o un depredador y su presa, la evolución de una de ellas causa adaptaciones en la otra; estos cambios en la segunda especie causan, a su vez, nuevas adaptaciones en la primera. Este ciclo de selección y respuesta recibe el nombre de coevolución.[229] Un ejemplo es la producción de tetradotoxina por parte del tritón de Oregón y la evolución de la resistencia a esta toxina en su predador, la serpiente de jarretera. En esta pareja predador-presa, la carrera armamentista evolutiva ha tenido como resultado una elevada producción de toxina en el tritón, y un aumento correspondiente de resistencia a ella en la serpiente.[230]
Especiación
La especiación (o cladogénesis) es el proceso por el cual una especie diverge en dos o más especies descendientes.[231] Los biólogos evolutivos ven las especies como fenómenos estadísticos y no como categorías o tipos. Este planteamiento es contrario al aún muy arraigado concepto clásico de especie como una clase de organismos ejemplificados por un «espécimen tipo» que posee todas las características comunes a dicha especie. En su lugar, una especie se concibe ahora como un linaje que comparte un único acervo genético y evoluciona independientemente. Según esta descripción, los límites entre especies son difusos, a pesar de que se utilizan propiedades tanto genéticas como morfológicas para ayudar a diferenciar entre linajes estrechamente relacionados.[232] De hecho, la definición exacta del término «especie» está todavía en discusión, particularmente para organismos basados en células procariotas;[233] es lo que se denomina «problema de las especies».[234] Diversos autores han propuesto una serie de definiciones basadas en criterios diferentes, pero la aplicación de una u otra es finalmente una cuestión práctica, dependiendo en cada caso concreto de las particularidades del grupo de organismos en estudio.[234] Actualmente, la unidad de análisis principal en biología es la población, un conjunto observable de individuos que interactúan, en lugar de la especie, un conjunto observable de individuos que se parecen entre sí.

La especiación ha sido observada en múltiples ocasiones tanto en condiciones de laboratorio controladas como en la naturaleza.[235] En los organismos que se reproducen sexualmente, la especiación es el resultado de un aislamiento reproductivo seguido de una divergencia genealógica. Hay cuatro modalidades de especiación. La más habitual en los animales es la especiación alopátrica, que tiene lugar en poblaciones que quedan geográficamente aisladas, como en el caso de la fragmentación de hábitat o las migraciones. En estas condiciones, la selección puede causar cambios muy rápidos en la apariencia y el comportamiento de los organismos.[236][237] Debido a los procesos de selección y deriva genética, la separación de poblaciones puede tener como resultado la aparición de descendientes que no se pueden reproducir entre ellos.[238]
La segunda modalidad de especiación es la especiación peripátrica, que tiene lugar cuando poblaciones pequeñas quedan aisladas en un nuevo medio. Se diferencia de la especiación alopátrica en que las poblaciones aisladas son numéricamente mucho menores que la población madre. Esto causa una especiación rápida por medio de la aceleración de la deriva genética y la selección en un acervo génico pequeño, proceso conocido como el efecto fundador.[239]
La tercera modalidad de especiación es la especiación parapátrica. Se parece a la especiación peripátrica en que una pequeña población coloniza un nuevo hábitat, pero se diferencia en que no hay ninguna separación física entre las dos poblaciones. En cambio, la especiación es el resultado de la evolución de mecanismos que reducen el flujo génico entre ambas poblaciones.[231] Generalmente, esto ocurre cuando ha habido un cambio drástico en el medio dentro del hábitat de la especie madre. Un ejemplo es la hierba Anthoxanthum odoratum, que puede sufrir una especiación parapátrica en respuesta a contaminación metálica localizada proveniente de minas.[240] En este caso, evolucionan plantas con una resistencia a niveles altos de metales en el suelo; el aislamiento reproductivo ocurre porque la selección favorece una época de floración distinta a la de las especie madre para las nuevas plantas, que no pueden perder entonces por hibridación los genes que les otorgan la resistencia. La selección en contra de los híbridos puede reforzarse por la diferenciación de los rasgos que promueven la reproducción entre miembros de la misma especie, así como por el aumento de las diferencias de apariencia en el área geográfica en la que la se solapan.[241]
Finalmente, en la especiación simpátrica, las especies divergen sin que haya aislamiento geográfico o cambios en el hábitat. Esta modalidad es rara, pues incluso una pequeña cantidad de flujo génico puede eliminar las diferencias genéticas entre partes de una población.[242] En general, en los animales, la especiación simpátrica requiere la evolución de diferencias genéticas y un apareamiento no aleatorio, para que se pueda desarrollar un aislamiento reproductivo.[243]
Un tipo de especiación simpátrica es el cruce de dos especies relacionadas para producir una nueva especie híbrida. Esto no es habitual en los animales, porque los cromosomas homólogos de progenitores de especies diferentes no pueden aparearse con éxito durante la meiosis. Es más habitual en las plantas, que duplican a menudo su número de cromosomas para formar poliploides.[244] Esto permite a los cromosomas de cada especie parental formar una pareja complementaria durante la meiosis, ya que los cromosomas de cada padre ya son representados por una pareja.[245] Un ejemplo de este tipo de especiación tuvo lugar cuando las especies vegetales Arabidopsis thaliana y Arabidopsis arenosa se cruzaron para producir la nueva especie Arabidopsis suecica.[246] Esto tuvo lugar hace aproximadamente 20 000 años,[247] y el proceso de especiación ha sido reproducido en el laboratorio, lo que ha permitido estudiar los mecanismos genéticos implicados en este proceso.[248] De hecho, la duplicación de cromosomas dentro de una especie puede ser una causa habitual de aislamiento reproductivo, pues la mitad de los cromosomas duplicados quedarán sin pareja cuando se aparean con los de organismos no duplicados.[234]
Los episodios de especiación son importantes en la teoría del equilibrio puntuado, que explica la presencia en el registro fósil de rápidos momentos de especiación intercalados con periodos relativamente largos de estasis, durante los que las especies permanecen prácticamente sin modificar.[249] En esta la teoría, la especiación está relacionada con la evolución rápida, y la selección natural y la deriva genética actúan de forma particularmente intensa sobre los organismos que sufren una especiación en hábitats nuevos o pequeñas poblaciones. Como resultado de ello, los períodos de estasis del registro fósil corresponden a la población madre, y los organismos que sufren especiación y evolución rápida se encuentran en poblaciones pequeñas o hábitats geográficamente restringidos, por lo que raramente quedan preservados en forma de fósiles.[250]
Extinción
La extinción es la desaparición de una especie entera. No es un acontecimiento inusual,[251] y, de hecho, la práctica totalidad de especies animales y vegetales que han vivido en la Tierra están actualmente extinguidas,[252] por lo que parece que la extinción es el destino final de todas las especies.[253] Las extinciones tienen lugar continuamente durante la historia de la vida, aunque el ritmo de extinción aumenta drásticamente en los ocasionales eventos de extinción.[254] La extinción del Cretácico-Terciario, durante la cual se extinguieron los dinosaurios, es la más conocida, pero la anterior extinción Permo-Triásica fue aún más severa, causando la extinción de casi el 96 % de las especies.[254] La extinción del Holoceno todavía dura y está asociada con la expansión de la humanidad por el globo terrestre en los últimos milenios. El ritmo de extinción actual es de 100 a 1000 veces mayor que el ritmo medio, y hasta un 30 % de las especies pueden desaparecer a mediados del siglo XXI.[255] Las actividades humanas son actualmente la causa principal de la extinción;[256] es posible que el calentamiento global la acelere aún más en el futuro.[257]
Las causas de la extinción determinan su impacto en la evolución.[254] Las mayoría de las extinciones, que tienen lugar continuamente, podrían ser el resultado de la competencia entre especies por recursos limitados (exclusión competitiva).[2] Si la competencia entre especies altera la probabilidad de extinción, se podría considerar la selección de especies como un nivel de la selección natural.[178] Las extinciones masivas intermitentes también son importantes, pero en lugar de actuar como fuerza selectiva, reducen drásticamente la diversidad de manera indiscriminada y promueven explosiones de rápida evolución y especiación en los supervivientes.[258]
Microevolución y macroevolución
Microevolución es un término usado para referirse a cambios en pequeña escala de las frecuencias génicas en una población durante el transcurso de varias generaciones. La adaptación de los insectos al uso de plaguicidas o la variación del color de piel de los humanos son ejemplos de microevolución.[259][260] Estos cambios pueden deberse a varios procesos: mutación, flujo génico, deriva génica o selección natural.[259] La genética de poblaciones es una rama de la biología evolutiva aplica métodos bioestadísticos al estudio de los procesos de la microevolución,
Los cambios a mayor escala, desde la especiación hasta las grandes transformaciones evolutivas ocurridas en largos períodos, son comúnmente denominados macroevolución. La evolución de los anfibios a partir de un grupo de peces óseos es un ejemplo de macroevolución. Los biólogos no acostumbran hacer una separación absoluta entre macroevolución y microevolución, pues consideran que macroevolución es simplemente microevolución acumulada durante escalas de tiempo grandes.[261] Una minoría de teóricos, sin embargo, considera que los mecanismos de la teoría sintética para la microevolución no bastan para hacer esa extrapolación y que se necesitan otros mecanismos. La teoría de los equilibrios puntuados, propuesta por Gould y Eldredge, intenta explicar ciertas tendencias macroevolutivas que se observan en el registro fósil.[262]
Ampliación de la síntesis moderna
En las últimas décadas se ha hecho evidente que los patrones y los mecanismos evolutivos son mucho más variados que los que fueran postulados por los pioneros de la Biología evolutiva (Darwin, Wallace o Weismann) y los arquitectos de la teoría sintética (Dobzhansky, Mayr y Huxley, entre otros). Los nuevos conceptos e información en la biología molecular del desarrollo, la sistemática, la geología y el registro fósil de todos los grupos de organismos necesitan ser integrados en lo que se ha denominado «síntesis evolutiva ampliada». Los campos de estudio mencionados muestran que los fenómenos evolutivos no pueden ser comprendidos solamente a través de la extrapolación de los procesos observados a nivel de las poblaciones y especies modernas.[263][264][265][266][72] En las próximas secciones se presentan los aspectos considerados como la ampliación de la síntesis moderna.
Paleobiología y tasas de evolución
| Grupo de organismos | Longevidad promedio
(en millones de años) |
|---|---|
| Bivalvos y gasterópodos marinos | 10 a 14 |
| Foraminíferos bénticos y planctónicos | 20 a 30 |
| Diatomeas marinas | 25 |
| Trilobites (extinguidos). | > 1 |
| Amonites (extinguidos). | 5 |
| Peces de agua dulce | 3 |
| Serpientes | >2 |
| Mamíferos | 1 a 2 |
| Briófitas | > 20 |
| Plantas superiores. Hierbas | 3 a 4 |
| Angiospermas. Árboles y arbustos | 27 a 34 |
| Gimnospermas. Coníferas y cícadas | 54 |
En el momento en que Darwin propuso su teoría de evolución, caracterizada por modificaciones pequeñas y sucesivas, el registro fósil disponible era todavía muy fragmentario y no se habían hallado fósiles previos al período Cámbrico. El dilema de Darwin, o sea, la inexistencia aparente de registros fósiles del Precámbrico, fue utilizado como el principal argumento en contra de su propuesta de que todos los organismos de la Tierra provienen de un antepasado común.[2]
Además de la ausencia de fósiles antiguos, a Darwin también le preocupaba la carencia de formas intermedias o enlaces conectores en el registro fósil, lo cual desafiaba su visión gradualística de la especiación y de la evolución. De hecho en tiempos de Darwin, con la excepción de Archaeopteryx, que muestra una mezcla de características de ave y de reptil, virtualmente no se conocían otros ejemplos de formas intermedias o eslabones perdidos, como se los denominó coloquialmente.[2]
Incluso en 1944, cuando se publicó el libro de Simpson Tempo and mode in evolution, no se conocían todavía fósiles del Precámbrico y solo se disponía de unos pocos ejemplos de formas intermedias en el registro fósil que enlazaran las formas antiguas con las modernas. Desde entonces los científicos han explorado el período Precámbrico con detalle y se sabe que la vida es mucho más antigua de lo que se creía en los tiempos de Darwin. También se sabe que esas antiguas formas de vida fueron los ancestros de todos los organismos aparecidos posteriormente en el planeta. Asimismo desde finales del siglo XX se han descubierto, descrito y analizado una gran cantidad de ejemplos representativos de formas fósiles intermedias que enlazan a los principales grupos de vertebrados e, incluso, fósiles de las primeras plantas con flor.[42][43] Como resultado de estos y otros avances científicos, ha surgido una nueva disciplina de la Paleontología, denominada Paleobiología.[42][263][270][271][272][273][274]
Un ejemplo de forma transicional entre los peces y los anfibios es el género extinto Panderichthys, que habitó la tierra hace unos 370 millones de años y es el enlace intermedio entre Eustenopteron (género de peces de 380 millones de años) y Acanthostega (anfibios de hace 363 millones de años).[275] Entre los animales terrestres surgió el género Pederpes hace 350 millones de años, que conecta a los principales anfibios acuáticos del Devónico superior con los tetrápodos tempranos.[276] Asimismo, la historia evolutiva de varios grupos de organismos extintos, tales como los dinosaurios, ha sido reconstruida con notable detalle.[277] Un ejemplo de eslabón entre los reptiles y los mamíferos es el Thrinaxodon, un reptil con características de mamífero que habitó el planeta hace 230 millones de años.[278] El Microraptor, un dromeosáurido con cuatro alas que podía planear y que vivió hace 126 millones de años, representa un estado intermedio entre los terópodos y las primitivas aves voladoras como Archaeopteryx.[279] Una forma transicional entre los mamíferos terrestres y la vaca marina es Pezosiren, un sirénido cuadrúpedo primitivo con adaptaciones terrestres y acuáticas que vivió hace 50 millones de años.[280] Los mamíferos terrestres con pezuñas y las ballenas se hallan conectados a través de los géneros extintos Ambulocetus y Rodhocetus que habitaron el planeta hace 48 a 47 millones de años.[281] Para finalizar esta enumeración de ejemplos de formas transicionales, el ancestro común de los chimpancés y de los seres humanos es el género Sahelanthropus, un homínido con aspecto de mono que exhibía un mosaico de caracteres de chimpancé y de hominino y que habitó África hace 7 a 5 millones de años.[282]
En su libro Variation and evolution in plants (1950), Stebbins también se lamentaba por la ausencia de un registro fósil que permitiera comprender el origen de las primeras plantas con flores, las angiospermas. De hecho, el propio Darwin caracterizó al origen de las angiospermas como un «abominable misterio». No obstante, este vacío de conocimiento está siendo rápidamente completado con los descubrimientos realizados desde fines del siglo XX y hasta la actualidad. En 1998 se descubrió en China, en los estratos provenientes del Jurásico Superior (de más de 125 millones de años de antigüedad), un fósil de un eje con frutos, que se ha denominado Archaefructus[283] Este descubrimiento, que permite establecer la edad de las angiospermas más antiguas, hizo mundialmente famosa a la Formación Yixian, donde fue hallado este fósil. En la misma formación se encontraron el fósil de otra angiosperma, Sinocarpus,[284] y, en 2007, una flor que presenta la organización típica de las angiospermas, con la presencia de tépalos, estambres y gineceo. Esta especie ha sido bautizada como Euanthus (del griego, «flor verdadera») por sus descubridores, y demuestra que en el Cretácico inferior ya existían flores como las de las angiospermas actuales.[285]
Causas ambientales de las extinciones masivas

Darwin no solo consideró el origen, sino también la disminución y la desaparición de las especies; propuso que la competencia interespecífica por recursos limitados era una causa importante de la extinción de poblaciones y especies: durante el tiempo evolutivo, las especies superiores surgirían para reemplazar a especies menos adaptadas. Esta perspectiva ha cambiado en los últimos años, con una mejor comprensión de las causas de las extinciones masivas, episodios de la historia de la Tierra, donde parecen no cumolirse las «reglas» de la selección natural.[254][2] Mayr planteó la nueva perspectiva en su libro Animal species and evolution, en el que señaló que la extinción debe considerarse como uno de los fenómenos evolutivos más conspicuos. Mayr discutió las causas de los eventos de extinción y especuló que la aparición de nuevas enermedades, la invasión de un ecosistema por otras especies o los cambios en el ambiente biótico pueden ser los responsables:
«Las causas reales de la extinción de cualquier especie de fósil presumiblemente siempre seguirán siendo inciertas... Es cierto, sin embargo, que cualquier evento grave de extinción está siempre correlacionado con un trastorno ambiental importante» (Mayr, 1963).[286])
Esta hipótesis, no sustentada por hechos probados cuando fue propuesta, ha adquirido desde entonces un considerable apoyo. El término «extinción masiva», mencionado por Mayr[287] sin una definición asociada, se utiliza cuando una gran cantidad de especies se extinguen en un plazo geológicamente breve; los eventos pueden estar relacionados con una causa única o con una combinación de causas, y las especies extintas son plantas y animales de todo tamaño, tanto marinos como terrestres.[271] Al menos han ocurrido cinco extinciones masivas: la extinción masiva del Cámbrico-Ordovícico, las extinciones masivas del Ordovícico-Silúrico, la extinción masiva del Devónico, la extinción masiva del Pérmico-Triásico y la extinción masiva del Cretácico-Terciario.[288]
La extinción biológica que se produjo en el Pérmico-Triásico hace unos 250 millones de años representa el más grave evento de extinción en los últimos 550 millones de años. Se estima que en este evento se extinguieron alrededor del 70 % de las familias de vertebrados terrestres, muchas gimnospermas leñosas y más del 90 % de las especies oceánicas. Se han propuesto varias causas para explicar este evento, como el vulcanismo, el impacto de un asteroide o un cometa, la anoxia oceánica y el cambio ambiental. No obstante, es aparente en la actualidad que las gigantescas erupciones volcánicas, que tuvieron lugar durante un intervalo de tiempo de sólo unos pocos cientos de miles de años, fueron la causa principal de la catástrofe de la biosfera durante el Pérmico tardío.[289] El límite Cretácico-Terciario registra el segundo mayor evento de extinción masivo. Esta catástrofe mundial acabó con el 70 % de todas las especies, entre las cuales los dinosaurios son el ejemplo más popularmente conocido. Los pequeños mamíferos sobrevivieron para heredar los nichos ecológicos vacantes, lo que permitió el ascenso y la radiación adaptativa de los linajes que en última instancia se convertirían en Homo sapiens[272] y otras especies actuales. Los paleontólogos han propuesto numerosas hipótesis para explicar este evento; las más aceptadas en la actualidad son las del impacto de un asteroide[290] y la de fenómenos de vulcanismo.[272]
En resumen, la hipótesis de los trastornos ambientales como causas de las extinciones masivas ha sido confirmada, lo cual indica que si bien gran parte de historia de la evolución puede ser gradual, de vez en cuando ciertos acontecimientos catastróficos han marcado su ritmo de fondo. Es evidente que los pocos «afortunados sobrevivientes» determinan los subsecuentes patrones de evolución en la historia de la vida.[2][254]
Selección sexual y altruismo

Determinadas características en una especie son sexualmente atractivas aunque carezcan de otro significado adaptativo. Por ejemplo, la atracción de las hembras de algunas especies de aves por los machos más capaces de hinchar los cuellos, ha traído como resultado —en el transcurso de las generaciones― la selección de machos que pueden hinchar los cuellos hasta un nivel extraordinario. Darwin concluyó que si bien la selección natural guía el curso de la evolución, la selección sexual influye su curso aunque no parezca existir ninguna razón evidente para ello. Los argumentos de Darwin a favor de la selección sexual aparecen en el capítulo cuarto de El origen de las especies y, muy especialmente, en The Descent of Man, and Selection in Relation to Sex de 1871. En ambos casos, se esgrime la analogía con el mundo artificial:
[La selección sexual] no depende de una lucha por la existencia sino de una lucha entre los machos por la posesión de las hembras; el resultado no es la muerte del competidor que no ha tenido éxito, sino el tener poca o ninguna descendencia. La selección sexual es, por lo tanto, menos rigurosa que la selección natural. Generalmente, los machos más vigorosos, aquellos que están mejor adaptados a los lugares que ocupan en la naturaleza, dejarán mayor progenie. Pero en muchos casos la victoria no dependerá del vigor sino de las armas especiales exclusivas del sexo masculino[...] Entre las aves, la pugna es habitualmente de carácter más pacífico. Todos los que se han ocupado del asunto creen que existe una profunda rivalidad entre los machos de muchas especies para atraer por medio del canto a las hembras. El tordo rupestre de Guayana, las aves del paraíso y algunas otras se congregan, y los machos, sucesivamente, despliegan sus magníficos plumajes y realizan extraños movimientos ante las hembras que, colocadas como espectadoras, eligen finalmente el compañero más atractivo.Darwin 1859:136-137)
En su libro The Descent of Man describió numerosos ejemplos, tales como la cola del pavo real y de la melena del león. Darwin argumentó que la competencia entre los machos es el resultado de la selección de los rasgos que incrementan el éxito del apareamiento de los machos competidores, rasgos que podrían, sin embargo, disminuir las posibilidades de supervivencia del individuo. De hecho, los colores brillantes hacen a los animales más visibles a los depredadores, el plumaje largo de los machos de pavo real y de las aves del paraíso, o la enorme cornamenta de los ciervos son cargas incómodas en el mejor de casos. Darwin sabía que no era esperable que la selección natural favoreciera la evolución de tales rasgos claramente desventajosos, y propuso que los mismos surgieron por selección sexual,
la cual depende no de una lucha por la existencia en relación con otros seres orgánicos o condiciones externas, sino de una lucha entre los individuos de un sexo, generalmente los machos, por la posesión del otro sexo. Darwin, 1871.
Para Darwin, la selección sexual incluía fundamentalmente dos fenómenos: la preferencia de las hembras por ciertos machos ―selección intersexual, femenina, o epigámica― y, en las especies polígamas, las batallas de los machos por el harén más grande ―selección intrasexual―. En este último caso, el tamaño corporal grande y la musculatura proporcionan ventajas en el combate, mientras que en el primero, son otros rasgos masculinos, como el plumaje colorido y el complejo comportamiento de cortejo los que se seleccionan a favor para aumentar la atención de las hembras. Las ideas de Darwin en este sentido no fueron ampliamente aceptadas y los defensores de la teoría sintética (Dobzhansky, Mayr y Huxley) en gran medida ignoraron el concepto de selección sexual.
El estudio de la selección sexual sólo cobró impulso en la era postsíntesis.[291] Se ha argumentado que, según propuso Wallace, los machos con plumaje brillante demuestran de ese modo su buena salud y su alta calidad como parejas sexuales. De acuerdo con esta hipótesis de la «selección sexual de los buenos genes», la elección de pareja masculina por parte de las hembras ofrece una ventaja evolutiva.[81] Esta perspectiva ha recibido apoyo empírico en las últimas décadas. Por ejemplo, se ha hallado una asociación, aunque pequeña, entre la supervivencia de la descendencia y los caracteres sexuales secundarios masculinos en un gran número de taxones, tales como aves, anfibios, peces e insectos).[292] Además, las investigaciones con mirlos han proporcionado la primera evidencia empírica de que existe una correlación entre un carácter sexual secundario y un rasgo que incrementa la supervivencia ya que los machos con los más brillantes colores presentan un sistema inmune más fuerte.[293] Así, la selección femenina podría promover la salud general de las poblaciones en esta especie. Estos y otros datos son coherentes con el concepto de que la elección de la hembra influye en los rasgos de los machos e, incluso, que puede ser beneficiosa para la especie en formas que no tienen ninguna relación directa con el éxito del apareamiento.[291]

En el mismo contexto y desde la publicación del Origen de las especies, se ha argumentado que el comportamiento altruista, los actos abnegados realizados en beneficio de los demás, es incompatible con el principio de la selección natural. Sin embargo, el comportamiento altruista, como el cuidado de las crías por los padres y el mutualismo, se ha observado y documentado en todo el reino animal, desde los invertebrados hasta en los mamíferos.[294][295][296][297] Una de las formas más notorias de altruismo se produce en ciertos insectos eusociales, como las hormigas, abejas y avispas, que tienen una clase de trabajadoras estériles. La cuestión general de la evolución del altruismo, de la sociabilidad de ciertos insectos o de la existencia de abejas u hormigas obreras que no dejan descendientes ha sido contestada por la teoría de la aptitud inclusiva, también llamada teoría de selección familiar.[298] De acuerdo con el principio de Darwin/Wallace la selección natural actúa sobre las diferencias en el éxito reproductivo (ER) de cada individuo, donde ER es el número de descendientes vivos producidos por ese individuo durante toda la vida. Hamilton (1972) amplió esta idea e incluyó los efectos de ER de los familiares del individuo: la aptitud inclusiva es el ER de cada individuo, más el ER de sus familiares, cada uno devaluado por el correspondiente grado de parentesco. Numerosos estudios en una gran variedad de especies animales han demostrado que el altruismo no está en conflicto con la teoría evolutiva. Por esta razón, es necesario realizar una modificación y ampliación de la visión tradicional de que la selección opera sobre un solo organismo aislado en una población: el individuo aislado ya no parece tener una importancia central desde el punto de vista evolutivo, sino como parte de una compleja red familiar.[299][300][301]
Macroevolución, monstruos prometedores y equilibrio puntuado
Cuando se define macroevolución como el proceso responsable del surgimiento de los taxones de rango superior, se está utilizando un lenguaje metafórico. En sentido estricto, solo «surgen» nuevas especies, ya que la especie es el único taxón que posee estatus ontológico.[302] La macroevolución da cuenta de la emergencia de discontinuidades morfológicas importantes entre grupos de especies, razón por la cual se las clasifica como grupos marcadamente diferenciados, es decir, pertenecientes a unidades taxonómicas distintas y de alto rango. Es en los mecanismos que explican el surgimiento de estas discontinuidades que las diferentes concepciones y aproximaciones disciplinarias se contraponen.
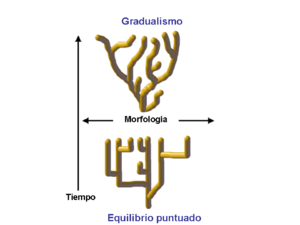
El gradualismo es el modelo macroevolucionista ortodoxo. Explica la macroevolución como el producto de un cambio lento, de la acumulación de muchos pequeños cambios en el transcurso del tiempo. Este cambio gradual debería reflejarse en el registro fósil con la aparición de numerosas formas de transición entre los grupos de organismos. Sin embargo, el registro no es abundante en formas intermedias. Los gradualistas atribuyen esta discrepancia entre su modelo y las pruebas halladas a la imperfección del propio registro geológico —según Darwin, el registro geológico es una narración de la que se han perdido algunos volúmenes y muchas páginas—. El modelo del equilibrio puntuado, propuesto en 1972 por N. Eldredge y S. J. Gould, sostiene en cambio que el registro fósil es un fiel reflejo de lo que en realidad ocurrió. Las especies aparecen repentinamente en los estratos geológicos, se las encuentra en ellos por 5 a 10 millones de años sin grandes cambios morfológicos y luego desaparecen abruptamente del registro, sustituidas por otra especie emparentada, pero distinta. Eldredge y Gould utilizan los términos estasis e interrupción, respectivamente, para designar estos períodos. Según su modelo, las interrupciones abruptas en el registro fósil de una especie reflejarían el momento en que esta fue reemplazada por una pequeña población periférica ―en la cual el ritmo de evolución habría sido más rápido― que compitió con la especie originaria y terminó por sustituirla. De acuerdo con este patrón, la selección natural no solo opera dentro de la población, sino también entre especies, y los cambios cualitativamente importantes en los organismos ocurrirían en períodos geológicos relativamente breves separados por largos períodos de equilibrio.
En biología evolutiva, un monstruo prometedor es un organismo con un fenotipo profundamente mutante que tiene el potencial para establecer un nuevo linaje evolutivo. El término se utiliza para describir un evento de especiación saltacional que puede ser el origen de nuevos grupos de organismos. La frase fue acuñada por el genetista alemán Richard Goldschmidt, quien pensaba que los cambios pequeños y graduales que dan lugar a la microevolución no pueden explicar la macroevolución. La relevancia evolutiva de los monstruos prometedores ha sido rechazada o puesta en duda por muchos científicos que defienden la Teoría sintética de evolución biológica.[303] En su obra The material basis of evolution (La base material de la evolución), Goldschmidt escribió que «el cambio desde una especie a otra no es un cambio que no implica más y más cambios atomísticos, sino una modificación completa del patrón principal o del sistema de reacción principal en uno nuevo, el que, más tarde puede nuevamente producir variación intraespecífica por medio de micromutaciones».[304]
La tesis de Goldschmidt fue universalmente rechazada y ampliamente ridiculizada dentro la comunidad científica, que favorecía las explicaciones neodarwinianas de R. A. Fisher, J. B. S. Haldane y Sewall Wright.[305]
No obstante, varias líneas de evidencia sugieren que los monstruos prometedores juegan un papel significativo durante el origen de innovaciones clave y de planes corporales noveles por evolución saltacional, más que por evolución gradual.[303] Stephen Jay Gould expuso en 1977 que los genes o secuencias reguladoras ofrecían cierto apoyo a los postulados de Goldschmidt. De hecho, arguyó que los ejemplos de evolución rápida no minan el darwinismo ―como Goldschmidt suponía― pero tampoco merecen un descrédito inmediato, como muchos neodarwinistas pensaban.[306] Gould insistió que la creencia de Charles Darwin en el gradualismo no fue jamás un componente esencial de su teoría de evolución por selección natural. Thomas Henry Huxley también advirtió a Darwin que había sobrecargado su trabajo con una innecesaria dificultad al adoptar sin reservas el principio Natura non facit saltum.[307] Huxley temía que ese supuesto podría desalentar a aquellos naturalistas que creían que los cataclismos y los grandes saltos evolutivos jugaban un papel significativo en la historia de la vida. En este sentido, Gould escribió:
Como un darwinista, deseo defender el postulado de Goldschmidt de que la macroevolución no es simplemente la microevolución extrapolada y que las grandes transiciones estructurales pueden ocurrir rápidamente sin una serie suave de estados intermedios... En su libro de 1940, Goldschmidt específicamente invoca los genes para el desarrollo como potenciales fabricantes de monstruos prometedores.[306]
La síntesis de la biología del desarrollo y la teoría de la evolución

Aunque Darwin discutió en detalle la biología del desarrollo —antes llamada embriología—, esta rama de la biología no contribuyó a la síntesis evolutiva. Ernst Mayr, en su ensayo What was the evolutionary synthesis? («Qué fue la síntesis evolutiva?»),[14] explicó que varios de los embriólogos del período en que surgió la síntesis moderna tenían una postura contraria a la teoría evolutiva:
«Los representantes de algunas disciplinas biológicas, por ejemplo, de la biología del desarrollo, ofrecieron una fuerte resistencia a la síntesis. No se les dejó fuera de la síntesis, como algunos de ellos ahora reclaman, simplemente no querían unirse».
En las dos últimas décadas, sin embargo, la biología del desarrollo y la biología evolutiva se han unido para formar una nueva rama de la investigación biológica llamada Biología evolutiva del desarrollo o, coloquialmente, «Evo-devo», que explora cómo han evolucionado los procesos del desarrollo y cómo ha surgido la organización de las diversas partes del cuerpo de los organismos antiguos y actuales.[308]
El principal descubrimiento responsable de la integración de la biología del desarrollo con la teoría de la evolución fue el de un grupo de genes reguladores, la familia de genes homeóticos (genes HOX). Estos genes codifican proteínas de unión al ADN (factores de transcripción) que influyen profundamente en el desarrollo embrionario.[309] Por ejemplo, la supresión de las extremidades abdominales de los insectos está determinada por los cambios funcionales en una proteína llamada Ultrabithorax, que es codificada por un gen Hox.[310] La familia de genes Hox existe en los artrópodos (insectos, crustáceos, quelicerados, miriápodos), cordados (peces, anfibios, reptiles, aves, mamíferos), y hay análogos entre las especies de plantas y hongos.[309] Los genes HOX influyen en la morfogénesis de los embriones de los vertebrados al expresarse en diferentes regiones a lo largo del eje anteroposterior del cuerpo. Esta familia de genes es homóloga tanto funcional como estructuralmente al complejo homeótico (HOM-C) de Drosophila melanogaster. Sobre la base de la comparación de genes de varios taxones, se ha logrado reconstruir la evolución de los grupos de genes HOX en vertebrados. Los 39 genes que comprenden la familia de genes HOX en humanos y ratones, por ejemplo, están organizados en cuatro complejos genómicos localizados en diferentes cromosomas, HOXA en el brazo corto del cromosoma 7, HOXB en el 17, HOXC en el 12 y HOXD en el 2, y cada uno de ellos comprende de 9 a 11 genes acomodados en una secuencia homóloga a la del genoma de D. melanogaster.[311] Aunque el ancestro común del ratón y del humano vivió hace alrededor de 75 millones de años, la distribución y arquitectura de sus genes HOX son idénticas.[312] Por lo tanto, la familia de genes HOX es muy antigua y aparentemente muy conservada, lo que tiene profundas implicaciones para la evolución de los patrones y procesos de desarrollo.[312][308][309]
Microbiología y transferencia horizontal de genes
Las primeras teorías evolucionistas prácticamente ignoraron la microbiología, debido a la escasez de rasgos morfológicos y la falta de un concepto de especie particularmente entre los procariotas.[313] Los recientes avances en el estudio de la genómica microbiana han contribuido a comprender mejor la fisiología y ecología de estos organismos y a facilitar la investigación de su la taxonomía y evolución.[314] Estos estudios han revelado niveles de diversidad totalmente inesperados entre los microbios.[315][316]
Particularmente importante fue el descubrimiento en 1959 en Japón de la transferencia horizontal de genes.[317] El intercambio de material genético entre diferentes especies de bacterias ha desempeñado un papel señalado en la propagación de la resistencia a los antibióticos,[318] y, al albor de un mejor conocimiento de los genomas, ha surgido la teoría de que la importancia de la transferencia horizontal de material genético en la evolución no se limita a los microorganismos, sino que alcanza a todos los seres vivos.[319] Los altos niveles de transferencia horizontal de genes han llevado a cuestionar el árbol genealógico de los organismos:[23][24] en efecto, como parte de la teoría endosimbiótica del origen de los orgánulos, la transferencia horizontal de genes fue un paso crítico en la evolución de eucariotas como los hongos, las plantas y los animales.[320][49]
Endosimbiosis y el origen de las células eucariotas
La evolución de los primeros eucariontes desde la condición antecedente procariota ha recibido una considerable atención por parte de los científicos.[321] Este acontecimiento clave en la historia de la vida se produjo hace alrededor de 2000 a 1400 millones de años durante el Proterozoico temprano. Dos hipótesis mutuamente no excluyentes se han enunciado para explicar el origen de los eucariotas: la endosimbiosis y la autogénesis. La hipótesis (también llamada teoría) de la endosimbiosis indica que la evolución de las primeras células eucariotas es el resultado de la incorporación permanente de lo que alguna vez fueron células procariotas fisiológicamente diferentes y autónomas, dentro de una arquea como célula hospedadora. De acuerdo con este concepto, las mitocondrias han evolucionado de una cierta forma de antigua proteobacteria aerobia, mientras que los cloroplastos evolucionaron de alguna forma de procariota del tipo de las cianobacterias.
La explicación de estos procesos simbiogenéticos seguiría estos pasos: en un principio, un individuo entraría en contacto con una bacteria, al comienzo esa relación podría ser parasitaria, pero con el tiempo ambos individuos podrían llegar a una relación mutualista, el hospedador encontraría ventajas en las características y especialidades del hospedado. De no llegar a este punto la selección natural penalizaría esta relación, disminuyendo paulatinamente el número de estos individuos en el conjunto de la población; por el contrario, una relación fructífera, se vería favorecida por la selección natural y los individuos implicados proliferarían. Finalmente la estrecha relación de ambos se vería plasmada en la herencia genética del individuo resultante; este individuo portaría parte o el conjunto de los dos genomas originales.[322][323] En contraste, la hipótesis autogénica sostiene que las mitocondrias y los cloroplastos ―así como otros orgánulos eucariotas tales como el retículo endoplasmático― se desarrollaron como consecuencia de las presiones de selección para la especialización fisiológica dentro de una antigua célula procariota. Según esta hipótesis, la membrana de la célula hospedadora se habría invaginado para encapsular porciones fisiológicamente diferentes de la célula ancestral. Con el transcurso del tiempo, estas regiones unidas a la membrana se convirtieron en estructuras cada vez más especializadas hasta conformar los diferentes orgánulos que actualmente definen la célula eucariota.[324] No obstante, numerosos hechos, tales como la estructura de la membrana, el tipo de reproducción, la secuencia de ADN y la susceptibilidad a los antibióticos de los cloroplastos y de las mitocondrias tienden a sustentar la hipótesis simbiogenética.[325]
Variaciones en la expresión de los genes involucrados en la herencia
También existen formas de variación hereditaria que no están basadas en cambios de la información genética; pero sí en el proceso de decodificación de ella. El proceso que produce estas variaciones deja intacta la información genética y es con frecuencia reversible. Este proceso es llamado herencia epigenética que resulta de la trasmisión de secuencias de información no-ADN a través de la meiosis o mitosis; y puede incluir fenómenos como la metilación del ADN o la herencia estructural. Se sigue investigando si estos mecanismos permiten la producción de variaciones específicas beneficiosas en respuesta a señales ambientales. De ser éste el caso, algunas instancias de la evolución podrían ocurrir fuera del cuadro típicamente darwiniano, que evitaría cualquier conexión entre las señales ambientales y la producción de variaciones hereditarias; aunque recordando que indirectamente el origen del proceso en sí mismo, si estarían involucrados genes, como por ejemplo los genes de la enzima ADN-metiltransferasa, histonas, etc.
Experimentos y estudios sobre el proceso evolutivo
Observación directa del proceso evolutivo en bacterias
Al respecto, Richard Lenski, Profesor de la Michigan State University, lleva a cabo desde 1989 un experimento sobre evolución utilizando bacterias; debido a la rápida reproducción de estos microorganismos. En 1989, Lenski tomó de un congelador de –80 °C un vial que contenía una cepa de Escherichia coli; con el objetivo de observar si se producía alguna diferencia entre la cepa original y sus descendientes. En el experimento se establecieron subcultivos, los cuales se cultivan siempre en las mismas condiciones, y de los cuales cada 75 días se procede a tomar una muestra de cada uno de ellos y congelarla; procediendo del mismo modo para subcultivos de estos subcultivos. Cada 75 días transcurre unas 500 generaciones desde el comienzo del experimento, por lo que actualmente ya han pasado más de 40 000 generaciones desde la cepa original.
Tras 10 000 generaciones las bacterias ya mostraron bastantes diferencias con su ancestro. Las bacterias evolucionadas al compararlas con la cepa original, eran ahora más grandes y se dividían mucho más rápidamente en medio DM (medio de cultivo utilizado para el experimento). El "cambio evolutivo" más importante observado hasta el momento consistió en que uno de los subcultivos en la generación 31 500 de la E. Coli (que son citrato negativo) comenzaron a metabolizar el citrato, por lo que aprovechaban mucho mejor el medio DM y por ello pueden crecer más en este medio. Así, las E. coli de ese subcultivo evolucionaron en mutantes Citrato positivas. Otro cambio evolutivo importante es que un subcultivo, después de pasadas 20 000 generaciones, sufrió un cambio en su velocidad de mutación, provocando que estas mutaciones se incrementaran y se acumularan en su genoma (fenotipo hipermutable). Al crecer en un medio ambiente constante, la mayor parte de las nuevas mutaciones fueron de carácter neutral (mutaciones neutrales), pero también se comenzó a notar un incremento de las mutaciones beneficiosas en los descendientes de este subcultivo.[326]
Los resultados de Lenski propiciaron que se establecieran otros experimentos parecidos pero con diferentes condiciones: temperatura, otras fuentes de carbono, presencia de antibióticos; o con diferentes microorganismos: Pseudomonas fluorescens, Myxococcus xanthus, e incluso levaduras; y en todos ellos se encontraban resultados parecidos. Los microorganismos cambiaban, evolucionaban y se adaptaban a las condiciones del cultivo.
Simulación computarizada del proceso de evolución biológica
Salvo excepciones, la evolución biológica es un proceso demasiado lento para ser observado directamente. Para ello se recurre a disciplinas como la Paleontología, Biología evolutiva o Filogenia, entre otras áreas, para lo observación y el estudio indirecto de la evolución. Por ello en 1990 el ecólogo Thomas Ray enfrentó el problema desde otra vertiente; consciente de que era imposible observar en un corto periodo de tiempo los cambios evolutivos más complejos de los organismos vivos, decidió crear una herramienta informática capaz de simularla. Este programa informático se denomina Tierra.
Tierra es una simulación computarizada de la evolución biológica. La idea es simple: crear organismos digitales que vivan en un ambiente digital con recursos digitales. Los organismos son programas a los que se les confiere la característica principal de los organismos vivos: utilizar los recursos disponibles en su ambiente con el objetivo de reproducirse. El ambiente sería la memoria de la computadora y los recursos para proliferar el tiempo en el procesador. Por tanto estos organismos digitales competirán por un espacio en la memoria del ordenador y por un tiempo en el procesador que les capacita para la replicación.[327]
Impactos de la teoría de la evolución

En el siglo XIX, especialmente tras la publicación de El origen de las especies, la idea de que la vida había evolucionado fue un tema de intenso debate académico centrado en las implicaciones filosóficas, sociales y religiosas de la evolución. Hoy en día, el hecho de que los organismos evolucionan es indiscutible en la literatura científica, y la síntesis evolutiva moderna tiene una amplia aceptación entre los científicos.[2] Sin embargo, la evolución sigue siendo un concepto controvertido por algunos grupos religiosos.[329]
Mientras que muchas religiones y grupos religiosos han reconciliado sus creencias con la evolución por medio de diversos conceptos de evolución teísta, hay muchos creacionistas que creen que la evolución se contradice con el mito de creación en su religión.[330][331][332] Como fuera reconocido por el propio Darwin, el aspecto más controvertido de la biología evolutiva son sus implicaciones respecto a los orígenes del hombre.[84] En algunos países ―notoriamente en los Estados Unidos― esta tensión entre la ciencia y la religión ha alimentado la controversia creación―evolución, un conflicto religioso que aún dura centrado en la política y la educación pública.[333] Mientras que otros campos de la ciencia como la cosmología,[334] y las ciencias de la Tierra[335] también se contradicen con interpretaciones literales de muchos textos religiosos, la biología evolutiva se encuentra con una oposición significativamente mayor de muchos creyentes religiosos.
La evolución ha sido utilizada para apoyar posiciones filosóficas que promueven la discriminación y el racismo. Por ejemplo, las ideas eugenésicas de Francis Galton fueron desarrolladas para argumentar que el patrimonio génico humano debería ser mejorado a través de políticas de mejoramiento genético, incluyendo incentivos para que se reproduzcan aquellos que son considerados con «buenos» genes, y la esterilización forzosa, pruebas prenatales, contracepción e, incluso, la eliminación de las personas consideradas con «malos» genes.[336] Otro ejemplo de una extensión de la teoría de la evolución que actualmente es considerada infundada es el darwinismo social, un término referido a la teoría malthusiana del partido Whig, desarrollada por Herbert Spencer en frases publicitarias tales como «la supervivencia del más apto» y por otras afirmaciones acerca de que la desigualdad social, el racismo y el imperialismo se encuentran justificados por la teoría evolutiva. Sin embargo, los científicos y filósofos contemporáneos consideran que estas ideas no se hallan implícitas en la teoría evolutiva ni están respaldadas por la información disponible.[337][338][339]
A medida que se ha ido desarrollando la comprensión de los fenómenos evolutivos, ciertas posturas y creencias bien arraigadas se han visto revisadas, vulneradas o por lo menos cuestionadas. La aparición de la teoría evolutiva marcó un hito, no solo en su campo de pertinencia, al explicar los procesos que originan la diversidad del mundo vivo; sino también más allá del ámbito de las ciencias biológicas. Naturalmente, este concepto biológico choca con las explicaciones tradicionalmente creacionistas y fijistas de algunas posturas religiosas y místicas y ―de hecho― aspectos como el de la descendencia de un ancestro común, aún suscitan reacciones en algunas personas.[64] El impacto más importante de la teoría evolutiva se da a nivel de la historia del pensamiento moderno y la relación de este con la sociedad. Este profundo impacto se debe, en definitiva, a la naturaleza no teleológica de los mecanismos evolutivos: la evolución no sigue un fin u objetivo. Las estructuras y especies no «aparecen» por necesidad ni por designio divino sino que a partir de la variedad de formas existentes solo las más adaptadas se conservan en el tiempo.[340] Este mecanismo «ciego», independiente de un plan, de una voluntad divina o de una fuerza sobrenatural ha sido en consecuencia explorado en otras ramas del saber. La adopción de la perspectiva evolutiva para abordar problemas en otros campos se ha mostrado enriquecedora y muy vigente; sin embargo en el proceso también se han dado abusos ―atribuir un valor biológico a las diferencias culturales y cognitivas― o deformaciones de la misma ―como justificativo de posturas eugenéticas― las cuales han sido usadas como «Argumentum ad consequentiam» a través de la historia de las objeciones a la teoría de la evolución.[341]
Evolución y religión
Antes de que la geología se convirtiera en una ciencia, a principios del siglo XIX, tanto las religiones occidentales como los científicos descontaban o condenaban de manera dogmática y casi unánime cualquier propuesta que implicara que la vida es el resultado de un proceso evolutivo. Sin embargo, a medida que la evidencia geológica empezó a acumularse en todo el mundo, un grupo de científicos comenzó a cuestionar si una interpretación literal de la creación relatada en la Biblia judeo-cristiana podía reconciliarse con sus descubrimientos (y sus implicaciones). Algunos geólogos religiosos, como Dean William Auckland en Inglaterra, Edward Hitchcock en Estados Unidos y Hugo Millar en Escocia siguieron justificando la evidencia geológica y fósil sólo en términos de un Diluvio universal; pero una vez que Charles Darwin publicara su Origen de las especies en 1859 la opinión científica comenzó a alejarse rápidamente de la interpretación literal de la Biblia. Este debate temprano acerca de la validez literal de la Biblia no se llevó a cabo tras puertas cerradas, y desestabilizó la opinión educativa en ambos continentes. Eventualmente, instigó una contrarreforma que tomó la forma de un renacimiento religioso en ambos continentes entre 1857 y 1860.[342]
En los países o regiones en las que la mayoría de la población mantiene fuertes creencias religiosas, el creacionismo posee un atractivo mucho mayor que en los países donde la mayoría de la gente posee creencias seculares. Desde los años 1920 hasta el presente en los Estados Unidos, han ocurrido varios ataques religiosos a la enseñanza de la teoría evolutiva, particularmente por parte de cristianos fundamentalistas evangélicos o pentecostales. A pesar de las abrumadoras evidencias que avalan la teoría de la evolución,[343] algunos grupos interpretan en la Biblia que un ser divino creó directamente a los seres humanos, y a cada una de las otras especies, como especies separadas y acabadas. Este punto de vista es comúnmente llamado creacionismo, y sigue siendo defendido por algunos grupos integristas religiosos, particularmente los protestantes estadounidenses; principalmente a través de una forma de creacionismo llamada diseño inteligente. Los «lobbies» religiosos-creacionistas desean excluir la enseñanza de la evolución biológica de la educación pública de ese país; aunque actualmente es un fenómeno más bien local, ya que la enseñanza de base en ciencias es obligatoria dentro de los currículos, las encuestas revelan una gran sensibilidad del público estadounidense a este mensaje, lo que no tiene equivalente en ninguna otra parte del mundo. Uno de los episodios más conocidos de este enfrentamiento se produjo a finales de 2005 en Kansas, donde el Consejo de Educación del Estado de Kansas (en inglés: Kansas State Board of Education) decidió permitir que se enseñaran las doctrinas creacionistas como una alternativa de la teoría científica de la evolución.[344] Tras esta decisión se produjo una fuerte respuesta ciudadana, que tuvo una de sus consecuencias más conocidas en la creación de una parodia de religión, el pastafarismo, una invención de Bobby Henderson, licenciado en Física de la Universidad Estatal de Oregón, para demostrar irónicamente que no corresponde y es equivocado enseñar el diseño inteligente como teoría científica. Posteriormente, el Consejo de Educación del Estado de Kansas revocó su decisión en agosto de 2006.[345] Este conflicto educativo también ha afectado a otros países; por ejemplo, en el año 2005 en Italia hubo un intento de suspensión de la enseñanza de la teoría de la evolución.[346][347]
En respuesta a la aceptación científica de la teoría de la evolución, muchos religiosos y filósofos han tratado de unificar los puntos de vista científico y religioso, ya sea de manera formal o informal; a través de un «creacionismo pro-evolución». Así por ejemplo algunos religiosos han adoptado un enfoque creacionista desde la evolución teísta o el creacionismo evolutivo, y defienden que Dios provee una chispa divina que inicia el proceso de la evolución, y (o) donde Dios creó el curso de la evolución.
A partir de 1950 la Iglesia católica tomó una posición neutral con respecto a la evolución con la encíclica Humani generis del papa Pío XII.[348] En ella se distingue entre el alma, tal como fue creada por Dios, y el cuerpo físico, cuyo desarrollo puede ser objeto de un estudio empírico:
... el Magisterio de la Iglesia no prohíbe el que ―según el estado actual de las ciencias y la teología― en las investigaciones y disputas, entre los hombres más competentes de entrambos campos, sea objeto de estudio la doctrina del evolucionismo, en cuanto busca el origen del cuerpo humano en una materia viva preexistente ―pero la fe católica manda defender que las almas son creadas inmediatamente por Dios―. Mas todo ello ha de hacerse de manera que las razones de una y otra opinión ―es decir la defensora y la contraria al evolucionismo― sean examinadas y juzgadas seria, moderada y templadamente; y con tal que todos se muestren dispuestos a someterse al juicio de la Iglesia, a quien Cristo confirió el encargo de interpretar auténticamente las Sagradas Escrituras y defender los dogmas de la fe.[349]
Por otro lado, la encíclica no respalda ni rechaza la creencia general en la evolución debido a que se consideró que la evidencia en aquel momento no era convincente. Permite, sin embargo, la posibilidad de aceptarla en el futuro:
No pocos ruegan con insistencia que la fe católica tenga muy en cuenta tales ciencias; y ello ciertamente es digno de alabanza, siempre que se trate de hechos realmente demostrados; pero es necesario andar con mucha cautela cuando más bien se trate sólo de hipótesis, que, aun apoyadas en la ciencia humana, rozan con la doctrina contenida en la Sagrada Escritura o en la tradición.[350]
En 1996, Juan Pablo II afirmó que «la teoría de la evolución es más que una hipótesis» y recordó que «El Magisterio de la Iglesia está interesado directamente en la cuestión de la evolución, porque influye en la concepción del hombre».[351] El papa Benedicto XVI ha afirmado que «existen muchas pruebas científicas en favor de la evolución, que se presenta como una realidad que debemos ver y que enriquece nuestro conocimiento de la vida y del ser como tal. Pero la doctrina de la evolución no responde a todos los interrogantes y sobre todo no responde al gran interrogante filosófico: ¿de dónde viene todo esto y cómo todo toma un camino que desemboca finalmente en el hombre?».[352]
La reacción musulmana a la teoría de la evolución fue sumamente variada, desde aquellos que creían en una interpretación literal de la creación según el Corán, hasta la de muchos musulmanes educados que suscribieron a una versión de evolución teísta o guiada, en la que el Corán reforzaba más que contradecía a la ciencia. Esta última reacción se vio favorecida debido a que Al-Jahiz, un erudito musulmán del siglo IX, había propuesto un concepto similar al de la selección natural. [353]
Sin embargo, la aceptación de la evolución sigue siendo baja en el mundo musulmán ya que figuras prominentes rechazan la teoría evolutiva como una negación de Dios y como poco fiable para explicar el origen de los seres humanos.[353] Otras objeciones de los eruditos y escritores musulmanes reflejan en gran medida las presentadas en el mundo occidental.[354]
Independientemente de su aceptación por las principales jerarquías religiosas, las mismas objeciones iniciales a la teoría de Darwin siguen siendo utilizadas en contra de la teoría evolutiva actual. Las ideas de que las especies cambian con el tiempo a través de procesos naturales y que distintas especies comparten sus ancestros parece contradecir el relato del Génesis de la Creación. Los creyentes en la infalibilidad bíblica atacaron al darwinismo como una herejía. La teología natural del siglo XIX se caracterizó por la analogía del relojero de William Paley, un argumento de diseño todavía utilizado por el movimiento creacionista. Cuando la teoría de Darwin se publicó, las ideas de la evolución teísta se presentaron de modo de indicar que la evolución es una causa secundaria abierta a la investigación científica, al tiempo que mantenían la creencia en Dios como causa primera, con un rol no especificado en la orientación de la evolución y en la creación de los seres humanos.[355]
Otras teorías de la evolución y críticas científicas de la teoría sintética
Richard Dawkins, en su obra El gen egoísta de 1976, hizo la siguiente afirmación:
Hoy la teoría de la evolución está tan sujeta a la duda como la teoría de que la Tierra gira alrededor del Sol.[356]
La evolución biológica es un hecho aceptado desde el siglo XVIII, la teoría que la explica ha tratado de ser debatida científicamente.[357] La teoría vigente que explica la evolución biológica, denominada teoría de la síntesis evolutiva moderna (o simplemente teoría sintética), es el modelo actualmente aceptado por la comunidad científica para describir los fenómenos evolutivos; y aunque no existe hoy una sólida teoría alternativa desarrollada, algunos científicos[¿quién?] han reclamado la necesidad de realizar una reforma, ampliación o sustitución de la teoría sintética, con nuevos modelos capaces de integrar por ejemplo la biología del desarrollo o incorporar dentro de la teoría actual una serie de descubrimientos biológicos cuyo papel evolutivo se está debatiendo; tales como ciertos mecanismos hereditarios epigenéticos, la transferencia horizontal de genes, o propuestas como la existencia de múltiples niveles jerárquicos de selección o la plausibilidad de los fenómenos de asimilación genómica para explicar procesos macroevolutivos.
Los aspectos más criticados y debatidos de la teoría de la síntesis evolutiva moderna son: el gradualismo, que ha obtenido como respuesta el modelo del equilibrio puntuado de Niles Eldredge y Stephen Jay Gould;[358] la preponderancia de la selección natural frente a los motivos puramente estocásticos; la explicación al comportamiento del altruismo y el reduccionismo geneticista que evitaría las implicaciones holísticas y las propiedades emergentes a cualquier sistema biológico complejo.[359] A pesar de lo indicado, sin embargo, hay que considerar que el actual consenso científico es que la teoría misma (en sus fundamentos) no ha sido rebatida en el campo de la biología evolutiva, siendo solo perfeccionada; y por ello se la sigue considerando como la «piedra angular de la biología moderna».[360][361]
Otras hipótesis minoritarias
Entre otras hipótesis minoritarias, destaca la de la bióloga estadounidense Lynn Margulis, quién consideró que del mismo modo que las células eucariotas surgieron a través de la interacción simbiogenética de varias células procariotas, muchas otras características de los organismos y el fenómeno de especiación serían la consecuencia de interacciones simbiogenéticas similares. En su obra Captando Genomas. Una teoría sobre el origen de las especies Margulis argumentó que la simbiogénesis sería la fuerza principal en la evolución. Según su teoría, la adquisición y acumulación de mutaciones al azar no serían suficientes para explicar cómo se producen variaciones hereditarias, sino que ella postulo que las organelas, los organismos y las especies surgirían como el resultado de la simbiogénesis.[323] Mientras que la síntesis evolutiva moderna hace hincapié en la "competencia" como la principal fuerza detrás de la evolución, Margulis postulo a la cooperación como el motor del cambio evolutivo.[362] Argumenta que las bacterias, junto con otros microorganismos, ayudaron a crear las condiciones que se requieren para la vida, tales como el oxígeno. Margulis sostuvo que estos microorganismos constituirían un componente importante de la biomasa de la Tierra y que constituirían la principal razón por la que las condiciones actuales se mantienen. Afirmó, asimismo, que las bacterias son capaces de intercambiar genes con mayor rapidez y facilidad que los eucariotas, y debido a esto, son más versátiles, por lo que estas serían las artífices de la complejidad de los seres vivos.[323]
Igualmente, Máximo Sandín, por otra parte, rechazó vehementemente cualquiera de las versiones del darwinismo presentes en la actual teoría y propuso una hipótesis alternativa para explicar el hecho de la evolución. En primer lugar, justiprecia la obra de Lamarck, revisando las hipótesis o predicciones (conocidas como Lamarckismo) que fueran realizadas por este biólogo; y que actualmente según Sandín, se verían corroboradas por los hechos.[363][364] Por ejemplo, Sandín formuló su hipóteis a partir del hecho que las circunstancias ambientales pueden condicionar, no sólo la expresión de la información genética (fenómenos epigenéticos, control del splicing alternativo, estrés genómico…), sino la dinámica del proceso de desarrollo embrionario,[365][366][367][368] y postula que el cimiento fundamental de los ecosistemas es el equilibrio y no la competencia.[369][370] Conforme a sus ideas, él postula que se puede apreciar la tendencia de las formas orgánicas a una mayor complejidad,[371][372] y ello sería por la existencia de unas «leyes» (es decir, no «el azar») que gobiernan la variabilidad de los organismos;[373][374] y que la capacidad de estos cambios están, de alguna manera, inscritas en los organismos.[375][376] Habida cuenta que el 98,5% del genoma humano, por ejemplo, está compuesto por secuencias repetidas con función reguladora así como una notable cantidad de virus endógenos; y a partir de ello, Sandín propone en su hipótesis que esa conformación del genoma no puede ser el resultado del azar y de la selección natural. Rechazando además la tesis del "ADN egoísta" de Dawkins, Sandín sugiere que ello se produciría por la presión del medio ambiente, lo que provocaría que ciertos virus se inserten en el genoma o determinadas secuencias génicas se modifiquen y, como consecuencia, se generen organismos completamente nuevos; con sustanciales diferencias con respecto a sus predecesores. Según esta teoría, entonces, el mecanismo fundamental del cambio evolutivo no es la selección natural ni la mutación aleatoria; sino solo la capacidad de integración de los virus (en genomas ya existentes (mediante la transferencia horizontalde sus genes). Además, Sandín señala que el medio ambiente, y no las mutaciones aleatorias, provocarían que determinados grupos de seres vivos asuman muevas características, las que ―por otro lado― no serían cambios graduales sino cambios bruscos, en episodios específicos y sin fases intermedias.[377][357] Según Abdalla, la hipótesis sostenida por Sandín estaría fundamentada por una gran cantidad de datos científicos y abríría una nueva área de investigación en el campo de la biología.[357]
Véase también
- Evolución experimental
- Evolución humana
- Evolución convergente
- Sociedad Española de Biología Evolutiva (SESBE)
- Dinámica de sistemas
- Teoría de sistemas
- Gran Historia
Referencias
- ↑ Hall, B. K., Hallgrímsson, B. (2008). Strickberger's Evolution (4th edición). Jones & Bartlett. p. 762. ISBN 0763700665. Archivado desde el original el 28 de junio de 2012.
- ↑ a b c d e f g h i j k l m n Kutschera, U., Karl J. Niklas (2004). «The modern theory of biological evolution: an expanded synthesis». Naturwissenschaften (en inglés) 91 (6): 255-276. doi:10.1007/s00114-004-0515-y.
- ↑ Hall y Hallgrímsson, 2008
- ↑ Doolittle, W. Ford (febrero de 2000). «Uprooting the Tree of Life». Scientific American (Londres: Nature Publishing Group) 282 (2): 90-95. ISSN 0036-8733. PMID 10710791. doi:10.1038/scientificamerican0200-90.
- ↑ Glansdorff, Nicolas; Ying Xu, Bernard (9 de julio de 2008). «The Last Universal Common Ancestor: emergence, constitution and genetic legacy of an elusive forerunner». Biology Direct (Londres: BioMed Central) 3: 29. ISSN 1745-6150. PMC 2478661. PMID 18613974. doi:10.1186/1745-6150-3-29.
- ↑ Bonnett, Charles. 1762. Consideration sur les corps organisés. Marc Michel-Rey, Amsterdam.
- ↑ Richards, R. 1993. The meaning of evolution: the morphological construction and ideological reconstruction of Darwin's theory. The University of Chicago Press. ISBN 02267120396. Pág. 10.
- ↑ a b Mayr, Ernst (1982). The Growth of Biological Thought: Diversity, Evolution, and Inheritance (en inglés). Cambridge, Mass.: The Belknap Press of Harvard University Press. ISBN 0-674-36445-7.
- ↑ Darwin, C. (1872) On the origin of species (6.ª ed.). John Murray, Londres.
- ↑ a b Darwin, Charles (1859), On the origin of species by means of natural selection, or the preservation of favoured races in the struggle for life (1.ª edición), Londres: John Murray.
- ↑ BBC Mundo (10 de marzo de 2019): Al-Jahiz, el filósofo musulmán que tuvo la idea de la evolución 1.000 años antes que Darwin. BBC Mundo. Consultado el 10 de marzo de 2019.
- ↑ a b Darwin, Charles; Wallace, Alfred Russel (1858). «On the tendency of species to form varieties; and on the perpetuation of varieties and species by natural means of selection». Journal of the Proceedings of the Linnean Society of London. Zoology 3: 45-62.
- ↑ Cordón, Faustino (1982). La evolución conjunta de los animales y su medio. Anthropos Editorial. p. 15. ISBN 9788485887064.
- ↑ a b Mayr, E. (1993) «What was the evolutionary synthesis?» Trends Ecol Evol 8:31-34.
- ↑ Mayr, E. (2001) What evolution is. Basic Books, Nueva York. ISBN 0-465-04426-3
- ↑ Mahner, M., Bunge, M. (1997) Foundations of biophilosophy. Springer, Berlín, Heidelberg, Nueva York. ISBN 3-540-61838-4
- ↑ Penny, D.; Poole, A. (1999). «The nature of the last universal common ancestor». Curr. Opin. Genet. Dev. 9 (6): 672-77. PMID 10607605. doi:10.1016/S0959-437X(99)00020-9.
- ↑ Bapteste, E.; Walsh, D. A. (2005). «Does the 'Ring of Life' ring true?». Trends Microbiol. 13 (6): 256-61. PMID 15936656. doi:10.1016/j.tim.2005.03.012.
- ↑ «Biogeography: Wallace and Wegener». Understanding evolution (en inglés). Universidad de Berkeley. Consultado el 12 de julio de 2018.
- ↑ «Homologies». Understanding evolution (en inglés). Universidad de Berkeley. Consultado el 14 de julio de 2018.
- ↑ «Learning about evolutionary history». Understanding evolution (en inglés). Universidad de Berkeley. Consultado el 17 de julio de 2018.
- ↑ Mayr, E. (1942). Systematics and the origin of species, Columbia University Press, New York.
- ↑ a b Doolittle, W. F.; Bapteste, E. (febrero de 2007). «Pattern pluralism and the Tree of Life hypothesis». Proc. Natl. Acad. Sci. U.S.A. 104 (7): 2043-9. PMC 1892968. PMID 17261804. doi:10.1073/pnas.0610699104.
- ↑ a b Kunin, V.; Goldovsky, L.; Darzentas, N.; Ouzounis, C. A. (2005). «The net of life: reconstructing the microbial phylogenetic network». Genome Res. 15 (7): 954-9. PMC 1172039. PMID 15965028. doi:10.1101/gr.3666505.
- ↑ Jablonski, D. (1999). «The future of the fossil record». Science 284 (5423): 2114-16. PMID 10381868. doi:10.1126/science.284.5423.2114.
- ↑ a b Mason, S. F. (1984). «Origins of biomolecular handedness». Nature 311 (5981): 19-23. PMID 6472461. doi:10.1038/311019a0.
- ↑ Wolf, Y. I.; Rogozin, I. B.; Grishin, N. V.; Koonin, E. V. (2002). «Genome trees and the tree of life». Trends Genet. 18 (9): 472-79. PMID 12175808. doi:10.1016/S0168-9525(02)02744-0.
- ↑ Varki, A.; Altheide, T. K. (2005). «Comparing the human and chimpanzee genomes: searching for needles in a haystack». Genome Res. 15 (12): 1746-58. PMID 16339373. doi:10.1101/gr.3737405.
- ↑ Kover, Paula. «The five most common misunderstandings about evolution». The Conversation (en inglés). Consultado el 17 de julio de 2018.
- ↑ Peretó, J. (2005). «Controversies on the origin of life». Int. Microbiol. 8 (1): 23-31. PMID 15906258.
- ↑ Alberts, J. y col. (2002). Molecular Biology of the Cell, 4.ª edición, Routledge, ISBN 0-8153-3218-1.
- ↑ Luisi, P. L., Ferri, F., Stano, P. (2006). «Approaches to semi-synthetic minimal cells: a review». Naturwissenschaften 93 (1): 1-13. PMID 16292523. doi:10.1007/s00114-005-0056-z.
- ↑ Trevors, J. T., Abel, D. L. (2004). «Chance and necessity do not explain the origin of life». Cell Biol. Int. 28 (11): 729-39. PMID 15563395. doi:10.1016/j.cellbi.2004.06.006.Forterre, P., Benachenhou-Lahfa, N., Confalonieri, F., Duguet, M., Elie, C., Labedan, B. (1992). «The nature of the last universal ancestor and the root of the tree of life, still open questions». BioSystems 28 (1-3): 15-32. PMID 1337989. doi:10.1016/0303-2647(92)90004-I.
- ↑ Cowen, R. (2000). History of Life (en inglés) (3rd edición). Blackwell Science. p. 6. ISBN 0632044446.
- ↑ Orgel, L. E. (octubre de 1994). «The origin of life on the earth» (PDF). Scientific American (en inglés) 271 (4): 76-83. PMID 7524147. doi:10.1038/scientificamerican1094-76. Consultado el 30 de agosto de 2008.
- ↑ Joyce, G. F. (2002). «The antiquity of RNA-based evolution». Nature 418 (6894): 214-21. PMID 12110897. doi:10.1038/418214a.
- ↑ Trevors, J. T., Psenner, R. (2001). «From self-assembly of life to present-day bacteria: a possible role for nanocells». FEMS Microbiol. Rev. 25 (5): 573-82. PMID 11742692. doi:10.1111/j.1574-6976.2001.tb00592.x.
- ↑ Beech, Martin (2016). The Pillars of Creation: Giant Molecular Clouds, Star Formation, and Cosmic Recycling (en inglés). Springer. pp. 239-241. ISBN 9783319487755.
- ↑ Ciccarelli, F. D., Doerks, T., von Mering, C., Creevey, C. J., Snel, B., Bork, P. (2006). «Toward automatic reconstruction of a highly resolved tree of life». Science 311 (5765): 1283-7. PMID 16513982.
- ↑ Shen, Y. y Buick, R. (2004). «The antiquity of microbial sulfate reduction». Earth-Science Reviews 64: 243-272. Archivado desde el original el 11 de agosto de 2011. Consultado el 16 de mayo de 2010.
- ↑ Schopf, J. W. (1993). «Microfossils of the Early Archean Apex chert: new evidence of the antiquity of life», en Science 260:640-646.
- ↑ a b c d Schopf, J. W. (1999). Cradle of life: the discovery of Earth’s earliest fossils. Princeton (Nueva Jersey): Princeton University Press. ISBN 0-691-00230-4.
- ↑ a b c d Knoll, A. H. (2003) Life on a young planet: the first three billion years of evolution on earth. Princeton University Press, Princeton, N.J. ISBN 0-691-12029-3
- ↑ Shen, Y., Buick, R., Canfield, D. E. (2001) Isotopic evidence for microbial sulphate reduction in the early Archean era. Nature 410:77-81.
- ↑ Lang, B., Gray, M., Burger, G. (1999). «Mitochondrial genome evolution and the origin of eukaryotes». Annu Rev Genet 33: 351-97. PMID 10690412. doi:10.1146/annurev.genet.33.1.351.
McFadden, G (1999). «Endosymbiosis and evolution of the plant cell». Curr Opin Plant Biol 2 (6): 513-19. PMID 10607659. doi:10.1016/S1369-5266(99)00025-4. - ↑ a b Cavalier-Smith, T. (2006). «Cell evolution and Earth history: stasis and revolution». Philos Trans R Soc Lond B Biol Sci 361 (1470): 969-1006. PMC 1578732. PMID 16754610. doi:10.1098/rstb.2006.1842.
- ↑ Schopf, J. (2006). «Fossil evidence of Archaean life». Philos Trans R Soc Lond B Biol Sci 361 (1470): 869-85. PMC 1578735. PMID 16754604. doi:10.1098/rstb.2006.1834.
Altermann, W., Kazmierczak, J. (2003). «Archean microfossils: a reappraisal of early life on Earth». Res Microbiol 154 (9): 611-17. PMID 14596897. doi:10.1016/j.resmic.2003.08.006. - ↑ Schopf, J. W. (1994). «Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic». Proc Natl Acad Sci U S a 91 (15): 6735-42. PMC 44277. PMID 8041691. doi:10.1073/pnas.91.15.6735.
- ↑ a b Dyall, S., Brown, M., Johnson, P. (2004). «Ancient invasions: from endosymbionts to organelles». Science 304 (5668): 253-57. PMID 15073369. doi:10.1126/science.1094884.
- ↑ Martin, W. (2005). «The missing link between hydrogenosomes and mitochondria». Trends Microbiol. 13 (10): 457-59. PMID 16109488. doi:10.1016/j.tim.2005.08.005.
- ↑ Knoll, A. H. (1999). «A new molecular window on early life.» Science 285:1025-1026.
- ↑ Martin, W., Russell, M. J. (2003). «On the origins of cells: a hypothesis for the evolutionary transitions from abiotic geochemistry to chemoautotrophic prokaryotes, and from prokaryotes to nucleated cells.» Philos Trans R Soc Lond B 358:59-85.
- ↑ DeLong, E., Pace, N. (2001). «Environmental diversity of bacteria and archaea». Syst Biol 50 (4): 470-8. PMID 12116647. doi:10.1080/106351501750435040.
- ↑ a b Narbonne, Guy (junio de 2006). «The Origin and Early Evolution of Animals». Department of Geological Sciences and Geological Engineering, Queen's University. Archivado desde el original el 24 de julio de 2015. Consultado el 10 de marzo de 2007.
- ↑ Droser y Gehling 2008. «Synchronous Aggregate Growth in an Abundant New Ediacaran Tubular Organism.» Science 319: 1660-1662.
- ↑ Kaiser, D. (2001). «Building a multicellular organism». Annu. Rev. Genet. 35: 103-23. PMID 11700279. doi:10.1146/annurev.genet.35.102401.090145.
- ↑ Valentine, J. W., Jablonski, D., Erwin, D. H. (1 de marzo de 1999). «Fossils, molecules and embryos: new perspectives on the Cambrian explosion». Development 126 (5): 851-9. PMID 9927587.
- ↑ Ohno, S. (1997). «The reason for as well as the consequence of the Cambrian explosion in animal evolution». J. Mol. Evol. 44 supl. 1: S23-7. PMID 9071008. doi:10.1007/PL00000055.
- ↑ Valentine, J., Jablonski, D. (2003). «Morphological and developmental macroevolution: a paleontological perspective». Int. J. Dev. Biol. 47 (7-8): 517-22. PMID 14756327.
- ↑ Waters, E. R. (2003). «Molecular adaptation and the origin of land plants». Mol. Phylogenet. Evol. 29 (3): 456-63. PMID 14615186. doi:10.1016/j.ympev.2003.07.018.
- ↑ Gayo Marcio Censorino De Die Natali, IV, 7. Versión en latin, traducción al francés. Consultada el 10 de julio de 2011.
- ↑ Couprie, Dirk L. «Anaximander». Internet Encyclopedia of Philosophy. Consultado el 9 de julio de 2011..
- ↑ Campbell, Gordon. «Empedocles». Internet Encyclopedia of Philosophy. Consultado el 9 de julio de 2011.
- ↑ a b Singer, Charles (1931). A Short History of Biology. Clarendon Press.
- ↑ Johnston, Ian (1999). «Section Three: The Origins of Evolutionary Theory». ... And Still We Evolve: A Handbook on the History of Modern Science. Liberal Studies Department, Malaspina University College. Consultado el 11 de agosto de 2007.
- ↑ a b Lovejoy, Arthur (1936). The Great Chain of Being: A Study of the History of an Idea (en inglés). Harvard University Press. ISBN 0-674-36153-9.
- ↑ Chan, Wing-Tsit (1963). A Source Book In Chinese Philosophy. Princeton University Press. p. 204. ISBN 0-691-01964-9.
- ↑ Needham, Joseph; Ronan, Colin Alistair (1995). The Shorter Science and Civilisation in China: An Abridgement of Joseph Needham's Original Text, Vol. 1. Cambridge University Press. ISBN 0-521-29286-7.
- ↑ «The first theory of evolution is 600 years older than Darwin]». The Vintage News. 27 de agosto de 2016.
- ↑ Sapp, Jan (2003). Genesis: The Evolution of Biology (en inglés). Oxford University Press. ISBN 0-19-515618-8.
- ↑ Province, W. B.; Ernst, Mayr, ed. (1998). The Evolutionary Synthesis: Perspectives on the Unification of Biology (en inglés). Harvard University Press. ISBN 0-674-27225-0.
- ↑ a b c Gould, Stephen Jay (2002). The Structure of Evolutionary Theory (en inglés). Belknap Press of Harvard University Press. ISBN 0-674-00613-5.
- ↑ Moya, A. (1989): Sobre la estructura de la teoría de la evolución. Editorial Anthropos, S. A. Servicio Editorial de la Universidad del País Vasco. Nueva ciencia, 5. 174 págs. ISBN 84-7658-154-8
- ↑ Racine, Valerie, "Essay: The Cuvier-Geoffroy Debate". Embryo Project Encyclopedia (2013-10-07). Essay: The Cuvier-Geoffroy Debate ISSN 1940-5030 Essay: The Cuvier-Geoffroy Debate
- ↑ Étienne Geoffroy Saint Hilaire Collection 1811-1844 Archivado el 7 de enero de 2014 en Wayback Machine.
- ↑ Browne, E. Janet (2002). Charles Darwin: Vol. 2 The Power of Place. Londres: Jonathan Cape. ISBN 0-7126-6837-3.
- ↑ Larson, Edward J. (2004). Evolution: The Remarkable History of a Scientific Theory (en inglés). Modern Library. ISBN 0-679-64288-9.
- ↑ Desmond, Adrian; Moore, James (1994). Darwin: The Life of a Tormented Evolutionist (en inglés). W. W. Norton & Company. ISBN 0393311503.
- ↑ Bowler, Peter J. (2003). Evolution: The History of an Idea (en inglés) (3.ª edición). University of California Press. ISBN 0-52023693-9.
- ↑ Cowan, Ian (11 de julio de 2008). «A Trumpery Affair How Wallace stimulated Darwin to publish and be damned». Proceedings of the Symposium: Wallace and Darwin's Theory of Natural Selection 150 years on - current views.
- ↑ a b Dawkins, R. (2002) «The reading of the Darwin-Wallace papers commemorated – in the Royal Academy of Arts». Linnean, 18:17-24.
- ↑ Kutschera, U. (2003) «A comparative analysis of the Darwin–Wallace papers and the development of the concept of natural selection.» Theory Biosci., 122:343-359.
- ↑ a b Wallace, A. R. (1889) Darwinism. An exposition of the theory of natural selection with some of its applications. MacMillan, London.
- ↑ a b Bowler, Peter J. (2003). Evolution: The History of an Idea (3.ª edición). University of California Press. ISBN 0-520-23693-9.
- ↑ Romanes GJ (1895) Darwin and after Darwin, vol 2. Open Court, Chicago.
- ↑ Weismann, A. (1892) Das Keimplasma. Eine Theorie der Vererbung. Fischer, Jena, Alemania.
- ↑ Mayr, E. (1982) The growth of biological thought: diversity, evolution and inheritance. Harvard University Press, Cambridge, Mass. ISBN 0-674-36446-5
- ↑ Junker, T. (2004) Die zweite Darwinsche Revolution. Geschichte des Synthetischen Darwinismus in Deutschland 1924 bis 1950. Marburg: Basilisken-Presse. Acta Biohistorica, 8. 635 págs. ISBN 3-925347-67-4
- ↑ a b Reif, W.-E., Junker, T., Hoßfeld, U. (2000). «The synthetic theory of evolution: general problems and the German contribution to the synthesis.» Theory Biosci., 119:41-91.
- ↑ «Ask an expert». Nature Education (en inglés). 11 de septiembre de 2012.
- ↑ Huxley, J. S. (1942) Evolution: the modern synthesis. Londres: Allen and Unwin.
- ↑ Ruse, M. (1996) Monad to man: the concept of progress in evolutionary biology. Cambridge, Mass.: Harvard University Press ISBN 0-674-03248-9
- ↑ Huxley, 1942. Óp. cit. p. 13.
- ↑ «Evolution is a change in the genetic composition of populations. The study of the mechanisms of evolution falls within the province of population genetics» (Dobzhansky, 1951).
- ↑ Mayr, E. (1992) «A local flora and the biological species concept». Am. J. Bot., 79:222-238.
- ↑ Beurton, P. J. (2002) «Ernst Mayr through time on the biological species concept: a conceptual analysis». Theory Biosci., 121:81-98.
- ↑ Dobzhansky, 1937, p. 312.
- ↑ Mayr, E. y Diamond, J. (2001) The birds of northern Melanesia. Speciation, Ecology and Biogeography. New York: Oxford University Press. 548 págs. ISBN 978-0-19-514170-2
- ↑ Howard, D. J. y Berlocher, S. H. (eds.) (1998) Endless forms: species and speciation. Nueva York: Oxford University Press
- ↑ Wu, R., Lin, M. (2006). «Functional mapping: how to map and study the genetic architecture of dynamic complex traits». Nat. Rev. Genet. 7 (3): 229-37. PMID 16485021. doi:10.1038/nrg1804.
- ↑ a b Harwood, A. J.; Harwood, J. (1998). «Factors affecting levels of genetic diversity in natural populations». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 353 (1366): 177-86. PMC 1692205. PMID 9533122. doi:10.1098/rstb.1998.0200.
- ↑ Draghi J, Turner P (2006). «DNA secretion and gene-level selection in bacteria». Microbiology (Reading, Engl.) 152 (Pt 9): 2683-8. PMID 16946263. doi:10.1099/mic.0.29013-0.
*Mallet, J. (2007). «Hybrid speciation». Nature 446 (7133): 279-83. PMID 17361174. doi:10.1038/nature05706. - ↑ Butlin, R. K., Tregenza, T. (1998). «Levels of genetic polymorphism: marker loci versus quantitative traits». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 353 (1366): 187-98. PMC 1692210. PMID 9533123. doi:10.1098/rstb.1998.0201.
- ↑ Wetterbom, A., Sevov, M., Cavelier, L., Bergström, T. F. (2006). «Comparative genomic analysis of human and chimpanzee indicates a key role for indels in primate evolution». J. Mol. Evol. 63 (5): 682-90. PMID 17075697. doi:10.1007/s00239-006-0045-7.
- ↑ a b Bertram, J. (2000). «The molecular biology of cancer». Mol. Aspects Med. 21 (6): 167-223. PMID 11173079. doi:10.1016/S0098-2997(00)00007-8.
- ↑ a b Aminetzach, Y. T., Macpherson, J. M., Petrov, D. A. (2005). «Pesticide resistance via transposition-mediated adaptive gene truncation in Drosophila». Science 309 (5735): 764-7. PMID 16051794. doi:10.1126/science.1112699.
- ↑ Burrus, V., Waldor, M. (2004). «Shaping bacterial genomes with integrative and conjugative elements». Res. Microbiol. 155 (5): 376-86. PMID 15207870. doi:10.1016/j.resmic.2004.01.012.
- ↑ Sawyer, S. A., Parsch, J., Zhang, Z., Hartl, D. L. (2007). «Prevalence of positive selection among nearly neutral amino acid replacements in Drosophila». Proc. Natl. Acad. Sci. U.S.A. 104 (16): 6504-10. PMC 1871816. PMID 17409186. doi:10.1073/pnas.0701572104.
- ↑ a b Barbadilla, Antonio, «La genética de poblaciones», Universidad Autónoma de Barcelona.
- ↑ Sniegowski, P., Gerrish, P., Johnson, T., Shaver, A. (2000). «The evolution of mutation rates: separating causes from consequences». Bioessays 22 (12): 1057-66. PMID 11084621. doi:10.1002/1521-1878(200012)22:12<1057::AID-BIES3>3.0.CO;2-W.
- ↑ Drake, J. W., Holland, J. J. (1999). «Mutation rates among RNA viruses». Proc. Natl. Acad. Sci. U.S.A. 96 (24): 13910-3. PMC 24164. PMID 10570172. doi:10.1073/pnas.96.24.13910.
- ↑ Holland, J., Spindler, K., Horodyski, F., Grabau, E., Nichol, S., VandePol, S. (1982). «Rapid evolution of RNA genomes». Science 215 (4540): 1577-85. PMID 7041255. doi:10.1126/science.7041255.
- ↑ Hastings, P. J.; Lupski, J. R.; Rosenberg, S. M.; Ira, G. (2009). «Mechanisms of change in gene copy number». Nature Reviews. Genetics 10 (8): 551-564. PMID 19597530. doi:10.1038/nrg2593.
- ↑ Carroll, Sean B.; Jennifer K. Grenier; Scott D. Weatherbee. (2005). From DNA to Diversity: Molecular Genetics and the Evolution of Animal Design. Second Edition. Oxford: Blackwell Publishing. ISBN 1-4051-1950-0.
- ↑ Harrison, P., Gerstein, M. (2002). «Studying genomes through the aeons: protein families, pseudogenes and proteome evolution». J Mol Biol 318 (5): 1155-74. PMID 12083509. doi:10.1016/S0022-2836(02)00109-2.
- ↑ Gregory TR, Hebert PD (1999). «The modulation of DNA content: proximate causes and ultimate consequences». Genome Res. 9 (4): 317-24. PMID 10207154. doi:10.1101/gr.9.4.317 (inactivo 2009-11-14).
- ↑ Hurles M (julio de 2004). «Gene duplication: the genomic trade in spare parts». PLoS Biol. 2 (7): E206. PMC 449868. PMID 15252449. doi:10.1371/journal.pbio.0020206.
- ↑ Bowmaker, J. K. (1998). «Evolution of colour vision in vertebrates». Eye (London, En) 12 (Pt 3b): 541-7. PMID 9775215.
- ↑ Liu, N., Okamura, K., Tyler, D. M. (2008). «The evolution and functional diversification of animal microRNA genes». Cell Res. 18 (10): 985-96. PMC 2712117. PMID 18711447. doi:10.1038/cr.2008.278.
- ↑ Siepel, A. (octubre de 2009). «Darwinian alchemy: Human genes from noncoding DNA». Genome Res. 19 (10): 1693-5. PMC 2765273. PMID 19797681. doi:10.1101/gr.098376.109.
- ↑ Orengo CA, Thornton, J. M. (2005). «Protein families and their evolution-a structural perspective». Annu. Rev. Biochem. 74: 867-900. PMID 15954844. doi:10.1146/annurev.biochem.74.082803.133029.
- ↑ Long, M., Betrán, E., Thornton, K., Wang, W. (noviembre de 2003). «The origin of new genes: glimpses from the young and old». Nat. Rev. Genet. 4 (11): 865-75. PMID 14634634. doi:10.1038/nrg1204.
- ↑ Wang, M., Caetano-Anollés, G. (2009). «The evolutionary mechanics of domain organization in proteomes and the rise of modularity in the protein world». Structure 17 (1): 66-78. PMID 19141283. doi:10.1016/j.str.2008.11.008.
- ↑ Zhang, J., Wang, X., Podlaha, O. (2004). «Testing the chromosomal speciation hypothesis for humans and chimpanzees». Genome Res. 14 (5): 845-51. PMC 479111. PMID 15123584. doi:10.1101/gr.1891104.
- ↑ Ayala, F. J., Coluzzi, M. (2005). «Chromosome speciation: humans, Drosophila, and mosquitoes». Proc. Natl. Acad. Sci. U.S.A. 102 (Supl 1): 6535-42. PMC 1131864. PMID 15851677. doi:10.1073/pnas.0501847102. Archivado desde el original el 17 de noviembre de 2011. Consultado el 21 de enero de 2010.
- ↑ Hurst, G. D., Werren, J. H. (2001). «The role of selfish genetic elements in eukaryotic evolution». Nat. Rev. Genet. 2 (8): 597-606. PMID 11483984. doi:10.1038/35084545.
- ↑ Häsler, J., Strub, K. (2006). «Alu elements as regulators of gene expression». Nucleic Acids Res. 34 (19): 5491-7. PMC 1636486. PMID 17020921. doi:10.1093/nar/gkl706.
- ↑ Radding, C. (1982). «Homologous pairing and strand exchange in genetic recombination». Annu. Rev. Genet. 16: 405-37. PMID 6297377. doi:10.1146/annurev.ge.16.120182.002201.
- ↑ Agrawal, A. F. (2006). «Evolution of sex: why do organisms shuffle their genotypes?». Curr. Biol. 16 (17): R696. PMID 16950096. doi:10.1016/j.cub.2006.07.063.
- ↑ Peters, A. D., Otto, S. P. (2003). «Liberating genetic variance through sex». Bioessays 25 (6): 533-7. PMID 12766942. doi:10.1002/bies.10291.
- ↑ Goddard, M. R., Godfray, H. C., Burt, A. (2005). «Sex increases the efficacy of natural selection in experimental yeast populations». Nature 434 (7033): 636-40. PMID 15800622. doi:10.1038/nature03405.
- ↑ Crow, J. F., & Kimura, M. 1965. Evolution in sexual and asexual populations. Amer.Nat., 99, 439-450.
- ↑ Fontaneto, D., Herniou, E. A., Boschetti, C. (abril de 2007). «Independently evolving species in asexual bdelloid rotifers». PLoS Biol. 5 (4): e87. PMC 1828144. PMID 17373857. doi:10.1371/journal.pbio.0050087. Resumen divulgativo.
- ↑ Clausen, J. 1954. Partial apomixis as an equilibrium system in evolution. Caryologia Suppl.,6, 469-479.
- ↑ Lien, S., Szyda, J., Schechinger, B., Rappold, G., Arnheim, N. (febrero de 2000). «Evidence for heterogeneity in recombination in the human pseudoautosomal region: high resolution analysis by sperm typing and radiation-hybrid mapping». Am. J. Hum. Genet. 66 (2): 557-66. PMC 1288109. PMID 10677316. doi:10.1086/302754.
- ↑ Barton, N. H. (2000). «Genetic hitchhiking». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 355 (1403): 1553-1562. PMC 1692896. PMID 11127900. doi:10.1098/rstb.2000.0716. Consultado el 23 de septiembre de 2009.
- ↑ Muller, H. (1964). «The relation of recombination to mutational advance». Mutat. Res. 106: 2-9. PMID 14195748.
- ↑ Charlesworth, B., Charlesworth, D. (noviembre de 2000). «The degeneration of Y chromosomes». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 355 (1403): 1563-72. PMC 1692900. PMID 11127901. doi:10.1098/rstb.2000.0717.
- ↑ a b Otto, S. (1 de julio de 2003). «The advantages of segregation and the evolution of sex». Genetics 164 (3): 1099-118. PMC 1462613. PMID 12871918.
- ↑ Maynard Smith, J. (1978). The Evolution of Sex. Cambridge University Press. ISBN 0-521-29302-2.
- ↑ Doncaster, C. P., Pound, G. E., Cox, S. J. (marzo de 2000). «The ecological cost of sex». Nature 404 (6775): 281-5. PMID 10749210. doi:10.1038/35005078.
- ↑ Butlin, R. (abril de 2002). «Evolution of sex: The costs and benefits of sex: new insights from old asexual lineages». Nat. Rev. Genet. 3 (4): 311-7. PMID 11967555. doi:10.1038/nrg749.
- ↑ Salathé, M., Kouyos, R. D., Bonhoeffer, S. (agosto de 2008). «The state of affairs in the kingdom of the Red Queen». Trends Ecol. Evol. (Amst.) 23 (8): 439-45. PMID 18597889. doi:10.1016/j.tree.2008.04.010.
- ↑ Stoltzfus, A. (2006). «Mutationism and the dual causation of evolutionary change». Evol. Dev. 8 (3): 304-17. PMID 16686641. doi:10.1111/j.1525-142X.2006.00101.x.
- ↑ a b Bright, Kerry (2006). «Causes of evolution». Teach Evolution and Make It Relevant. National Science Foundation. Archivado desde el original el 7 de octubre de 2010. Consultado el 30 de diciembre de 2007.
- ↑ O'Neil, Dennis (2008). «Hardy-Weinberg Equilibrium Model». The synthetic theory of evolution: An introduction to modern evolutionary concepts and theories. Behavioral Sciences Department, Palomar College. Consultado el 6 de enero de 2008.
- ↑ Packer, C., Gilbert, D. A., Pusey, A. E. y O'Brieni, S. J. (junio de 1991). «A molecular genetic analysis of kinship and cooperation in African lions». Nature 351: 562-65. doi:10.1038/351562a0.
- ↑ Morjan, C. y Rieseberg, L. (2004). «How species evolve collectively: implications of gene flow and selection for the spread of advantageous alleles». Mol. Ecol. 13 (6): 1341-56. PMC 2600545. PMID 15140081. doi:10.1111/j.1365-294X.2004.02164.x.
- ↑ Baker, H. G. (1959). «Reproductive methods as factors in speciation in flowering plants». Cold. Spring Harbor Symp. Quant. Biol 24: 177-191.
- ↑ Barton, N. y Bengtsson, B. O. (1986). «The barrier to genetic exchange between hybridising populations». Heredity 57: 357-376.
- ↑ Strickberger, M. (1978). Genética. Barcelona: Omega. pp. 874-879. ISBN 84-282-0369-5.
- ↑ Futuyma, D. (1998). Evolutionary biology (3.ª edición). Sunderland: Sinauer Associates, Inc.
- ↑ Short, R. V. (1975). «The contribution of the mule to scientific thought». J. Reprod. Fertil. Suppl. (23): 359-64. PMID 1107543.
- ↑ Gross, B., Rieseberg, L. (2005). «The ecological genetics of homoploid hybrid speciation». J. Hered. 96 (3): 241-52. PMC 2517139. PMID 15618301. doi:10.1093/jhered/esi026.
- ↑ Burke JM, Arnold ML (2001). «Genetics and the fitness of hybrids». Annu. Rev. Genet. 35: 31-52. PMID 11700276. doi:10.1146/annurev.genet.35.102401.085719.
- ↑ Vrijenhoek RC (2006). «Polyploid hybrids: multiple origins of a treefrog species». Curr. Biol. 16 (7): R245. PMID 16581499. doi:10.1016/j.cub.2006.03.005.
- ↑ Wendel J (2000). «Genome evolution in polyploids». Plant Mol. Biol. 42 (1): 225-49. PMID 10688139. doi:10.1023/A:1006392424384.
- ↑ Sémon M, Wolfe KH (2007). «Consequences of genome duplication». Curr Opin Genet Dev 17 (6): 505-12. PMID 18006297. doi:10.1016/j.gde.2007.09.007.
- ↑ Comai L (2005). «The advantages and disadvantages of being polyploid». Nat. Rev. Genet. 6 (11): 836-46. PMID 16304599. doi:10.1038/nrg1711.
- ↑ Soltis, P., Soltis, D. (junio de 2000). «The role of genetic and genomic attributes in the success of polyploids». Proc. Natl. Acad. Sci. U.S.A. 97 (13): 7051-7. PMC 34383. PMID 10860970. doi:10.1073/pnas.97.13.7051.
- ↑ a b Whitlock, M. (1 de junio de 2003). «Fixation probability and time in subdivided populations». Genetics 164 (2): 767-79. PMC 1462574. PMID 12807795.
- ↑ Ohta, T. (2002). «Near-neutrality in evolution of genes and gene regulation». Proc. Natl. Acad. Sci. U.S.A. 99 (25): 16134-7. PMC 138577. PMID 12461171. doi:10.1073/pnas.252626899. Archivado desde el original el 8 de junio de 2008. Consultado el 4 de febrero de 2010.
- ↑ a b Hurst, L. D. (febrero de 2009). «Fundamental concepts in genetics: genetics and the understanding of selection». Nat. Rev. Genet. 10 (2): 83-93. PMID 19119264. doi:10.1038/nrg2506.
- ↑ a b c Orr, H. A. (agosto de 2009). «Fitness and its role in evolutionary genetics». Nat. Rev. Genet. 10 (8): 531-9. PMC 2753274. PMID 19546856. doi:10.1038/nrg2603.
- ↑ Haldane, J. (1959). «The theory of natural selection today». Nature 183 (4663): 710-3. PMID 13644170. doi:10.1038/183710a0.
- ↑ Lande, R. y Arnold, S. J. (1983). «The measurement of selection on correlated characters». Evolution 37 (6): 1210-26. doi:10.2307/2408842.
- ↑ Futuyma, Douglas J. (2005). Evolution. Sunderland, Massachusetts: Sinauer Associates, Inc. ISBN 0-87893-187-2.
- ↑ Miller, Ken (agosto de 1999). «The Peppered Moth». Consultado el 13 de abril de 2011.
- ↑ Dollo, L (1893). Les lois de l'évolution. Bull. Soc. Belge Geol. Pal. Hydr, VII:164-166.
- ↑ Goldberg, Emma E.; Boris Igić (2008). «On phylogenetic tests of irreversible evolution». Evolution 62 (11): 2727-2741. PMID 18764918. doi:10.1111/j.1558-5646.2008.00505.x.
- ↑ Collin, Rachel; Maria Pia Miglietta (2008). «Reversing opinions on Dollo's Law». Trends in Ecology & Evolution 23 (11): 602-609. PMID 18814933. doi:10.1016/j.tree.2008.06.013.
- ↑ Dawkins, Richard (1996) [1986]. The Blind Watchmaker. Nueva York: W. W. Norton & Company, Inc. ISBN 0-393-31570-3.
- ↑ Hoekstra, H.; Hoekstra, J.; Berrigan, D.; Vignieri, S.; Hoang, A.; Hill, C.; Beerli, P. y Kingsolver, J. (julio de 2001). «Strength and tempo of directional selection in the wild». Proc. Natl. Acad. Sci. U.S.A. 98 (16): 9157-60. PMC 55389. PMID 11470913. doi:10.1073/pnas.161281098.
- ↑ Felsenstein (1 de noviembre de 1979). «Excursions along the Interface between Disruptive and Stabilizing Selection». Genetics 93 (3): 773-95. PMC 1214112. PMID 17248980.
- ↑ Andersson, M. y Simmons, L. (2006). «Sexual selection and mate choice». Trends Ecol. Evol. (Amst.) 21 (6): 296-302. PMID 16769428. doi:10.1016/j.tree.2006.03.015.
- ↑ Kokko, H.; Brooks, R.; McNamara, J. y Houston, A. (2002). «The sexual selection continuum». Proc. Biol. Sci. 269 (1498): 1331-40. PMC 1691039. PMID 12079655. doi:10.1098/rspb.2002.2020.
- ↑ Hunt, J.; Brooks, R.; Jennions, M.; Smith, M.; Bentsen, C. y Bussière, L. (2004). «High-quality male field crickets invest heavily in sexual display but die young». Nature 432 (7020): 1024-1027. PMID 15616562. doi:10.1038/nature03084.
- ↑ a b Gould, S. J. (febrero de 1998). «Gulliver's further travels: the necessity and difficulty of a hierarchical theory of selection». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 353 (1366): 307-314. PMC 1692213. PMID 9533127. doi:10.1098/rstb.1998.0211.
- ↑ Mayr, E. (1997). «The objects of selection». Proc. Natl. Acad. Sci. U.S.A. 94 (6): 2091-2094. PMC 33654. PMID 9122151. doi:10.1073/pnas.94.6.2091.
- ↑ Maynard Smith, J. (1998). «The units of selection». Novartis Found. Symp. 213: 203-211; discusión 211-217. PMID 9653725.
- ↑ Hickey, D. A. (1992). «Evolutionary dynamics of transposable elements in prokaryotes and eukaryotes». Genetica 86 (1-3): 269-274. PMID 1334911. doi:10.1007/BF00133725.
- ↑ Gould, S. J. y Lloyd, E. A. (1999). «Individuality and adaptation across levels of selection: how shall we name and generalize the unit of Darwinism?». Proc. Natl. Acad. Sci. U.S.A. 96 (21): 11904-9. PMC 18385. PMID 10518549. doi:10.1073/pnas.96.21.11904.
- ↑ Lande, R. «Fisherian and Wrightian theories of speciation». Genome, vol. 31, 1, pp. 221-27.
- ↑ Otto, S., Whitlock, M. (1997). «The probability of fixation in populations of changing size». Genetics 146 (2): 723-33. PMID 9178020.
- ↑ Nei, M. (2005). «Selectionism and neutralism in molecular evolution». Mol. Biol. Evol. 22 (12): 2318-42. PMID 16120807. doi:10.1093/molbev/msi242.
- ↑ Kimura, M. (1991). «The neutral theory of molecular evolution: a review of recent evidence». Jpn. J. Genet. 66 (4): 367-86. PMID 1954033. doi:10.1266/jjg.66.367. Archivado desde el original el 11 de diciembre de 2008.
- ↑ Kimura, M. (1989). «The neutral theory of molecular evolution and the world view of the neutralists». Genome 31 (1): 24-31. PMID 2687096.
- ↑ Adaptation. In: Oxford Dictionary of Science.
- ↑ Bowler P. J. 2003. Evolution: the history of an idea. California. pág. 10
- ↑ Orr, H. (2005). «The genetic theory of adaptation: a brief history». Nat. Rev. Genet. 6 (2): 119-27. PMID 15716908. doi:10.1038/nrg1523.
- ↑ Patterson C. 1999. Evolution. Natural History Museum, Londres. Pág. 1.
- ↑ Williams, G. C. 1966. Adaptation and natural selection: a critique of some current evolutionary thought. Princeton. «La adaptación evolutiva es un fenómeno de enorme importancia en la biología.» pág. 5.
- ↑ Huxley, J. 1942. Evolution: the modern synthesis. Allen & Unwin, Londres. Pág. 449.
- ↑ Kocher, T. D. (2004). «Adaptive evolution and explosive speciation: the cichlid fish model». Nat. Rev. Genet. 5 (4): 288-98. PMID 15131652. doi:10.1038/nrg1316. Archivado desde el original el 20 de julio de 2011.
- ↑ Hardin, G. (1960). «The competitive exclusion principle». Science 131 (3409): 1292-7. PMID 14399717. doi:10.1126/science.131.3409.1292.
- ↑ Price, P. W. 1980. The evolutionary biology of parasites. Princeton.
- ↑ Dobzhansky, T.; Hecht, M. K.; Steere, W. C. (1968). «On some fundamental concepts of evolutionary biology». Evolutionary biology volume 2 (1st edición). New York: Appleton-Century-Crofts. pp. 1-34.
- ↑ Dobzhansky, T. (1970). Genetics of the evolutionary process. N.Y.: Columbia. pp. 4-6, 79-82, 84-87. ISBN 0-231-02837-7.
- ↑ Dobzhansky, T. (1956). «Genetics of natural populations XXV. Genetic changes in populations of Drosophila pseudoobscura and Drosphila persimilis in some locations in California». Evolution 10 (1): 82-92. JSTOR 2406099. doi:10.2307/2406099.
- ↑ Blount, Z. D., Borland, C. Z., Lenski, R. E (2008). «Inaugural Article: Historical contingency and the evolution of a key innovation in an experimental population of Escherichia coli». Proc. Natl. Acad. Sci. U.S.A. 105 (23): 7899-906. PMC 2430337. PMID 18524956. doi:10.1073/pnas.0803151105.
- ↑ Okada, H., Negoro, S., Kimura, H., Nakamura, S. (1983). «Evolutionary adaptation of plasmid-encoded enzymes for degrading nylon oligomers». Nature 306 (5939): 203-6. PMID 6646204. doi:10.1038/306203a0.
- ↑ Ohno, S. (1984). «Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence». Proc. Natl. Acad. Sci. U.S.A. 81 (8): 2421-5. PMC 345072. PMID 6585807. doi:10.1073/pnas.81.8.2421.
- ↑ Copley, S. D. (2000). «Evolution of a metabolic pathway for degradation of a toxic xenobiotic: the patchwork approach». Trends Biochem. Sci. 25 (6): 261-5. PMID 10838562. doi:10.1016/S0968-0004(00)01562-0.
- ↑ Crawford, R. L., Jung, C. M., Strap, J. L. (2007). «The recent evolution of pentachlorophenol (PCP)-4-monooxygenase (PcpB) and associated pathways for bacterial degradation of PCP». Biodegradation 18 (5): 525-39. PMID 17123025. doi:10.1007/s10532-006-9090-6.
- ↑ Nakajima, A., Sugimoto, Y., Yoneyama, H., Nakae, T. (2002). «High-level fluoroquinolone resistance in Pseudomonas aeruginosa due to interplay of the MexAB-OprM efflux pump and the DNA gyrase mutation». Microbiol. Immunol. 46 (6): 391-5. PMID 12153116.
- ↑ Colegrave, N., Collins, S. (2008). «Experimental evolution: experimental evolution and evolvability». Heredity 100 (5): 464-70. PMID 18212804. doi:10.1038/sj.hdy.6801095.
- ↑ Kirschner, M., Gerhart, J. (1998). «Evolvability». Proc. Natl. Acad. Sci. U.S.A. 95 (15): 8420-7. PMC 33871. PMID 9671692. doi:10.1073/pnas.95.15.8420.
- ↑ a b Bejder L, Hall BK (2002). «Limbs in whales and limblessness in other vertebrates: mechanisms of evolutionary and developmental transformation and loss». Evol. Dev. 4 (6): 445-58. PMID 12492145. doi:10.1046/j.1525-142X.2002.02033.x.
- ↑ Young, Nathan M.; Hallgrímsson, B (2005). «Serial homology and the evolution of mammalian limb covariation structure». Evolution 59 (12): 2691-704. PMID 16526515. doi:10.1554/05-233.1. Consultado el 24 de septiembre de 2009.
- ↑ Hall, Brian K. (2003). «Descent with modification: the unity underlying homology and homoplasy as seen through an analysis of development and evolution». Biological Reviews of the Cambridge Philosophical Society 78 (3): 409-433. PMID 14558591. doi:10.1017/S1464793102006097.
- ↑ Shubin, Neil; Tabin, C.; Carroll, S. (2009). «Deep homology and the origins of evolutionary novelty». Nature 457 (7231): 818-823. PMID 19212399. doi:10.1038/nature07891.
- ↑ a b c Fong, D., Kane, T., Culver, D. (1995). «Vestigialization and Loss of Nonfunctional Characters». Ann. Rev. Ecol. Syst. 26 (4): 249-68. PMID 12733778. doi:10.1146/annurev.es.26.110195.001341.
- ↑ Zhang, Z., Gerstein, M. (2004). «Large-scale analysis of pseudogenes in the human genome». Curr. Opin. Genet. Dev. 14 (4): 328-35. PMID 15261647. doi:10.1016/j.gde.2004.06.003.
- ↑ Jeffery, W. R. (2005). «Adaptive evolution of eye degeneration in the Mexican blind cavefish». J. Hered. 96 (3): 185-96. PMID 15653557. doi:10.1093/jhered/esi028.
- ↑ Maxwell, E. E., Larsson, H. C. (2007). «Osteology and myology of the wing of the Emu (Dromaius novaehollandiae) and its bearing on the evolution of vestigial structures». J. Morphol. 268 (5): 423-41. PMID 17390336. doi:10.1002/jmor.10527.
- ↑ Silvestri, A. R., Singh, I. (2003). «The unresolved problem of the third molar: would people be better off without it?». Journal of the American Dental Association (1939) 134 (4): 450-5. PMID 12733778. doi:10.1146/annurev.es.26.110195.001341. Archivado desde el original el 23 de agosto de 2014.
- ↑ Darwin, Charles. (1872) The Expression of the Emotions in Man and Animals John Murray, London.
- ↑ Gray, Peter (2007). Psychology (5.ª edición). Worth Publishers. p. 66. ISBN 0-7167-0617-2.
- ↑ Behavior Development in Infants por Evelyn Dewey, citando el estudio "Reflexes and other motor activities in newborn infants: a report of 125 cases as a preliminary study of infant behavior" publicado en Bull. Neurol. Inst. New York, 1932, Vol. 2, pp. 1-56.
- ↑ Coyne, Jerry (2009). Why Evolution is True. Penguin Group. pp. 85-86. ISBN 978-0-670-02053.
- ↑ Anthony Stevens (1982). Archetype: A Natural History of the Self. Routledge & Kegan Paul. p. 87. ISBN 0-7100-0980-1.
- ↑ a b Gould, 2002, pp. 1235-6
- ↑ Hoffman, M.; Taylor, B. E.; Harris, M. B. (2016). «Evolution of lung breathing from a lungless primitive vertebrate». Respiratory physiology & neurobiology 224: 11-16. ISSN 1569-9048. PMID 26476056. doi:10.1016/j.resp.2015.09.016. Consultado el 27 de enero de 2019.
- ↑ Johnson, N. A., Porter, A. H. (2001). «Toward a new synthesis: population genetics and evolutionary developmental biology». Genetica. 112-113: 45-58. PMID 11838782. doi:10.1023/A:1013371201773.
- ↑ Baguñà, J., Garcia-Fernàndez, J. (2003). «Evo-Devo: the long and winding road». Int. J. Dev. Biol. 47 (7-8): 705-13. PMID 14756346.
*Love, A. C. (2003). «Evolutionary Morphology, Innovation and the Synthesis of Evolutionary and Developmental Biology». Biology and Philosophy 18 (2): 309-345. doi:10.1023/A:1023940220348. - ↑ Allin, E. F. (1975). «Evolution of the mammalian middle ear». J. Morphol. 147 (4): 403-37. PMID 1202224. doi:10.1002/jmor.1051470404.
- ↑ Harris, M. P., Hasso, S. M., Ferguson, M. W., Fallon, J. F. (2006). «The development of archosaurian first-generation teeth in a chicken mutant». Curr. Biol. 16 (4): 371-7. PMID 16488870. doi:10.1016/j.cub.2005.12.047.
- ↑ Carroll, S. B. (2008). «Evo-devo and an expanding evolutionary synthesis: a genetic theory of morphological evolution». Cell 134 (1): 25-36. PMID 18614008. doi:10.1016/j.cell.2008.06.030.
- ↑ Wade, M. J. (2007). «The co-evolutionary genetics of ecological communities». Nat. Rev. Genet. 8 (3): 185-95. PMID 17279094. doi:10.1038/nrg2031.
- ↑ Geffeney, S., Brodie, E. D., Ruben, P. C., Brodie, E. D. (2002). «Mechanisms of adaptation in a predator-prey arms race: TTX-resistant sodium channels». Science 297 (5585): 1336-9. PMID 12193784. doi:10.1126/science.1074310.
*Brodie, E. D., Ridenhour, B. J., Brodie, E. D. (2002). «The evolutionary response of predators to dangerous prey: hotspots and coldspots in the geographic mosaic of coevolution between garter snakes and newts». Evolution 56 (10): 2067-82. PMID 12449493.
*Sean B. Carroll (21 de diciembre de 2009). «Remarkable Creatures - Clues to Toxins in Deadly Delicacies of the Animal Kingdom». New York Times. - ↑ a b Gavrilets, S. (2003). «Perspective: models of speciation: what have we learned in 40 years?». Evolution 57 (10): 2197-2215. PMID 14628909. doi:10.1554/02-727.
- ↑ Queiroz, K. de (diciembre de 2007). «Species concepts and species delimitation». Syst. Biol. 56 (6): 879-886. PMID 18027281. doi:10.1080/10635150701701083.
- ↑ Fraser, C., Alm, E. J., Polz, M. F., Spratt, B. G. y Hanage, W. P. (febrero de 2009). «The bacterial species challenge: making sense of genetic and ecological diversity». Science 323 (5915): 741-746. PMID 19197054. doi:10.1126/science.1159388.
- ↑ a b c Queiroz, K. de (mayo de 2005). «Ernst Mayr and the modern concept of species». Proc. Natl. Acad. Sci. U.S.A. 102 (Supl. 1): 6600-6607. PMC 1131873. PMID 15851674. doi:10.1073/pnas.0502030102.
- ↑ Rice, W. R.; Hostert (1993). «Laboratory experiments on speciation: what have we learned in 40 years». Evolution 47 (6): 1637-1653. doi:10.2307/2410209. Consultado el 19 de mayo de 2008.
*Jiggins, C. D., Bridle, J. R. (2004). «Speciation in the apple maggot fly: a blend of vintages?». Trends Ecol. Evol. (Amst.) 19 (3): 111-4. PMID 16701238. doi:10.1016/j.tree.2003.12.008.
*Boxhorn, J. (1995). «Observed Instances of Speciation». TalkOrigins Archive. Consultado el 26 de diciembre de 2008.
*Weinberg, J. R., Starczak, V. R., Jorg, D. (1992). «Evidence for Rapid Speciation Following a Founder Event in the Laboratory». Evolution 46 (4): 1214-20. doi:10.2307/2409766. - ↑ Herrel, A., Huyghe, K., Vanhooydonck, B., Backeljau, T., Breugelmans, K., Grbac, I.; Van Damme, R.; Irschick, D. J. (2008). «Rapid large-scale evolutionary divergence in morphology and performance associated with exploitation of a different dietary resource». Proceedings of the National Academy of Sciences 105 (12): 4792-5. PMC 2290806. PMID 18344323. doi:10.1073/pnas.0711998105.
- ↑ Losos, J. B., Warhelt, K. I., Schoener, T. W. (1997). «Adaptive differentiation following experimental island colonization in Anolis lizards». Nature 387 (6628): 70-3. doi:10.1038/387070a0.
- ↑ Hoskin CJ, Higgle M, McDonald KR, Moritz C (2005). «Reinforcement drives rapid allopatric speciation». Nature 437: 1353-356. doi:10.1038/nature04004.
- ↑ Templeton AR (1 de abril de 1980). «The theory of speciation via the founder principle». Genetics 94 (4): 1011-38. PMC 1214177. PMID 6777243.
- ↑ Antonovics, J. (2006). «Evolution in closely adjacent plant populations X: long-term persistence of prereproductive isolation at a mine boundary». Heredity 97 (1): 33-7. PMID 16639420. doi:10.1038/sj.hdy.6800835.
- ↑ Nosil, P., Crespi, B., Gries, R., Gries, G. (2007). «Natural selection and divergence in mate preference during speciation». Genetica 129 (3): 309-27. PMID 16900317. doi:10.1007/s10709-006-0013-6.
- ↑ Savolainen, V., Anstett, M-C., Lexer, C., Hutton, I., Clarkson, J. J., Norup, M. V., Powell, M. P., Springate, D., Salamin, N., Baker, W. Jr. (2006). «Sympatric speciation in palms on an oceanic island». Nature 441 (7090): 210-3. PMID 16467788. doi:10.1038/nature04566.
*Barluenga, M., Stölting, K. N., Salzburger, W., Muschick, M., Meyer, A. (2006). «Sympatric speciation in Nicaraguan crater lake cichlid fish». Nature 439 (7077): 719-23. PMID 16467837. doi:10.1038/nature04325. - ↑ Gavrilets S (2006). «The Maynard Smith model of sympatric speciation». J. Theor. Biol. 239 (2): 172-82. PMID 16242727. doi:10.1016/j.jtbi.2005.08.041.
- ↑ Wood, T. E., Takebayashi, N., Barker, M. S., Mayrose, I., Greenspoon, P. B., Rieseberg, L. H. (agosto de 2009). «The frequency of polyploid speciation in vascular plants». Proc. Natl. Acad. Sci. U.S.A. 106 (33): 13875-9. PMC 2728988. PMID 19667210. doi:10.1073/pnas.0811575106.
- ↑ Hegarty, M. F., Hiscock, S. J. (2008). «Genomic clues to the evolutionary success of polyploid plants». Current Biology 18 (10): 435-44. doi:10.1016/j.cub.2008.03.043.
- ↑ Jakobsson, M., Hagenblad, J., Tavaré, S. (2006). «A unique recent origin of the allotetraploid species Arabidopsis suecica: Evidence from nuclear DNA markers». Mol. Biol. Evol. 23 (6): 1217-31. PMID 16549398. doi:10.1093/molbev/msk006.
- ↑ Säll, T., Jakobsson, M., Lind-Halldén, C., Halldén, C. (2003). «Chloroplast DNA indicates a single origin of the allotetraploid Arabidopsis suecica». J. Evol. Biol. 16 (5): 1019-29. PMID 14635917. doi:10.1046/j.1420-9101.2003.00554.x.
- ↑ Bomblies, K., Weigel, D. (2007). «Arabidopsis-a model genus for speciation». Curr Opin Genet Dev 17 (6): 500-4. PMID 18006296. doi:10.1016/j.gde.2007.09.006.
- ↑ Eldredge, N. & S. J. Gould, 1972.«Punctuated equilibria: an alternative to phyletic gradualism» En: T. J. M. Schopf (ed.), Models in Paleobiology. San Francisco: Freeman Cooper. pp. 82-115.
- ↑ Gould, S. J. (1994). «Tempo and mode in the macroevolutionary reconstruction of Darwinism». Proc. Natl. Acad. Sci. U.S.A. 91 (15): 6764-71. PMC 44281. PMID 8041695. doi:10.1073/pnas.91.15.6764.
- ↑ Benton, M. J. (1995). «Diversification and extinction in the history of life». Science 268 (5207): 52-8. PMID 7701342. doi:10.1126/science.7701342.
- ↑ Raup, D. M. (1986). «Biological extinction in earth history». Science 231: 1528-33. PMID 11542058. doi:10.1126/science.11542058.
- ↑ Avise, J. C., Hubbell, S. P., Ayala, F. J. (agosto de 2008). «In the light of evolution II: Biodiversity and extinction». Proc. Natl. Acad. Sci. U.S.A. 105 (Supl. 1): 11453-7. PMC 2556414. PMID 18695213. doi:10.1073/pnas.0802504105.
- ↑ a b c d e Raup, D. M. (1994). «The role of extinction in evolution». Proc. Natl. Acad. Sci. U.S.A. 91 (15): 6758-63. PMC 44280. PMID 8041694. doi:10.1073/pnas.91.15.6758.
- ↑ Novacek, M. J.; Cleland, E. E. (2001). «The current biodiversity extinction event: scenarios for mitigation and recovery». Proc. Natl. Acad. Sci. U.S.A. 98 (10): 5466-70. PMC 33235. PMID 11344295. doi:10.1073/pnas.091093698.
- ↑ Pimm, S., Raven, P., Peterson, A., Sekercioglu, C. H., Ehrlich, P. R. (2006). «Human impacts on the rates of recent, present, and future bird extinctions». Proc. Natl. Acad. Sci. U.S.A. 103 (29): 10941-6. PMC 1544153. PMID 16829570. doi:10.1073/pnas.0604181103.
*Barnosky, A. D., Koch, P. L., Feranec, R. S., Wing, S. L., Shabel, A. B. (2004). «Assessing the causes of late Pleistocene extinctions on the continents». Science 306 (5693): 70-5. PMID 15459379. doi:10.1126/science.1101476. - ↑ Lewis, O. T. (2006). «Climate change, species-area curves and the extinction crisis» (PDF). Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1465): 163-71. PMC 1831839. PMID 16553315. doi:10.1098/rstb.2005.1712.
- ↑ Jablonski, D. (mayo de 2001). «Lessons from the past: evolutionary impacts of mass extinctions». Proc. Natl. Acad. Sci. U.S.A. 98 (10): 5393-8. PMC 33224. PMID 11344284. doi:10.1073/pnas.101092598.
- ↑ a b «La microevolución». Understanding evolution. Universidad de Berkeley. Consultado el 17 de marzo de 2019.
- ↑ Ros Aragones, Joan; Wilson, Edward O. (2012). «Capítulo 10». La conquista social de la Tierra. Penguin Random House Grupo Editorial España. ISBN 9788499922447. OCLC 932507445.
- ↑ «La macroevolución». Understanding evolution. Universidad de Berkeley. Consultado el 17 de marzo de 2019.
- ↑ Ayala, Francisco José (2006). «¿Es necesaria una nueva síntesis evolutiva?». En Amparo Latorre, Andrés Moya, ed. La evolución de un evolucionista : escritos seleccionados (Edición digital edición). Universitat de València. ISBN 9788437086828. OCLC 992115199.
- ↑ a b Carroll RL (1997) Patterns and processes of vertebrate evolution. Cambridge University Press, Cambridge.
- ↑ Carroll RL (2000) Towards a new evolutionary synthesis. Trends Ecol Evol 15:27-32.
- ↑ Carroll RL (2002) Evolution of the capacity to evolve. J Evol Biol 15:911-921.
- ↑ Fleagle, J. G. (2001) T«he unfused synthesis.» Evol Anthropol 10:191.
- ↑ Levin DA (2000) The origin, expansion, and demise of plant species. Oxford University Press, Oxford. ISBN 0-19-512729-3
- ↑ Stanley SM (1979) Macroevolution: pattern and process. WH Freeman, San Francisco. ISBN 0 7167 1092 7
- ↑ Nilsson, D-E., Pelger, S. (1994) A pessimistic estimate of the time required for an eye to evolve. Proc R Soc Lond B 256:53-58
- ↑ Benton MJ, Pearson PN (2001) Speciation in the fossil record. Trends Ecol Evol 16:405-411.
- ↑ a b Benton MJ, Harper DAT (1997) Basic palaeontology. Addison Wesley Longman, Essex. ISBN 0-582-22857-3
- ↑ a b c Benton, M. J. (1997) Vertebrate palaeontology (2nd edn). Chapman and Hall, London. ISBN 0-04-566001-8
- ↑ Briggs, D. E. G., Crowther, P. R. (eds) (1990) Palaeobiology: a synthesis. Blackwell Science, Oxford. ISBN 0-632-02525-5
- ↑ Cowen R (2000) History of life. (3.ª ed.). Blackwell Science, Oxford. ISBN 0-632-04501-9
- ↑ Ahlberg, P. E., Clack, J. A., Luksevics, E. (1996) Rapid braincase evolution between Panderichthys and the earliest tetrapods. Nature 381:61-64.
- ↑ Clack JA (2002) An early tetrapod from Romers gap. Nature 418:72-76.
- ↑ Sereno, P. C. (1999) The evolution of dinosaurs Archivado el 22 de julio de 2011 en Wayback Machine.. Science 284:2137-2147.
- ↑ Rubidge, B. S., Sidor, C. A. (2001) Evolutionary patterns among Permo-Triassic therapsids. Annu Rev Ecol Syst 32:449-480.
- ↑ Xu, X., Zhou, Z., Wang, X., Kuang, X., Zhang, F., Du, X. (2003) Fourwinged dinosaurs from China. Nature 421:335-340.
- ↑ Domning, D. P. (2001) «The earliest known fully quadrupedal sirenian». Nature, 413:625-627.
- ↑ Thewissen, J. G. M. y Williams, E. M. (2002) «The early radiation of cetacea (mammalia): evolutionary pattern and developmental correlations». Annu. Rev. Ecol. Syst., 33:73-90.
- ↑ Wood, B. (2002) «Hominid revelations from Chad». Nature, 418:133-135.
- ↑ Ge Sun, David L. Dilcher, Shaoling Zheng, Zhekun Zhou. 1998. In Search of the First Flower: A Jurassic Angiosperm, Archaefructus, from Northeast China. Science Vol. 282. no. 5394, pp. 1692 - 1695
- ↑ G. Sun, Q. Ji, D.L. Dilcher et al., Archaefructaceae, a New Basal Angiosperm Family. Science 296, 899.
- ↑ Xin Wang, Shaolin Zheng- 2007. The Earliest Perfect Flower: a perfect flower typical of angiosperms, including androecium, gynoecium and tepals, from western Liaoning, China marks the earliest record of undoubted flowers in the early Cretaceous (>125 Ma). Nature Precedings: hdl:10101/npre.2007.1320.1: Posted 15 Nov 2007
- ↑ Mayr, E. 1963. Óp. cit. p. 620
- ↑ Mayr, E. 1963. Op.Cit. p. 617.
- ↑ Sole, R. V., & Newman, M., 2002. «Extinctions and Biodiversity in the Fossil Record - Volume Two, The Earth system: biological and ecological dimensions of global environment change» pp. 297-391, Encyclopedia of Global Enviromental Change. John Wilely & Sons.
- ↑ Benton, M. J., Twitchett, R. J. (2003) «How to kill (almost) all life: the end-Permian extinction event.» Trends Ecol Evol 18:358-365.
- ↑ Alvarez, L. W. (1983). «Experimental evidence that an asteroid impact led to the extinction of many species 65 million years ago» (PDF (4,5 Mb)). Proceedings of the National Academy of Sciences (en inglés) 80 (2): 627-642. Consultado el 4 de octubre de 2009.
- ↑ a b Andersson, M. (1994). Sexual selection. Princeton, N.J.: Princeton University Press. ISBN 0-691-03344-7.
- ↑ Møller, A. P., Alatalo, R. V. (1999) Good-genes effects in sexual selection. Proc R Soc Lond B 266:85-91.
- ↑ Pennisi, E. (2003) Colourful males flaunt their health. Science 300:29-30.
- ↑ Krebs, J. R., Davies NB (1993) An introduction to behavioural ecology (3rd edn). Blackwell Science, Oxford. ISBN 0-632-00666-8
- ↑ Kutschera U, Wirtz P (2001) The evolution of parental care in freshwater leeches. Theory Biosci 120:115-137.
- ↑ Trillmich, F., Diesel, R. (2002) «Parental care.» In: Bateson PPG, Alleva E (eds) Encyclopedia of biology. Academic Press, New York, pp 313-325.
- ↑ Clutton-Brock T (2002) Breeding together: kin selection and mutualism in cooperative vertebrates. Science 296:69-72.
- ↑ Hamilton, W. D. (1972) Altruism and related phenomena, mainly in social insects. Annu Rev Ecol Syst 3:193-232.
- ↑ Hölldobler B, Wilson EO (1990) The ants. Harvard University Press, Cambridge, Mass. ISBN 0-674-04075-9
- ↑ Griffin AS, West SA (2002) Kin selection: fact and fiction. Trends Ecol Evol 17:15-2.
- ↑ Jost, J. (2003) On the notion of fitness, or: the selfish ancestor. Theory Biosci 121:331-350.
- ↑ Eldredge, N. y Cracraft, J. (1980). Phylogenetic patterns and the evolutionary process. Nueva York: Columbia University Press. pp. 249-250. ISBN 0231083785.
- ↑ a b Theissen, G. (2006). «The proper place of hopeful monsters in evolutionary biology». Theory Biosci. 124 (3-4): 349-69.
- ↑ Goldschmidt, R. (1940) The Material Basis of Evolution. New Haven, CT: Yale University Press, pp. 205-206.
- ↑ Gould, S. J. (1982). «The uses of heresey; an introduction to Richard Goldschmidt's The Material Basis of Evolution.» pp. xiii-xlii. New Haven: Yale University Press.
- ↑ a b Gould, S. J. (1977). «The Return of Hopeful Monsters». Natural History 86 (6 (junio/julio)): 22-30.
- ↑ Huxley, T. H. (1859). Letter to Charles Darwin. Archivado el 31 de enero de 2008 en Wayback Machine. Nov. 23, 1859.
- ↑ a b Hall, B. K. (1999) Evolutionary developmental biology. Kluwer Academic, Dordrecht. ISBN 0-412-27550-3
- ↑ a b c Arthur, W. (2002) The emerging conceptual framework of evolutionary developmental biology. Nature 415:757-764.
- ↑ Ronshaugen, M., McGinnis, N., McGinnis, W. (2002) Hox protein mutation and macroevolution of the insect body plan. Nature 415:914-917.
- ↑ Daumas Nunes y cols., 2003. Homeobox genes: a molecular link between development and cancer. Pesqui. Odontol. Bras. vol.17 no.1 São Paulo Jan./Mar
- ↑ a b Meyer, A. (1998) Hox gene variation and evolution. Nature 391: 225-228.
- ↑ Gevers, D., Cohan, F. M., Lawrence, J. G., et al. (2005). «Opinion: Re-evaluating prokaryotic species». Nat.Rev.Microbiol. 3 (9): 733-9. PMID 16138101. doi:10.1038/nrmicro1236.
- ↑ Coenye, T., Gevers, D., Van de Peer, Y., Vandamme, P., Swings, J. (2005). «Towards aprokaryotic genomic taxonomy». FEMS Microbiol.Rev. 29 (2): 147-67. PMID 15808739. doi:10.1016/j.femsre.2004.11.004.
- ↑ Whitman, W., Coleman, D., Wiebe, W. (1998). «Prokaryotes: the unseenmajority». ProcNatl Acad SciUSA 95 (12): 6578-83. PMC 33863. PMID 9618454. doi:10.1073/pnas.95.12.6578.
- ↑ Schloss, P., Handelsman, J. (2004). «Status of the microbialcensus». MicrobiolMol BiolRev 68 (4): 686-91. PMC 539005. PMID 15590780. doi:10.1128/MMBR.68.4.686-691.2004.
- ↑ Ochiai, K., Yamanaka, T., Kimura, K., Sawada, O. (1959). «Inheritance of drug resistance (and its transfer)between Shigella strains and Between Shigella and E.colistrains». Hihon Iji Shimpor 1861: 34. (en japonés)
- ↑ «Lateral gene transfer and the nature of bacterial innovation» (PDF). Nature (en inglés) 405: 299-304. 2000. doi:10.1038/35012500.
- ↑ de la Cruz, F., Davies, J. (2000). «Horizontal gene transferand the origin of species: lessons from bacteria». TrendsMicrobiol. 8 (3): 128-33. PMID 10707066. doi:10.1016/S0966-842X(00)01703-0.
- ↑ Poole, A., Penny, D. (2007). «Evaluating hypotheses for the origin of eukaryotes». Bioessays 29 (1): 74-84. PMID 17187354. doi:10.1002/bies.20516.
- ↑ Martin, W., Borst, P. (2003) «Secondary loss of chloroplasts in trypanosomes.» Proc Natl Acad Sci USA 100:765-767.
- ↑ Margulis, L. (1993). Symbiosis in cell evolution: microbial communities in the Archean and Proterozoic eons (en inglés) (2.ª edición). Nueva York: WH Freeman.
- ↑ a b c Margulis, Lynn; Dorion Sagan (2003). Captando Genomas. Una teoría sobre el origen de las especies.. Ernst Mayr (prólogo). David Sempau (trad.). (1.ª edición). Barcelona: Editorial Kairós. ISBN 84-7245-551-3.
- ↑ Niklas KJ (1997) The evolutionary biology of plants. University of Chicago Press, Chicago
- ↑ Martin W, Hoffmeister M, Rotte C, Henze K (2001). «An overview of endosymbiotic models for the origins of eukaryotes, their ATP-producing organelles (mitochondria and hydrogenosomes), and their heterotrophic lifestyle». Biol Chem (en inglés) 382: 1521-1539. Archivado desde el original el 28 de noviembre de 2012.
- ↑ Lenski, Richard. «Experimental evolution». Michigan State University.
- ↑ Moreno, Daniel (2013). «Programa Tierra. Simulación computarizada de la evolución biológica», Journal of Feelsynapsis (JoF). ISSN 2254-3651. 2013. (8): págs. 20-23.
- ↑ Browne, Janet (2003). Charles Darwin: The Power of Place. London: Pimlico. pp. 376-379. ISBN 0-7126-6837-3.
- ↑ Dennett, D. (1995). Darwin's Dangerous Idea: Evolution and the Meanings of Life. Simon & Schuster. ISBN 978-0684824710.
Para la percepción científica y social de la evolución en los siglos XIX y XX, consúltese Johnston, Ian C. «History of Science: Origins of Evolutionary Theory». And Still We Evolve. Liberal Studies Department, Malaspina University College. Consultado el 24 de mayo de 2007.
*Zuckerkandl E (2006). «Intelligent design and biological complexity». Gene 385: 2-18. PMID 17011142. doi:10.1016/j.gene.2006.03.025. - ↑ Scott, E. C., Matzke, N. J. (mayo de 2007). «Biological design in science classrooms». Proc. Natl. Acad. Sci. U.S.A. 104, supl. 1: 8669-76. PMC 1876445. PMID 17494747. doi:10.1073/pnas.0701505104.
- ↑ Ross, M. R. (2005). «Who Believes What? Clearing up Confusion over Intelligent Design and Young-Earth Creationism» (PDF). Journal of Geoscience Education 53 (3): 319. Consultado el 28 de abril de 2008.
- ↑ Hameed, Salman (12 de diciembre de 2008). «Science and Religion: Bracing for Islamic Creationism». Science 322 (5908): 1637-1638. PMID 19074331. doi:10.1126/science.1163672. Archivado desde el original el 10 de noviembre de 2014. Consultado el 2009.
- ↑ Spergel, D. N.; Scott, E. C.; Okamoto, S. (2006). «Science communication. Public acceptance of evolution». Science 313 (5788): 765-66. PMID 16902112. doi:10.1126/science.1126746.
- ↑ Spergel, D. N.; Verde, L.; Peiris, H. V.; Komatsu, E.; Nolta, M. R.; Bennett, C. L.; Halpern, M.; Hinshaw, G. et al. (2003). «First-Year Wilkinson Microwave Anisotropy Probe (WMAP) Observations: Determination of Cosmological Parameters». The Astrophysical Journal Supplement Series 148: 175-94. doi:10.1086/377226.
- ↑ Wilde SA, Valley JW, Peck WH, Graham CM (2001). «Evidence from detrital zircons for the existence of continental crust and oceans on the Earth 4.4 Gyr ago». Nature 409 (6817): 175-78. PMID 11196637. doi:10.1038/35051550.
- ↑ Kevles, D. (1998). In the Name of Eugenics: Genetics and the Uses of Human Heredity. Harvard University Press. ISBN 978-0674445574.
- ↑ Midgley, M. (2004). The Myths we Live By. Routledge. p. 62. ISBN 978-0415340779.
- ↑ Allhoff, F. (2003). «Evolutionary ethics from Darwin to Moore». History and philosophy of the life sciences 25 (1): 51-79. PMID 15293515. doi:10.1080/03919710312331272945.
- ↑ Gowaty, Patricia Adair (1997). Feminism and evolutionary biology: boundaries, intersections, and frontiers. London: Chapman & Hall. ISBN 0-412-07361-7.
- ↑ Smocovitis, Vassiliki, Betty (1996). Unifying Biology: The Evolutionary Synthesis and Evolutionary Biology (en inglés). Princeton University Press. ISBN 0-691-03343-9.
- ↑ Bayrakdar, Mehmet. Al Jahiz and the Rise of Biological Evolution (en inglés).
- ↑ van Wyhe, John (2002-7). «Charles Darwin: gentleman naturalist: A biographical sketch». The Complete Work of Charles Darwin Online. University of Cambridge. Consultado el 10 de julio de 2011.
- ↑ Gee, Henry; Rory Howlett, Philip Campbell (enero de 2009). «15 Evolutionary Gems» (PDF) (en inglés). Nature. Consultado el 2 de mayo de 2009.
- ↑ «Kansas Education Board First to Back 'Intelligent Design'». The Washington Post (en inglés). 9 de noviembre de 2005.
«En Kansas se enseñará la teoría creacionista». La Nación. 10 de noviembre de 2005. - ↑ Bhattarcharjee, Y. (2006). «Evolution Trumps Intelligent Design in Kansas Vote». Science (en inglés) 313 (5788): 743. doi:10.1126/science.313.5788.743.
- ↑ «El gobierno de Berlusconi pretendió eliminar la teoría de Darwin de los colegios italianos». Archivado desde el original el 29 de agosto de 2011. Consultado el 2 de mayo de 2009.
- ↑ Algañaraz, Julio. «La ciencia de la evolución por Berlusconi: Ola de protestas de científicos y premios nobel italianos». Clarín. Consultado el 2 de mayo de 2009.
- ↑ Pío XII. 12 de agosto de 1950. Carta Encíclica Humani Generis del Sumo Pontífice Pío XII. Sobre las falsas opiniones contra los fundamentos de la doctrina católica..
- ↑ Pio XII, Enc. Humani Generis, 29.
- ↑ Pio XII, Enc. Humani Generis, 28.
- ↑ Juan Pablo II. 1996. Mensaje a los miembros de la Academia Pontificia de las Ciencias
- ↑ Benedicto XVI. 2007. Encuentro del Santo Padre Benedicto XVI con los párrocos y sacerdotes de las diócesis de Belluno-Feltre y Treviso. Libreria Editrice Vaticana (en línea) Fecha acceso 10-3-2008
- ↑ a b Abdul Majid (2002). «The Muslim Responses To Evolution». Islamic Research Foundation International, Inc. Archivado desde el original el 13 de febrero de 2010. Consultado el 10 de julio de 2011.
- ↑ Adnan Oktar (1999). «The Evolution Deceit». Consultado el 24 de mayo de 2010.
- ↑ «Darwin and design: historical essay» (en inglés). Darwin Correspondence Project. Archivado desde el original el 21 de octubre de 2014. Consultado el 17 de agosto de 2011.
- ↑ El gen egoísta, Richard Dawkins Barcelona: Salvat, 2.ª edición, 2000, 407 páginas, ISBN 84-345-0178-3
- ↑ a b c Abdalla, M. (2006). «La crisis latente del darwinismo». En Asclepio, revista de Historia de la Medicina y de la Ciencia, LVIII, págs. 43-94.
- ↑ Eldredge, N. y Gould, S. J. (1972): «Punctuated equilibria: an alternative to phyletic gradualism.» En: Schopf, T. J. M. (ed.): Models in Paleobiology. San Francisco. Freeman Cooper and Co.: 82-115.
- ↑ Goodwin, B. (1994): How the leopard changed its spots. Phoenix Giants [Las manchas del leopardo. La evolución de la complejidad. Tusquets Editores, Metatemas, 51. 307 págs. Barcelona (1998) ISBN 84-8310-563-2]
- ↑ Overton, William (1982). «McLean v. Arkansas Board of Education». The TalkOrigins Archive. Consultado el 24 de marzo de 2007.
- ↑ Colby, C (1996). «Introduction to Evolutionary Biology». The TalkOrigins Archive. Consultado el 24 de marzo de 2007.
- ↑ Margulis L, Bermudes D (1985). «Symbiosis as a mechanism of evolution: status of cell symbiosis theory». Symbiosis 1: 101-24. PMID 11543608.
- ↑ Sandín, M. (2005). «La transformación de la evolución.» Boletín de la Real Sociedad Española de Historia Natural. Sección Biológica. Tomo 100(1-4), 139-167.
- ↑ Sandín, M. 2009. En busca de la Biología. Reflexiones sobre la evolución Archivado el 16 de agosto de 2011 en Wayback Machine.. Asclepio, LXI-1
- ↑ Jablonka, E., M. Lachman y M. Lamb. 1992. Evidence, mechanisms and models for the inheritance of acquired characters. Journal of Theoretical Biology 158: 245-268.
- ↑ Workman, C. T., Mak, H. C., McCuine, S., Tagne, J. B., Agarwal, M. Ozier, O. Begley, T. J., Samson, L. D., Ideker, L. T. (2006). A Systems Approach to Mapping DNA Damage Response Pathways. Science Vol. 312. no. 5776, pp. 1054 - 1059 /
- ↑ Hall, B. K. (2003). Evo-Devo: evolutionary developmental mechanisms (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última).. Int. J. Dev. Biol. 47: 491-495.
- ↑ Richards, E. J. (2006). «Opinion: Inherited epigenetic variation - revisiting soft inheritance». Nature Reviews Genetics (en inglés) 7: 395-401. doi:10.1038/nrg1834.
- ↑ Prouls, S. R., Promislow, D. E. L. y Phillips, P. C. (junio de 2005). «Network thinking in ecology and Evolution». Trends in Ecology and Evolution 20 (6). doi:10.1016/j.tree.2005.04.004.
- ↑ Bascompte, J., Jordano, P. & Olesen, J. M. (2006): Asymmetric Coevolutionary Networks Facilitate Biodiversity Maintenance Science Vol 312 21 pp.431-433
- ↑ Barabási. A. L. & Oltvai, Z. N. (2004). Network biology: understanding the cell's functional organization. Nature Reviews Genetics 5, 101-113 (2004); doi:10.1038/nrg1272
- ↑ Bornholdt, S. (2005). Less Is More in Modeling Large Genetic Networks. Science 310, 451
- ↑ Lôning, W. W. y Saedler, H. (2003). «Chromosome Rearrangements and Transposable Elements». Annual Reviews of Genetics (en inglés) 36 (1): 389-416. doi:10.1146/annurev.genet.36.040202.092802.
- ↑ Flores, M. y colaboradores. (2007). Recurrent DNA inversion rearrangements in the human genome. PNAS, vol. 104 no. 15 6099-6106
- ↑ Wagner, G. P., Amemiya, C. & Ruddle, F. (2003). Hox cluster duplications and the opportunity for evolutionary novelties. PNAS vol. 100 no. 25, 14603-14606
- ↑ Mulley, J. F., Chi-Hua C. & Holland, P. W. H. (2006). Breakup of a homeobox cluster after genome duplication in teleosts. PNAS vol. 103 no. 27, 10369-10372
- ↑ Sandín, M. (1997). «Teoría Sintética: Crisis y Revolución». Arbor (623-624): 269-303.
Bibliografía
- Avise, J. C. (1994): Molecular markers, natural history and evolution. New York, Chapman and Hall. 511 págs. ISBN 0-412-03771-8 [Relato de los descubrimientos evolutivos realizados gracias a los estudios moleculares, con especial atención a los aspectos conservacionistas].
- Ayala, F.J. (1994): La teoría de la evolución. De Darwin a los últimos avances de la genética. Ediciones Temas de Hoy, S.A. Colección Fin de Siglo / Serie Mayor, 60. 237 págs. Madrid ISBN 84-7880-457-9
- Ayala, F. J. y Valentine, J. W. (1983 [1979]): La evolución en acción. Teoría y procesos de la evolución orgánica. Editorial Alhambra, S.A. Alhambra Universidad. 412 págs. Madrid ISBN 84-205-0981-7
- Buskes, Chris (2008 [2009]): La herencia de Darwin. La evolución en nuestra visión del mundo. Herder Editorial. 541 págs. Barcelona ISBN 978-84-254-2621-6
- Cabello, M. y Lope, S. (1987): Evolución. Editorial Alhambra, S.A. Biblioteca de Recursos Didácticos Alhambra, 12. 114 págs. Madrid ISBN 84-205-1535-3
- Carter, G. S. (1958 [1959]). Cien años de evolución. Editorial Taurus, S.A. Ser y Tiempo, 5. 223 págs. Madrid
- Castrodeza, C. (1988): Teoría Histórica de la Selección Natural. Editorial Alhambra, S.A. Exedra, 160. 284 págs. Madrid ISBN 84-205-1740-2
- Crusafont, M., Meléndez, B. y Aguirre, E. (Eds.) (1966): La evolución. La Editorial Católica, S.A. Biblioteca de Autores Cristianos [B.A.C.]. Sección VI (Filosofía), 258. 1014 págs. Madrid ISBN 978-84-220-0676-3 (de la 4.ª ed., 1986).
- Richard Dawkins. El gen egoísta. Barcelona: Salvat Editores, S.A., 2.ª edición, 2000, 407 páginas, ISBN 84-345-0178-3
- Devillers, C. y Chaline, J. (1989 [1993]): La teoría de la evolución. Estado de la cuestión a la luz de los conocimientos científicos actuales. Ediciones Akal, S.A. Ciencia Hoy, 5. 383 págs. Madrid ISBN 84-7600-989-5
- Dobzhansky, Th., Ayala, F. J., Stebbins, G. L. y Valentine, J. W. (1977 [1979]): Evolución. Barcelona: Omega. 558 págs. ISBN 84-282-0568-X
- Gould, S. J. (2002 [2004]): La estructura de la teoría de la evolución. Barcelona: Tusquets (Metatemas), 82. 1426 págs. ISBN 84-8310-950-6
- Limoges, C. (1970 [1976]):La selección natural. Ensayo sobre la primera constitución de un concepto (1837-1859). Siglo Veintiuno Editores, S.A. Ciencia y Técnica. 183 págs. México D.F.
- Maynard Smith, J. (1972 [1979]): Acerca de la Evolución. H. Blume Ediciones. 136 págs. Madrid ISBN 84-7214-182-9
- Milner, R. (1990 [1995]): Diccionario de la evolución. La humanidad a la búsqueda de sus orígenes. Barcelona: Biblograf (Vox). 684 págs. ISBN 84-7153-871-7
- Moya, A. (1989): Sobre la estructura de la teoría de la evolución. Editorial Anthropos, S.A. - Servicio Editorial de la Univ. del País Vasco. Nueva ciencia, 5. 174 págs. ISBN 84-7658-154-8
- Templado, J. (1974): Historia de las teorías evolucionistas. Editorial Alhambra, S.A. Exedra, 100. 170 págs. Madrid ISBN 84-205-0900-0
- VV. AA. (1978 [1979]): Evolución. Barcelona: Labor (Libros de Investigación y Ciencia). 173 págs. ISBN 84-335-5002-0
- VV. AA. (1982): Charles R. Darwin: La evolución y el origen del hombre. Revista de Occidente, Extraordinario IV, 18-19: 1-235 ISSN 0034-8635
Enlaces externos
 Wikimedia Commons alberga una categoría multimedia sobre Evolución biológica.
Wikimedia Commons alberga una categoría multimedia sobre Evolución biológica. Wikiquote alberga frases célebres de o sobre Evolución biológica.
Wikiquote alberga frases célebres de o sobre Evolución biológica. Wikiversidad alberga proyectos de aprendizaje sobre Evolución biológica.
Wikiversidad alberga proyectos de aprendizaje sobre Evolución biológica.
En español:
- «La evolución es un hecho y una teoría», artículo en el sitio web Chile Skeptic.
- «La evolución biológica como un hecho y como una teoría», artículo de Stephen Jay Gould en el sitio web Sin Dioses.
- «La evolución biológica», artículo de Antonio Barbadilla donde expone los conceptos de evolución y selección natural.
- Evolución y ambiente.
- Collado González, Santiago: «Teoría de la evolución», artículo en el sitio web Philosophica: Enciclopedia filosófica online
- «Comprendiendo la evolución».
En inglés:
- Un estudio para saber de dónde venimos y adónde vamos (en inglés)
- LANCELET AND ANSCENTRAL BODY (Artículo: evolución de los cordados, herencia genética, descubrimientos recientes) PDF (en inglés)



