Ensembl
| Ensembl | ||
|---|---|---|
 | ||
 | ||
| Tipo | Base de datos biológicos y en línea del Proyecto Ensembl | |
| Sede central | Instituto Europeo de Bioinformática (EBI) | |
| Sitio web | https://www.ensembl.org | |
| Yates, et al. (2020)[1] | ||
Ensembl es un proyecto de investigación bioinformática que trata de "desarrollar un sistema de software que produzca y mantenga anotaciones automáticas en los genomas eucariotas seleccionados". Funciona como una colaboración entre el Wellcome Trust Sanger Institute y el Instituto Europeo de Bioinformática, una división del Laboratorio Europeo de Biología Molecular. Toda la información y software generados en el proyecto es de libre uso y acceso.[2][3]
La mayoría del software producido y utilizado se escribe en el lenguaje de programación Perl, y se basa en las librerías BioPerl. La Application programming interface de Perl puede utilizarse fácilmente en otros proyectos genómicos, por ejemplo en la anotación de genes o listas de clones. También hay disponible una API para Java.
Historia[editar]
El genoma humano está compuesto de 3 mil millones de bases, que codifican aproximadamente para 20 000 - 25 000 genes. Sin embargo, el genoma por si sólo es de poca utilidad sin identificar la localización y relaciones entre genes individuales. Una opción es anotarlos manualmente, donde un equipo de investigadores puede localizar genes mediante datos experimentales extraídos de revistas científicas y datos públicos. Este procedimiento manual es un proceso lento y tedioso. La alternativa es la anotación automática, en la que se realiza la búsqueda de patrones mediante herramientas computacionales desde proteínas al ADN.[4][5] En 1999 se lanzó el proyecto Ensembl en respuesta a la inminente finalización del Proyecto Genoma Humano, con los objetivos iniciales de anotar automáticamente el genoma humano, integrar esta anotación con datos biológicos disponibles y hacer público todo este conocimiento.[6]
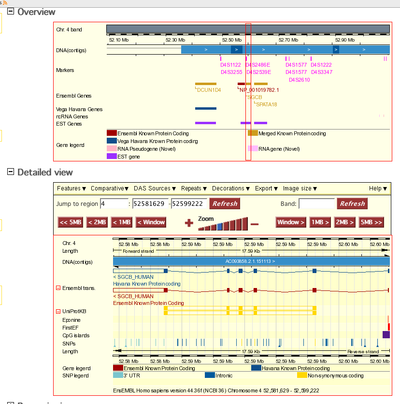
En el proyecto Ensembl, los datos de secuenciación son procesados por el sistema de anotación de genes (una colección de pipelines escritos en Perl), el cual predice un conjunto de genes y los guarda en una base de datos en MySQL para posteriores análisis y visualización. Ensembl publica estos datos a toda la comunidad científica mundial. Todos los datos y código producido por el proyecto Ensembl están disponibles para su descarga, existiendo también un servidor de acceso remoto para acceder a estos.[7] Además, es posible visualizar gran parte de los datos generados computacionalmente en el sitio web de Ensembl.
El proyecto se ha expandido con el tiempo, incluyendo nuevas especies (organismos modelo relevantes como el ratón, Drosophila melanogaster y el pez cebra), así como un mayor repertorio de datos genómicos, incluyendo variantes genéticas y elementos reguladores. Desde abril de 2009, el proyecto hermano Ensembl Genomes ha expandido el alcance de Ensembl hacia organismos invertebrados, tanto animales, plantas, hongos, bacterias como protistas, centrándose en describir el contexto taxonómico y evolutivo de genes, mientras que el proyecto original continúa enfocado en organismos vertebrados.[8][9]
A fecha de 2020, Ensembl guardaba más de 50 000 genomas entre las bases de datos de Ensembl y Ensembl Genomes, incluyendo algunas innovadoras características como Rapid Release, un sitio web diseñado para publicar más rápidamente datos de anotación de genomas, y COVID-19, un portal de acceso al genoma de referencia del virus SARS-CoV-2.
Genomas anotados[editar]
Los genomas anotados incluyen los vertebrados más completos, y organismos modelo seleccionados. A fecha de 2022, hay 271 especies registradas en la base de datos, incluyendo:[10]
Este servicio se utiliza por los biólogos moleculares y bioinformáticos de todo el mundo que trabajan con genomas de las especies listadas. Las predicciones de codificación, control y otros elementos en los genomas pueden compararse con datos de investigaciones primarias y con fuentes primarias de conocimiento genómico actualizado (bases de datos biológicas). La sintenia es de valor educativo en los colegios.
Aplicaciones[editar]
En una investigación realizada en 2014 se empleó Ensembl para el análisis genómico de conejo en busca de cambios fenotípicos durante su domesticación, es así que se realizó el ensamblaje del genoma que junto con la secuenciación de RNA de conejo y datos de ortólogos humanos, se obtuvieron regiones no traducidas (UTRs) (168,286 características distintas), regiones no codificantes de RNA (n=9666), y no elementos no codificantes conservados (2.518.476 características distintas). Esta información permitió agrupar las muestras para el análisis de la secuenciación genómica y sus modificaciones durante la domesticación de los conejos[11]
Acceso libre/espejos[editar]
Todos los datos del proyecto Ensembl, así como el software, son de acceso libre, estando disponibles para toda la comunidad científica bajo una licencia CC BY 4.0. Actualmente, el sitio web de Ensembl tiene cuatro espejos diferentes en el mundo para mejorar su servicio.
| Sitios web espejo oficiales |
|---|
| Servidor de Reino Unido (Instituto Sanger) ---- Sitio web principal |
| Servidor de la costa oeste de EE. UU. (Amazon AWS) ---- Espejo en la nube |
| Servidor de la costa este de EE. UU. (Amazon AWS) ---- Espejo en la nube |
| Servidor de Asia (Amazon AWS) ---- Espejo en la nube, en Singapur |
Véase también[editar]
Referencias[editar]
- ↑ Yates A. D. (January 2020). «Ensembl 2020». Nucleic Acids Res. 48 (D1): D682-D688. PMC 7145704. PMID 31691826. doi:10.1093/nar/gkz966.
- ↑ «Ensembl 2011». Nucleic Acids Research (en inglés). Consultado el 2 de mayo de 2021.
- ↑ Flicek, Paul; Aken, Bronwen L.; Ballester, Benoit; Beal, Kathryn; Bragin, Eugene; Brent, Simon; Chen, Yuan; Clapham, Peter et al. (2010-1). «Ensembl’s 10th year». Nucleic Acids Research 38 (Database issue): D557-D562. ISSN 0305-1048. PMC 2808936. PMID 19906699. doi:10.1093/nar/gkp972. Consultado el 25 de junio de 2021.
- ↑ «Medical definition of Genome Annotation» (en inglés). 29 de marzo de 2021. Archivado desde el original el 14 de junio de 2021. Consultado el 7 de agosto de 2022.
- ↑ Curwen, Val; Eyras, Eduardo; Andrews, T. Daniel; Clarke, Laura; Mongin, Emmanuel; Searle, Steven M. J.; Clamp, Michele (1 de mayo de 2004). «The Ensembl Automatic Gene Annotation System». Genome Research (en inglés) 14 (5): 942-950. ISSN 1088-9051. PMC 479124. PMID 15123590. doi:10.1101/gr.1858004. Consultado el 7 de agosto de 2022.
- ↑ Hubbard, T. (1 de enero de 2002). «The Ensembl genome database project». Nucleic Acids Research 30 (1): 38-41. ISSN 1362-4962. PMC 99161. PMID 11752248. doi:10.1093/nar/30.1.38. Consultado el 7 de agosto de 2022.
- ↑ Ruffier, Magali; Kähäri, Andreas; Komorowska, Monika; Keenan, Stephen; Laird, Matthew; Longden, Ian; Proctor, Glenn; Searle, Steve et al. (1 de enero de 2017). «Ensembl core software resources: storage and programmatic access for DNA sequence and genome annotation». Database 2017. ISSN 1758-0463. PMC 5467575. PMID 28365736. doi:10.1093/database/bax020. Consultado el 7 de agosto de 2022.
- ↑ Hubbard, T. J. P.; Aken, B. L.; Ayling, S.; Ballester, B.; Beal, K.; Bragin, E.; Brent, S.; Chen, Y. et al. (2009-01). «Ensembl 2009». Nucleic Acids Research (en inglés) 37 (Database issue): D690-697. ISSN 1362-4962. PMC 2686571. PMID 19033362. doi:10.1093/nar/gkn828. Consultado el 7 de agosto de 2022.
- ↑ Howe, Kevin L.; Contreras-Moreira, Bruno; De Silva, Nishadi; Maslen, Gareth; Akanni, Wasiu; Allen, James; Alvarez-Jarreta, Jorge; Barba, Matthieu et al. (8 de enero de 2020). «Ensembl Genomes 2020-enabling non-vertebrate genomic research». Nucleic Acids Research (en inglés) 48 (D1): D689-D695. ISSN 1362-4962. PMC 6943047. PMID 31598706. doi:10.1093/nar/gkz890. Consultado el 7 de agosto de 2022.
- ↑ «Species List». uswest.ensembl.org. Consultado el 14 de agosto de 2022.
- ↑ Carneiro, Miguel; Rubin, Carl-Johan; Di Palma, Federica; Albert, Frank W.; Alföldi, Jessica; Martinez Barrio, Alvaro; Pielberg, Gerli; Rafati, Nima et al. (29 de agosto de 2014). «Rabbit genome analysis reveals a polygenic basis for phenotypic change during domestication». Science (New York, N.Y.) 345 (6200): 1074-1079. ISSN 1095-9203. PMC 5421586. PMID 25170157. doi:10.1126/science.1253714. Consultado el 30 de septiembre de 2023.
