Diferencia entre revisiones de «Bacteria»
traducido de la wikipedia inglesa el apartado "Interacciones con otros organismos" |
traducido de la wikipedia inglesa el apartado "Utilidad económica de las bacterias" |
||
| Línea 161: | Línea 161: | ||
En el suelo, los microorganismos que habitan la [[rizosfera]] (la zona que incluye la superficie de la raíz y la tierra que se adhiere a ella) realizan la [[fijación de nitrógeno]], convirtiendo el gas nitrógeno en compuestos nitrogenados.<ref>{{cite journal |author=Barea J, Pozo M, Azcón R, Azcón-Aguilar C |title=Microbial co-operation in the rhizosphere | url=http://jxb.oxfordjournals.org/cgi/content/full/56/417/1761 |journal=J Exp Bot |volume=56 |issue=417 |pages=1761–78 |year=2005 |pmid=15911555}}</ref> Esto proporciona a muchas plantas que no pueden fijar el gas nitrógeno ellas mismas, una forma fácilmente absorbible de nitrógeno. |
En el suelo, los microorganismos que habitan la [[rizosfera]] (la zona que incluye la superficie de la raíz y la tierra que se adhiere a ella) realizan la [[fijación de nitrógeno]], convirtiendo el gas nitrógeno en compuestos nitrogenados.<ref>{{cite journal |author=Barea J, Pozo M, Azcón R, Azcón-Aguilar C |title=Microbial co-operation in the rhizosphere | url=http://jxb.oxfordjournals.org/cgi/content/full/56/417/1761 |journal=J Exp Bot |volume=56 |issue=417 |pages=1761–78 |year=2005 |pmid=15911555}}</ref> Esto proporciona a muchas plantas que no pueden fijar el gas nitrógeno ellas mismas, una forma fácilmente absorbible de nitrógeno. |
||
Muchas otras bacterias se encuentran como simbiontes en seres humanos y en otros organismos. Por ejemplo, en el tracto digestivo proliferan unas mil especies bacterianas. Sintetizan vitaminas tales como [[ácido fólico]], [[vitamina K]] y [[biotina]]. También fermentan los carbohidratos complejos indigeribles y convierten las proteínas de la leche en ácido láctico (por ejemplo, ''[[ |
Muchas otras bacterias se encuentran como simbiontes en seres humanos y en otros organismos. Por ejemplo, en el tracto digestivo proliferan unas mil especies bacterianas. Sintetizan vitaminas tales como [[ácido fólico]], [[vitamina K]] y [[biotina]]. También fermentan los carbohidratos complejos indigeribles y convierten las proteínas de la leche en ácido láctico (por ejemplo, ''[[Lactobacillus]]'').<ref>{{cite journal |author=O'Hara A, Shanahan F |title=The gut flora as a forgotten organ |journal=EMBO Rep |volume=7 |issue=7 |pages=688–93 |year=2006 |pmid=16819463}}</ref><ref>{{cite journal |author=Zoetendal E, Vaughan E, de Vos W |title=A microbial world within us |journal=Mol Microbiol |volume=59 |issue=6 |pages=1639–50 |year=2006 |pmid=16553872}}</ref><ref>{{cite journal |author=Gorbach S |title=Lactic acid bacteria and human health |journal=Ann Med |volume=22 |issue=1 |pages=37–41 |year=1990|id = PMID 2109988}}</ref> Además, la presencia de esta flora intestinal inhibe el crecimiento de bacterias potencialmente patógenas (generalmente por exclusión competitiva). Muchas veces estas bacterias beneficiosas se venden como suplementos dietéticos [[probiótico]]s.<ref>{{cite journal |author=Salminen S, Gueimonde M, Isolauri E |title=Probiotics that modify disease risk | url=http://jn.nutrition.org/cgi/content/full/135/5/1294 |journal=J Nutr |volume=135 |issue=5 |pages=1294–8 |year=2005 |pmid=15867327}}</ref> |
||
===Patógenos=== |
===Patógenos=== |
||
| Línea 200: | Línea 200: | ||
== Utilidad económica de las bacterias == |
== Utilidad económica de las bacterias == |
||
Muchas industrias dependen en parte o enteramente de la acción bacteriana. Gran cantidad de sustancias quimicas importantes como alcohol |
Muchas industrias dependen en parte o enteramente de la acción bacteriana. Gran cantidad de sustancias quimicas importantes como [[alcohol etílico]], [[ácido acético]], [[alcohol butílico]] y [[acetona]] son producidas por bacterias especificas. Se tienen que emplear bacterias para el curado de [[tabaco]], el curtido de [[cuero]]s, [[caucho]], [[algodón]], etc. Las bacterias (a menudo ''[[Lactobacillus]]'') junto con [[levadura]]s y [[moho]]s, se han utilizado durante miles de años para la preparación de alimentos fermentados tales como [[queso]], [[mantequilla]], [[encurtido]]s, [[salsa de soja]], [[chucrut]], [[vinagre]], [[vino]] y [[yogur]]. <ref>{{cite journal |author=Johnson M, Lucey J |title=Major technological advances and trends in cheese |journal=J Dairy Sci |volume=89 |issue=4 |pages=1174–8 |year=2006 |pmid=16537950}}</ref><ref>{{cite journal |author=Hagedorn S, Kaphammer B |title=Microbial biocatalysis in the generation of flavor and fragrance chemicals |journal=Annu Rev Microbiol |volume=48 |issue=|pages=773–800 |year=|pmid=7826026}}</ref> |
||
Las bacterias tienen una capacidad notable para degradar una gran variedad de compuestos orgánicos, por lo que se utilizan en el reciclado de basura y en [[biorremediación]]. Las bacterias capaces de degradar los hidrocarburos son de uso frecuente en la limpieza de los vertidos de petróleo.<ref>{{cite journal |author=Cohen Y |title=Bioremediation of oil by marine microbial mats |journal=Int Microbiol |volume=5 |issue=4 |pages=189–93 |year=2002 |pmid=12497184}}</ref> Así por ejemplo, después del vertido del petrolero [[Exxon Valdez]] en 1989, en algunas playas de Alaska se usaron fertilizantes con objeto de promover el crecimiento de estas bacterias naturales. Estos esfuerzos fueron eficaces en las playas en las que la capa de petróleo no era demasiado espesa. Las bacterias también se utilizan para la biorremediación de basuras tóxicas industriales.<ref>{{cite journal |author=Neves L, Miyamura T, Moraes D, Penna T, Converti A |title=Biofiltration methods for the removal of phenolic residues |journal=Appl Biochem Biotechnol |volume=129–132 |issue=|pages=130–52 | year =|id = PMID 16915636}}</ref> En la industria química, las bacterias son utilizadas en la producción de productos químicos [[Enantiómero|enantioméricamente]] puros para uso farmacéutico o agroquímico.<ref>{{cite journal |author=Liese A, Filho M |title=Production of fine chemicals using biocatalysis |journal=Curr Opin Biotechnol |volume=10 |issue=6 |pages=595–603 |year=1999 |pmid=10600695}}</ref> |
|||
Las bacterias también pueden ser utilizadas para el control biológico de parásitos en lugar de los pesticidas. Esto implica comúnmente la especie ''[[Bacillus thuringiensis]]'' (también llamado BT), una bacteria de suelo Gram-positiva. Las subespecies de esta bacteria se utilizan como insecticidas específicos para [[lepidóptero]]s.<ref>{{cite journal |author=Aronson A, Shai Y |title=Why Bacillus thuringiensis insecticidal toxins are so effective: unique features of their mode of action |journal=FEMS Microbiol Lett |volume=195 |issue=1 |pages=1–8 |year=2001|id = PMID 11166987}}</ref> Debido a su especificidad, estos pesticidas se consideran respectuosos con el medio ambiente, con poco o ningún efecto sobre los seres humanos, fauna y la mayoría de los insectos beneficiosos, como por ejemplo, los [[polinización|polinizadores]].<ref>{{cite journal |author=Bozsik A |title=Susceptibility of adult Coccinella septempunctata (Coleoptera: Coccinellidae) to insecticides with different modes of action |journal=Pest Manag Sci |volume=62 |issue=7 |pages=651–4 |year=2006 |pmid=16649191}}</ref><ref>{{cite journal |author=Chattopadhyay A, Bhatnagar N, Bhatnagar R |title=Bacterial insecticidal toxins |journal=Crit Rev Microbiol |volume=30 |issue=1 |pages=33–54 |year=2004 |pmid=15116762}}</ref> |
|||
Las bacterias son herramientas básicas en los campos de la biología, genética y bioquímica moleculares debido a su capacidad para crecer rápidamente y a la facilidad relativa con la que pueden ser manipuladas. Realizando modificaciones en el ADN bacteriano y examinando los [[fenotipo]]s que resultan, los científicos pueden determinar la función de [[gen]]es, [[enzima]]s y [[ruta metabólica|rutas metabólicas]], y después estos conocimientos se pueden trasladar a organismos más complejos.<ref>{{cite journal |author=Serres M, Gopal S, Nahum L, Liang P, Gaasterland T, Riley M |title=A functional update of the Escherichia coli K-12 genome | url=http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=11574054 |journal=Genome Biol |volume=2 |issue=9 |pages=RESEARCH0035 |year=2001 |pmid=11574054}}</ref> La comprensión de la bioquímica celular, que require cantidades enormes de datos de [[cinética enzimática]] y de expresión de genes, permitirá realizar modelos matemáticos de organismos enteros. Esto es realizable en alguna bacteria bien estudiada, por ejemplo, actualmente está siendo desarrollado y probado el modelo del metabolismo de ''[[Escherichia coli]]''.<ref>{{cite journal |author=Almaas E, Kovács B, Vicsek T, Oltvai Z, Barabási A |title=Global organization of metabolic fluxes in the bacterium Escherichia coli |journal=Nature |volume=427 |issue=6977 |pages=839–43 |year=2004 |pmid=14985762}}</ref><ref>{{cite journal |author=Reed J, Vo T, Schilling C, Palsson B |title=An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR) | url=http://genomebiology.com/2003/4/9/R54 |journal=Genome Biol |volume=4 |issue=9 |pages=R54 |year=2003|id = PMID 12952533}}</ref> Esta comprensión del metabolismo y genética bacterianos permite a la biotecnología la modificación de las bacterias para que produzcan proteínas terapéuticas, tales como [[insulina]], [[factor de crecimiento|factores de crecimiento]] y [[anticuerpo]]s.<ref>{{cite journal |author=Walsh G |title=Therapeutic insulins and their large-scale manufacture |journal=Appl Microbiol Biotechnol |volume=67 |issue=2 |pages=151–9 |year=2005 |pmid=15580495}}</ref><ref>{{cite journal |author=Graumann K, Premstaller A |title=Manufacturing of recombinant therapeutic proteins in microbial systems |journal=Biotechnol J |volume=1 |issue=2 |pages=164–86 |year=2006 |pmid=16892246}}</ref> |
|||
== Enlaces externos == |
== Enlaces externos == |
||
Revisión del 18:24 14 jun 2007
Las bacterias son microorganismos unicelulares. Tienen típicamente algunos micrómetros de largo (entre 0,5 y 5 μm) y se presentan en diversas formas incluyendo esferas, barras, y espirales. Generalmente poseen una pared celular similar a la de plantas u hongos, pero compuesta por peptidoglicanos. Muchos antibióticos son efectivos sólo contra las bacterias ya que inhiben la formación de esta pared celular. Muchas bacterias disponen de cilios o flagelos y son móviles.
Del estudio de las bacterias se encarga la bacteriología, una rama de la microbiología. Las bacterias son los organismos más abundantes del planeta. Son ubícuas, encontrándose en todo habitat de la tierra, creciendo en el suelo, manantiales calientes ácidos, desechos radioactivos,[1] en el mar y en las profundidades de la corteza de terrestre. Algunas bacterias pueden incluso sobrevivir en el frío y vacío extremos del espacio exterior. Hay típicamente 40 millones de células bacterianas en un gramo de tierra y un millón de células bacterianas en un mililitro de agua dulce. En total, hay aproximadamente 5×1030 bacterias en el mundo.[2]
Las bacterias son imprescindibles para el reciclaje de los nutrientes pues muchos pasos importantes de los ciclos nutrientes dependen de bacterias. Como ejemplo podemos citar la fijación del nitrógeno de la atmósfera. Sin embargo, solamente la mitad de los filos de bacterias tienen especies que se pueden cultivar en el laboratorio,[3] por lo que una gran parte de las especies de bacterias existentes todavía no ha sido descrita.
Hay aproximadamente 10 veces tantas células bacterianas como células humanas en el cuerpo humano, con una gran cantidad de bacterias en la piel y en el tracto digestivo.[4] Aunque el efecto protector del sistema inmune hace que la gran mayoría de estas bacterias sean inofensivas o beneficiosas, algunas bacterias patógenas pueden causar enfermedades infecciosas, incluyendo cólera, sífilis, ántrax, lepra y peste bubónica.
Las enfermedades bacterianas mortales más comunes son las infecciones respiratorias, con una mortalidad solo para la tuberculosis de cerca de 2 millones de personas al año, sobre todo en el África Subsahariana.[5] En los países desarrollados se utilizan antibióticos para tratar las infecciones bacterianas y también se usan extensamente en la agricultura y la ganadería, lo que ocasiona que se esté generalizando la resistencia de las bacterias a los antibióticos. En la industria, las bacterias son importantes en procesos tales como el tratamiento de aguas residuales, la producción de queso y yogur, y en la fabricación de antibióticos y de otros productos químicos.[6]
Las bacterias son procariotas. Al contrario que los animales y que otros eucariotas, las células bacterianas no tienen núcleo ni membranas que delimiten sus orgánulos internos. Aunque el término bacteria incluye tradicionalmente a todos los procariotes, actualmente la taxonomía y la nomenclatura científica los divide en dos grupos. Estos dominios evolutivos se denominan Bacteria y Archaea.[7] La división se justifica en las grandes diferencias que presentan ambos grupos a nivel bioquímico.
Historia de la bacteriología
La primera bacteria fue observada por Anton van Leeuwenhoek en 1683 usando un microscopio de lente simple diseñado por él. El nombre de bacteria fue introducido más tarde, por Ehrenberg en 1828, derivado del griego βακτηριον significando bastón pequeño. Louis Pasteur (1822-1895) y Robert Koch (1843-1910) describieron el papel de la bacteria como causa de enfermedades. La Bacteriología es una rama de la Microbiología que tiene por objeto de estudio la taxonomía, estructura y fisiología de las bacterias.
Estructura de la célula bacteriana

Las bacterias son organismos relativamente sencillos. Sus dimensiones son muy reducidas, unos 2 μm de ancho por 7-8 μm de longitud en la forma cilíndrica (bacilo) de tamaño medio; aunque son muy frecuentes las especies de 0,5-1,5 μm.
Carecen de un núcleo delimitado por una membrana aunque presentan un nucleoide, una estructura elemental que contiene una gran molécula circular de ADN. El citoplasma carece de los orgánulos delimitados por membranas y de las formaciones protoplasmáticas propios de las células eucariotas. En el citoplasma se aprecian plásmidos, pequeñas moléculas circulares de ADN que coexisten con el nucleoide, contienen genes y son comúnmente usados por las bacterias en la "reprodución sexual". El citoplasma también contiene vacuolas (gránulos que contienen sustancias de reserva) y ribosomas (utilizados en la síntesis de proteínas).
Una membrana celular compuesta de lípidos rodea el citoplasma y, al igual que las células de las plantas, la mayoría posee una pared celular a base de carbohidratos. El espacio comprendido entre la membrana celular y la pared celular se denomina periplasma. Algunas bacterias presentan una cápsula y otras son capaces de evolucionar a esporas, formas durmientes capaces de resistir condiciones extremas. Entre las formaciones exteriores propias de la célula bacteriana destacan los flagelos y los pilis.
Estructuras intracelulares
La célula bacteriana está rodeada por una membrana lípida denominada membrana celular. Esta membrana actúa como barrera de contención para los nutrientes, proteínas y demás componentes esenciales del citoplasma celular. Puesto que las bacterias son procariotas, no tienen membranas que limiten los orgánulos citoplasmáticos y por ello presentan pocas estructuras intracelulares. Carecen de núcleo celular, mitocondrias, cloroplastos y de los otros orgánulos presentes en las células eucariotas, tales como el aparato de Golgi y el retículo endoplasmático.[8]
Muchas importantes reacciones bioquímicas que tienen lugar en las células se producen por la existencia de gradientes de concentración a ambos lados de una membrana. Este gradiente crea una diferencia potencial análoga a la de una batería eléctrica y permite a la célula, por ejemplo, el transporte de electrones y la obtención de energía. La ausencia de membranas internas en las bacterias significa que estas reacciones tienen que producirse a través de la propia membrana celular, entre el citoplasma y el espacio periplasmático.[9]
Las bacterias no tienen membranas que delimiten un núcleo. El material genético está típicamente organizado en un solo cromosoma circular situado en el citoplasma dentro de un cuerpo de forma irregular denominado nucleoide.[10] El nucleoide contiene el cromosoma junto con las proteínas asociadas y ARN. Como todos los organismos vivos, las bacterias contienen ribosomas para la producción de proteínas, pero su estructura es diferente al de los ribosomas de Eukarya y Archaea.[11] El orden Planctomycetes es una excepción a la ausencia general de membranas internas en las bacterias, pues una membrana rodea su nucleoide y tiene varias estructuras celulares delimitadas por membranas.[12]
Algunas bacterias producen vacuolas, gránulos intracelulares para el almacenaje de sustancias, por ejemplo glucógeno,[13] polifosfato,[14] sulfuro[15] o polihidroxialcanoatos.[16] Ciertas especies bacterianas, tales como las fotosintéticas cyanobacterias, producen vesículas internas de gas que utilizan regular su flotabilidad y así alcanzar la intensidad de luz y/o los niveles nutrientes óptimos.[17]
Estructuras extracelulares
Las bacterias disponen de una pared celular que rodea a su membrana celular. Las paredes celulares bacterianas están hechas de peptidoglicano (llamado antiguamente mureína). Esta sustancia está compuesta por cadenas de polisacárido enlazadas por peptidos inusuales que contienen aminoácidos D.[18] Estos aminoácidos no se encuentran en las proteínas, por lo que protegen a la pared de la mayoría de las peptidasas. Las paredes celulares bacterianas son distintas de las que tienen plantas y hongos, que están hechas de celulosa y quitina, respectivamente.[19] Son también distintas a las paredes celulares de Archaea, que no contienen peptidoglicano. El antibiótico penicilina puede matar a muchas bacterias inhibiendo un paso de la síntesis del peptidoglicano.[19]
Existen dos diferentes tipos de pared celular bacteriana, denominadas Gram-positiva y Gram-negativa, respectivamente. Estos nombres provienen de la reacción de las pared celular a la tinción de Gram, un método tradicionalmente empleado para la clasificación de las especies bacterianas.[20] Las bacterias Gram-positivas tienen una pared celular gruesa que contiene numerosas capas de peptidoglicano y ácido teicoico. En cambio, las bacterias Gram-negativas tienen una pared relativamente fina consistente en unas pocas capas de peptidoglicano rodeadas por una segunda membrana lípida que contiene lipopolisacáridos y lipoproteínas.
La mayoría de las bacterias tienen paredes celulares Gram-negativas; solamente son Gram-positivas Firmicutes y Actinobacteria. Estos dos grupos eran antiguamente conocidos como bacterias Gram-positivas de contenido GC bajo y bacterias Gram-positivas de contenido GC alto, respectivamente.[21] Estas diferencias en la estructura de la pared celular dan lugar a diferencias en la susceptibilidad antibiótica. Por ejemplo, la vancomicina puede matar solamente a bacterias Gram-positivas y es ineficaz contra patógenos Gram-negativos, tales como Haemophilus influenzae o Pseudomonas aeruginosa.[22]
Muchas bacterias tienen una capa S de moléculas de proteína de estructura rígida que cubre la pared celular.[23] Esta capa proporciona protección química y física para la superficie celular y puede actuar como una barrera de difusión macromolecular. Las capas S tienen diversas (aunque todavía no bien comprendidas) funciones. Por ejemplo, en Campylobacter actúan como factores de virulencia y en Bacillus stearothermophilus contienen enzimas superficiales.[24]

Los flagelos son estructuras rígidas hechas de proteínas usados para el movimiento. Tienen un diámetro aproximado de 20 nm y una longitud de hasta 20 μm. Los flagelos son impulsados por la energía obtenida de la transferencia de iones. Esta transferencia es impulsada por el gradiente electroquímico que existe entre ambos lados de la membrana celular.[25]
Las fimbrias son filamentos finos de proteínas que se distribuyen sobre la superficie de la célula. Tienen un diámetro aproximado de 2-10 nm y una longitud de hasta varios μm. Cuando se observan a través del microscopio electrónico se asemejan a pelos finos. Las fimbrias ayudan a la adherencia de las bacterias a las superficies sólidas o a otras células y son esenciales para la virulencia de algunos patógenos.[26] Los pilis son apéndices celulares ligeramente mayores que las fimbrias y se utilizan para la transferencia de material genético entre bacterias en un proceso denominado conjugación bacteriana.[27]
Muchas bacterias producen una cápsula que recubre sus células. Estas cápsulas varían en complejidad estructural: desde una simple capa de moco compuesta de un polímero extracelular hasta una cápsula altamente estructurada o glicocalix. Estas estructuras protegen a las bacterias pues dificultan que sean ingeridas por células eucariotas tales como macrófagos.[28] Las cápsulas pueden también actuar como antígenos y estar implicadas en el reconocimiento bacterial, así como ayudar a la adherencia superficial y a la formación de biopelículas. [29]
La formación de estas estructuras extracelulares depende del sistema de secreción bacteriano. Este sistema transfiere proteínas desde el citoplasma al periplasma o al espacio que rodea a la célula. Se conocen muchos tipos de sistemas de secreción, que son a menudo esenciales para la virulencia de los patógenos, por lo que han sido y son extensamente estudiados.[30]
Endosporas

Ciertos géneros de bacterias Gram-positivas, tales como Bacillus, Clostridium, Sporohalobacter, Anaerobacter y Heliobacterium, pueden formar endosporas.[31] Las endosporas son estructuras durmientes altamente resistentes cuya función primaria es sobrevivir cuando las condiciones ambientales son adversas. En casi todos los casos, las endosporas no forman parte de un proceso reproductivo, aunque Anaerobacter puede formar hasta siete endosporas a partir de una célula.[32] Las endosporas tienen una base central de citoplasma que contiene ADN y ribosomas, rodeada por una corteza y protegida por una cubierta impermeable y rígida.
Las endosporas no presentan un metabolismo detectable y pueden sobrevivir a condiciones físicas y químicas extremas, tales como altos niveles de luz ultravioleta, rayos gamma, detergentes, desinfectantes, calor, presión y desecación.[33] En este estado durmiente, las bacterias pueden seguir viviendo durante millones de años,[34][35] e incluso pueden sobrevivir en la radiación y vacío del espacio exterior.[36] Las endosporas pueden también causar enfermedades. Por ejemplo, puede contraerse carbunco por la inhalación de endosporas de Bacillus anthracis y tétanos por la contaminación de las heridas con endosporas de Clostridium tetani.[37]
Clasificación morfológica de bacterias

La forma de las bacterias es muy variada y, a menudo, una misma especie adopta distintos tipos morfológicos, es lo que se conoce como pleomorfismo. De todas forma, podemos distinguir tres tipos fundamentales de bacterias:
- Coco: de forma esférica
- Diplococo: cocos en grupos de dos
- Tetracoco: cocos en grupos de cuatro
- Estreptococo: cocos en cadenas
- Estafilococo: cocos en agrupaciones irregulares o en racimo
- Bacilo: en forma de bastoncillo
- Formas helicoidales
- Vibrio: ligeramente curvados y en forma de coma
- Espirilo: en forma helicoidal rígida
- Espiroqueta: en forma de tirabuzón (helicoidal flexible)
Ejemplos:
Metabolismo
En contraste con los organismos superiores, las bacterias exhiben una gran variedad de tipos metabólicos.[38] La distribución de estos tipos metabólicos dentro de un grupo de bacterias se ha utilizado tradicionalmente para definir su taxonomía, pero estos rasgos no corresponden a menudo con las clasificaciones genéticas modernas.[39] El metabolismo bacteriano se clasifica en base de tres criterios importantes: el origen del carbono, la fuente de energía y los donadores de electrones. Un criterio adicional para clasificar a los microorganismos que respiran es el receptor de electrones usado en la respiración.[40]
Según la fuente del carbono, las bacterias se pueden clasificar como heterótrofas cuando usan compuestos orgánicos, o como autótrofas cuando el carbono celular se obtiene mediante la fijación del dióxido de carbono. Las bacterias autótrofas típicas son las cyanobacterias fotosintéticas, las bacterias verdes del azufre y algunas bacterias púrpura. Pero hay también muchas otras especies quimiolitotrofas, por ejemplo, las bacterias nitrificantes y oxidantes del azufre.[41] Según la fuente de energía, las bacterias pueden ser fototrofas, es decir, que emplean la luz a través de la fotosíntesis, o quimiotrofas, es decir, que obtienen energía a partir de sustancias químicas que son oxidadas principalmente a expensas del oxígeno o de otros receptotes de electrones alternativos (respiración aerobia/anaerobia).
Finalmente, las bacterias se pueden clasificar como litotrofas o organotrofas según utilicen como donadores de electrones compuestos inorgánicos u orgánicos, respectivamente. Los organismos quimiotrofos usan donadores de electrones para la conservación de energía (durante la respiración aerobia/anaerobia o durante la fermentación) y para las reacciones biosintéticas (por ejemplo, para la fijación del dióxido de carbono), mientras que los organismos fototrofos los utilizan solamente para propósitos biosintéticos. Los organismos que respiran usan compuestos químicos como fuente de energía, tomando electrones del substrato reducido y transfiriéndolos a un receptor terminal de electrones en una reacción redox. Esta reacción desprende energía que se puede utilizar para sintetizar ATP y conducir el metabolismo. En los organismos aerobios, el oxígeno se utiliza como receptor de electrones. En los organismos anaerobios se utilizan como receptores de electrones otros compuestos inorgánicos tales como los nitratos, sulfatos o el dióxido de carbono. Esto conduce a los procesos ecológicamente importantes de la desnitrificación, reducción del sulfato y acetogénesis, respectivamente.
Otra posibilidad de los quimiotrofos en ausencia de receptores de electrones es la fermentación. En la fermentación, los electrones tomados de los substratos reducidos se transfieren a intermediarios oxidados para generar productos de fermentación reducidos (por ejemplo, lactato, etanol, hidrógeno, butirato). La fermentación es posible porque el contenido de energía de los substratos es mayor que el de los productos, lo que permite que los organismos sinteticen ATP y conduzcan su metabolismo.[42][43]
Estos procesos son también importantes en las respuestas biológicas a la contaminación. Por ejemplo, las bacterias reductoras de sulfato son en gran parte responsables de la producción de formas altamente tóxicas de mercurio (metil- y dimetil-mercurio) en el ambiente.[44] Los organismos anaerobios utilizan la fermentación para generar energía, secretando subproductos metabólicos (tales como el etanol en la elaboración de la cerveza) como productos de desecho. Los anaerobios facultativos pueden elegir entre la fermentación y diversos receptores terminales de electrones dependiendo de las condiciones ambientales en las cuales se encuentren.
Las bacterias litotrofas pueden utilizar compuestos inorgánicos como fuente de la energía. Los donantes de electrones inorgánicos más comunes son el hidrógeno, monóxido de carbono, amoníaco (que conduce a la nitrificación), hierro ferroso y otros iones de metales reducidos, y varios compuestos de azufre reducidos. Inusualmente, las bacterias metanotrofas pueden usar gas metano tanto como fuente de electrones como substrato para el anabolismo del carbono.[45] En la fototrofía y quimiolitotrofía aerobias, se utiliza el oxígeno como receptor terminal de electrones, mientras que bajo condiciones anaerobias se utilizan compuestos inorgánicos. La mayoría de los organismos litótrofos son autótrofos, mientras que los organismos organótrófos son heterótrofos.
Además de la fijación del dióxido de carbono mediante la fotosíntesis, algunas bacterias también fijan el gas nitrógeno usando la encima nitrogenasa. Esta característica es ambientalmente importante y se puede encontrar en bacterias de casi todos los tipos metabólicos enumerados anteriormente, pero no es universal.[46]
Reproducción
En las bacterias, el aumento en el tamaño de las células (crecimiento) y la reproducción por división celular están íntimamente ligadas, como en la mayor parte de los organismos unicelulares. Las bacterias crecen hasta un tamaño fijo y después se reproducen por fisión binaria, una forma de reproducción asexual.[47] En condiciones apropiadas, una bacteria Gram-positiva puede dividirse cada 20 – 30 minutos y una Gram-negativa cada 15 – 20 minutos, y en alrededor de 16 horas su número puede ascender a unos 5.000 millones (aproximadamente el número de personas que habitan la Tierra). Bajo condiciones óptimas, algunas bacterias pueden crecer y dividirse extremadamente rápido, tanto como cada 9,8 minutos.[48] En la división celular se producen dos células hijas idénticas.
Algunas bacterias, todavía reproduciéndose asexualmente, forman estructuras reproductivas más complejas que facilitan la dispersión de las células hijas recién formadas. Ejemplos incluyen la formación de cuerpos fructíferos (esporangios) en Myxobacteria, la formación de hifas en Streptomyces y la gemación. En la gemación una célula forma una protuberancia que a continuación se separa y produce una nueva célula hija.
Genética
La mayoría de las bacterias disponen de un único cromosoma circular cuyo tamaño puede ir desde sólo 160.000 pares de bases en la bacteria endosimbionte Candidatus Carsonella ruddii[49] a los 12.200.000 pares de bases de la bacteria del suelo Sorangium cellulosum.[50] Las spirochaetes del género Borrelia son una notable excepción a esta regla pues, por ejemplo, Borrelia burgdorferi, la causa del Enfermedad de Lyme, contiene un único cromosoma lineal.[51] Las bacterias pueden también presentar plásmidos, pequeñas móleculas extra-cromosómicas de ADN que pueden contener genes responsables de la resistencia a los antibióticos o factores de virulencia. Otro tipo de ADN bacteriano proviene de la integración de material genético de virus (bacteriófagos). Existen muchos tipos de bacteriófagos, algunos simplemente infectan y rompen las células huésped bacterianas, mientras que otros se insertan en el cromosoma bacteriano. Un bacteriófago puede insertar genes en la bacteria y contribuir a su fenotipo. Por ejemplo, en la evolución de Escherichia coli O157:H7 y Clostridium botulinum, los genes tóxicos aportados por un bacteriófago convirtieron a una inocensiva bacteria ancestral en un patógeno letal.[52][53]
Las bacterias, como organismos asexuales que son, heredan copias idénticas de genes, es decir, son clones. Sin embargo, todas las bacterias pueden evolucionar por la selección de cambios en su ADN debidos a recombinación genética o a mutaciones. Las mutaciones provienen de errores durante la réplica del ADN o por exposición a mutagénicos. Las tasas de mutación varían extensamente entre las diversas especies de bacterias e incluso entre diferentes clones de una misma especie de bacteria.[54] Los cambios genéticos en los genomas bacterianos se producen bien por mutaciones ocurridas al azar durante la replicación como por mutaciones dirigidas por estrés, en donde los genes implicados en algún proceso que limita el crecimiento tienen una mayor tasa de mutación.[55]
Las bacterias también pueden transferirse material genético entre células. Esto puede realizarse en tres formas principales. En primer lugar, las bacterias pueden recoger ADN exógeno del ambiente en un proceso denominado transformación. Los genes también se pueden transferir por un proceso del transducción mediante un bacteriófago que introduce ADN extraño en el cromosoma bacteriano. El tercer método de transferencia de genes es por conjugación bacteriana, en donde el ADN se transfiere a través del contacto directo entre células. Esta adquisición de genes de otras bacterias o del ambiente se denomina transferencia de genes horizontal y puede ser común en condiciones naturales[56] La transferencia de genes es especialmente importante en la resistencia a los antibióticos, pues permite una rápida diseminación de los genes responsables de la resistencia entre diferentes patógenos.[57]
Interacciones con otros organismos
A pesar de su simplicidad evidente, las bacterias pueden formar asociaciones complejas con otros organismos. Estas asociaciones se pueden clasificar como parasitismo, mutualismo y comensalismo.
Comensales
Debido a su pequeño tamaño, las bacterias comensales son ubícuas y crecen sobre animales y plantas exactamente igual a como crecerían sobre cualquier otra superficie. Así, por ejemplo, grandes poblaciones de estos organismos son las causantes del mal olor corporal y su crecimiento puede aumentar con calor y sudor.
Mutualistas
Ciertas bacterias forman asociaciones íntimas con otros organismos, que les son imprescindibles para su supervivencia. Una de estas asociaciones mutualistas es la transferencia de hidrógeno entre especies. Se produce entre grupos de bacterias anaerobias que consumen ácidos orgánicos tales como ácido butírico o ácido propiónico y producen hidrógeno y las archaea metanógenas que consumen este hidrógeno.[58] Las bacterias en esta asociación no pueden consumir los ácidos orgánicos cuando el hidrógeno se acumula a su alrededor. Solamente la asociación íntima con las archaea mantiene la concentración de hidrógeno lo bastante baja para permitir que las bacterias crezcan.
En el suelo, los microorganismos que habitan la rizosfera (la zona que incluye la superficie de la raíz y la tierra que se adhiere a ella) realizan la fijación de nitrógeno, convirtiendo el gas nitrógeno en compuestos nitrogenados.[59] Esto proporciona a muchas plantas que no pueden fijar el gas nitrógeno ellas mismas, una forma fácilmente absorbible de nitrógeno.
Muchas otras bacterias se encuentran como simbiontes en seres humanos y en otros organismos. Por ejemplo, en el tracto digestivo proliferan unas mil especies bacterianas. Sintetizan vitaminas tales como ácido fólico, vitamina K y biotina. También fermentan los carbohidratos complejos indigeribles y convierten las proteínas de la leche en ácido láctico (por ejemplo, Lactobacillus).[60][61][62] Además, la presencia de esta flora intestinal inhibe el crecimiento de bacterias potencialmente patógenas (generalmente por exclusión competitiva). Muchas veces estas bacterias beneficiosas se venden como suplementos dietéticos probióticos.[63]
Patógenos
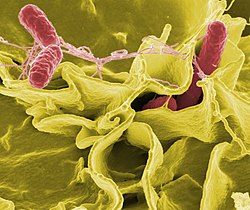
Las bacterias patógenas son una de las principales causas de las enfermedades y de la mortalidad humana, causando infecciones tales como tétanos, fiebre tifoidea, difteria, sífilis, cólera, intoxicaciones alimentarias, lepra y tuberculosis. Puede ocurrir que, para una enfermedad médicamente conocida, su causa patógenica se descubra solamente después de muchos años, como fue caso de la úlcera péptica y Helicobacter pylori. Las enfermedades bacterianas son también importantes en la agricultura y en la ganadería, por ejemplo, el carbunco.
Cada especie de patógeno tiene un espectro característico de interacciones con sus huéspedes humanos. Algunos organismos, tales Staphylococcus o Streptococcus, pueden causar infecciones de la piel, pulmonía, meningitis e incluso sepsis, una respuesta inflamatoria sistémica que produce shock, vasodilatación masiva y muerte.[64] Sin embargo, estos organismos son también parte de la flora humana normal y se encuentran generalmente en la piel o nariz sin causar ninguna enfermedad.
Otros organismos causan invariablemente enfermedades en los seres humanos. Por ejemplo, Rickettsia, parásitos intracelulares obligados, capaces de crecer y reproducirse solamente dentro de las células de otros organismos. Una especie de Rickettsia causa tifus, mientras que otra ocasiona la fiebre de las Montañas Rocosas. Chlamydiae, otro filo de parásitos obligados intracelulares, contiene especies que causan pulmonía, infecciones urinarias y pueden estar implicadas en las enfermedades cardíacas coronarias.[65] Finalmente, ciertas especies tales como Pseudomonas aeruginosa, Burkholderia cenocepacia y Mycobacterium aviumaeruginosa son patógenos oportunistas y causan enfermedades principalmente en las personas que sufren inmunosupresión o fibrosis quística.[66][67]
Las infecciones bacterianas se pueden tratar con antibióticos, que se clasifican como bactericidas si matan bacterias, o bacterioestáticos si solo detienen el crecimiento bacteriano. Existen muchos tipos de antibióticos y cada tipo inhibe un proceso que difiere en el patógeno con respecto al huésped. Ejemplos de antibióticos de toxicidad selectiva son el cloranfenicol y la puromicina, que inhiben el ribosoma bacteriano, pero no el ribosoma eucariota que es estructuralmente diferente.[68] Los antibióticos se utilizan para tratar enfermedades humanas y en la ganaderia intensiva para promover el crecimiento animal. Esto último puede contribuir al rápido desarrollo de la resistencia antibiótica de las poblaciones bacterianas.[69] Las infecciones se pueden prevenir con medidas antisépticas tales como la esterilización de la piel antes de las inyecciones y con el cuidado apropiado de los catéteres. Los instrumentos quirúrgicos y dentales también son esterilizados para prevenir la contaminación e infección por bacterias. Los desinfectantes se utilizan para matar a bacterias u otros patógenos que se depositan sobre las superficies para prevenir la contaminación y reducir el riesgo de infección.
Taxonomía y evolución

Las bacterias forman uno de los tres dominios en los que se dividen los seres vivos. En los antiguos sistemas taxonómicos, las bacterias formaban un subreino del reino Monera.
Los antepasados de las bacterias modernas fueron los primeros microorganismos unicelulares que se desarrollaron sobre la tierra, hace aproximadamente 4.000 millones años. Durante cerca de 3.000 millones de años más, todos los organismos siguieron siendo microscópicos, siendo Bacteria y Archaea las formas dominantes de vida.[70][71] Aunque existen fósiles bacterianos, por ejemplo los estromatolitos, al no conservar su morfología distintiva no se pueden emplear para estudiar la historia de la evolución bacteriana, o el origen de una especie bacteriana en particular. Sin embargo, las secuencias genéticas sí se pueden utilizar para reconstruir la filogenética bacteriana, y estos estudios indican que las bacterias fueron las primeras en separarse del linaje Archaea/Eukarya.[72] El ancestro común más reciente de bacterias y archaeas fue probablemente un hipertermófilo que vivió entre 2500 y 3200 millones de años atrás.[73][74]
Las bacterias también han estado implicadas en la segunda gran divergencia evolutiva, la que separó Archaea de Eukarya. En este caso, los eucariontes surgieron por la asociación simbiótica de una bacteria con el antepasado de las células eucariotas, que posiblemente estaba relacionado con archaea.[75][76] Ello implicó la endosimbiosis de una proteobacteria alfa dentro del citoplasma de una célula proto-eucariota para formar las mitocondrias o hidrogenosomas, que todavía se pueden encontrar en todos los Eukarya (aunque a veces en una forma altamente reducida, por ejemplo en los protozoos amitocondriales). Después e independientemente, una segunda endosimbiosis por parte de algún eucarionte mitocondrial de un organismo similar a las cianobacterias condujo a la formación de los cloroplastos de algas y plantas. Se conocen incluso algunos grupos de algas que se han originado claramente de acontecimientos posteriores de endosimbiosis por parte de eucariotas heterótrofos que ingirieron algas eucariotas que se han convertido en plastos de segunda generación.[77][78]
Tinción de Gram
La técnica de tinción de membranas de bacterias de Gram ha supuesto un antes y un después en el campo de la medicina, y consiste en teñir con tintes específicos diversas muestras de baterías en portaobjetos para saber si se han teñido o no con dicho tinte.
Cuando se han adicionado los tintes específicos en las muestras, quitando el sobrante pasados unos minutos para evitar confusiones, hay que limpiarlas con unas gotas de alcohol etílico. La función del alcohol es la de eliminar el tinte de las bacterias, y es aquí donde se reconocen las bacterias que se han tomado: Si la bacteria conserva el tinte, es Gram positiva, posee una membrana más gruesa constituida por varias decenas de capas de diversos componentes proteínicos; en el caso de que el tinte no se mantenga, la bacteria es Gram negativa, la cual solo posee una membrana simple. La función biológica que posee ésta técnica es la de fabricar antibióticos específicos para esas bacterias.
Tinción empleada en microbiología para la visualización de bacterias en muestras clínicas. También se emplea como primer paso en la diferenciación bacteriana, considerándose bacteria Gram positivas a las bacterias que se visualizan de color violeta y gram negativas a las que se visualizan de color rojo.
En estudio de muestras clínicas suele ser un estudio fundamental por cumplir varias funciones:
- Identificación preliminar de la bacteria causal de la infección.
- Consideración de la calidad de la muestra biológica para el estudio, es decir permite apreciar el número de células inflamatorias así como de células epiteliales. A mayor número de células inflamatorias en cada campo del microscopio, más probabilidad de que la flora que crezca en los medios de cultivo sea la representativa del lugar de la infección. A mayor número de células epiteliales sucede los contrario, mayor probabilidad de contaminación con flora saprófita y la flora aislada en los medios de cultivos no es representativa del lugar de la infección.
- Utilidad como control calidad del aislamiento bacteriano. Los morfotipos bacterianos identificados en la tinción de Gram se deben de corresponder con aislamientos bacterianos realizados en los cultivos. Si se observan mayor número de formas bacterianas que las aisladas hay que reconsiderar los medios de cultivos empleados así como la atmósfera de incubación.
Utilidad económica de las bacterias
Muchas industrias dependen en parte o enteramente de la acción bacteriana. Gran cantidad de sustancias quimicas importantes como alcohol etílico, ácido acético, alcohol butílico y acetona son producidas por bacterias especificas. Se tienen que emplear bacterias para el curado de tabaco, el curtido de cueros, caucho, algodón, etc. Las bacterias (a menudo Lactobacillus) junto con levaduras y mohos, se han utilizado durante miles de años para la preparación de alimentos fermentados tales como queso, mantequilla, encurtidos, salsa de soja, chucrut, vinagre, vino y yogur. [79][80]
Las bacterias tienen una capacidad notable para degradar una gran variedad de compuestos orgánicos, por lo que se utilizan en el reciclado de basura y en biorremediación. Las bacterias capaces de degradar los hidrocarburos son de uso frecuente en la limpieza de los vertidos de petróleo.[81] Así por ejemplo, después del vertido del petrolero Exxon Valdez en 1989, en algunas playas de Alaska se usaron fertilizantes con objeto de promover el crecimiento de estas bacterias naturales. Estos esfuerzos fueron eficaces en las playas en las que la capa de petróleo no era demasiado espesa. Las bacterias también se utilizan para la biorremediación de basuras tóxicas industriales.[82] En la industria química, las bacterias son utilizadas en la producción de productos químicos enantioméricamente puros para uso farmacéutico o agroquímico.[83]
Las bacterias también pueden ser utilizadas para el control biológico de parásitos en lugar de los pesticidas. Esto implica comúnmente la especie Bacillus thuringiensis (también llamado BT), una bacteria de suelo Gram-positiva. Las subespecies de esta bacteria se utilizan como insecticidas específicos para lepidópteros.[84] Debido a su especificidad, estos pesticidas se consideran respectuosos con el medio ambiente, con poco o ningún efecto sobre los seres humanos, fauna y la mayoría de los insectos beneficiosos, como por ejemplo, los polinizadores.[85][86]
Las bacterias son herramientas básicas en los campos de la biología, genética y bioquímica moleculares debido a su capacidad para crecer rápidamente y a la facilidad relativa con la que pueden ser manipuladas. Realizando modificaciones en el ADN bacteriano y examinando los fenotipos que resultan, los científicos pueden determinar la función de genes, enzimas y rutas metabólicas, y después estos conocimientos se pueden trasladar a organismos más complejos.[87] La comprensión de la bioquímica celular, que require cantidades enormes de datos de cinética enzimática y de expresión de genes, permitirá realizar modelos matemáticos de organismos enteros. Esto es realizable en alguna bacteria bien estudiada, por ejemplo, actualmente está siendo desarrollado y probado el modelo del metabolismo de Escherichia coli.[88][89] Esta comprensión del metabolismo y genética bacterianos permite a la biotecnología la modificación de las bacterias para que produzcan proteínas terapéuticas, tales como insulina, factores de crecimiento y anticuerpos.[90][91]
Enlaces externos
 Wikispecies tiene un artículo sobre Bacteria.
Wikispecies tiene un artículo sobre Bacteria. Wikcionario tiene definiciones y otra información sobre bacteria.
Wikcionario tiene definiciones y otra información sobre bacteria.- Bacterias: Cómo transformaron la Tierra
- agronlin.tripod.com; datos sobre bacterias
- Crea un cultivo de bacterias
Referencias
- ↑ Fredrickson J, Zachara J, Balkwill D, et al (2004). «Geomicrobiology of high-level nuclear waste-contaminated vadose sediments at the hanford site, Washington state». Appl Environ Microbiol 70 (7): 4230 - 41. PMID 15240306.
- ↑ Whitman W, Coleman D, Wiebe W (1998). «Prokaryotes: the unseen majority». Proc Natl Acad Sci U S A 95 (12): 6578 - 83. PMID 9618454.
- ↑ Rappé M, Giovannoni S. «The uncultured microbial majority». Annu Rev Microbiol 57: 369 - 94. PMID 14527284.
- ↑ Sears C (2005). «A dynamic partnership: Celebrating our gut flora». Anaerobe 11 (5): 247 - 51. PMID 16701579.
- ↑ 2002 WHO mortality data Accessed 20 January 2007
- ↑ Ishige T, Honda K, Shimizu S (2005). «Whole organism biocatalysis». Curr Opin Chem Biol 9 (2): 174 - 80. PMID 15811802.
- ↑ Woese C, Kandler O, Wheelis M (1990). «Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya». Proc Natl Acad Sci U S A 87 (12): 4576 - 9. PMID 2112744.
- ↑ Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Harold F (1972). «Conservation and transformation of energy by bacterial membranes». Bacteriol Rev 36 (2): 172-230. PMID 4261111.
- ↑ Thanbichler M, Wang S, Shapiro L (2005). «The bacterial nucleoid: a highly organized and dynamic structure». J Cell Biochem 96 (3): 506-21. PMID 15988757.
- ↑ Poehlsgaard J, Douthwaite S (2005). «The bacterial ribosome as a target for antibiotics». Nat Rev Microbiol 3 (11): 870-81. PMID 16261170.
- ↑ Fuerst J (2005). «Intracellular compartmentation in planctomycetes». Annu Rev Microbiol 59: 299-328. PMID 15910279.
- ↑ Yeo M, Chater K (2005). «The interplay of glycogen metabolism and differentiation provides an insight into the developmental biology of Streptomyces coelicolor». Microbiology 151 (Pt 3): 855-61. PMID 15758231.
- ↑ Shiba T, Tsutsumi K, Ishige K, Noguchi T (2000). «Inorganic polyphosphate and polyphosphate kinase: their novel biological functions and applications». Biochemistry (Mosc) 65 (3): 315-23. PMID 10739474.
- ↑ Brune DC. (1995). «Isolation and characterization of sulfur globule proteins from Chromatium vinosum and Thiocapsa roseopersicina». Arch Microbiol 163 (6): 391-99. PMID 7575095.
- ↑ Kadouri D, Jurkevitch E, Okon Y, Castro-Sowinski S. (2005). «Ecological and agricultural significance of bacterial polyhydroxyalkanoates». Crit Rev Microbiol 31 (2): 55-67. PMID 15986831.
- ↑ Walsby A (1994). «Gas vesicles». Microbiol Rev 58 (1): 94-144. PMID 8177173.
- ↑ van Heijenoort J (2001). «Formation of the glycan chains in the synthesis of bacterial peptidoglycan». Glycobiology 11 (3): 25R - 36R. PMID 11320055.
- ↑ a b Koch A (2003). «Bacterial wall as target for attack: past, present, and future research». Clin Microbiol Rev 16 (4): 673 - 87. PMID 14557293.
- ↑ Gram, HC (1884). «Über die isolierte Färbung der Schizomyceten in Schnitt- und Trockenpräparaten». Fortschr. Med. 2: 185–189.
- ↑ Hugenholtz P (2002). «Exploring prokaryotic diversity in the genomic era». Genome Biol 3 (2): REVIEWS0003. PMID 11864374.
- ↑ Walsh F, Amyes S (2004). «Microbiology and drug resistance mechanisms of fully resistant pathogens.». Curr Opin Microbiol 7 (5): 439-44. PMID 15451497.
- ↑ Engelhardt H, Peters J (1998). «Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions». J Struct Biol 124 (2 - 3): 276-302. PMID 10049812.
- ↑ Beveridge T, Pouwels P, Sára M, Kotiranta A, Lounatmaa K, Kari K, Kerosuo E, Haapasalo M, Egelseer E, Schocher I, Sleytr U, Morelli L, Callegari M, Nomellini J, Bingle W, Smit J, Leibovitz E, Lemaire M, Miras I, Salamitou S, Béguin P, Ohayon H, Gounon P, Matuschek M, Koval S (1997). «Functions of S-layers». FEMS Microbiol Rev 20 (1 - 2): 99 - 149. PMID 9276929.
- ↑ Kojima S, Blair D. «The bacterial flagellar motor: structure and function of a complex molecular machine». Int Rev Cytol 233: 93 - 134. PMID 15037363.
- ↑ Beachey E (1981). «Bacterial adherence: adhesin-receptor interactions mediating the attachment of bacteria to mucosal surface». J Infect Dis 143 (3): 325 - 45. PMID 7014727.
- ↑ Silverman P (1997). «Towards a structural biology of bacterial conjugation». Mol Microbiol 23 (3): 423 - 9. PMID 9044277.
- ↑ Stokes R, Norris-Jones R, Brooks D, Beveridge T, Doxsee D, Thorson L (2004). «The glycan-rich outer layer of the cell wall of Mycobacterium tuberculosis acts as an antiphagocytic capsule limiting the association of the bacterium with macrophages». Infect Immun 72 (10): 5676 - 86. PMID 15385466.
- ↑ Daffé M, Etienne G (1999). «The capsule of Mycobacterium tuberculosis and its implications for pathogenicity». Tuber Lung Dis 79 (3): 153 - 69. PMID 10656114.
- ↑ Finlay B, Falkow S (1997). «Common themes in microbial pathogenicity revisited». Microbiol Mol Biol Rev 61 (2): 136 - 69. PMID 9184008.
- ↑ Nicholson W, Munakata N, Horneck G, Melosh H, Setlow P (2000). «Resistance of Bacillus endospores to extreme terrestrial and extraterrestrial environments». Microbiol Mol Biol Rev 64 (3): 548 - 72. PMID 10974126.
- ↑ Siunov A, Nikitin D, Suzina N, Dmitriev V, Kuzmin N, Duda V. «Phylogenetic status of Anaerobacter polyendosporus, an anaerobic, polysporogenic bacterium». Int J Syst Bacteriol. 49 Pt 3: 1119 - 24. PMID 10425769.
- ↑ Nicholson W, Fajardo-Cavazos P, Rebeil R, Slieman T, Riesenman P, Law J, Xue Y (2002). «Bacterial endospores and their significance in stress resistance». Antonie Van Leeuwenhoek 81 (1 - 4): 27 - 32. PMID 12448702.
- ↑ Vreeland R, Rosenzweig W, Powers D (2000). «Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal». Nature 407 (6806): 897 - 900. PMID 11057666.
- ↑ Cano R, Borucki M (1995). «Revival and identification of bacterial spores in 25- to 40-million-year-old Dominican amber». Science 268 (5213): 1060 - 4. PMID 7538699.
- ↑ Nicholson W, Schuerger A, Setlow P (2005). «The solar UV environment and bacterial spore UV resistance: considerations for Earth-to-Mars transport by natural processes and human spaceflight». Mutat Res 571 (1 - 2): 249 - 64. PMID 15748651.
- ↑ Hatheway C (1990). «Toxigenic clostridia». Clin Microbiol Rev 3 (1): 66 - 98. PMID 2404569.
- ↑ Nealson K (1999). «Post-Viking microbiology: new approaches, new data, new insights». Orig Life Evol Biosph 29 (1): 73-93. PMID 11536899.
- ↑ Xu J (2006). «Microbial ecology in the age of genomics and metagenomics: concepts, tools, and recent advances». Mol Ecol 15 (7): 1713-31. PMID 16689892.
- ↑ Zillig W (1991). «Comparative biochemistry of Archaea and Bacteria». Curr Opin Genet Dev 1 (4): 544-51. PMID 1822288.
- ↑ Hellingwerf K, Crielaard W, Hoff W, Matthijs H, Mur L, van Rotterdam B (1994). «Photobiology of bacteria». Antonie Van Leeuwenhoek 65 (4): 331 - 47. PMID 7832590.
- ↑ Zumft W (1997). «Cell biology and molecular basis of denitrification». Microbiol Mol Biol Rev 61 (4): 533 - 616. PMID 9409151.
- ↑ Drake H, Daniel S, Küsel K, Matthies C, Kuhner C, Braus-Stromeyer S (1997). «Acetogenic bacteria: what are the in situ consequences of their diverse metabolic versatilities?». Biofactors 6 (1): 13 - 24. PMID 9233536.
- ↑ Morel, FMM; Kraepiel AML, Amyot M (1998). «The chemical cycle and bioaccumulation of mercury». Annual Review of Ecological Systems 29: 543—566.
- ↑ Dalton H (2005). «The Leeuwenhoek Lecture 2000 the natural and unnatural history of methane-oxidizing bacteria». Philos Trans R Soc Lond B Biol Sci 360 (1458): 1207 - 22. PMID 16147517.
- ↑ Zehr J, Jenkins B, Short S, Steward G (2003). «Nitrogenase gene diversity and microbial community structure: a cross-system comparison». Environ Microbiol 5 (7): 539 - 54. PMID 12823187.
- ↑ Koch A (2002). «Control of the bacterial cell cycle by cytoplasmic growth». Crit Rev Microbiol 28 (1): 61 - 77. PMID 12003041.
- ↑ Eagon R. «Pseudomonas natriegens, a marine bacterium with a generation time of less than 10 minutes». J Bacteriol 83: 736 - 7. PMID 13888946.
- ↑ Nakabachi A, Yamashita A, Toh H, Ishikawa H, Dunbar H, Moran N, Hattori M (2006). «The 160-kilobase genome of the bacterial endosymbiont Carsonella». Science 314 (5797): 267. PMID 17038615.
- ↑ Pradella S, Hans A, Spröer C, Reichenbach H, Gerth K, Beyer S (2002). «Characterisation, genome size and genetic manipulation of the myxobacterium Sorangium cellulosum So ce56». Arch Microbiol 178 (6): 484-92. PMID 12420170.
- ↑ Hinnebusch J, Tilly K (1993). «Linear plasmids and chromosomes in bacteria». Mol Microbiol 10 (5): 917-22. PMID 7934868.
- ↑ Brüssow H, Canchaya C, Hardt W (2004). «Phages and the evolution of bacterial pathogens: from genomic rearrangements to lysogenic conversion». Microbiol Mol Biol Rev 68 (3): 560-602. PMID 15353570.
- ↑ Perna N, Mayhew G, Pósfai G, Elliott S, Donnenberg M, Kaper J, Blattner F (1998). «Molecular evolution of a pathogenicity island from enterohemorrhagic Escherichia coli O157:H7». Infect Immun 66 (8): 3810-7. PMID 9673266.
- ↑ Denamur E, Matic I (2006). «Evolution of mutation rates in bacteria». Mol Microbiol 60 (4): 820 - 7. PMID 16677295.
- ↑ Wright B (2004). «Stress-directed adaptive mutations and evolution». Mol Microbiol 52 (3): 643 - 50. PMID 15101972.
- ↑ Davison J (1999). «Genetic exchange between bacteria in the environment». Plasmid 42 (2): 73 - 91. PMID 10489325.
- ↑ Hastings P, Rosenberg S, Slack A (2004). «Antibiotic-induced lateral transfer of antibiotic resistance». Trends Microbiol 12 (9): 401 - 4. PMID 15337159.
- ↑ Stams A, de Bok F, Plugge C, van Eekert M, Dolfing J, Schraa G (2006). «Exocellular electron transfer in anaerobic microbial communities». Environ Microbiol 8 (3): 371–82. PMID 16478444.
- ↑ Barea J, Pozo M, Azcón R, Azcón-Aguilar C (2005). «Microbial co-operation in the rhizosphere». J Exp Bot 56 (417): 1761–78. PMID 15911555.
- ↑ O'Hara A, Shanahan F (2006). «The gut flora as a forgotten organ». EMBO Rep 7 (7): 688–93. PMID 16819463.
- ↑ Zoetendal E, Vaughan E, de Vos W (2006). «A microbial world within us». Mol Microbiol 59 (6): 1639–50. PMID 16553872.
- ↑ Gorbach S (1990). «Lactic acid bacteria and human health». Ann Med 22 (1): 37–41. PMID 2109988.
- ↑ Salminen S, Gueimonde M, Isolauri E (2005). «Probiotics that modify disease risk». J Nutr 135 (5): 1294–8. PMID 15867327.
- ↑ Fish D. «Optimal antimicrobial therapy for sepsis». Am J Health Syst Pharm. 59 Suppl 1: S13–9. PMID 11885408.
- ↑ Belland R, Ouellette S, Gieffers J, Byrne G (2004). «Chlamydia pneumoniae and atherosclerosis». Cell Microbiol 6 (2): 117–27. PMID 14706098.
- ↑ Heise E. «Diseases associated with immunosuppression». Environ Health Perspect 43: 9–19. PMID 7037390.
- ↑ Saiman, L. «Microbiology of early CF lung disease». Paediatr Respir Rev.volume=5 Suppl A: S367–369. Parámetro desconocido
|yar=ignorado (ayuda) PMID 14980298 - ↑ Yonath A, Bashan A (2004). «Ribosomal crystallography: initiation, peptide bond formation, and amino acid polymerization are hampered by antibiotics». Annu Rev Microbiol 58: 233–51. PMID 15487937.
- ↑ Khachatourians G (1998). «Agricultural use of antibiotics and the evolution and transfer of antibiotic-resistant bacteria». CMAJ 159 (9): 1129-36. PMID 9835883.
- ↑ Schopf J (1994). «Disparate rates, differing fates: tempo and mode of evolution changed from the Precambrian to the Phanerozoic». Proc Natl Acad Sci U S A 91 (15): 6735-42. PMID 8041691.
- ↑ DeLong E, Pace N (2001). «Environmental diversity of bacteria and archaea». Syst Biol 50 (4): 470-78. PMID 12116647.
- ↑ Brown J, Doolittle W (1997). «Archaea and the prokaryote-to-eukaryote transition». Microbiol Mol Biol Rev 61 (4): 456-502. PMID 9409149.
- ↑ Di Giulio M (2003). «The universal ancestor and the ancestor of bacteria were hyperthermophiles». J Mol Evol 57 (6): 721-30. PMID 14745541.
- ↑ Battistuzzi F, Feijao A, Hedges S. «A genomic timescale of prokaryote evolution: insights into the origin of methanogenesis, phototrophy, and the colonization of land.». BMC Evol Biol 4: 44. PMID 15535883.
- ↑ Poole A, Penny D (2007). «Evaluating hypotheses for the origin of eukaryotes». Bioessays 29 (1): 74-84. PMID 17187354.
- ↑ Dyall S, Brown M, Johnson P (2004). «Ancient invasions: from endosymbionts to organelles». Science 304 (5668): 253 - 7. PMID 15073369.
- ↑ Lang B, Gray M, Burger G. «Mitochondrial genome evolution and the origin of eukaryotes». Annu Rev Genet 33: 351-97. PMID 10690412.
- ↑ McFadden G (1999). «Endosymbiosis and evolution of the plant cell». Curr Opin Plant Biol 2 (6): 513-9. PMID 10607659.
- ↑ Johnson M, Lucey J (2006). «Major technological advances and trends in cheese». J Dairy Sci 89 (4): 1174-8. PMID 16537950.
- ↑ Hagedorn S, Kaphammer B. «Microbial biocatalysis in the generation of flavor and fragrance chemicals». Annu Rev Microbiol 48: 773-800. PMID 7826026.
- ↑ Cohen Y (2002). «Bioremediation of oil by marine microbial mats». Int Microbiol 5 (4): 189–93. PMID 12497184.
- ↑ Neves L, Miyamura T, Moraes D, Penna T, Converti A. «Biofiltration methods for the removal of phenolic residues». Appl Biochem Biotechnol. 129–132: 130-52. PMID 16915636.
- ↑ Liese A, Filho M (1999). «Production of fine chemicals using biocatalysis». Curr Opin Biotechnol 10 (6): 595–603. PMID 10600695.
- ↑ Aronson A, Shai Y (2001). «Why Bacillus thuringiensis insecticidal toxins are so effective: unique features of their mode of action». FEMS Microbiol Lett 195 (1): 1-8. PMID 11166987.
- ↑ Bozsik A (2006). «Susceptibility of adult Coccinella septempunctata (Coleoptera: Coccinellidae) to insecticides with different modes of action». Pest Manag Sci 62 (7): 651-4. PMID 16649191.
- ↑ Chattopadhyay A, Bhatnagar N, Bhatnagar R (2004). «Bacterial insecticidal toxins». Crit Rev Microbiol 30 (1): 33-54. PMID 15116762.
- ↑ Serres M, Gopal S, Nahum L, Liang P, Gaasterland T, Riley M (2001). «A functional update of the Escherichia coli K-12 genome». Genome Biol 2 (9): RESEARCH0035. PMID 11574054.
- ↑ Almaas E, Kovács B, Vicsek T, Oltvai Z, Barabási A (2004). «Global organization of metabolic fluxes in the bacterium Escherichia coli». Nature 427 (6977): 839-43. PMID 14985762.
- ↑ Reed J, Vo T, Schilling C, Palsson B (2003). «An expanded genome-scale model of Escherichia coli K-12 (iJR904 GSM/GPR)». Genome Biol 4 (9): R54. PMID 12952533.
- ↑ Walsh G (2005). «Therapeutic insulins and their large-scale manufacture». Appl Microbiol Biotechnol 67 (2): 151–9. PMID 15580495.
- ↑ Graumann K, Premstaller A (2006). «Manufacturing of recombinant therapeutic proteins in microbial systems». Biotechnol J 1 (2): 164–86. PMID 16892246.

