Diferencia entre revisiones de «Archaea»
Avance. |
¿Palet? Duuuuuda. |
||
| Línea 73: | Línea 73: | ||
=== Pared celular y flagelos === |
=== Pared celular y flagelos === |
||
La mayoría de arqueas tienen una pared celular, las excepciones son ''[[Thermoplasma]]'' y ''[[Ferroplasma]]''.<ref>{{cite journal |author=Golyshina OV, Pivovarova TA, Karavaiko GI, ''et al'' |title=Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea |journal=Int. J. Syst. Evol. Microbiol. |volume=50 Pt 3 |pages=997–1006 |year=2000 |month=Maig |pmid=10843038 |url=http://ijs.sgmjournals.org/cgi/pmidlookup?view=long&pmid=10843038}}</ref> En la mayoría de arqueas, la pared se compone de [[proteína]]s de capa de superficie, que forman una [[capa S]].<ref>{{cite journal |author=Sára M, Sleytr UB |title=S-Layer proteins |journal=J. Bacteriol. |volume=182 |issue=4 |pages=859–68 |year=2000 |pmid=10648507 |url=http://jb.asm.org/cgi/pmidlookup?view=long&pmid=10648507 |doi=10.1128/JB.182.4.859-868.2000}}</ref> Una capa S es una agrupación rígida de moléculas proteínicas que cubren el exterior de la célula como una [[cota de malla]]<ref>{{cite journal |author=Engelhardt H, Peters J |title=Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions |journal=J Struct Biol |volume=124 |issue=2–3 |pages=276–302 |year=1998|pmid = 10049812 |doi=10.1006/jsbi.1998.4070}}</ref> Esta capa ofrece una producción química y física, y puede servir de barrera, impidiendo que entren en contacto [[macromolécula]]s con la membrana celular.<ref name=Kandler1998/> A diferencia de las bacterias, la mayoría de arqueas carecen de [[peptidoglicano]] en la pared celular.<ref name="Howland">{{Ref-llibre |last=Howland |first=John L. |year=2000 |title=The Surprising Archaea: Discovering Another Domain of Life |pages=32 |location=Oxford |publisher=Oxford University Press |isbn=0-19-511183-4 }}</ref> La excepción es el [[pseudopeptidoglicano]], que se encuentra en las archaeas metanógenas, pero este[[ polímero]] es diferente del peptidoglicano bacteriano ya que carece de [[aminoácido]]s y [[ácido N-acetilmurámico]].<ref name=Kandler1998>{{cite journal | authors=Kandler, O.; König, H. |year=1998 |title=Cell wall polymers in Archaea (Archaebacteria) |journal=Cellular and Molecular Life Sciences (CMLS) |volume=54 |issue=4 |pages=305–308 |doi=10.1007/s000180050156 |url=http://www.springerlink.com/index/PXMTKQ8WH8X650ED.pdf}}</ref> |
|||
La mayoría de arqueas tienen una pared celular, las excepciones son [[Thermoplasma]] y [[Ferroplasma]]. |
|||
Las arqueas también tienen [[flagelo]]s, que funcionan de una manera parecida a los [[flagelo bacteriano|flagelos bacterianos]] —son largas colas que se mueven por motores rotatorios situados en la base de los flagelos. Estos motores son impulsados por el gradiente de protones de la membrana. Sin embargo, los flagelos arquobacterianos son notablemente diferentes en su composición y su desarrollo.<ref name=Thomas>{{cite journal |author=Thomas NA, Bardy SL, Jarrell KF |title=The archaeal flagellum: a different kind of prokaryotic motility structure |journal=FEMS Microbiol. Rev. |volume=25 |issue=2 |pages=147–74 |year=2001 |pmid=11250034 |doi=10.1111/j.1574-6976.2001.tb00575.x}}</ref> Cada tipo de flagelo evolucionó de un antepasado diferente, el flagelo bacteriano evolucionó de un sistema de secreción de tipo III, mientras que los flagelos arquobacterianos parecen haber evolucionado de los ''pili'' bacterianos de tipo IV.<ref>{{cite journal |author=Ng SY, Chaban B, Jarrell KF |title=Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications |journal=J. Mol. Microbiol. Biotechnol. |volume=11 |issue=3–5 |pages=167–91 |year=2006 |pmid=16983194 |doi=10.1159/000094053}}</ref> A diferencia del flagelo bacteriano, que es un palet vacío y que está formado por subunidades que se mueven por la cavidad central y luego se añaden a la punta del flagelo, los flagelos arquobacterianos se sintetizan mediante la adición de subunidades en la su base.<ref>{{cite journal |author=Bardy SL, Ng SY, Jarrell KF |title=Prokaryotic motility structures |journal=Microbiology (Reading, Engl.) |volume=149 |issue=Pt 2 |pages=295–304 |year=2003 |month=Febrer |pmid=12624192 |url=http://mic.sgmjournals.org/cgi/pmidlookup?view=long&pmid=12624192 |doi=10.1099/mic.0.25948-0}}</ref> |
|||
== Genética == |
== Genética == |
||
Revisión del 00:15 19 may 2010
| Archaea | ||
|---|---|---|
| Rango temporal: Arcaico - Holoceno | ||
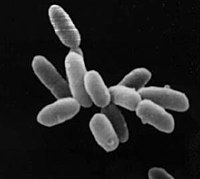 Halobacteria sp. cepa NRC-1, cada célula mide aproximadamente 5 μm de longitud. | ||
| Taxonomía | ||
| Dominio: |
Archaea Woese, Kandler & Wheelis, 1990 | |
| Filos | ||
Las arqueas o arqueobacterias son un grupo de microorganismos unicelulares pertenecientes al dominio Archaea. El término archibacteria es una denominación desestimada. Las arqueas, como las bacterias, son procariotas que carecen de núcleo celular o cualquier otro orgánulo dentro de las células. En el pasado, se las consideró un grupo inusual de bacterias, pero como tienen una historia evolutiva independiente y presentan muchas diferencias en su bioquímica respecto al resto de formas de vida, actualmente se las clasifica como un dominio distinto en el sistema de tres dominios. En este sistema, presentado por Carl Woese, las tres ramas evolutivas principales son las arqueas, las bacterias y las eucariotas. Las arqueas están subdivididas en cuatro filos, de los cuales dos, Crenarchaeota y Euryarchaeota, son estudiados más intensivamente. La clasificación de las arqueas sigue siendo difícil, pues la inmensa mayoría de estos organismos no han sido estudiados en el laboratorio y sólo se les ha detectado mediante el análisis de sus ácidos nucleicos en muestras del medio ambiente.
En general, las arqueas y bacterias son bastante similares en forma y en tamaño, aunque algunas arqueas tienen formas muy inusuales, como las células planas y cuadradas de Haloquadra walsbyi. A pesar de esta semejanza visual con las bacterias, las arqueobacterias poseen genes y varias rutas metabólicas que son más cercanas a las de las eucariotas, notablemente, en las enzimas implicadas en la transcripción y la traducción. Otros aspectos de la bioquímica de las arqueobacterias son únicos, como los éter lípidos de sus membranas celulares. Las arqueas explotan una variedad de recursos mucho mayores que las eucariotas, desde compuestos orgánicos familiares como los azúcares, hasta el uso de amoníaco, iones de metales o incluso hidrógeno como nutrientes. Las arqueas tolerantes de la sal (las halobacterias) utilizan la luz solar como fuente de energía, y otras especies de arqueas fijan carbono, sin embargo, a diferencia de las plantas y las cianobacterias, no se conoce ninguna especie de arquea que sea capaz de ambas cosas. Las arqueas se reproducen asexualmente y se dividen por fisión binaria, fragmentación o gemación, a diferencia de las bacterias y las eucariotas, no se conoce ninguna especie de arquea que forme esporas.
Originalmente, las arqueas eran vistas como extremófilos que vivían en ambientes severos, como aguas termales y lagos salados, pero posteriormente se les ha observado en una gran variedad de hábitats, como suelos, océanos y humedales. Las archaea son especialmente numerosas en los océanos, y las que se encuentran en el plancton podrían ser uno de los grupos de organismos más abundantes del planeta. Estas procariotas son actualmente consideradas una parte importante de la vida en la Tierra y podrían jugar un papel importante tanto en el ciclo del carbono como en el ciclo del nitrógeno. No se conocen ejemplos claros de patógenos o parásitos arquobacterianos, pero suelen ser mutualistas o comensales. Son ejemplos las arqueas metanógenas que viven en el intestino de los humanos y los rumiantes, donde están presentes en grandes cantidades y contribuyen a digerir el alimento. Las arqueas tienen su importancia en la tecnología, hay metanógenos que son utilizados para producir biogás y como parte del proceso de depuración, y las enzimas de arqueas extremófilas capaces de resistir a temperaturas elevadas y solventes orgánicos son explotados en la biotecnología.
Clasificación

Un nuevo dominio
A principios del siglo XX, las procariotas eran consideradas un único grupo de organismos y clasificados según su bioquímica, morfología y metabolismo. Por ejemplo, los microbiólogos intentaban clasificar los microorganismos según la estructura de su pared celular, su forma, y las sustancias que consumen.[1] Sin embargo, en 1965 se propuso un nuevo sistema,[2] utilizando las secuencias genéticas de estos organismos para averiguar qué procariotas están realmente relacionadas entre sí. Este método, conocido como filogenia, es el método principal utilizado actualmente.
Las arqueas fueron clasificados por primera vez como un grupo separado de procariotas en 1977 por Carl Woese y George E. Fox en árboles filogenéticos basados en las secuencias de genes de ARN ribosomal (ARNr).[3] Estos dos grupos fueron originalmente denominados arqueas y eubaceris y tratados como reinos o subreinos, que Woese y Fox denominaron "reinos originales". Woese argumentó que este grupo de procariotas es un tipo de vida fundamentalmente distinto. Para enfatizar esta diferencia, ambos dominios fueron posteriormente llamados Archaea y Eubacteria.[4] El nombre científico Archaea proviene del griego antiguo ἀρχαῖα, que significa "los antiguos".[5] El término "arquea" proviene de la combinación de esta raíz y del término griego baktērion, que significa "pequeño bastón".
Originalmente, sólo se clasificaron los metanógenos en este nuevo dominio, y las arqueas eran consideradas extremófilos que sólo viven en hábitats tales como aguas termales y lagos salados. A finales del siglo XX, los microbiólogos se dieron cuenta de que las Archaea son un grupo de organismos grandes y diversos ampliamente distribuidos en la naturaleza, y que son comunes en hábitats mucho menos extremos, como suelos y océanos.[6] Esta nueva toma de conciencia de la importancia y la omnipresencia vino del uso de la reacción en cadena de la polimerasa para detectar procariotas en muestras de agua o suelo a partir, de únicamente, sus ácidos nucleicos. Esto permite detectar e identificar organismos que no pueden ser cultivados en el laboratorio, debido a su complejidad.[7][8]
Clasificación actual

La clasificación de las arqueas, y las procariotas en general, es un campo en constante movimiento y controvertido. Los sistemas actuales de clasificación intentan organizar las arqueas en grupos de organismos que comparten rasgos estructurales y antepasados comunes.[9] Estas clasificaciones se basan significativamente en el uso de secuencias de genes de ARN ribosomal para revelar las relaciones entre los organismos (Análisis moleculares de ADN).[10] La mayoría de especies de arqueas cultivables y bien investigadas son miembros de dos filos principales, los Euryarchaeota y Crenarchaeota. Se han propuesto tentativamente otros grupos. Por ejemplo, la peculiar especie Nanoarchaeum equitans, que fue descubierta en 2003, se le ha atribuido su propio filo, el de las Nanoarchaeota.[11] También se ha propuesto el nuevo filo Korarchaeota, que contiene un número reducido de inusuales especies termófilas que comparten rasgos de los dos filos principales, pero que son más cercanas a los Crenarchaeota.[12][13] Otras especies de arqueas recientemente descubiertas sólo tienen una relación distante con cualquiera de estos grupos, como los Nanoorganismos arqueobacterianos acidófilos de Richmond Mine (ARMAN), que fueron descubiertos en 2006.[14]
La clasificación de las arqueas en especies también es controvertida. En biología, una especie es un grupo de organismos relacionados. Una definición popular de especie entre los animales es un conjunto de organismos que pueden reproducirse entre ellos y que están reproductivamente aislados de otro grupos de organismos (es decir, no pueden reproducirse con otras especies).[15] Sin embargo, los esfuerzos por clasificar las procariotas tales como las arqueas en especies se complican debido a que son asexuales y que presentan un alto nivel de transferencia horizontal de genes entre linajes. Este tema es controvertido, por ejemplo, algunos datos sugieren que en arqueas como Ferroplasma, se pueden agrupar células individuales en poblaciones de genoma muy similar y que raramente transfieren genes a grupos más divergentes de células.[16] Algunos argumentan que estos grupos de células son análogos a especies. Por otra parte, estudios de Halorubrum descubrieron un intercambio genético significativo entre estas poblaciones.[17] Estos resultados han llevado a pensar que clasificar estos grupos de organismos como especies tendría poco sentido práctico.[18]
El conocimiento actual sobre la diversidad de las arqueas es fragmentario, y no se puede estimar con ningún tipo de precisión el número total de especies de arqueas.[10] Incluso las estimaciones del número total de filos arqueobacterianos varían entre 18 y 23, de los cuales sólo ocho tienen representantes que han cultivado y estudiado directamente. Muchos de estos grupos hipotéticos son conocidos únicamente a partir de una sola secuencia de ARNr, lo que indica que la diversidad de estos organismos permanece completamente desconocida.[19] El problema de cómo estudiar y clasificar microbios no cultivados también se da en las bacterias.[20]
Origen y evolución


Las archaea son una forma de vida muy antigua. Se han datado fósiles probables de estas células hace casi 3.500 millones de años,[22] y se han detectado restos de lípidos que podrían ser arqueobacterias o eucariotas en esquistos de hace 2.700 millones de años.[23] Como la mayoría de procariotas no tienen una morfología característica, la forma de los fósiles no sirve para identificarlos como arqueas. En cambio, fósiles químicos, en forma de los lípidos únicos de las arqueobacterias, ofrecen más información sobre estos compuestos que no aparecen en otros grupos de organismos.[24] Estos lípidos han sido detectados en rocas que datan del Precámbrico. Los restos más antiguos conocidos de estos lípidos de isopreno datan del cinturón de Isua, al oeste de Groelandia, que incluyen sedimentos formados hace 3.800 millones de años que son los más antiguos del mundo.[25] El origen de las arqueas parece extremadamente remoto en el tiempo, y el linaje arqueobacteriano podría ser el más antiguo de la Tierra.[26]
Woese argumentó que las bacterias, arqueas y eucariotas representan cada uno un linaje evolutivo distinto que divergieron hace muchos millones de años de una colonia ancestral de organismos.[27][28] Sin embargo, algunos biólogos han argumentado que las arqueas y eucariotas surgieron de un grupo de bacterias.[29] Thomas Cavalier-Smith propuso el clado Neomura para representar esta teoría. Neomura significa "paredes nuevas" y se refiere a la teoría de que las arqueas y eucariotas habrían derivado de bacterias que (entre otras adaptaciones) habían sustituido las paredes de peptidoglicano por otra de glicoproteína. Es posible que el último antepasado común de las bacterias y arqueas fuera un termófilo, lo que presenta la posibilidad de que las temperaturas bajas sean "ambientes extremos" por las arqueas, y que los organismos que viven en ambientes más fríos aparecieran más tarde en la historia de la vida en la Tierra.[30] Como las arqueas y las bacterias no están relacionadas más estrechamente unas con otras que con las eucariotas, se ha argumentado que el término "procariota" no tiene un sentido evolutivo auténtico y que habría que descartarlo por completo.[31]
La relación entre archaea y eucariotas es un problema importante, hasta la actualidad. Aparte de las similitudes en la estructura y las funciones celulares que se discuten más abajo, muchos árboles genéticos agrupan los dos clados. Algunos de los primeros análisis incluso sugerían que la relación entre las eucariotas y el filo arqueobacteriano de las Euryarchaeota es más cercana que la relación entre las eucariotas y Crenarchaeota.[32] Sin embargo, actualmente se considera más verosímil que el antepasado de las eucariotas pudo divergir de las arqueobacterias.[33][34] Los descubrimiento de genes similares a las de arqueas en algunos grupos de bacterias, como Thermotogae maritima, hace que estas relaciones sean difíciles de determinar, pues ha tenido lugar una transferencia horizontal de genes.[35] Algunos han sugerido que las eucariotas surgieron de la fusión de una arquea y una bacteria, que se convirtieron en el núcleo y el citoplasma, lo que explica varias semejanzas genéticas pero presenta dificultades a la hora de explicar la estructura celular.[36]
Morfología

Las arqueas individuales tienen diámetros comprendidos entre 0,1 μm y más de 15 μm y se presentan en diversas formas, siendo comunes esferas, barras, espirales y placas.[37] El grupo Crenarchaeota incluye otras morfologías tales como células lobuladas irregularmente en Sulfolobus, finos filamentos de menos de medio μm de diámetro en Thermofilum y barras casi perfectamente rectangulares en Thermoproteus y Pyrobaculum.[38] Recientemente, se ha descubierto en piscinas hipersalinas una especie de forma cuadrada y plana (como un sello de correos) denominada Haloquadra walsbyi.[39] Estas formas inusuales probablemente se mantienen tanto por la pared celular como por un citoesqueleto procariótico, pero estas estructuras celulares, en contraste con las bacterianas, son poco conocidas.[40] En las células de las arqueas se han identificado, sin embargo, proteínas relacionadas con los componentes del citoesqueleto,[41] así como filamentos.[42]
Algunas especies forman agregados o filamentos celulares de hasta 200 μm de longitud[37] y pueden ser miembros prominentes de las comunidades de microbios que conforman biopelículas.[43] Un ejemplo particularmente elaborado de colonias multicelulares lo constituyen las arqueas del género Pyrodictium. En este caso, las células producen arreglos de tubos largos, delgados y huecos, denominados cánulas, que se conectan formando densas colonias arbustivas.[44] La función de estas cánulas se desconoce, pero pueden permitir que las células se comuniquen o intercambien nutrientes con sus vecinos.[45] También se pueden formar colonias por asociación entre especies diferentes. Por ejemplo, en una comunidad que fue descubierta en 2001 en un humedal alemán, había colonias blancas y redondas de una nueva especie de arquea del filo euryarchaeota esparcidas a lo largo de filamentos delgados que pueden medir hasta 15 cm de largo; estos filamentos se componen de una especie particular de bacterias.[46]
Estructura celular
Las archaea son similares a las bacterias en su estructura celular general, pero la composición y organización de algunas de estas estructuras diferencian de las arqueas. Como las bacterias, las archaea carecen de membranas interiores, de modo que sus células no contienen orgánulos.[31] También se parecen a las bacterias en que su membrana celular está habitualmente delimitada por una pared celular y en el que nadan por medio de uno o más flagelos.[47] En su estructura general, las arqueas parecen especialmente a las bacterias Gram positivas, pues la mayoría tienen una única membrana plasmática y pared celular, y carecen de espacio periplasmático, la excepción de esta regla general es la arquea Ignicoccus, que tiene un espacio periplasmático particularmente grande que contiene vesículas limitadas por membranas, y que queda cerrado por una membrana exterior.[48]
Membrana celular
Las membranas arqueobacterianas se componen de moléculas que difieren mucho de las que se encuentran en otras formas de vida, lo que es una prueba de que las arqueas sólo tienen una relación distante con las bacterias y eucariotas.[49] En todos los organismos, las membranas celulares se componen de moléculas conocidas como fosfolípidos. Estas moléculas tienen una parte polar que se disuelve en el agua (el "jefe" de fosfatos), y una parte "grasa" no polar que no se disuelve en el agua (la "cola" de lípidos). Estas partes diferentes quedan conectadas por un grupo glicerol. En el agua, los fosfolípidos se aglomeran, con los jefes de fosfatos polares hacia el agua y las colas lipídicas no polares lejos de ella. Esto hace que se estructuren en capas. La estructura principal de la membrana celular es una doble capa de estos fosfolípidos, que recibe el nombre de bicapa lipídica.
Los fosfolípidos de las membranas arqueobacterianas son inusuales en cuatro cosas. Primeramente, las bacterias tienen membranas compuestas principalmente de lípidos éster con glicerol, mientras que las arqueas las tienen compuestas de lípidos éter con glicerol.[50] La diferencia entre estos dos tipos de fosfolípidos es el tipo de enlace que une los lípidos en el grupo glicerol. En los lípidos éster hay un enlace éster, mientras que en el resto de lípidos hay un enlace éter. Los enlaces éter tienen una resistencia química superior a la de los enlaces éster, lo que podría contribuir a la capacidad de algunas archaeas de sobrevivir a temperaturas extremas o en ambientes muy ácidos o alcalinos.[51] Las bacterias y eucariotas también contienen algunos lípidos éter, pero a diferencia de las arqueobacterias, estos lípidos no forman una parte importante de sus membranas.
En segundo lugar, los lípidos arquobacterianos son únicos porque la estereoquímica del grupo glicerol es la inversa de la que se observa en otros organismos. El grupo glicerol puede existir en dos formas que son la imagen inversa la una de la otra, y que se pueden denominar formas "derechista" e "izquierdista", en lenguaje químico se les denomina enantiómeros. Del mismo modo que una mano derecha no entra fácilmente en un guante para la mano izquierda, una molécula de glicerol derechista generalmente no puede ser utilizada o creada por enzimas adaptados por la forma izquierdista. Esto sugiere que las arqueas utilizan enzimas completamente diferentes para sintetizar sus fosfolípidos de los que utilizan las bacterias y eucariotas; como estas enzimas se desarrollaron muy al principio de la historia de la vida, esto sugiere a su vez que las arqueas se separaron muy pronto de los otros dos dominios.[49]
En tercer lugar, las colas lipídicas de los fosfolípidos de las arqueobacterias tienen una composición química diferente a las de otros organismos. Los lípidos arquobacterianos se basan en la cadena lateral isoprenoide y son largas cadenas con múltiples ramas laterales y, a veces, incluso anillos de ciclopropano o ciclohexano.[52] Esto contrasta con los ácidos grasos que hay en las membranas de otros organismos, que tienen cadenas rectas sin ramas ni anillos. Aunque los isoprenoides desempeñan un papel importante en la bioquímica de muchos organismos, sólo las arqueas los utilizan para producir fosfolípidos. Estas cadenas con ramas podrían ayudar a evitar que las membranas arqueobacterianas tengan fugas a altas temperaturas.[53]
Finalmente, en algunas arqueas la bicapa lipídica es sustituida por una única monocapa. De hecho, las arqueas fusionan las colas de dos moléculas fosfolipídicas independientes en una única molécula con dos cabezas polares, esta fusión podría hacer su membrana más rígida y más apta para resistir ambientes severos.[54] Por ejemplo, todos los lípidos de Ferroplasma son de este tipo, lo que se cree que ayuda a este organismo a sobrevivir en los medios extraordinariamente ácidos en que habita.
Pared celular y flagelos
La mayoría de arqueas tienen una pared celular, las excepciones son Thermoplasma y Ferroplasma.[55] En la mayoría de arqueas, la pared se compone de proteínas de capa de superficie, que forman una capa S.[56] Una capa S es una agrupación rígida de moléculas proteínicas que cubren el exterior de la célula como una cota de malla[57] Esta capa ofrece una producción química y física, y puede servir de barrera, impidiendo que entren en contacto macromoléculas con la membrana celular.[58] A diferencia de las bacterias, la mayoría de arqueas carecen de peptidoglicano en la pared celular.[59] La excepción es el pseudopeptidoglicano, que se encuentra en las archaeas metanógenas, pero estepolímero es diferente del peptidoglicano bacteriano ya que carece de aminoácidos y ácido N-acetilmurámico.[58]
Las arqueas también tienen flagelos, que funcionan de una manera parecida a los flagelos bacterianos —son largas colas que se mueven por motores rotatorios situados en la base de los flagelos. Estos motores son impulsados por el gradiente de protones de la membrana. Sin embargo, los flagelos arquobacterianos son notablemente diferentes en su composición y su desarrollo.[47] Cada tipo de flagelo evolucionó de un antepasado diferente, el flagelo bacteriano evolucionó de un sistema de secreción de tipo III, mientras que los flagelos arquobacterianos parecen haber evolucionado de los pili bacterianos de tipo IV.[60] A diferencia del flagelo bacteriano, que es un palet vacío y que está formado por subunidades que se mueven por la cavidad central y luego se añaden a la punta del flagelo, los flagelos arquobacterianos se sintetizan mediante la adición de subunidades en la su base.[61]
Genética

Las arqueas por lo general tienen un único cromosoma circular al igual que las bacterias.[63] Estos varían en tamaño desde 5.751.492 pares de bases en Methanosarcina acetivorans,[64] el mayor genoma secuenciado hasta la fecha, hasta 490.885 pares de bases en Nanoarchaeum equitans, el genoma más pequeño conocido que puede contener sólo 537 genes codificadores de proteínas. Las arqueas también presentan plásmidos[65] que se pueden propagar por contacto físico entre células, en un proceso que puede ser similar a la conjugación bacteriana.[66][67] Los plásmidos son cada vez más importantes como herramientas genéticas pues permiten la realización de estudios genéticos en Archaea.[68]
Al igual que los bacteriófagos que infectan bacterias, existen virus que se replican en las arqueas. Son virus ADN bicatenarios que parecen no estar relacionados con otros virus y que pueden tener una gran variedad de formas inusuales, como botellas o ganchos.[69] Estos virus han sido estudiados en más detalle en los termófilos, en particular en los órdenes Sulfolobales y Thermoproteales.[70] La defensa contra estos virus pueden implicar la interferencia de ARN por secuencias de ADN repetitivas en el genoma de las arqueas.[71][72]
Las arqueas son genéticamente distintas a otros organismos, con hasta un 15% de proteínas exclusivas codificadas por el genoma de cualquiera arquea.[73] Los genes que son compartidos entre Archaea, Bacteria y Eukarya forman un núcleo de funciones de la célula, relacionados principalmente con la transcripción, traducción y metabolismo de nucleótidos.[74] La mayoría de los genes exclusivos de las arqueas no tienen una función conocida, pero de los que tienen una función identificada, la mayoría participan en la metanogénesis. Otros elementos característicos de los genomas de las arqueas son la organización de genes de función relacionada, tales como las enzimas que catalizan las etapas de la misma ruta metabólica, nuevos operones y grandes diferencias en genes ARNt y sus aminoacil ARNt sintetasas.[74]
La transcripción y traducción en Archaea son más similares a Eukarya que a Bacteria, por ejemplo en las subunidades y secuencias de la ARN polimerasa II y en los ribosomas.[63] Las funciones e interacciones de la ARN polimerasa en la transcripción en Archaea también parece estar relacionada con la de Eukarya, con un similar ensamblado de proteínas (factores de transcripción genéricos) dirigiendo la unión de la ARN polimerasa a un promotor de gen. Sin embargo, muchos otros factores de transcripción en las arqueas son similares a los de bacterias.[75]
Reproducción
Las arqueas se reproducen asexualmente por fisión binaria o múltiple, fragmentación o gemación. No se produce meiosis, de manera que si una especie de arquea existe en más de una forma, todas tienen el mismo número de cromosomas (tienen el mismo cariotipo).[37] La división celular está controlada como parte de un complejo ciclo celular, donde el cromosoma se replica, las copias se separan y luego la célula se divide.[76] Los detalles del ciclo celular sólo han sido investigados en el género Sulfolobus, siendo similares a los de bacterias y eucariontes: los cromosomas se replican desde múltiples puntos de partida (origen de replicación) usando ADN polimerasas que son similares a las enzimas equivalentes eucarióticas.[77] Sin embargo, las proteínas que dirigen la división celular, como la proteína FtsZ que forma un anillo contráctil alrededor de la célula, parecen estar más relacionadas con sus equivalentes bacterianos.[76]
No se forman endosporas en ninguna especie de arquea,[78] aunque algunas especies de Halobacteria pueden alternar entre fenotipos y crecer como diferentes tipos de células, incluidas estructuras de paredes gruesas que son resistentes al choque osmótico y que les permiten sobrevivir a bajas concentraciones de sal.[79] No se trata de estructuras reproductivas, pero es posible que ayuden a estas especies a dispersarse en nuevos hábitats.
Clasificación
La sistemática de las arqueas no está completa y las diferencias entre algunos grupos son muy grandes.
Se conocen unos 100 géneros y unas 300 especies de arqueas. Se proponen cuatro filos (cada uno de los cuales se puede incluir en un reino del mismo nombre), comprendiendo los dos primeros la mayor parte de las especies conocidas:
- Crenarchaeota. Comprende a la mayoría de las especies hipertermófilas. Muchas son autótrofas quimiosintéticas, constituyendo los únicos productores primarios en ciertos hábitats inhóspitos. Recientes análisis basados en secuencias de ARNr indican que Crenarchaeota también se puede distribuir extensamente en ambientes de baja temperatura tales como mares, sedimentos y suelos.
- Euryarchaeota. Comprende a las que se encuentran en concentraciones salinas altas y que pueden obtener su energía de la luz, sin ningún pigmento clorofílico. También se clasifican dentro de este clado los metanógenos que habitan los sedimentos acuosos y en el tracto digestivo de diversos animales. Análisis de secuencias de ARNr sugieren que miembros de este grupo también se encuentran en gran número en las aguas oceánicas.
- Korarchaeota. Es un pequeño grupo de hipertermófilas (encontradas en el parque Yellowstone), filogenéticamente diferentes a las Crenarchaeotas y que se consideran las arqueas más primitivas.
- Nanoarchaeota. Es un taxón especialmente creado para la especie Nanoarchaeum equitans, descubierta en 2002 y cuyas relaciones de parentesco son inciertas.[80]
Se debe tomar en cuenta que, aunque los dos primeros filos están firmemente establecidos, los dos restantes comprenden pocas especies mal conocidas y son dudosos. Dado el escaso conocimiento actual sobre las especies de este dominio, el número de filos puede aumentar con rapidez. Muchos de estos hipotéticos grupos son conocidos por una única secuencia de ARNr, lo que indica que la gran mayoría de la diversidad de estos organismos sigue siendo completamente desconocida.[81][82] Este problema de como abordar el estudio y clasificación de microbios no cultivados es común en todos los procariontes.[83] Recientemente (en 2006) se han descubierto tres nuevos grupos de Euryarchaeota en el ambiente extremadamente ácido en una mina del Norte de California, denominados provisionalmente ARMAN (Archaeal Richmond Mine Acidophilic Nanoorganisms).[84]
Uso de Archaea en tecnología e industria
Las arqueas extremófilas, en particular las resistentes a las altas temperaturas o a los extremos de acidez y alcalinidad, son una importante fuente de enzimas que puede funcionar bajo estas duras condiciones.[85][86] Estas enzimas tienen una amplia gama de usos. Por ejemplo, las ADN polimerasas termoestables, como la ADN polimerasa Pfu de Pyrococcus furiosus, han revolucionado la biología molecular, al permitir el uso de la reacción en cadena de la polimerasa como método simple y rápido para la clonación del ADN.
En la industria, las amilasas, galactosidasas y pululanasas de otras especies de Pyrococcus realizan su función a más de 100 °C, lo que permite la elaboración de alimentos a altas temperaturas, tales como leche baja en lactosa y suero de leche.[87] Las enzimas de estas arqueas termófilas también tienden a ser muy estables en solventes orgánicos, por lo que pueden utilizarse en una amplia gama de procesos respetuosos con el medio ambiente para la síntesis de compuestos orgánicos.[86]
En contraste con la amplia gama de aplicaciones de las enzimas, la utilización en biotecnología de los organismos en sí mismos es más reducida. Sin embargo, las arqueas metanógeneas son una parte vital del tratamiento de aguas residuales, realizando la digestión anaeróbica de los residuos y produciendo biogás.[88] Las arqueas acidófilas son también prometedores en minería para la extracción de metales tales como oro, cobalto y cobre.[89]
Una nueva clase de antibióticos potencialmente útiles se derivan de este grupo de organismos. Ocho de esas sustancias ya han sido caracterizadas, pero podría haber muchas más, especialmente en Halobacteria. El descubrimiento de nuevas sustancias depende de la recuperación de estos organismos del medio ambiente y de su cultivo.[90]
Referencias
- ↑ Staley JT (2006). «The bacterial species dilemma and the genomic-phylogenetic species concept». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1475): 1899-909. PMID 17062409. doi:10.1098/rstb.2006.1914.
- ↑ Zuckerkandl E, Pauling L (1965). «Molecules as documents of evolutionary history». J. Theor. Biol. 8 (2): 357-66. PMID 5876245. doi:10.1016/0022-5193(65)90083-4.
- ↑ Woese C, Fox G (1977). «Phylogenetic structure of the prokaryotic domain: the primary kingdoms». Proc Natl Acad Sci USA 74 (11): 5088-90. PMID 270744. doi:10.1073/pnas.74.11.5088.
- ↑ Woese CR, Kandler O, Wheelis ML (1990). «Towards a natural system of organisms: proposal for the domains Archaea, Bacteria, and Eucarya». Proc. Natl. Acad. Sci. U.S.A. 87 (12): 4576-9. PMID 2112744. doi:10.1073/pnas.87.12.4576.
- ↑ archaea. (2008). Al Merriam-Webster Online Dictionary. Consultado el 1 de julio del 2008, de [1]
- ↑ DeLong EF (1998). «Everything in moderation: archaea as 'non-extremophiles'». Curr. Opin. Genet. Dev. 8 (6): 649-54. PMID 9914204. doi:10.1016/S0959-437X(98)80032-4.
- ↑ Theron J, Cloete TE (2000). «Molecular techniques for determining microbial diversity and community structure in natural environments». Crit. Rev. Microbiol. 26 (1): 37-57. PMID 10782339. doi:10.1080/10408410091154174.
- ↑ Schmidt TM (2006). «The maturing of microbial ecology». Int. Microbiol. 9 (3): 217-23. PMID 17061212.
- ↑ Gevers D, Dawyndt P, Vandamme P, et al. (2006). «Stepping stones towards a new prokaryotic taxonomy». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1475): 1911-6. PMID 17062410. doi:10.1098/rstb.2006.1915.
- ↑ a b Robertson CE, Harris JK, Spear JR, Pace NR (2005). «Phylogenetic diversity and ecology of environmental Archaea». Curr. Opin. Microbiol. 8 (6): 638-42. PMID 16236543.
- ↑ Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO. (2002). «A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont». Nature 417 (6884): 27-8. PMID 11986665. doi:10.1038/417063a.
- ↑ Barns SM, Delwiche CF, Palmer JD, Pace NR (1996). «Perspectives on archaeal diversity, thermophily and monophyly from environmental rRNA sequences». Proc. Natl. Acad. Sci. U.S.A. 93 (17): 9188-93. PMID 8799176. doi:10.1073/pnas.93.17.9188.
- ↑ Elkins JG, Podar M, Graham DE, et al. (Juny de 2008). «A korarchaeal genome reveals insights into the evolution of the Archaea». Proc. Natl. Acad. Sci. U.S.A. 105 (23): 8102-7. PMID 18535141. doi:10.1073/pnas.0801980105.
- ↑ Baker, B.J., Tyson, G.W., Webb, R.I., Flanagan, J., Hugenholtz, P. and Banfield, J.F. (2006). «Lineages of acidophilic Archaea revealed by community genomic analysis. Science». Science 314 (6884): 1933 - 1935. PMID 17185602. doi:10.1126/science.1132690.
- ↑ de Queiroz K (2005). «Ernst Mayr and the modern concept of species». Proc. Natl. Acad. Sci. U.S.A. 102 Suppl 1: 6600-7. PMID 15851674. doi:10.1073/pnas.0502030102.
- ↑ Eppley JM, Tyson GW, Getz WM, Banfield JF (2007). «Genetic exchange across a species boundary in the archaeal genus ferroplasma». Genetics 177 (1): 407-16. PMID 17603112. doi:10.1534/genetics.107.072892.
- ↑ Papke RT, Zhaxybayeva O, Feil EJ, Sommerfeld K, Muise D, Doolittle WF (2007). «Searching for species in haloarchaea». Proc. Natl. Acad. Sci. U.S.A. 104 (35): 14092-7. PMID 17715057. doi:10.1073/pnas.0706358104.
- ↑ Kunin V, Goldovsky L, Darzentas N, Ouzounis CA (2005). «The net of life: reconstructing the microbial phylogenetic network». Genome Res. 15 (7): 954-9. PMID 15965028. doi:10.1101/gr.3666505.
- ↑ Hugenholtz P (2002). «Exploring prokaryotic diversity in the genomic era». Genome Biol. 3 (2): REVIEWS0003. PMID 11864374. doi:10.1186/gb-2002-3-2-reviews0003.
- ↑ Rappé MS, Giovannoni SJ (2003). «The uncultured microbial majority». Annu. Rev. Microbiol. 57: 369-94. PMID 14527284. doi:10.1146/annurev.micro.57.030502.090759.
- ↑ Ciccarelli FD, Doerks T, von Mering C, Creevey CJ, Snel B, Bork P (2006). «Toward automatic reconstruction of a highly resolved tree of life». Science 311 (5765): 1283-7. PMID 165139821
|pmid=incorrecto (ayuda). - ↑ Schopf J (2006). «Fossil evidence of Archaean life». Philos Trans R Soc Lond B Biol Sci 361 (1470): 869-85. PMID 16754604. doi:10.1098/rstb.2006.1834.
- ↑ Brocks JJ, Logan GA, Buick R, Summons RE (1999). «Archean molecular fossils and the early rise of eukaryotes». Science 285 (5430): 1033-6. PMID 10446042. doi:10.1126/science.285.5430.1033.
- ↑ Chappe B, Albrecht P, Michaelis W (Juliol de 1982). «Polar Lipids of Archaebacteria in Sediments and Petroleums». Science 217 (4554): 65-66. PMID 17739984. doi:10.1126/science.217.4554.65.
- ↑ Hahn, Jürgen; Pat Haug (1986). «Traces of Archaebacteria in ancient sediments». System Applied Microbiology 7 (Archaebacteria '85 Proceedings): 178-83.
- ↑ Wang M, Yafremava LS, Caetano-Anollés D, Mittenthal JE, Caetano-Anollés G (2007). «Reductive evolution of architectural repertoires in proteomes and the birth of the tripartite world». Genome Res. 17 (11): 1572-85. PMID 17908824. doi:10.1101/gr.6454307.
- ↑ Woese CR, Gupta R (1981). «Are archaebacteria merely derived 'prokaryotes'?». Nature 289 (5793): 95-6. PMID 6161309. doi:10.1038/289095a0.
- ↑ Woese C (1998). «The universal ancestor». Proc. Natl. Acad. Sci. U.S.A. 95 (12): 6854-9. PMID 9618502. doi:10.1073/pnas.95.12.6854.
- ↑ Gupta RS (2000). «The natural evolutionary relationships among prokaryotes». Crit. Rev. Microbiol. 26 (2): 111-31. PMID 10890353. doi:10.1080/10408410091154219.
- ↑ Gribaldo S, Brochier-Armanet C (2006). «The origin and evolution of Archaea: a state of the art». Philos. Trans. R. Soc. Lond., B, Biol. Sci. 361 (1470): 1007-22. PMID 16754611. doi:10.1098/rstb.2006.1841.
- ↑ a b Woese CR (Març de 1994). «There must be a prokaryote somewhere: microbiology's search for itself». Microbiol. Rev. 58 (1): 1-9. PMC 372949. PMID 8177167.
- ↑ Lake JA (Gener de 1988). «Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences». Nature 331 (6152): 184-6. PMID 3340165. doi:10.1038/331184a0.
- ↑ Gouy M, Li WH (Maig de 1989). «Phylogenetic analysis based on rRNA sequences supports the archaebacterial rather than the eocyte tree». Nature 339 (6220): 145-7. PMID 2497353. doi:10.1038/339145a0.
- ↑ Yutin N, Makarova KS, Mekhedov SL, Wolf YI, Koonin EV (Maig de 2008). «The deep archaeal roots of eukaryotes». Mol. Biol. Evol. 25: 1619. PMID 18463089. doi:10.1093/molbev/msn108.
- ↑ Nelson KE, Clayton RA, Gill SR, et al. (1999). «Evidence for lateral gene transfer between Archaea and bacteria from genome sequence of Thermotoga maritima». Nature 399 (6734): 323-9. PMID 10360571. doi:10.1038/20601.
- ↑ Lake JA. (1988). «Origin of the eukaryotic nucleus determined by rate-invariant analysis of rRNA sequences». Nature 331 (6152): 184-6. PMID 3340165. doi:10.1038/331184a0.
- ↑ a b c Krieg, Noel (2005). Bergey’s Manual® of Systematic Bacteriology. USA: Springer. pp. 21-26. ISBN 978-0-387-24143-2.
- ↑ Barns, Sue and Burggraf, Siegfried. (1997) Crenarchaeota. Version 01 January 1997. in The Tree of Life Web Project
- ↑ Walsby AE (2005). «Archaea with square cells». Trends Microbiol. 13 (5): 193-5. PMID 15866034.
- ↑ Hixon WG, Searcy DG (1993). «Cytoskeleton in the archaebacterium Thermoplasma acidophilum? Viscosity increase in soluble extracts». BioSystems 29 (2-3): 151-60. PMID 8374067.
- ↑ Hara F, Yamashiro K, Nemoto N, et al (2007). «An actin homolog of the archaeon Thermoplasma acidophilum that retains the ancient characteristics of eukaryotic actin». J. Bacteriol. 189 (5): 2039-45. PMID 17189356. doi:10.1128/JB.01454-06.
- ↑ Trent JD, Kagawa HK, Yaoi T, Olle E, Zaluzec NJ (1997). «Chaperonin filaments: the archaeal cytoskeleton?». Proc. Natl. Acad. Sci. U.S.A. 94 (10): 5383-8. PMID 9144246.
- ↑ Hall-Stoodley L, Costerton JW, Stoodley P (2004). «Bacterial biofilms: from the natural environment to infectious diseases». Nat. Rev. Microbiol. 2 (2): 95-108. PMID 15040259.
- ↑ Nickell S, Hegerl R, Baumeister W, Rachel R (2003). «Pyrodictium cannulae enter the periplasmic space but do not enter the cytoplasm, as revealed by cryo-electron tomography». J. Struct. Biol. 141 (1): 34-42. PMID 12576018.
- ↑ Horn C, Paulmann B, Kerlen G, Junker N, Huber H (1999). «In vivo observation of cell division of anaerobic hyperthermophiles by using a high-intensity dark-field microscope». J. Bacteriol. 181 (16): 5114-8. PMID 10438790. doi:10.1073/pnas.241636498v1.
- ↑ Rudolph C, Wanner G, Huber R (Maig de 2001). «Natural communities of novel archaea and bacteria growing in cold sulfurous springs with a string-of-pearls-like morphology». Appl. Environ. Microbiol. 67 (5): 2336-44. PMC 92875. PMID 11319120. doi:10.1128/AEM.67.5.2336-2344.2001.
- ↑ a b Thomas NA, Bardy SL, Jarrell KF (2001). «The archaeal flagellum: a different kind of prokaryotic motility structure». FEMS Microbiol. Rev. 25 (2): 147-74. PMID 11250034. doi:10.1111/j.1574-6976.2001.tb00575.x.
- ↑ Rachel R, Wyschkony I, Riehl S, Huber H (Març de 2002). «The ultrastructure of Ignicoccus: evidence for a novel outer membrane and for intracellular vesicle budding in an archaeon». Archaea 1 (1): 9-18. PMID 15803654.
- ↑ a b Koga Y, Morii H (2007). «Biosynthesis of ether-type polar lipids in archaea and evolutionary considerations». Microbiol. Mol. Biol. Rev. 71 (1): 97-120. PMID 17347520. doi:10.1128/MMBR.00033-06.
- ↑ De Rosa M, Gambacorta A, Gliozzi A (1986). «Structure, biosynthesis, and physicochemical properties of archaebacterial lipids». Microbiol. Rev. 50 (1): 70-80. PMID 3083222.
- ↑ Albers SV, van de Vossenberg JL, Driessen AJ, Konings WN (Setembre de 2000). «Adaptations of the archaeal cell membrane to heat stress». Front. Biosci. 5: D813-20. PMID 10966867. doi:10.2741/albers.
- ↑ Damsté JS, Schouten S, Hopmans EC, van Duin AC, Geenevasen JA (Octubre de 2002). «Crenarchaeol: the characteristic core glycerol dibiphytanyl glycerol tetraether membrane lipid of cosmopolitan pelagic crenarchaeota». J. Lipid Res. 43 (10): 1641-51. PMID 12364548. doi:10.1194/jlr.M200148-JLR200.
- ↑ Koga Y, Morii H (Novembre de 2005). «Recent advances in structural research on ether lipids from archaea including comparative and physiological aspects». Biosci. Biotechnol. Biochem. 69 (11): 2019-34. PMID 16306681. doi:10.1271/bbb.69.2019.
- ↑ Hanford MJ, Peeples TL (Gener de 2002). «Archaeal tetraether lipids: unique structures and applications». Appl. Biochem. Biotechnol. 97 (1): 45-62. PMID 11900115. doi:10.1385/ABAB:97:1:45.
- ↑ Golyshina OV, Pivovarova TA, Karavaiko GI, et al (Maig de 2000). «Ferroplasma acidiphilum gen. nov., sp. nov., an acidophilic, autotrophic, ferrous-iron-oxidizing, cell-wall-lacking, mesophilic member of the Ferroplasmaceae fam. nov., comprising a distinct lineage of the Archaea». Int. J. Syst. Evol. Microbiol. 50 Pt 3: 997-1006. PMID 10843038.
- ↑ Sára M, Sleytr UB (2000). «S-Layer proteins». J. Bacteriol. 182 (4): 859-68. PMID 10648507. doi:10.1128/JB.182.4.859-868.2000.
- ↑ Engelhardt H, Peters J (1998). «Structural research on surface layers: a focus on stability, surface layer homology domains, and surface layer-cell wall interactions». J Struct Biol 124 (2–3): 276-302. PMID 10049812. doi:10.1006/jsbi.1998.4070.
- ↑ a b Kandler, O.; König, H. (1998). «Cell wall polymers in Archaea (Archaebacteria)». Cellular and Molecular Life Sciences (CMLS) 54 (4): 305-308. doi:10.1007/s000180050156.
- ↑ Plantilla:Ref-llibre
- ↑ Ng SY, Chaban B, Jarrell KF (2006). «Archaeal flagella, bacterial flagella and type IV pili: a comparison of genes and posttranslational modifications». J. Mol. Microbiol. Biotechnol. 11 (3–5): 167-91. PMID 16983194. doi:10.1159/000094053.
- ↑ Bardy SL, Ng SY, Jarrell KF (Febrer de 2003). «Prokaryotic motility structures». Microbiology (Reading, Engl.) 149 (Pt 2): 295-304. PMID 12624192. doi:10.1099/mic.0.25948-0.
- ↑ Xiang X, Chen L, Huang X, Luo Y, She Q, Huang L (2005). «Sulfolobus tengchongensis spindle-shaped virus STSV1: virus-host interactions and genomic features». J. Virol. 79 (14): 8677-86. PMID 15994761. doi:10.1128/JVI.79.14.8677-8686.2005.
- ↑ a b Allers T, Mevarech M (2005). «Archaeal genetics - the third way». Nat. Rev. Genet. 6 (1): 58-73. PMID 15630422.
- ↑ Baliga NS, Bonneau R, Facciotti MT, et al (2004). «Genome sequence of Haloarcula marismortui: a halophilic archaeon from the Dead Sea». Genome Res. 14 (11): 2221-34. PMID 15520287. doi:10.1101/gr.2700304.
- ↑ Waters E, Hohn MJ, Ahel I, et al (2003). «The genome of Nanoarchaeum equitans: insights into early archaeal evolution and derived parasitism». Proc. Natl. Acad. Sci. U.S.A. 100 (22): 12984-8. PMID 14566062. doi:10.1073/pnas.1735403100.
- ↑ Schleper C, Holz I, Janekovic D, Murphy J, Zillig W (1995). «A multicopy plasmid of the extremely thermophilic archaeon Sulfolobus effects its transfer to recipients by mating». J. Bacteriol. 177 (15): 4417-26. PMID 7635827.
- ↑ Sota M; Top EM (2008). «Horizontal Gene Transfer Mediated by Plasmids». Plasmids: Current Research and Future Trends. Caister Academic Press. ISBN 978-1-904455-35-6.
- ↑ Lipps G (2008). «Archaeal Plasmids». Plasmids: Current Research and Future Trends. Caister Academic Press. ISBN 978-1-904455-35-6.
- ↑ Prangishvili D, Forterre P, Garrett RA (2006). «Viruses of the Archaea: a unifying view». Nat. Rev. Microbiol. 4 (11): 837-48. PMID 17041631.
- ↑ Prangishvili D, Garrett RA (2004). «Exceptionally diverse morphotypes and genomes of crenarchaeal hyperthermophilic viruses». Biochem. Soc. Trans. 32 (Pt 2): 204-8. PMID 15046572.
- ↑ Mojica FJ, Díez-Villaseñor C, García-Martínez J, Soria E (2005). «Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements». J. Mol. Evol. 60 (2): 174-82. PMID 15791728.
- ↑ Makarova KS, Grishin NV, Shabalina SA, Wolf YI, Koonin EV (2006). «A putative RNA-interference-based immune system in prokaryotes: computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action». Biol. Direct 1: 7. PMID 16545108.
- ↑ Graham DE, Overbeek R, Olsen GJ, Woese CR (2000). «An archaeal genomic signature». Proc. Natl. Acad. Sci. U.S.A. 97 (7): 3304-8. PMID 10716711. doi:10.1073/pnas.050564797.
- ↑ a b Gaasterland T (1999). «Archaeal genomics». Curr. Opin. Microbiol. 2 (5): 542-7. PMID 10508726.
- ↑ Aravind L, Koonin EV (1999). «DNA-binding proteins and evolution of transcription regulation in the archaea». Nucleic Acids Res. 27 (23): 4658-70. PMID 10556324.
- ↑ a b Bernander R (1998). «Archaea and the cell cycle». Mol. Microbiol. 29 (4): 955-61. PMID 9767564.
- ↑ Kelman LM, Kelman Z (2004). «Multiple origins of replication in archaea». Trends Microbiol. 12 (9): 399-401. PMID 153371581
|pmid=incorrecto (ayuda). - ↑ Onyenwoke RU, Brill JA, Farahi K, Wiegel J (2004). «Sporulation genes in members of the low G+C Gram-type-positive phylogenetic branch ( Firmicutes)». Arch. Microbiol. 182 (2-3): 182-92. PMID 15340788.
- ↑ Kostrikina NA, Zvyagintseva IS, Duda VI. (1991). «Cytological peculiarities of some extremely halophilic soil archaeobacteria». Arch. Microbiol. 156 (5): 344-49. doi:10.1007/BF00248708.
- ↑ Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO. (2002). «A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont.». Nature 417 (6884): 27 - 8. PMID 11986665.
- ↑ Robertson CE, Harris JK, Spear JR, Pace NR (2005). «Phylogenetic diversity and ecology of environmental Archaea». Curr. Opin. Microbiol. 8 (6): 638-42. PMID 16236543.
- ↑ Hugenholtz P (2002). «Exploring prokaryotic diversity in the genomic era». Genome Biol. 3 (2): REVIEWS0003. PMID 11864374. doi:10.1186/gb-2002-3-2-reviews0003.
- ↑ Rappé MS, Giovannoni SJ (2003). «The uncultured microbial majority». Annu. Rev. Microbiol. 57: 369-94. PMID 14527284.
- ↑ Baker, B.J., Tyson, G.W., Webb, R.I., Flanagan, J., Hugenholtz, P. and Banfield, J.F. (2006). «Lineages of acidophilic Archaea revealed by community genomic analysis. Science». Science 314 (6884): 1933 - 1935. Texto « DOI: 10.1126/science.1132690» ignorado (ayuda)
- ↑ Breithaupt H (2001). «The hunt for living gold. The search for organisms in extreme environments yields useful enzymes for industry». EMBO Rep. 2 (11): 968-71. PMID 11713183. doi:10.1093/embo-reports/kve238.
- ↑ a b Egorova K, Antranikian G (2005). «Industrial relevance of thermophilic Archaea». Curr. Opin. Microbiol. 8 (6): 649-55. PMID 16257257.
- ↑ Synowiecki J, Grzybowska B, Zdziebło A (2006). «Sources, properties and suitability of new thermostable enzymes in food processing». Crit Rev Food Sci Nutr 46 (3): 197-205. PMID 16527752.
- ↑ Schiraldi C, Giuliano M, De Rosa M (2002). «Perspectives on biotechnological applications of archaea». Archaea 1 (2): 75-86. PMID 15803645.
- ↑ Norris PR, Burton NP, Foulis NA (2000). «Acidophiles in bioreactor mineral processing». Extremophiles 4 (2): 71-6. PMID 10805560.
- ↑ Shand RF; Leyva KJ (2008). «Archaeal Antimicrobials: An Undiscovered Country». Archaea: New Models for Prokaryotic Biology. Caister Academic Press. ISBN 978-1-904455-27-1.
