Genética mitocondrial humana
| Genoma mitocondrial humano | ||
|---|---|---|
 El genoma mitocondrial humano de 16.569 pb de largo con los genes codificantes de proteínas (rojo, naranja, amarillo), ARN ribosómico (azul) y genes de transferencia de ARN (blanco). Región de control del ADNmt no codificando en gris. | ||
| Características | ||
| Longitud (bp) | 16,569 | |
| Número de genes |
13 (genes codificantes) 24 (genes no codificantes) | |
| Tipo | ADN mitocondrial | |
| Listas de genes | ||
| HGNC | Lista de genes | |
| NCBI | Lista de genes | |
| Visualizadores externos | ||
| Ensembl | Cromosoma MT | |
| Entrez | Cromosoma MT | |
| NCBI | Cromosoma MT | |
| UCSC | Cromosoma M | |
| Secuencias enteras de ADN | ||
| RefSeq | NC_012920 (FASTA) | |
| GenBank | J01415 (FASTA) | |

La genética mitocondrial humana es el estudio del ADN contenido en las mitocondrias humanas. El genoma mitocondrial humano es la totalidad de la información hereditaria contenida en las mitocondrias humanas. Las mitocondrias son pequeñas estructuras dentro de las células que generan energía para que la célula la use y, por lo tanto, se las conoce como las "centrales eléctricas" de la célula.[3]
En los humanos, como en la mayoría de los organismos multicelulares, el ADN mitocondrial —ADNmt— se hereda únicamente de la madre, y se transmite independientemente del ADN nuclear —ADNn—. Sin embargo, existen hipótesis que afirman que la transmisión del mtDNA paterno en humanos puede ocurrir bajo ciertas circunstancias.[4]
Por lo tanto, la herencia mitocondrial no es mendeliana, ya que esta supone que la mitad del material genético del cigoto deriva de cada progenitor.
El ochenta por ciento del ADN mitocondrial codifica ARN mitocondrial y, por lo tanto, la mayoría de las mutaciones conducen a problemas funcionales.
Debido a que proporcionan 30 moléculas de ATP por molécula de glucosa en contraste con las 2 moléculas de ATP producidas por la glucólisis, las mitocondrias son esenciales para todos los organismos superiores para mantener la vida. Las enfermedades mitocondriales son trastornos genéticos transportados en el ADN mitocondrial, o ADN nuclear que codifica componentes mitocondriales. Ligeros problemas con cualquiera de las numerosas enzimas utilizadas por la mitocondria pueden ser devastadores para la célula y, a su vez, para el organismo.
Cantidad[editar]
En los seres humanos, el ADN mitocondrial (ADNmt) forma moléculas circulares cerradas que contienen 16 569[5][6] pares de bases de ADN,[7] y cada una de estas moléculas normalmente contiene un conjunto completo de genes mitocondriales. Cada mitocondria humana contiene, en promedio, aproximadamente 5 de estas moléculas de mtDNA, con una cantidad que oscila entre 1 y 15.[7] Cada célula humana contiene aproximadamente 100 mitocondrias, lo que da un número total de moléculas de mtDNA por célula humana de aproximadamente 500.[7] La cantidad de mitocondrias por célula también varía según el tipo de célula, siendo algunos ejemplos:
- Eritrocitos : 0 mitocondrias por célula.[8]
- Linfocitos : 3 mitocondrias por célula.[9]
- Óvulo : los óvulos maduros en metafase II pueden contener 100 000 mitocondrias y entre 50 000 y 1 500 000 copias del genoma mitocondrial (que corresponde a hasta el 90 % del ADN del óvulo).[10]
Patrones de herencia[editar]
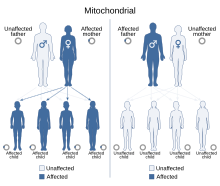

Debido a que las enfermedades mitocondriales (enfermedades debidas al mal funcionamiento de las mitocondrias) se pueden heredar tanto por vía materna como cromosómica, la forma en que se transmiten de generación en generación puede variar mucho según la enfermedad. Las mutaciones genéticas mitocondriales que ocurren en el ADN nuclear pueden ocurrir en cualquiera de los cromosomas (dependiendo de la especie). Las mutaciones heredadas a través de los cromosomas pueden ser autosómicas dominantes o recesivas y también pueden ser dominantes o recesivas ligadas al sexo. La herencia cromosómica sigue las leyes mendelianas normales, a pesar de que el fenotipo de la enfermedad puede estar enmascarado.
Debido a las formas complejas en que el ADN mitocondrial y nuclear se "comunican" e interactúan, incluso la herencia aparentemente simple es difícil de diagnosticar. Una mutación en el ADN cromosómico puede cambiar una proteína que regula (aumenta o disminuye) la producción de otra determinada proteína en la mitocondria o el citoplasma; esto puede conducir a síntomas leves, si los hubiere, perceptibles. Por otro lado, algunas mutaciones devastadoras del mtDNA son fáciles de diagnosticar debido a su daño generalizado en los tejidos musculares, neurales y/o hepáticos (entre otros tejidos de alta energía y dependientes del metabolismo) y porque están presentes en la madre y todos los demás. la descendencia
El número de moléculas de mtDNA afectadas heredadas por una descendencia específica puede variar mucho porque:
- la mitocondria dentro del ovocito fertilizado es con lo que tendrá que comenzar la nueva vida (en términos de ADNmt),
- el número de mitocondrias afectadas varía de una célula a otra (en este caso, el ovocito fertilizado) dependiendo tanto del número que heredó de su célula madre como de los factores ambientales que pueden favorecer el ADN mitocondrial mutante o de tipo salvaje ,
- el número de moléculas de mtDNA en las mitocondrias varía entre dos y diez.
Es posible, incluso en nacimientos de gemelos, que un bebé reciba más de la mitad de moléculas de ADNmt mutantes mientras que el otro gemelo puede recibir solo una pequeña fracción de moléculas de ADNmt mutantes con respecto al tipo salvaje (dependiendo de cómo los gemelos se dividen entre sí y cómo muchas mitocondrias mutantes están a cada lado de la división). En algunos casos, algunas mitocondrias o una mitocondria del espermatozoide ingresa al ovocito, pero las mitocondrias paternas se descomponen activamente.
Genes[editar]
Los genes del genoma mitocondrial humano son los siguientes.
Cadena de transporte de electrones y humanina[editar]
Originalmente se creía incorrectamente que el genoma mitocondrial contenía solo 13 genes codificadores de proteínas, todos ellos codificantes de proteínas de la cadena de transporte de electrones. Sin embargo, en 2001, se descubrió una decimocuarta proteína biológicamente activa llamada humanina, y se descubrió que estaba codificada por el gen mitocondrial MT-RNR2, que también codifica parte del ribosoma mitocondrial (hecho de ARN):
| Nº del complejo | Categoría | Gen | Posiciones en el mitogenoma | Hilo |
|---|---|---|---|---|
| I | NADH deshidrogenasa | |||
| MT-ND1 | 3.307–4.262 | L | ||
| MT-ND2 | 4.470–5.511 | L | ||
| MT-ND3 | 10.059–10.404 | L | ||
| MT-ND4L | 10.470–10.766 | L | ||
| MT-ND4 | 10.760–12.137 (superposición con MT-ND4L) | L | ||
| MT-ND5 | 12.337–14.148 | L | ||
| MT-ND6 | 14.149–14.673 | H | ||
| III | Coenzima Q - citocromo c reductasa / Citocromo b | MT-CYB | 14.747–15.887 | L |
| IV | Citocromo c oxidasa | MT-CO1 | 5.904–7.445 | L |
| MT-CO2 | 7.586–8.269 | L | ||
| MT-CO3 | 9.207–9.990 | L | ||
| V | ATP sintasa | MT-ATP6 | 8.527–9.207 (superposición con MT-ATP8) | L |
| MT-ATP8 | 8.366–8.572 | L | ||
| — | Humanina | MT-RNR2 | — | — |
A diferencia del resto de proteínas, la humanina no permanece en la mitocondria e interactúa con el resto de la célula y los receptores celulares. La humanina puede proteger las células del cerebro al inhibir la apoptosis. A pesar de su nombre, también existen versiones de humanin en otros animales, como rattin en ratas.
ARNr[editar]
Los siguientes genes codifican ARNr:
| Subunidad | ARNr | Gen | Posiciones en el mitogenoma | Hilo |
|---|---|---|---|---|
| Pequeño (SSU) | 12 | MT-RNR1 | 648–1.601 | L |
| Grande (LSU) | 16 | MT-RNR2 | 1.671–3.229 | L |
ARNt[editar]
Los siguientes genes codifican los ARNt :
| Aminoácido | 3-Letras | 1-Letra | ADM MT | Posiciones | Hilo |
|---|---|---|---|---|---|
| Alanina | Ala | A | MT-TA | 5,587–5,655 | H |
| Arginina | Arg | R | MT-TR | 10,405–10,469 | L |
| Asparagina | Asn | N | MT-TN | 5,657–5,729 | H |
| Ácido aspártico | Asp | D | MT-TD | 7,518–7,585 | L |
| Cisteína | Cys | C | MT-TC | 5,761–5,826 | H |
| Ácido glutámico | Glu | E | MT-TE | 14,674–14,742 | H |
| Glutamina | Gln | Q | MT-TQ | 4,329–4,400 | H |
| Glicina | Gly | G | MT-TG | 9,991–10,058 | L |
| Histidina | His | H | MT-TH | 12,138–12,206 | L |
| Isoleucina | Ile | I | MT-TI | 4,263–4,331 | L |
| Leucina | Leu (UUR) | L | MT-TL1 | 3,230–3,304 | L |
| Leucina | Leu (CUN) | L | MT-TL2 | 12,266–12,336 | L |
| Lisina | Lys | K | MT-TK | 8,295–8,364 | L |
| Metionina | Met | M | MT-TM | 4,402–4,469 | L |
| Fenilalanina | Phe | F | MT-TF | 577–647 | L |
| Prolina | Pro | P | MT-TP | 15,956–16,023 | H |
| Serina | Ser (UCN) | S | MT-TS1 | 7,446–7,514 | H |
| Serina | Ser (AGY) | S | MT-TS2 | 12,207–12,265 | L |
| Treonina | Thr | T | MT-TT | 15,888–15,953 | L |
| Triptófano | Trp | W | MT-TW | 5,512–5,579 | L |
| Tirosina | Tyr | Y | MT-TY | 5,826–5,891 | H |
| Valina | Val | V | MT-TV | 1,602–1,670 | L |
Localización de los genes[editar]
El ADN mitocondrial tradicionalmente tenía las dos hebras de ADN designadas como la hebra pesada y la ligera, debido a sus densidades flotantes durante la separación en gradientes de cloruro de cesio,[11][12] que se encontró que estaba relacionado con el contenido relativo de nucleótidos G+T de la hebra[13] Sin embargo, la confusión del etiquetado de estas hebras está muy extendida y parece originarse con una identificación de la hebra de codificación mayoritaria como la pesada en un artículo influyente de 1999.[14][13] En los seres humanos, la cadena ligera del ADNmt lleva 28 genes y la cadena pesada del ADNmt lleva solo 9 genes.[13][15] Ocho de los 9 genes de la hebra pesada codifican moléculas de ARNt mitocondrial. El ADNmt humano consta de 16.569 pares de nucleótidos. Toda la molécula está regulada por una sola región reguladora que contiene los orígenes de replicación de las cadenas pesada y ligera. Se ha mapeado toda la molécula de ADN mitocondrial humano.
Variantes del código genético[editar]
El código genético es, en su mayor parte, universal, con pocas excepciones:[16] la genética mitocondrial incluye algunos de estos. Para la mayoría de los organismos, los " codones de terminación " son "UAA", "UAG" y "UGA". En las mitocondrias de vertebrados, "AGA" y "AGG" también son codones de parada, pero no "UGA", que codifica para triptófano. "AUA" codifica para isoleucina en la mayoría de los organismos pero para metionina en ARNm mitocondrial de vertebrados.
Existen muchas otras variaciones entre los códigos usados por otros mtRNA/tRNA mitocondriales, que resultaron no ser dañinos para sus organismos, y que pueden usarse como una herramienta (junto con otras mutaciones entre el mtDNA/RNA de diferentes especies) para determinar proximidad relativa de ascendencia común de especies relacionadas. (Cuanto más relacionadas estén las dos especies, más mutaciones de ADNmt/ARN serán iguales en su genoma mitocondrial).
Usando estas técnicas, se estima que las primeras mitocondrias surgieron hace alrededor de 1.500 millones de años. Una hipótesis generalmente aceptada es que las mitocondrias se originaron como un procariota aeróbico en una relación simbiótica dentro de un eucariota anaeróbico.
Replicación, reparación, transcripción y traducción[editar]
La replicación mitocondrial está controlada por genes nucleares y es específicamente adecuada para producir tantas mitocondrias como esa célula en particular necesite en ese momento.
La transcripción mitocondrial en humanos se inicia a partir de tres promotores, H1, H2 y L (promotores de cadena pesada 1, cadena pesada 2 y cadena ligera). El promotor H2 transcribe casi toda la cadena pesada y el promotor L transcribe toda la cadena ligera. El promotor H1 provoca la transcripción de las dos moléculas de ARNr mitocondrial.[17]
Cuando la transcripción tiene lugar en la hebra pesada, se crea una transcripción policistrónica. La hebra ligera produce pequeños transcritos, que pueden usarse como cebadores, o un transcrito largo. La producción de cebadores se produce mediante el procesamiento de transcripciones de cadenas ligeras con la RNasa mitocondrial MRP (Procesamiento de ARN mitocondrial). El requisito de transcripción para producir cebadores vincula el proceso de transcripción con la replicación del mtDNA. Los transcritos completos se cortan en moléculas funcionales de ARNt, ARNr y ARNm.
El proceso de iniciación de la transcripción en las mitocondrias involucra tres tipos de proteínas: la ARN polimerasa mitocondrial ( POLRMT ), el factor de transcripción mitocondrial A (TFAM) y los factores de transcripción mitocondrial B1 y B2 (TFB1M, TFB2M). POLRMT, TFAM y TFB1M o TFB2M se ensamblan en los promotores mitocondriales y comienzan la transcripción. Se desconocen los eventos moleculares reales que intervienen en la iniciación, pero estos factores constituyen la maquinaria de transcripción basal y se ha demostrado que funcionan in vitro.
La traducción mitocondrial aún no se comprende muy bien. Las traducciones in vitro aún no han tenido éxito, probablemente debido a la dificultad de aislar suficiente mt mRNA, mt rRNA funcional y posiblemente debido a los complicados cambios que sufre el mRNA antes de traducirse.
Polimerasa del ADN mitocondrial[editar]
La polimerasa del ADN mitocondrial (Pol gamma, codificada por el gen POLG ) se utiliza en la copia del ADNmt durante la replicación. Debido a que las dos hebras ( pesada y liviana ) de la molécula circular de mtDNA tienen diferentes orígenes de replicación, se replica en un modo de bucle D. Una hebra comienza a replicarse primero, desplazando a la otra hebra. Esto continúa hasta que la replicación alcanza el origen de la replicación en la otra hebra, momento en el cual la otra hebra comienza a replicarse en la dirección opuesta. Esto da como resultado dos nuevas moléculas de mtDNA. Cada mitocondria tiene varias copias de la molécula de mtDNA y el número de moléculas de mtDNA es un factor limitante en la fisión mitocondrial. Una vez que la mitocondria tiene suficiente ADNmt, área de membrana y proteínas de membrana, puede sufrir una fisión (muy similar a la que usan las bacterias) para convertirse en dos mitocondrias. La evidencia sugiere que las mitocondrias también pueden fusionarse e intercambiar (en una forma de cruce ) material genético entre sí. Las mitocondrias a veces forman grandes matrices en las que se producen constantemente intercambios de fusión, fisión y proteínas. ADNmt compartido entre las mitocondrias (a pesar de que pueden fusionarse)
Daños y errores de transcripción[editar]
El ADN mitocondrial es susceptible al daño de los radicales libres de oxígeno debido a los errores que ocurren durante la producción de ATP a través de la cadena de transporte de electrones. Estos errores pueden ser causados por trastornos genéticos, cáncer y variaciones de temperatura. Estos radicales pueden dañar las moléculas de mtDNA o cambiarlas, lo que dificulta que la polimerasa mitocondrial las replique. Ambos casos pueden conducir a deleciones, reordenamientos y otras mutaciones. La evidencia reciente ha sugerido que las mitocondrias tienen enzimas que corrigen el mtDNA y corrigen las mutaciones que pueden ocurrir debido a los radicales libres. Se cree que una ADN recombinasa que se encuentra en células de mamíferos también está implicada en un proceso de reparación de recombinación. Las deleciones y mutaciones debidas a los radicales libres se han asociado con el proceso de envejecimiento. Se cree que los radicales provocan mutaciones que conducen a proteínas mutantes, que a su vez generan más radicales. Este proceso lleva muchos años y está asociado con algunos procesos de envejecimiento que intervienen en los tejidos dependientes de oxígeno, como el cerebro, el corazón, los músculos y los riñones. Los procesos de automejoramiento como estos son posibles causas de enfermedades degenerativas, como el Parkinson, el Alzheimer y la enfermedad de las arterias coronarias.
Errores de replicación del ADNmt mediados cromosómicamente[editar]
Debido a que el crecimiento y la fisión mitocondrial están mediados por el ADN nuclear, las mutaciones en el ADN nuclear pueden tener una amplia gama de efectos en la replicación del ADNmt. A pesar de que los loci de algunas de estas mutaciones se han encontrado en los cromosomas humanos, todavía no se han aislado los genes y proteínas específicos implicados. Las mitocondrias necesitan una determinada proteína para someterse a la fisión. Si esta proteína (generada por el núcleo) no está presente, las mitocondrias crecen pero no se dividen. Esto conduce a mitocondrias gigantes e ineficientes. Los errores en los genes cromosómicos o sus productos también pueden afectar la replicación mitocondrial más directamente al inhibir la polimerasa mitocondrial e incluso pueden causar mutaciones en el mtDNA directa o indirectamente. Las mutaciones indirectas son causadas con mayor frecuencia por radicales creados por proteínas defectuosas hechas de ADN nuclear.
Enfermedades mitocondriales[editar]
Contribución del genoma mitocondrial versus el genoma nuclear[editar]
En total, la mitocondria alberga alrededor de 3000 tipos diferentes de proteínas, pero solo alrededor de 13 de ellas están codificadas en el ADN mitocondrial. La mayoría de los 3000 tipos de proteínas están involucradas en una variedad de procesos además de la producción de ATP, como la síntesis de porfirina. Solo alrededor del 3% de ellos codifican proteínas de producción de ATP. Esto significa que la mayor parte de la información genética que codifica la composición proteica de las mitocondrias se encuentra en el ADN cromosómico y está involucrada en procesos distintos a la síntesis de ATP. Esto aumenta las posibilidades de que se produzca una mutación que afectará a una mitocondria en el ADN cromosómico, que se hereda con un patrón mendeliano. Otro resultado es que una mutación cromosómica afectará a un tejido específico debido a sus necesidades específicas, ya sean requisitos de alta energía o una necesidad de catabolismo o anabolismo de un neurotransmisor o ácido nucleico específico. Debido a que cada mitocondria lleva varias copias del genoma mitocondrial (de 2 a 10 en humanos), las mutaciones mitocondriales se pueden heredar por vía materna mediante mutaciones del ADNmt que están presentes en las mitocondrias dentro del ovocito antes de la fecundación o (como se indicó anteriormente) a través de mutaciones en el cromosomas
Presentación[editar]
Las enfermedades mitocondriales varían en severidad desde asintomáticas hasta fatales, y se deben más comúnmente a mutaciones heredadas en lugar de adquiridas en el ADN mitocondrial. Una mutación mitocondrial dada puede causar varias enfermedades según la gravedad del problema en las mitocondrias y el tejido en el que se encuentran las mitocondrias afectadas. Por el contrario, varias mutaciones diferentes pueden presentarse como la misma enfermedad. Esta caracterización casi específica del paciente de las enfermedades mitocondriales (consulte Medicina personalizada ) hace que sea muy difícil reconocerlas, diagnosticarlas y rastrearlas con precisión. Algunas enfermedades son observables en el nacimiento o incluso antes (muchas causan la muerte), mientras que otras no se manifiestan hasta la edad adulta tardía (trastornos de aparición tardía). Esto se debe a que el número de mitocondrias mutantes frente a las de tipo salvaje varía entre las células y los tejidos, y cambia continuamente. Debido a que las células tienen múltiples mitocondrias, diferentes mitocondrias en la misma célula pueden tener diferentes variaciones del mtDNA. Esta condición se conoce como heteroplasmia. Cuando un determinado tejido alcanza una cierta proporción de mitocondrias mutantes frente a las de tipo salvaje, se presentará una enfermedad. La proporción varía de una persona a otra y de un tejido a otro (según sus requisitos específicos de energía, oxígeno y metabolismo, y los efectos de la mutación específica). Las enfermedades mitocondriales son muy numerosas y diferentes. Aparte de las enfermedades causadas por anomalías en el ADN mitocondrial, se sospecha que muchas enfermedades están asociadas en parte a disfunciones mitocondriales, como la diabetes mellitus,[18] formas de cáncer[19] y enfermedades cardiovasculares, acidosis láctica,[20] formas específicas de miopatía,[21] osteoporosis,[22] enfermedad de Alzheimer,[23] enfermedad de Parkinson,[24] accidente cerebrovascular,[25] infertilidad masculina[26] y que también se cree que desempeñan un papel en el proceso de envejecimiento.[27]
Uso en medicina forense[editar]
El mtDNA humano también se puede utilizar para ayudar a identificar individuos.[28] Los laboratorios forenses ocasionalmente usan la comparación de mtDNA para identificar restos humanos, y especialmente para identificar restos óseos más antiguos no identificados. Aunque a diferencia del ADN nuclear, el mtDNA no es específico de un individuo, se puede utilizar en combinación con otras pruebas (pruebas antropológicas, pruebas circunstanciales y similares) para establecer la identificación. El mtDNA también se utiliza para excluir posibles coincidencias entre personas desaparecidas y restos no identificados.[29] Muchos investigadores creen que el ADNmt es más adecuado para la identificación de restos óseos más antiguos que el ADN nuclear debido a que el mayor número de copias de ADNmt por célula aumenta la posibilidad de obtener una muestra útil y porque es posible la coincidencia con un pariente vivo incluso si existen numerosas copias maternas. generaciones separan a los dos.
Ejemplos[editar]
Los restos del forajido estadounidense Jesse James se identificaron mediante una comparación entre el ADNmt extraído de sus restos y el ADNmt del hijo de la bisnieta de línea femenina de su hermana.[30]
De manera similar, los restos de Alexandra Feodorovna (Alix de Hesse), última emperatriz de Rusia, y sus hijos fueron identificados por comparación de su ADN mitocondrial con el del príncipe Felipe, duque de Edimburgo, cuya abuela materna era la hermana de Alexandra , Victoria de Hesse.[31]
Del mismo modo, para identificar los restos del emperador Nicolás II, se comparó su ADN mitocondrial con el de James Carnegie, tercer duque de Fife, cuya bisabuela materna , Alexandra de Dinamarca (reina Alexandra), era hermana de la madre de Nicolás II , Dagmar de Dinamarca (emperatriz María Feodorovna).[32][33]
Y también con los restos del rey Ricardo III.[34]
Referencias[editar]
- ↑ Zhang D, Keilty D, Zhang ZF, Chian RC (2017). «Mitochondria in oocyte aging: current understanding.». Facts Views Vis Obgyn 9 (1): 29-38. PMC 5506767. PMID 28721182.
- ↑ Shuster RC, Rubenstein AJ, Wallace DC (1988). «Mitochondrial DNA in anucleate human blood cells.». Biochem Biophys Res Commun 155 (3): 1360-5. PMID 3178814. doi:10.1016/s0006-291x(88)81291-9.
- ↑ «Mitochondria – cell powerhouses». Science Learning Hub. 20 de julio de 2011. Consultado el 3 de mayo de 2023.
- ↑ Schwartz, Marianne; Vissing, John (22 de agosto de 2002). «Paternal Inheritance of Mitochondrial DNA». New England Journal of Medicine 347 (8): 576-580. PMID 12192017. doi:10.1056/NEJMoa020350.
- ↑ Anderson, S.; Bankier, A. T.; Barrell, B. G.; de Bruijn, M. H. L.; Coulson, A. R.; Drouin, J.; Eperon, I. C.; Nierlich, D. P. et al. (April 1981). «Sequence and organization of the human mitochondrial genome». Nature 290 (5806): 457-465. Bibcode:1981Natur.290..457A. PMID 7219534. doi:10.1038/290457a0.
- ↑ «Untitled». Archivado desde el original el 13 de agosto de 2011. Consultado el 13 de junio de 2012.
- ↑ a b c Satoh, M; Kuroiwa, T (September 1991). «Organization of multiple nucleoids and DNA molecules in mitochondria of a human cell». Experimental Cell Research 196 (1): 137-140. PMID 1715276. doi:10.1016/0014-4827(91)90467-9.
- ↑ Shuster RC, Rubenstein AJ, Wallace DC (1988). «Mitochondrial DNA in anucleate human blood cells.». Biochem Biophys Res Commun 155 (3): 1360-5. PMID 3178814. doi:10.1016/s0006-291x(88)81291-9.
- ↑ Nagy G, Barcza M, Gonchoroff N, Phillips PE, Perl A (2004). «Nitric oxide-dependent mitochondrial biogenesis generates Ca2+ signaling profile of lupus T cells.». J Immunol 173 (6): 3676-83. PMC 4034140. PMID 15356113. doi:10.4049/jimmunol.173.6.3676.
- ↑ Zhang D, Keilty D, Zhang ZF, Chian RC (2017). «Mitochondria in oocyte aging: current understanding.». Facts Views Vis Obgyn 9 (1): 29-38. PMC 5506767. PMID 28721182.
- ↑ Zimmerman, Earl G.; Akins, Darrin R.; Planz, John V.; Schurr, Michael J. (September 1988). «A rapid procedure for isolating mitochondrial DNA». Gene Analysis Techniques 5 (5): 102-104. PMID 2847966. doi:10.1016/0735-0651(88)90004-0.
- ↑ Welter, Cornelius; Meese, Eckart; Blin, Nikolaus (1988). «Rapid step-gradient purification of mitochondrial DNA». Molecular Biology Reports 13 (2): 117-120. PMID 3221842. doi:10.1007/BF00539059.
- ↑ a b c Barroso Lima, Nicholas Costa; Prosdocimi, Francisco (17 de febrero de 2018). «The heavy strand dilemma of vertebrate mitochondria on genome sequencing age: number of encoded genes or G + T content?». Mitochondrial DNA Part A 29 (2): 300-302. PMID 28129726. doi:10.1080/24701394.2016.1275603.
- ↑ Taanman, Jan-Willem (February 1999). «The mitochondrial genome: structure, transcription, translation and replication». Biochimica et Biophysica Acta (BBA) - Bioenergetics 1410 (2): 103-123. PMID 10076021. doi:10.1016/s0005-2728(98)00161-3.
- ↑ Anderson, S.; Bankier, A. T.; Barrell, B. G.; de Bruijn, M. H. L.; Coulson, A. R.; Drouin, J.; Eperon, I. C.; Nierlich, D. P. et al. (1981). «Sequence and organization of the human mitochondrial genome». Nature 290 (5806): 457-65. Bibcode:1981Natur.290..457A. PMID 7219534. doi:10.1038/290457a0.
- ↑ «The Genetic Codes». www.ncbi.nlm.nih.gov. National Center for Biotechnology Information. Consultado el 16 de marzo de 2019.
- ↑ Asin-Cayuela, Jordi; Gustafsson, Claes M. (2007). «Mitochondrial transcription and its regulation in mammalian cells». Trends in Biochemical Sciences 32 (3): 111-17. PMID 17291767. doi:10.1016/j.tibs.2007.01.003.
- ↑ Tanaka, Masashi; Fuku, Noriyuki; Nishigaki, Yutaka; Matsuo, Hitoshi; Segawa, Tomonori; Watanabe, Sachiro; Kato, Kimihiko; Yoko, Kiyoshi et al. (February 2007). «Women With Mitochondrial Haplogroup N9a Are Protected Against Metabolic Syndrome». Diabetes 56 (2): 518-521. PMID 17259400. doi:10.2337/db06-1105.
- ↑ Theodoratou, Evropi; Din, Farhat V.N.; Farrington, Susan M.; Cetnarskyj, Roseanne; Barnetson, Rebecca A.; Porteous, Mary E.; Dunlop, Malcolm G.; Campbell, Harry et al. (February 2010). «Association between common mtDNA variants and all-cause or colorectal cancer mortality». Carcinogenesis 31 (2): 296-301. PMID 19945968. doi:10.1093/carcin/bgp237.
- ↑ Goto, Y (September 1993). «[MELAS (mitochondrial myopathy, encephalopathy lactic acidosis, and stroke-like episodes): clinical features and mitochondrial DNA mutations].». Nihon Rinsho. Japanese Journal of Clinical Medicine 51 (9): 2373-8. PMID 8411715.
- ↑ Ahuja, Abhimanyu S. (21 de mayo de 2018). «Understanding mitochondrial myopathies: a review». PeerJ 6: e4790. PMC 5967365. PMID 29844960. doi:10.7717/peerj.4790.
- ↑ Angireddy, Rajesh; Kazmi, Hasan Raza; Srinivasan, Satish; Sun, Li; Iqbal, Jameel; Fuchs, Serge Y.; Guha, Manti; Kijima, Takashi et al. (August 2019). «Cytochrome c oxidase dysfunction enhances phagocytic function and osteoclast formation in macrophages». The FASEB Journal 33 (8): 9167-9181. PMC 6662975. PMID 31063702. doi:10.1096/fj.201900010RR.
- ↑ Carrieri, Giuseppina; Bonafè, Massimiliano; De Luca, Maria; Rose, Giuseppina; Varcasia, Ottavia; Bruni, Amalia; Maletta, Raffaele; Nacmias, Benedetta et al. (March 2001). «Mitochondrial DNA haplogroups and APOE4 allele are non-independent variables in sporadic Alzheimer's disease». Human Genetics 108 (3): 194-198. PMID 11354629. doi:10.1007/s004390100463.
- ↑ Martín-Jiménez, Rebeca; Lurette, Olivier; Hebert-Chatelain, Etienne (1 de agosto de 2020). «Damage in Mitochondrial DNA Associated with Parkinson's Disease». DNA and Cell Biology 39 (8): 1421-1430. PMID 32397749. doi:10.1089/dna.2020.5398.
- ↑ Chinnery, Patrick F; Elliott, Hannah R; Syed, Anila; Rothwell, Peter M (May 2010). «Mitochondrial DNA haplogroups and risk of transient ischaemic attack and ischaemic stroke: a genetic association study». The Lancet Neurology 9 (5): 498-503. PMC 2855429. PMID 20362514. doi:10.1016/S1474-4422(10)70083-1.
- ↑ Ruiz-Pesini, Eduardo; Lapeña, Ana-Cristina; Díez-Sánchez, Carmen; Pérez-Martos, Acisclo; Montoya, Julio; Alvarez, Enrique; Díaz, Miguel; Urriés, Antonio et al. (September 2000). «Human mtDNA Haplogroups Associated with High or Reduced Spermatozoa Motility». The American Journal of Human Genetics 67 (3): 682-696. PMC 1287528. PMID 10936107. doi:10.1086/303040.
- ↑ Courtenay, Monique D.; Gilbert, John R.; Jiang, Lan; Cummings, Anna C.; Gallins, Paul J.; Caywood, Laura; Reinhart-Mercer, Lori; Fuzzell, Denise et al. (February 2012). «Mitochondrial Haplogroup X is associated with successful aging in the Amish». Human Genetics 131 (2): 201-208. PMC 4834861. PMID 21750925. doi:10.1007/s00439-011-1060-3.
- ↑ Brown, W. M. (1 de junio de 1980). «Polymorphism in mitochondrial DNA of humans as revealed by restriction endonuclease analysis.». Proceedings of the National Academy of Sciences 77 (6): 3605-3609. Bibcode:1980PNAS...77.3605B. PMC 349666. PMID 6251473. doi:10.1073/pnas.77.6.3605.
- ↑ «Paleo-DNA Laboratory – Forensic Services». Archivado desde el original el 13 de marzo de 2012. Consultado el 13 de junio de 2012.
- ↑ Stone, Anne C.; Starrs, James E.; Stoneking, Mark (1 de enero de 2001). «Mitochondrial DNA Analysis of the Presumptive Remains of Jesse James». Journal of Forensic Sciences 46 (1): 173-6. PMID 11210907. doi:10.1520/JFS14932J.
- ↑ Gill, Peter; Ivanov, Pavel L.; Kimpton, Colin; Piercy, Romelle; Benson, Nicola; Tully, Gillian; Evett, Ian; Hagelberg, Erika et al. (February 1994). «Identification of the remains of the Romanov family by DNA analysis». Nature Genetics 6 (2): 130-135. PMID 8162066. doi:10.1038/ng0294-130.
- ↑ Gill, Peter; Ivanov, Pavel L.; Kimpton, Colin; Piercy, Romelle; Benson, Nicola; Tully, Gillian; Evett, Ian; Hagelberg, Erika et al. (February 1994). «Identification of the remains of the Romanov family by DNA analysis». Nature Genetics 6 (2): 130-135. PMID 8162066. doi:10.1038/ng0294-130.
- ↑ Ivanov, Pavel L.; Wadhams, Mark J.; Roby, Rhonda K.; Holland, Mitchell M.; Weedn, Victor W.; Parsons, Thomas J. (April 1996). «Mitochondrial DNA sequence heteroplasmy in the Grand Duke of Russia Georgij Romanov establishes the authenticity of the remains of Tsar Nicholas II». Nature Genetics 12 (4): 417-420. PMID 8630496. doi:10.1038/ng0496-417.
- ↑ Ashdown-Hill, John (2013). The Last Days of Richard III and the Fate of His DNA. History Press. ISBN 978-0-7524-9205-6.
Bibliografía[editar]
- Li, Xiangqi; Liu, Lianyong; Xi, Qian; Zhao, Xuemei; Fang, Mingshuang; Ma, Junhua; Zhu, Zhaohui; Wang, Xing et al. (2016). «Short-term serum deprivation causes no significant mitochondrial DNA mutation in vascular smooth muscle cells revealed by a new next generation sequencing technology». Acta Biochimica et Biophysica Sinica 48 (9): 862-4. PMID 27261779. doi:10.1093/abbs/gmw059.
Enlaces externos[editar]
 Wikimedia Commons alberga una categoría multimedia sobre Genética mitocondrial humana.
Wikimedia Commons alberga una categoría multimedia sobre Genética mitocondrial humana.- National Institutes of Health. «Mitochondrial DNA». Genetics Home Reference. Consultado el 6 de mayo de 2017.
- «Societat Catalana de Neurologia». Archivado desde el original el 18 de noviembre de 2005. Consultado el 5 de diciembre de 2005.
- «MITOMAP Genome». Archivado desde el original el 8 de abril de 2005. Consultado el 5 de diciembre de 2005.
