Diferencia entre revisiones de «Metabolismo»
m +s |
+nucleótidos +coenzimas |
||
| Línea 51: | Línea 51: | ||
Los [[carbohidrato]]s son [[aldehído]]s o [[cetona|cetonas]] con [[grupo hidroxilo|grupos hidroxilo]] que pueden existir como cadenas o anillos. Los carbohidratos son las moléculas biológicas más abundantes, y tienen varios roles; algunos son el almacenamiento de [[energía]] ([[almidón]] y [[glucógeno]]) y componentes estructurales ([[celulosa]] en las plantas, [[quitina]] en los animales). <ref name=Nelson/> Los carbohidratos básicos son llamados [[monosacárido]]s e incluyen [[galactosa]], [[fructosa]], y más importante [[glucosa]]. Los monosacáridos se pueden sintetizar y formar [[polisacárido]]s.<ref>{{cite journal |author=Raman R, Raguram S, Venkataraman G, Paulson J, Sasisekharan R |title=Glycomics: an integrated systems approach to structure-function relationships of glycans |journal=Nat Methods |volume=2 |issue=11 |pages=817-24 |year=2005 |pmid=16278650}}</ref> |
Los [[carbohidrato]]s son [[aldehído]]s o [[cetona|cetonas]] con [[grupo hidroxilo|grupos hidroxilo]] que pueden existir como cadenas o anillos. Los carbohidratos son las moléculas biológicas más abundantes, y tienen varios roles; algunos son el almacenamiento de [[energía]] ([[almidón]] y [[glucógeno]]) y componentes estructurales ([[celulosa]] en las plantas, [[quitina]] en los animales). <ref name=Nelson/> Los carbohidratos básicos son llamados [[monosacárido]]s e incluyen [[galactosa]], [[fructosa]], y más importante [[glucosa]]. Los monosacáridos se pueden sintetizar y formar [[polisacárido]]s.<ref>{{cite journal |author=Raman R, Raguram S, Venkataraman G, Paulson J, Sasisekharan R |title=Glycomics: an integrated systems approach to structure-function relationships of glycans |journal=Nat Methods |volume=2 |issue=11 |pages=817-24 |year=2005 |pmid=16278650}}</ref> |
||
=== Nucleótidos === |
|||
{{AP|Nucleótido}} |
|||
Los polímeros [[ADN]] y [[ARN]] son cadenas de los [[nucleótido]]s. Estas moléculas son críticas para el almacenamiento y uso de [[gen|información genética]] por el proceso de [[transcripción (genética)|transcripción]] y [[síntesis proteica|biosíntesis de proteínas]]<ref name=Nelson/>. Esta información es protegida por mecanismo de [[reparación del ADN]] y propagada por [[replicación del ADN]]. Algunos [[virus]] tienen un [[genoma]] de ARN, por ejemplo el [[HIV]], y utilizan retrotranscripción para crear ADN a partir de su genoma viral de ARN.<ref>{{cite journal |author=Sierra S, Kupfer B, Kaiser R |title=Basics of the virology of HIV-1 and its replication |journal=J Clin Virol |volume=34 |issue=4 |pages=233-44 |year=2005 |pmid=16198625}}</ref>; estos virus son llamados [[retrovirus]]. El ARN en [[ribozima]]s como los [[ribosoma]]s es similar a las enzimas y puede catabolizar reacciones químicas. [[Nucleósido]]s individuales son hechos al unir [[bases nitrogenadas]] con [[ribosa]]. Estas bases son anillos que contienen [[nitrógeno]], clasificado como [[purina]] y [[pirimidina]]s. Los nucleótidos también actúan como coenzimas en reacciones metabólicas de transferencia en grupo.<ref name=Wimmer>{{cite journal |author=Wimmer M, Rose I |title=Mechanisms of enzyme-catalyzed group transfer reactions |journal=Annu Rev Biochem |volume=47 |issue= |pages=1031-78 |year=1978 |pmid=354490}}</ref> |
|||
=== Coenzimas === |
|||
{{AP|Coenzima}} |
|||
[[Image:ATP-3D-vdW.png|thumb|right|250px|Estructura de una [[coenzima]], [[adenosín trifosfato]].]] |
|||
El metabolismo conlleva un gran arraigo de reacciones químicas, pero muchas por mayoría caen en algunos tipos básicos de [[Grupo funcional|reacción de transferencia en grupo]].<ref>{{cite journal |author=Mitchell P |title=The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems |journal=Eur J Biochem |volume=95 |issue=1 |pages=1-20 |year=1979 |pmid=378655}}</ref> Esta química común permite a células utilizar los intermedios metabólicos para logra grupos químicos entre diferentes reacciones.<ref name=Wimmer/> Este grupo de transferencia intermediaria son las [[coenzima]]s. Cada clase de reacción de grupo es llevada a cabo por una coenzima particular, que es un substrato para un grupo de enzimas que lo producen, y un grupo de enzimas que lo consumen. Estas coenzimas son, por ende, continuamente hechas, consumidas y luego recicladas.<ref name=Dimroth>{{cite journal |author=Dimroth P, von Ballmoos C, Meier T |title=Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series |url=http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=16607397 |journal=EMBO Rep |volume=7 |issue=3 |pages=276-82 |year=2006 |pmid=16607397}}</ref> |
|||
<!-- |
|||
These coenzymes are therefore continuously being made, consumed and then recycled.<ref name=Dimroth>{{cite journal |author=Dimroth P, von Ballmoos C, Meier T |title=Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series |url=http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=16607397 |journal=EMBO Rep |volume=7 |issue=3 |pages=276-82 |year=2006 |pmid=16607397}}</ref> |
|||
The most central coenzyme is [[adenosine triphosphate]] (ATP), the universal energy currency of cells. This [[nucleotide]] is used to transfer chemical energy between different chemical reactions. There is only a small amount of ATP in cells, but as it is continuously regenerated, the human body can use about its own weight in ATP per day.<ref name=Dimroth/> ATP acts as a bridge between catabolism and anabolism, with catabolic reactions generating ATP and anabolic reactions consuming it. It also serves as a carrier of phosphate groups in [[phosphorylation]] reactions. |
|||
A [[vitamin]] is an organic compound needed in small quantities that cannot be made in the cells. In human [[nutrition]], most vitamins function as coenzymes after modification; for example, all water-soluble vitamins are phosphorylated or are coupled to nucleotides when they are used in cells.<ref>{{Citation | last1 = Coulston | first1 = Ann | last2 = Kerner | first2 = John | last3 = Hattner | first3 = JoAnn | last4 = Srivastava | first4 = Ashini | contribution = Nutrition Principles and Clinical Nutrition | title = Stanford School of Medicine Nutrition Courses | publisher = SUMMIT | year = 2006 }}</ref> [[Nicotinamide adenine dinucleotide]] (NADH), a derivative of vitamin B<sub>3</sub> ([[niacin]]), is an important coenzyme that acts as a hydrogen acceptor. Hundreds of separate types of [[dehydrogenase]]s remove electrons from their substrates and [[redox|reduce]] NAD<sup>+</sup> into NADH. This reduced form of the coenzyme is then a substrate for any of the [[reductase]]s in the cell that need to reduce their substrates.<ref>{{cite journal |author=Pollak N, Dölle C, Ziegler M |title=The power to reduce: pyridine nucleotides--small molecules with a multitude of functions |journal=Biochem J |volume=402 |issue=2 |pages=205-18 |year=2007 |pmid=17295611}}</ref> Nicotinamide adenine dinucleotide exists in two related forms in the cell, NADH and NADPH. The NAD<sup>+</sup>/NADH form is more important in catabolic reactions, while NADP<sup>+</sup>/NADPH is used in anabolic reactions. |
|||
[[Image:1GZX Haemoglobin.png|thumb|300px|left|Structure of [[hemoglobin]]. The protein subunits are in red and blue, and the iron-containing [[heme]] groups in green. From {{PDB|1GZX}}.]] |
|||
===Minerals and cofactors=== |
|||
{{further|[[Physiology]], [[bioinorganic chemistry]] and [[Human iron metabolism|iron metabolism]]}} |
|||
Inorganic elements play critical roles in metabolism; some are abundant (e.g. [[sodium]] and [[potassium]]) while others function at minute concentrations. About 99% of mammals' mass are the elements [[carbon]], [[nitrogen]], [[calcium]], [[sodium]], [[chlorine]], [[potassium]], [[hydrogen]], [[oxygen]] and [[sulfur]].<ref name=Heymsfield>{{cite journal |author=Heymsfield S, Waki M, Kehayias J, Lichtman S, Dilmanian F, Kamen Y, Wang J, Pierson R |title=Chemical and elemental analysis of humans in vivo using improved body composition models |journal=Am J Physiol |volume=261 |issue=2 Pt 1 |pages=E190-8 |year=1991 |pmid=1872381}}</ref> The [[organic compound]]s (proteins, lipids and carbohydrates) contain the majority of the carbon and nitrogen and most of the oxygen and hydrogen is present as water.<ref name=Heymsfield/> |
|||
The abundant inorganic elements act as [[ion]]ic [[electrolyte]]s. The most important ions are [[sodium]], [[potassium]], [[calcium]], [[magnesium]], [[chloride]], [[phosphate]], and the organic ion [[bicarbonate]]. The maintenance of precise [[ion gradient|gradient]]s across cell membranes maintains [[osmotic pressure]] and [[pH]].<ref>{{cite journal |author=Sychrová H |title=Yeast as a model organism to study transport and homeostasis of alkali metal cations |url=http://www.biomed.cas.cz/physiolres/pdf/53%20Suppl%201/53_S91.pdf |journal=Physiol Res |volume=53 Suppl 1 |issue= |pages=S91-8 |year=2004 |pmid=15119939}}</ref> Ions are also critical for [[nerve]]s and [[muscle]]s, as [[action potential]]s in these tissues are produced by the exchange of electrolytes between the [[extracellular fluid]] and the [[cytosol]].<ref>{{cite journal |author=Levitan I |title=Modulation of ion channels in neurons and other cells |journal=Annu Rev Neurosci |volume=11 |issue= |pages=119-36 |year=1988 |pmid=2452594}}</ref> Electrolytes enter and leave cells through proteins in the [[cell membrane]] called [[ion channels]]. For example, [[muscle contraction]] depends upon the movement of calcium, sodium and potassium through ion channels in the cell membrane and [[T-tubule]]s.<ref>{{cite journal |author=Dulhunty A |title=Excitation-contraction coupling from the 1950s into the new millennium |journal=Clin Exp Pharmacol Physiol |volume=33 |issue=9 |pages=763-72 |year=2006 |pmid=16922804}}</ref> |
|||
The [[transition metal]]s are usually present as [[trace element]]s in organisms, with [[zinc]] and [[iron]] being most abundant.<ref>{{cite journal |author=Mahan D, Shields R |title=Macro- and micromineral composition of pigs from birth to 145 kilograms of body weight |url=http://jas.fass.org/cgi/reprint/76/2/506 |journal=J Anim Sci |volume=76 |issue=2 |pages=506-12 |year=1998 |pmid=9498359}}</ref><ref name=Husted>{{cite journal |author=Husted S, Mikkelsen B, Jensen J, Nielsen N |title=Elemental fingerprint analysis of barley (Hordeum vulgare) using inductively coupled plasma mass spectrometry, isotope-ratio mass spectrometry, and multivariate statistics |journal=Anal Bioanal Chem |volume=378 |issue=1 |pages=171-82 |year=2004 |pmid=14551660}}</ref> These metals are used in some proteins as cofactors and are essential for the activity of enzymes such as [[catalase]] and oxygen-carrier proteins such as [[hemoglobin]].<ref>{{cite journal |author=Finney L, O'Halloran T |title=Transition metal speciation in the cell: insights from the chemistry of metal ion receptors |journal=Science |volume=300 |issue=5621 |pages=931-6 |year=2003 |pmid=12738850}}</ref> These [[cofactors]] are bound tightly to a specific protein; although enzyme cofactors can be modified during catalysis, cofactors always return to their original state after catalysis has taken place. The metal micronutrients are taken up into organisms by specific transporters and bound to storage proteins such as [[ferritin]] or [[metallothionein]] when not being used.<ref>{{cite journal |author=Cousins R, Liuzzi J, Lichten L |title=Mammalian zinc transport, trafficking, and signals |url=http://www.jbc.org/cgi/content/full/281/34/24085 |journal=J Biol Chem |volume=281 |issue=34 |pages=24085-9 |year=2006 |pmid=16793761}}</ref><ref>{{cite journal |author=Dunn L, Rahmanto Y, Richardson D |title=Iron uptake and metabolism in the new millennium |journal=Trends Cell Biol |volume=17 |issue=2 |pages=93-100 |year=2007 |pmid=17194590}}</ref> --> |
|||
== Referencias == |
== Referencias == |
||
| Línea 58: | Línea 89: | ||
{{Wikiconcurso|3}} |
{{Wikiconcurso|3}} |
||
[[ar:التمثيل الغذائى]] |
|||
[[ca:Metabolisme]] |
|||
[[cs:Metabolismus]] |
|||
[[da:Stofskifte]] |
|||
[[de:Stoffwechsel]] |
|||
[[en:Metabolism]] |
|||
[[et:Metabolism]] |
|||
[[eo:Metabolo]] |
|||
[[fa:دگرگشت]] |
|||
[[fr:Métabolisme]] |
|||
[[ko:신진대사]] |
|||
[[io:Metabolio]] |
|||
[[id:Metabolisme]] |
|||
[[it:Metabolismo]] |
|||
[[he:מטבוליזם]] |
|||
[[lb:Metabolismus]] |
|||
[[mk:Метаболизам]] |
[[mk:Метаболизам]] |
||
[[nl:Stofwisseling]] |
[[nl:Stofwisseling]] |
||
[[ja:代謝]] |
|||
[[no:Stoffskifte]] |
|||
[[pl:Metabolizm]] |
[[pl:Metabolizm]] |
||
[[pt:Metabolismo]] |
[[pt:Metabolismo]] |
||
[[ro:Metabolism]] |
|||
[[ru:Метаболизм]] |
[[ru:Метаболизм]] |
||
[[simple:Metabolism]] |
[[simple:Metabolism]] |
||
[[sk: |
[[sk:Látková premena]] |
||
[[su:Métabolisme]] |
[[su:Métabolisme]] |
||
[[fi:Aineenvaihdunta]] |
|||
[[sv:Metabolism]] |
[[sv:Metabolism]] |
||
[[th:กระบวนการสร้างและสลาย]] |
[[th:กระบวนการสร้างและสลาย]] |
||
[[vi:Chuyển hóa]] |
[[vi:Chuyển hóa]] |
||
[[zh:新陈代谢]] |
|||
Revisión del 20:39 20 abr 2007
El metabolismo es el conjunto de reacciones químicas que ocurren en una célula. Estos procesos son la base de la vida, y permiten a las célula crecer, reproducirse, mantener sus estructuras, y responder a su ambiente. El metabolismo se divide en dos categorías. Reacciones catabólicas liberan y acarrean energía; un ejemplo es la degradación de comida en la respiración celular. Las reacciones anabólicas, en cambio, usan esta energía liberada para construir componentes de las célula como lo son las proteínas y los ácidos nucleicos.
Las reacciones químicas del metabolismo son organizadas en rutas metabólicas, donde un químico es transformado en otro por una secuencia de enzimas. Las enzimas son cruciales en el metabolismo porque permiten a las células transformar reacciones termodinámicas deseadas pero deseadas en favorables. Las enzimas también permiten la regulización de rutas metabólicas en respuesta al ambiente de la célula, o señales a otras células.
El metabolismo de un organismo determina que substancias encontrará nutritivas y cuales encontrará venenosas. Por ejemplo, algunas procariotas usan sulfuro de hidrógeno como nutriente, pero este gas es venenoso para animales.[1]. La velocidad del metabolismo, el rango metabólico, también influye en cuánto alimento el organismo va a requerir.
Una característica del metabolismo es la similitud de las rutas metabólicas básicas entre varias especies diferentes. Por ejemplo, los pasos químicos como el ciclo de Krebs es universal en todas las células vivas y diverso como la bacteria unicelular Escherichia coli y muchos organismos pluricelulares como el elefante.[2]. Esta estructura metabólica compartida es el resultado de estas rutas altamente efectivas, y su aparición temprana en la historia evolutiva.[3][4]
Bioquímicos principales
La mayor parte de las estructuras que componen a los animales, plantas y microbios son compuestos por tres moléculas básicsa: aminoácidos, hidratos de carbono y lípidos (también denominados grasas). Como estas moléculas son vitales para la vida, el metabolismo se centra en sintetizar estas moléculas, en la contrucción de células y tejidos, o en degradarlas y usarlas como recurso de energía, en la digestión y el uso del alimento. Muchos bioquímicos importantes pueden ser juntados para crear polímeros como el ADN (ácido desoxirribonucleico) y proteínas. Estas macromoléculas son esenciales en los organismos vivos. Polímeros conocidos se listan en la siguiente tabla:
| Tipo de molécula | Nombre de forma de monómero | Nombre de formas de polímero |
|---|---|---|
| Aminoácidos | Aminoácidos | Proteínas |
| Carbohidratos | Monosacáridos | Polisacáridos |
| Ácidos nucleicos | Nucleótidos | Polinucleótidos |
Aminoácidos y proteínas
Las proteínas están compuestas por los aminoácidos, arreglados en una cadena lineal y unidos por enlaces peptídicos. Muchas proteínas son las enzimas que catabolizan las reacciones químicas en el metabolismo. Otras proteínas tienen funciones estructurales o mecánicas, como las proteínas del citoesqueleto que forman un sistema de andamio para mantener la forma de la célula.[5][6]. Las proteínas también son partícipes en la comunicación celular, respuesta inmune, adhesión celular y el ciclo celular.[7]
Lípidos
Los lípidos son los bioquímicos más diversos. Sus usos estructurales básicos son ser parte de la membrana biológica como una membrana celular, o como recurso de energía.[7] Los lípidos son denominados hidrófobos, como moléculas biológicas que se disuelven en un solvente orgánico como la bencina o cloroformo.[8]. Las grasas son un grupo de compuestos que incluyen ácidos grasos y glicerol; una molécula de glicerol junto a a tres ácidos grasos éster es un triglicérido.[9] Hay variaciones en esta estrucutra básica, e incluyen columnas como grupos hidrófilos, que incluyen grupos fosfato en fosfolípidos. Esteroides como el colesterol son otra clase mayor de lípidos sintetizados en las células.[10]
Carbohidratos

Los carbohidratos son aldehídos o cetonas con grupos hidroxilo que pueden existir como cadenas o anillos. Los carbohidratos son las moléculas biológicas más abundantes, y tienen varios roles; algunos son el almacenamiento de energía (almidón y glucógeno) y componentes estructurales (celulosa en las plantas, quitina en los animales). [7] Los carbohidratos básicos son llamados monosacáridos e incluyen galactosa, fructosa, y más importante glucosa. Los monosacáridos se pueden sintetizar y formar polisacáridos.[11]
Nucleótidos
Los polímeros ADN y ARN son cadenas de los nucleótidos. Estas moléculas son críticas para el almacenamiento y uso de información genética por el proceso de transcripción y biosíntesis de proteínas[7]. Esta información es protegida por mecanismo de reparación del ADN y propagada por replicación del ADN. Algunos virus tienen un genoma de ARN, por ejemplo el HIV, y utilizan retrotranscripción para crear ADN a partir de su genoma viral de ARN.[12]; estos virus son llamados retrovirus. El ARN en ribozimas como los ribosomas es similar a las enzimas y puede catabolizar reacciones químicas. Nucleósidos individuales son hechos al unir bases nitrogenadas con ribosa. Estas bases son anillos que contienen nitrógeno, clasificado como purina y pirimidinas. Los nucleótidos también actúan como coenzimas en reacciones metabólicas de transferencia en grupo.[13]
Coenzimas
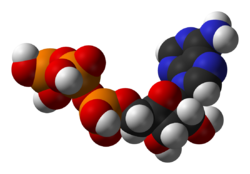
El metabolismo conlleva un gran arraigo de reacciones químicas, pero muchas por mayoría caen en algunos tipos básicos de reacción de transferencia en grupo.[14] Esta química común permite a células utilizar los intermedios metabólicos para logra grupos químicos entre diferentes reacciones.[13] Este grupo de transferencia intermediaria son las coenzimas. Cada clase de reacción de grupo es llevada a cabo por una coenzima particular, que es un substrato para un grupo de enzimas que lo producen, y un grupo de enzimas que lo consumen. Estas coenzimas son, por ende, continuamente hechas, consumidas y luego recicladas.[15]
Referencias
- ↑ Friedrich C (1998). «Physiology and genetics of sulfur-oxidizing bacteria». Adv Microb Physiol 39: 235-89. PMID 9328649.
- ↑ Smith E, Morowitz H (2004). «Universality in intermediary metabolism». Proc Natl Acad Sci U S A 101 (36): 13168-73. PMID 15340153.
- ↑ Ebenhöh O, Heinrich R (2001). «Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems». Bull Math Biol 63 (1): 21-55. PMID 11146883.
- ↑ Meléndez-Hevia E, Waddell T, Cascante M (1996). «The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution». J Mol Evol 43 (3): 293-303. PMID 8703096.
- ↑ Michie K, Löwe J (2006). «Dynamic filaments of the bacterial cytoskeleton». Annu Rev Biochem 75: 467-92. PMID 16756499.
- ↑ Educa.rcanaria.es
- ↑ a b c d Nelson, David L.; Michael M. Cox (2005). Lehninger Principles of Biochemistry. New York: W. H. Freeman and company. p. 841. ISBN 0-7167-4339-6.
- ↑ Fahy E, Subramaniam S, Brown H, Glass C, Merrill A, Murphy R, Raetz C, Russell D, Seyama Y, Shaw W, Shimizu T, Spener F, van Meer G, VanNieuwenhze M, White S, Witztum J, Dennis E (2005). «A comprehensive classification system for lipids». J Lipid Res 46 (5): 839-61. PMID 15722563.
- ↑ «Nomenclature of Lipids». IUPAC-IUB Commission on Biochemical Nomenclature (CBN). Consultado el 8 de marzo de 2007.
- ↑ Hegardt F (1999). «Mitochondrial 3-hydroxy-3-methylglutaryl-CoA synthase: a control enzyme in ketogenesis». Biochem J. 338 (Pt 3): 569-82. PMID 10051425.
- ↑ Raman R, Raguram S, Venkataraman G, Paulson J, Sasisekharan R (2005). «Glycomics: an integrated systems approach to structure-function relationships of glycans». Nat Methods 2 (11): 817-24. PMID 16278650.
- ↑ Sierra S, Kupfer B, Kaiser R (2005). «Basics of the virology of HIV-1 and its replication». J Clin Virol 34 (4): 233-44. PMID 16198625.
- ↑ a b Wimmer M, Rose I (1978). «Mechanisms of enzyme-catalyzed group transfer reactions». Annu Rev Biochem 47: 1031-78. PMID 354490.
- ↑ Mitchell P (1979). «The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems». Eur J Biochem 95 (1): 1-20. PMID 378655.
- ↑ Dimroth P, von Ballmoos C, Meier T (2006). «Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series». EMBO Rep 7 (3): 276-82. PMID 16607397.
