Orthocoronavirinae
| Orthocoronavirinae | ||
|---|---|---|
 | ||
| Taxonomía | ||
| Dominio: | Riboviria | |
| Reino: | Orthornavirae | |
| Filo: | Pisuviricota | |
| Clase: | Pisoniviricetes | |
| Orden: | Nidovirales | |
| Suborden: | Cornidovirineae | |
| Familia: | Coronaviridae | |
| Subfamilia: | Orthocoronavirinae | |
| Clasificación de Baltimore | ||
| Grupo: | IV (Virus ARN monocatenario positivo) | |
| Géneros[1] | ||
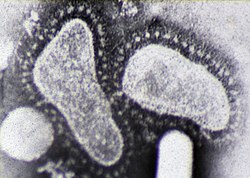
Orthocoronavirinae, comúnmente conocidos como coronavirus, es una subfamilia de virus ARN monocatenario positivos perteneciente a la familia Coronaviridae. Se subdivide en los géneros Alphacoronavirus, Betacoronavirus, Gammacoronavirus y Deltacoronavirus.[2][3] Estos incluyen genogrupos filogenéticamente similares de virus con una nucleocápside de simetría helicoidal con envoltura cuyos viriones pueden medir entre aproximadamente 50 y 200 nm de diámetro. Su material genético es el de mayor tamaño dentro de los virus de ARN, con genomas que van desde los 26 a 32 kilonucleótidos.[4][5] Se les llama coronavirus por la corona de puntas que se ve alrededor de la superficie del virus. Fue descrito por primera vez en 1965.[6]
Los coronavirus pueden infectar aves y mamíferos produciendo una serie de enfermedades respiratorias y digestivas, muchas de ellas letales trayendo como consecuencia serios perjuicios en la avicultura y la ganadería; también pueden infectar al ser humano causando enfermedades que van desde el resfriado común hasta enfermedades más graves, como bronquitis, bronquiolitis, neumonía, el síndrome respiratorio de Oriente Medio (MERS), el síndrome respiratorio agudo grave (SARS) y el COVID-19, entre otras. La mayoría de las personas se infectan con estos virus en algún momento de su vida.[2][7][3][8]
Hasta la fecha se han registrado cuarenta y cinco especies de coronavirus.[1] Varias especies son de reciente investigación[8] debido a que varias cepas particulares no habían sido identificadas previamente en humanos.[9] Existe poca información sobre la transmisión, gravedad e impacto clínico[9] y no existen tratamientos aprobados hasta la fecha,[8] sin embargo se pueden tratar varios de los síntomas, las opciones terapéuticas dependen del estado clínico de cada paciente.[8]
El género Alphacoronavirus —anteriormente conocido como Coronavirus grupo 1 (CoV-1)— incluye los subgrupos 1a y 1b, cuyos integrantes más representativos son el coronavirus humano 229E (HCoV-229E) y HCoV-NL63, así como la nueva especie alfacoronavirus 1 —incluyendo virus de la gastroenteritis transmisible porcina (TGEV)—, respectivamente. El género Betacoronavirus —anteriormente Betacoronavirus grupo 2 (Cov-2)— incluye varios subgrupos. Los más prominentes (subgrupos 2a y 2b) tienen como especies tipo las especies de coronavirus murino —incluido el virus de la hepatitis de ratón (MHV)– y el SARS-CoV, respectivamente. Los géneros Alphacoronavirus y Betacoronavirus provienen del pool genético que tiene a murciélagos como huésped. El género Gammacoronavirus incluye todos los coronavirus aviares identificados hasta 2009.[10][11]
Historia natural[editar]
El ancestro común más reciente del coronavirus se ha encontrado en el siglo IX a. C. Estudios realizados durante 1990 lograron datar los ancestros comunes más recientes de los géneros:[cita requerida]
- Betacoronavirus: 3300 a. C.
- Deltacoronavirus: 3000 a. C.
- Gammacoronavirus: 2800 a. C.
- Alphacoronavirus: 2400 a. C.
De acuerdo a estos estudios, en ese entonces el factor principal de la fuente del coronavirus era la sangre caliente, particularmente de los murciélagos y pájaros. El coronavirus bovino se separó de la especie equina coronavirus al final del siglo XVIII. El coronavirus bovino y el coronavirus humano OC43 se separaron en 1899. Otra estimación sugiere que el coronavirus humano OC43 divergió del coronavirus bovino en 1890. El coronavirus bovino y el canino respiratorio con el coronavirus divergieron de un ancestro común en 1951. El ancestro común más reciente del coronavirus humano OC43 ha sido fechado en la década de 1950. El síndrome respiratorio coronavirus de Oriente Medio, aunque relacionado con varias especies de murciélagos, parece haber divergido de estos hace varios siglos. El coronavirus de murciélago está más estrechamente relacionado con el coronavirus del SARS, del que se separó en 1986.[12][13][14][15][16][17][18][19][20][21][22]
Estructura[editar]

Las partes que conforman la estructura general de los coronavirus son, como en todos los virus animales, la envoltura y la nucleocápside. En el caso de los coronavirus, en la envoltura se encuentra una glucoproteína de membrana (M) de 20 a 35 kDa, que forma una matriz en contacto con la nucleocápside. Además se encuentra en la envoltura la glucoproteína S, de 180 a 220 kDa,[23] que forma las espículas, espigas o peplómeros responsables de la adhesión a la célula huésped. En el caso específico del coronavirus SARS, en sus espículas un dominio de unión para receptores definidos dirigen la adherencia del virus a su receptor celular, la enzima convertidora de angiotensina 2 (ACE-2).[24] Algunos coronavirus (específicamente los miembros de Betacoronavirus subgrupo A, también llamado subgénero Embecovirus[25]) tienen también en la superficie una proteína adicional más corta llamada esterasa-hemaglutinina.[26]
Replicación[editar]
La replicación de los coronavirus comienza con la entrada en la célula, momento en que pierde su envoltura, y el genoma de ARN se libera en el citoplasma. El genoma del coronavirus tiene un caperuza metilada en el extremo 5' (extremo cap'), y una cola poliadenilada (poly A) en el extremo 3', dándole un gran parecido al ARN mensajero eucariota. Esto permite que al ARN se le adhieran los ribosomas citoplasmáticos para su traducción. Los coronavirus tienen también una proteína replicasa codificada en su código genético, que le permite generar nuevas copias de su ARN sin necesidad de transcribirse a ADN, usando los recursos de la célula huésped. Esta replicasa es la primera proteína que se sintetiza ya que una vez que el gen que codifica la replicasa es traducido (síntesis proteica), el proceso se detiene por un codón de parada.[27] Esto se conoce como una transcripción anidada. Cuando el transcrito de ARNm solo codifica un gen, se conoce como monocistrónico. El genoma de ARN se replica a cadena negativa y de esta se forman copias positivas de la que se traduce una larga poliproteína, que deberá ser escindida en las distintas proteínas funcionales del virus. Los coronavirus tienen para ello una proteasa denominada Mpro o 3CLpro[28] que corta la poliproteína para dar lugar a las proteínas víricas (maduración de la poliproteína). Esta es una estrategia vírica para la economía genética, ya que le permite codificar un buen número de proteínas con un número pequeño de transcritos a la vez que mejora la tasa de fallos durante la ejecución de la ARN polimerasa.[29][27] Dicha proteasa es un objetivo de fármacos para impedir la replicación del virus.[30]
Humanos[editar]
Los coronavirus humanos fueron descritos por primera vez en la década de 1960 en cavidades nasales de pacientes con un resfriado común. Estos virus fueron nombrados posteriormente coronavirus humano 229E y OC43. Otros dos miembros de esta familia han sido identificados, el HCoV-NL63 en 2004 y HKU1 en 2005. Los cuales circulan globalmente en la población humana y causan aproximadamente un tercio de los casos de resfriado común. Al igual que otros tipos de virus pueden causar enfermedades más graves del sistema respiratorio como bronquitis o neumonía especialmente en personas con factores de riesgo, ancianos, niños y pacientes inmunodeprimidos. Además de afecciones respiratorias también pueden causar enfermedades intestinales y neurológicas.[31]
Existen registros de siete cepas de coronavirus relacionados con enfermedades respiratorias en humanos (HCoV):
- Coronavirus humano 229E
- Coronavirus humano OC43
- SARS-CoV
- Coronavirus humano NL63
- Coronavirus humano HKU1
- Coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV).
- SARS-CoV-2 (COVID-19).[32][33]
Después de la publicación del perfil de los brotes de SARS en 2003, resucitó entre los virólogos un interés por los coronavirus. Durante muchos años, los científicos sabían de solo dos coronavirus humanos (HCoV-229E y OC43-HCoV). El descubrimiento de SARS-CoV añadió un tercer coronavirus humano. A finales de 2004, tres laboratorios de investigación independientes informaron del descubrimiento de un cuarto coronavirus humano, nombrado NL63, NL, y coronavirus de New Haven simultáneamente por varios grupos de investigación. Los tres laboratorios siguen discutiendo sobre cuál de ellos descubrió el virus en primer lugar y por tanto tiene derecho a nombrarlo. A principios de 2005, un equipo de investigación de la Universidad de Hong Kong informó del hallazgo de un quinto coronavirus humano en dos pacientes con neumonía. Lo llamaron coronavirus humano HKU1. El brote de neumonía 2019-20 en Wuhan, China, llevó al hallazgo de un coronavirus nuevo, catalogado como 2019-nCoV por la OMS.[34][35][32][33]
Síndrome respiratorio agudo grave[editar]
En 2003, tras el brote del SARS (síndrome respiratorio agudo grave), que había comenzado en el año 2002 en Asia, y luego en otras partes del mundo, la Organización Mundial de la Salud (OMS) emitió un comunicado de prensa indicando que un coronavirus de nueva identificación por parte una serie de laboratorios era el agente causante del SARS. El virus fue nombrado oficialmente como coronavirus del SARS (SARS-CoV). Más de 8000 personas resultaron infectadas, alrededor del 10% de los cuales murieron.[26]
Síndrome respiratorio de Medio Oriente[editar]
En septiembre de 2012, se identificó un nuevo tipo de coronavirus, llamado inicialmente coronavirus nuevo 2012, y ahora con el nombre oficial coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV). La Organización Mundial de la Salud emitió una alerta mundial poco después. La actualización de la OMS el 28 de septiembre de 2012 declaró que no parecía que el virus se transmitiese fácilmente de persona a persona. Sin embargo, el 12 de mayo de 2013, un caso de transmisión de humano a humano en Francia fue confirmado por el Ministerio de Asuntos Sociales y de Salud de Francia. Además, los casos de transmisión de humano a humano han sido reportados por el Ministerio de Salud de Túnez. Dos casos confirmados parecen haber contraído la enfermedad de su difunto padre, quienes se enfermaron después de una visita a Catar y Arabia Saudita. A pesar de esto, parece que el virus tiene problemas para la difusión de humano a humano, ya que la mayoría de las personas que están infectadas no transmiten el virus.[36][37][38][39][40][41]
Para el 30 de octubre de 2013, había en Arabia Saudita 124 casos y 52 muertes. Después de que el Centro Médico Erasmus neerlandés secuenció el virus, le dio un nombre nuevo, coronavirus humano-Centro Médico Erasmus (HCoV-CEM). El nombre final para el virus es coronavirus del síndrome respiratorio de Oriente Medio (MERS-CoV). En mayo de 2014 se registraron los dos únicos casos de infección con MERS-CoV en los Estados Unidos, ocurrieron en trabajadores de la salud que trabajaron en Arabia Saudita y luego se desplazaron a Estados Unidos. Ambos individuos fueron hospitalizados temporalmente y luego dados de alta. En mayo de 2015, un brote de MERS-CoV se produjo en Corea del Sur, cuando un hombre que había viajado a Oriente Medio, visitó 4 hospitales diferentes en el área de Seúl para tratar su enfermedad. Esto provocó uno de los mayores brotes de MERS-CoV fuera del Medio Oriente. En diciembre de 2019, 2.468 casos de infección MERS-CoV habían sido confirmados por medio de pruebas de laboratorio, casos de los cuales 851 fueron mortales, una tasa de mortalidad de aproximadamente el 34,5%.[42][43][44][45]
SARS-CoV-2[editar]
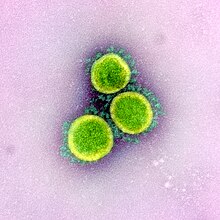
El coronavirus de tipo 2 causante del síndrome respiratorio agudo severo,[46] abreviado SARS-CoV-2 (del inglés severe acute respiratory syndrome coronavirus 2)[47] o SRAS-CoV-2,[46] es un tipo de coronavirus causante de la enfermedad por coronavirus de 2019,[48][49][50] cuya expansión mundial provocó la pandemia de COVID-19. Inicialmente fue llamado 2019-nCoV (en inglés: 2019-novel coronavirus, ‘nuevo coronavirus de 2019’) y también, ocasionalmente, HCoV-19 (en inglés: human coronavirus 2019).[51][52] Se descubrió y se aisló por primera vez en Wuhan, China. Tiene un origen zoonótico, es decir, que se transmitió de un huésped animal a uno humano.[53]
Es un clado dentro de la familia de los Coronaviridae, género Betacoronavirus, subgénero Sarbecovirus, especie virus SARS[54] (virus relacionado con el síndrome respiratorio agudo severo o grave).[55]
El genoma del virus está formado por una sola cadena de ARN, y se clasifica como un virus ARN monocatenario positivo. Su secuencia genética se ha aislado a partir de una muestra obtenida de un paciente afectado por neumonía en la ciudad china de Wuhan, aunque la falta de experimento de control doble ciego en la técnica de secuenciación publicada puede poner en cuestionamiento la validez científica de la técnica.[56][57][58][59][60][61] Se detectó por primera vez el 12 de noviembre de 2019. Puede producir el contagio de una persona a otra mediante las gotas de saliva expulsadas a través de la tos y el estornudo o al espirar (véase gotitas de Flügge).[62][63] Puede provocar enfermedad respiratoria aguda y neumonía grave en los seres humanos.[64]
Aunque previamente no había ningún tratamiento específico aprobado oficialmente, ya se habían desarrollado algunos antivirales existentes, así como el tratamiento con plasma convaleciente y la dexametasona, que parecen tener una mayor eficacia en el manejo de los síntomas o que parecen acortar el periodo de recuperación en poblaciones especiales.[65][66] En diciembre de 2020 comenzó una campaña de vacunación con las primeras vacunas experimentales autorizadas en emergencia, que se prolongó y mantuvo durante 2021 y 2022.[cita requerida] Fue Pfizer - BioNTech, con la vacuna Comirnaty, los laboratorios pioneros en patentar una vacuna[67] y posteriormente, los laboratorios Moderna y AstraZeneca se unieron a esta carrera por la vacuna.[68]Veterinaria[editar]
Los coronavirus han sido reconocidos como causantes de condiciones patológicas en veterinaria desde principios de 1970. A excepción de la bronquitis infecciosa aviar, las principales enfermedades relacionadas tienen principalmente una ubicación intestinal.
Los coronavirus infectan principalmente el tracto respiratorio y gastrointestinal superior de mamíferos y aves. Actualmente se conocen siete cepas del coronavirus que infectan a los humanos. Se cree que los coronavirus causan un porcentaje significativo de todos los resfriados comunes en personas adultas y niños. Los coronavirus causan resfriados con síntomas importantes; por ejemplo, fiebre, inflamación de las adenoides de la garganta, en los seres humanos principalmente en las temporadas de invierno y primavera temprana. Los coronavirus puede causar neumonía, ya sea neumonía viral directa o una neumonía bacteriana secundaria, y la bronquitis, ya sea bronquitis viral directa o una bronquitis bacteriana secundaria. El coronavirus humano más conocido fue descubierto en 2003, SARS-CoV que causa el síndrome respiratorio agudo grave (SARS), tiene una patogénesis única porque causa que infecciones de las vías respiratorias tanto superior como inferior. La importancia económica y el impacto de los coronavirus como agentes causantes del resfriado común son difíciles de evaluar debido a que, a diferencia de los rinovirus (otro virus del resfriado común), los coronavirus humanos son difíciles de cultivar en el laboratorio.[69][70]
Los coronavirus también causan una serie de enfermedades en los animales de granja y mascotas domesticadas, algunos de los cuales pueden ser graves y son una amenaza para la industria agrícola. En los pollos, el virus de la bronquitis infecciosa (IBV), es un coronavirus que afecta no solo las vías respiratorias, sino también el tracto urogenital. El virus puede propagarse a los diferentes órganos del cuerpo de la gallina. Los que tienen consecuencias económicamente significativas en los animales de granja incluyen el coronavirus porcino (coronavirus de la gastroenteritis transmisible, TGE) y el coronavirus bovino, el cual causa diarrea en los animales jóvenes. Coronavirus felino: existen dos formas, el coronavirus entérico felino es un patógeno de importancia clínica menor, pero la mutación espontánea de este virus puede provocar una peritonitis infecciosa felina (FIP), una enfermedad asociada con una alta mortalidad. Del mismo modo, hay dos tipos de coronavirus que infectan a los hurones: el coronavirus entérico de hurón causa un síndrome gastrointestinal conocido como epizoótica catarral enteritis (ECE), y una versión sistémica más letal del virus (como FIP en los gatos), conocido en hurones como coronavirus sistémico de hurón (FSC). Hay dos tipos de coronavirus canino (CoVC), uno que causa la enfermedad gastrointestinal leve y uno que causa enfermedad respiratoria. El virus de la hepatitis del ratón (MHV) es un coronavirus que causa una enfermedad epidémica murina con una mortalidad alta, especialmente entre las colonias de ratones de laboratorio.[71][72][73] El virus de la sialodacrioadenitis (sialo saliva, dacrio lágrima) es un coronavirus altamente infeccioso de ratas de laboratorio, que puede transmitirse entre individuos por contacto directo o indirectamente por las secreciones en aerosol. Las infecciones agudas tienen una alta morbilidad y tropismo para las glándulas salivales, lacrimales y harderiana.[74]
Uno relacionado con el coronavirus murciélago Rinolophus HKU2 llamado coronavirus del síndrome de diarrea aguda porcina (SADS-CoV) (del inglés swine acute diarrhea syndrome) causa diarrea en cerdos.[75]
Antes del descubrimiento de SARS-CoV, MHV había sido estudiado tanto in vivo como in vitro, así como a nivel molecular. Algunas cepas de MHV causaron una encefalitis desmielinizante progresiva en ratones a los que se ha utilizado como modelo murino para la esclerosis múltiple. Importantes esfuerzos de investigación se han centrado en dilucidar la patogénesis viral de estos coronavirus de animales, especialmente por los virólogos interesados en las enfermedades zoonóticas y veterinarios.[76]

[editar]
- Virus de la bronquitis infecciosa (IBV) causas infecciosa aviar bronquitis.
- Porcino coronavirus (transmisible gastroenteritis coronavirus de cerdos, TGEV).
- Coronavirus bovino (BCV), responsable de la profusa enteritis grave en los terneros jóvenes.
- Coronavirus felino (FCoV) provoca enteritis leve en los gatos, así como graves peritonitis infecciosa felina (otras variantes del mismo virus).
- Los dos tipos de canino coronavirus (CoVC) (una enteritis causando, la otra que se encuentra en las enfermedades respiratorias).
- Coronavirus aviar provoca enteritis en aves.
- Ferret entérica coronavirus provoca enteritis catarral epizoótica en hurones.
- Ferret sistémica coronavirus causa FIP-como el síndrome sistémico en hurones.
- Pantrópicas coronavirus canino.
- Coronavirus canino
Otra enfermedad veterinaria nueva, virus de la diarrea epidémica porcina (PED o PEDV), ha surgido en todo el mundo. Su importancia económica es aún poco clara, pero muestra una alta mortalidad en los lechones.[78][79][80]
Taxonomía[editar]

Los géneros, subgéneros y especies incluidas en Orthocoronavirinae son:[81]
- Alphacoronavirus
- Colacovirus
- Coronavirus de murciélago CDPHE15
- Decacovirus
- Coronavirus de murciélago HKU10
- Rhinolophus ferrumequinum alphacoronavirus HuB-2013
- Duvinacovirus
- Coronavirus humano 229E
- Luchacovirus
- Coronavirus de rata Lucheng Rn
- Minacovirus
- Hurón coronavirus
- Mink coronavirus 1
- Minunacovirus
- Miniopterus murciélago coronavirus 1
- Miniopterus murciélago coronavirus HKU8
- Myotacovirus
- Myotis ricketti alphacoronavirus Sax-2011
- Nyctacovirus
- Nyctalus velutinus alphacoronavirus SC-2013
- Pedacovirus
- Virus de la diarrea epidémica porcina
- Scotvilus murciélago coronavirus 512
- Rinacovirus
- Coronavirus murciélago Rhinolophus HKU2
- Setracovirus
- Coronavirus humano NL63
- Cepa de coronavirus de murciélago relacionada con NL63 BtKYNL63-9b
- Tegacovirus
- Alphacoronavirus 1 - especie tipo
- Colacovirus
- Betacoronavirus
- Embevirus
- Betacoronavirus 1
- Coronavirus Rattus de China HKU24
- Coronavirus humano HKU1
- Coronavirus murino - especie tipo
- Hibecovirus
- Bat Hp-betacoronavirus Zhejiang2013
- Merbecovirus
- Nobecovirus
- Rousettus murciélago coronavirus GCCDC1
- Coronavirus murciélago Rousettus HKU9
- Sarbecovirus
- Embevirus
- Deltacoronavirus
- Andecovirus
- Coronavirus Wigeon HKU20
- Buldecovirus
- Bulbul coronavirus HKU11 - especie tipo
- Coronavirus HKU15
- Coronavirus Munia HKU13
- Coronavirus de ojo blanco HKU16
- Herdecovirus
- Coronavirus de garza nocturna HKU19
- Moordecovirus
- Coronavirus de la polla de agua común HKU21
- Andecovirus
- Gammacoronavirus
- Cegacovirus
- Igacovirus
- Coronavirus aviar - especie tipo
Véase también[editar]
- Virus ARN
- Coronavirus humano OC43
- COVID-19
- SARS-CoV-2
- Pandemia de COVID-19
- Pandemia de enfermedad por coronavirus de 2020 en España
Bibliografía[editar]
- Thiel V, ed. (2007). Coronaviruses: Molecular and Cellular Biology (1st edición). Caister Academic Press. ISBN 978-1-904455-16-5.
Referencias[editar]
- ↑ a b «International Committee on Taxonomy of Viruses (ICTV)». talk.ictvonline.org (en inglés). Consultado el 6 de febrero de 2020.
- ↑ a b Schoeman", Dewald; Fielding, Burtram C (mayo de 2019). «Coronavirus envelope protein: current knowledge» [Proteína del envoltorio del coronavirus: conocimiento actual]. Virol J (en inglés) (BioMed Central) 16: 69. PMID 31133031. doi:10.1186/s12985-019-1182-0. Consultado el 22 de marzo de 2020.
- ↑ a b Cui, Jie; Li, Fang; Shi, Zheng-Li (diciembre de 2018). «Origin and evolution of pathogenic coronaviruses» [Origen y evolución de los coronavirus patógenos]. Nature Reviews Microbiology (en inglés) (Springer Nature Limited) 17: 181-192. Consultado el 22 de marzo de 2020.
- ↑ Woo, Patrick C. Y.; Huang, Yi; Lau, Susanna K. P.; Yuen, Kwok-Yung (24 de agosto de 2010). «Coronavirus Genomics and Bioinformatics Analysis». Viruses 2 (8): 1804-1820. ISSN 1999-4915. PMC 3185738. PMID 21994708. doi:10.3390/v2081803.
- ↑ Chen, Nanshan; Zhou, Min; Dong, Xuan; Qu, Jieming; Gong, Fengyun; Han, Yang; et al. (30 de enero de 2020). «Epidemiological and clinical characteristics of 99 cases of 2019 novel coronavirus pneumonia in Wuhan, China: a descriptive study». The Lancet (15 de febrero de 2020) 395 (10223): 507-513. doi:10.1016/S0140-6736(20)30211-7.
- ↑ «Cultivation of a Novel Type of Common-cold Virus in Organ Cultures».
- ↑ Tortorici, M. Alejandra; Veesler, David (2019). «Chapter Four - Structural insights into coronavirus entry» [Capítulo cuatro - Información estructural del coronavirus]. Advances in Virus Research (en inglés) (Elsevier Inc) 105: 93-116. doi:10.1016/bs.aivir.2019.08.002. Consultado el 21 de marzo de 2020.
- ↑ a b c d «Preguntas frecuentes sobre los nuevos coronavirus». www.who.int. Consultado el 25 de enero de 2020.
- ↑ a b «OMS | Infecciones por coronavirus». WHO. Consultado el 25 de enero de 2020.
- ↑ Lai, M. M. C.; Holmes, K. V. (2001). «Coronaviridae». En Knipe, D. M.; Howley, P. M.; Griffin, M. A., eds. Fields virology (4th edición). Philadelphia: Lippincott Williams & Wilkins. pp. 1163-85.
- ↑ Woo, P. C. Y.; Lau, S. K. P.; Lam, C. S. F.; Lau, C. C. Y.; Tsang, A. K. L.; Lau, J. H. N.; Bai, R.; Teng, J. L. L. et al. (2012). «Discovery of Seven Novel Mammalian and Avian Coronaviruses in the Genus Deltacoronavirus Supports Bat Coronaviruses as the Gene Source of Alphacoronavirus and Betacoronavirus and Avian Coronaviruses as the Gene Source of Gammacoronavirus and Deltacoronavirus». Journal of Virology 86 (7): 3995-4008. PMC 3302495. PMID 22278237. doi:10.1128/JVI.06540-11.
- ↑ «Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavirus». Journal of Virology 86 (7): 3995-4008. April 2012. PMC 3302495. PMID 22278237. doi:10.1128/JVI.06540-11.
- ↑ «Evolutionary dynamics of bovine coronaviruses: natural selection pattern of the spike gene implies adaptive evolution of the strains». The Journal of General Virology 94 (Pt 9): 2036-2049. September 2013. PMID 23804565. doi:10.1099/vir.0.054940-0.
- ↑ «Complete genomic sequence of human coronavirus OC43: molecular clock analysis suggests a relatively recent zoonotic coronavirus transmission event». Journal of Virology 79 (3): 1595-1604. February 2005. PMC 544107. PMID 15650185. doi:10.1128/jvi.79.3.1595-1604.2005.
- ↑ «Genetic characterization of Betacoronavirus lineage C viruses in bats reveals marked sequence divergence in the spike protein of pipistrellus bat coronavirus HKU5 in Japanese pipistrelle: implications for the origin of the novel Middle East respiratory syndrome coronavirus». Journal of Virology 87 (15): 8638-8650. August 2013. PMC 3719811. PMID 23720729. doi:10.1128/JVI.01055-13.
- ↑ «Molecular epidemiology of human coronavirus OC43 reveals evolution of different genotypes over time and recent emergence of a novel genotype due to natural recombination». Journal of Virology 85 (21): 11325-11337. November 2011. PMC 3194943. PMID 21849456. doi:10.1128/JVI.05512-11.
- ↑ «A case for the ancient origin of coronaviruses». Journal of Virology 87 (12): 7039-7045. June 2013. PMC 3676139. PMID 23596293. doi:10.1128/JVI.03273-12.
- ↑ «Evolutionary insights into the ecology of coronaviruses». Journal of Virology 81 (8): 4012-4020. April 2007. PMC 1866124. PMID 17267506. doi:10.1128/jvi.02605-06.
- ↑ «SARS-Coronavirus ancestor's foot-prints in South-East Asian bat colonies and the refuge theory». Infection, Genetics and Evolution 11 (7): 1690-702. October 2011. PMID 21763784. doi:10.1016/j.meegid.2011.06.021.
- ↑ «Evolutionary relationships between bat coronaviruses and their hosts». Emerging Infectious Diseases 13 (10): 1526-1532. October 2007. PMC 2851503. PMID 18258002. doi:10.3201/eid1310.070448.
- ↑ «Identification and characterization of a novel alpaca respiratory coronavirus most closely related to the human coronavirus 229E». Viruses 4 (12): 3689-3700. December 2012. PMC 3528286. PMID 23235471. doi:10.3390/v4123689.
- ↑ «Evidence supporting a zoonotic origin of human coronavirus strain NL63». Journal of Virology 86 (23): 12816-12825. December 2012. PMC 3497669. PMID 22993147. doi:10.1128/JVI.00906-12.
- ↑ «Coronavirus | Microbiología médica, 26e | AccessMedicina | McGraw Hill Medical». accessmedicina.mhmedical.com. Archivado desde el original el 21 de marzo de 2020. Consultado el 21 de marzo de 2020.
- ↑ Li, Fang, et. al. (2005). «Structure of SARS Coronavirus Spike Receptor-Binding Domain Complexed with Receptor». Science 309: 1864-1868.
- ↑ ICTV, julio de 2018
- ↑ a b «Structure of SARS coronavirus spike receptor-binding domain complexed with receptor». Science 309 (5742): 1864-1868. September 2005. Bibcode:2005Sci...309.1864L. PMID 16166518. doi:10.1126/science.1116480.
- ↑ a b «Coronaviruses: an overview of their replication and pathogenesis». Methods in Molecular Biology 1282. 2015. pp. 1-23. ISBN 978-1-4939-2437-0. PMC 4369385. PMID 25720466. doi:10.1007/978-1-4939-2438-7_1.
- ↑ Zhang, Linlin; Lin, Daizong; Sun, Xinyuanyuan; Curth, Ute; Drosten, Christian; Sauerhering, Lucie; Becker, Stephan; Rox, Katharina et al. (24 abr. 2020). «Crystal structure of SARS-CoV-2 main protease provides a basis for design of improved α-ketoamide inhibitors». Science. doi:10.1126/science.abb3405 – via www.science.org.
- ↑ «Homology-based identification of a mutation in the coronavirus RNA-dependent RNA polymerase that confers resistance to multiple mutagens». Journal of Virology 90 (16): 7415-7428. August 2016. PMC 4984655. PMID 27279608. doi:10.1128/JVI.00080-16.
- ↑ Anand, Kanchan; Ziebuhr, John; Wadhwani, Parvesh; Mesters, Jeroen R.; Hilgenfeld, Rolf (13 jun. 2003). «Coronavirus Main Proteinase (3CLpro) Structure: Basis for Design of Anti-SARS Drugs». Science. doi:10.1126/science.1085658 – via www.science.org.
- ↑ a b Lim, Yvonne Xinyi; Ng, Yan Ling; Tam, James P.; Liu, Ding Xiang (25 de julio de 2016). «Human Coronaviruses: A Review of Virus–Host Interactions». Diseases 4 (3): 26. ISSN 2079-9721. PMC 5456285. PMID 28933406. doi:10.3390/diseases4030026. «See Table 1.»
- ↑ a b «Laboratory testing of human suspected cases of novel coronavirus (nCoV) infection (2020)». Laboratory testing of human suspected cases of novel coronavirus. 10 de enero de 2020. Consultado el 20 de enero de 2020.
- ↑ a b «Novel Coronavirus 2019 (nCoV-2019), Wuhan, China». Archivado desde el original el 14 de enero de 2020. Consultado el 14 de enero de 2020.
- ↑ «Identification of a new human coronavirus». Nature Medicine 10 (4): 368-373. April 2004. PMID 15034574. doi:10.1038/nm1024. Archivado desde el original el 21 de agosto de 2007. Consultado el 12 de septiembre de 2019.
- ↑ «WHO Statement Regarding Cluster of Pneumonia Cases in Wuhan, China». www.who.int (en inglés). 9 de enero de 2020. Archivado desde el original el 14 de enero de 2020. Consultado el 10 de enero de 2020.
- ↑ CDC (2 de agosto de 2019). «MERS Transmission». Centers for Disease Control and Prevention (en inglés estadounidense). Archivado desde el original el 7 de diciembre de 2019. Consultado el 10 de diciembre de 2019.
- ↑ Nouveau coronavirus – Point de situation: Un nouveau cas d’infection confirmé Archivado el 8 de junio de 2013 en Wayback Machine. (Novel coronavirus – Status report: A new case of confirmed infection) 12 May 2013 social-sante.gouv.fr
- ↑ Doucleef, Michaeleen (26 de septiembre de 2012). «Scientists Go Deep On Genes Of SARS-Like Virus». Associated Press. Archivado desde el original el 27 de septiembre de 2012. Consultado el 27 de septiembre de 2012.
- ↑ Falco, Miriam (24 de septiembre de 2012). «New SARS-like virus poses medical mystery». CNN Health. Archivado desde el original el 1 de noviembre de 2013. Consultado el 16 de marzo de 2013.
- ↑ «New SARS-like virus found in Middle East». Al-Jazeera. 24 de septiembre de 2012. Archivado desde el original el 9 de marzo de 2013. Consultado el 16 de marzo de 2013.
- ↑ Kelland, Kate (28 de septiembre de 2012). «New virus not spreading easily between people: WHO». Reuters. Archivado desde el original el 24 de noviembre de 2012. Consultado el 16 de marzo de 2013.
- ↑ «Novel coronavirus infection - update». World Health Association. 22 de mayo de 2013. Archivado desde el original el 7 de junio de 2013. Consultado el 23 de mayo de 2013.
- ↑ CDC (2 de agosto de 2019). «MERS in the U.S.». Centers for Disease Control and Prevention (en inglés estadounidense). Archivado desde el original el 15 de diciembre de 2019. Consultado el 10 de diciembre de 2019.
- ↑ Sang-Hun, Choe (8 de junio de 2015). «MERS Virus's Path: One Man, Many South Korean Hospitals». The New York Times. Archivado desde el original el 15 de julio de 2017. Consultado el 1 de marzo de 2017.
- ↑ «Middle East respiratory syndrome coronavirus (MERS-CoV)». WHO. Archivado desde el original el 18 de octubre de 2019. Consultado el 10 de diciembre de 2019.
- ↑ a b «Los nombres de la enfermedad por coronavirus (COVID-19) y del virus que la causa». who.int. Organización Mundial de la Salud. Consultado el 3 de noviembre de 2020.
- ↑ «Enfermedad del coronavirus 2019 (COVID-19). urlarchivo=https://web.archive.org/web/20200318025404/https://www.cdc.gov/coronavirus/2019-ncov/faq-sp.html».
- ↑ «Coronavirus, claves de escritura». Fundéu BBVA. 29 de enero de 2020. Archivado desde el original el 30 de enero de 2020. Consultado el 25 de febrero de 2020.
- ↑ Organización Mundial de la Salud, ed. (11 de febrero de 2020). «WHO Director-General's remarks at the media briefing on 2019-nCoV on 11 February 2020». www.who.int (en inglés). Archivado desde el original el 12 de febrero de 2020. Consultado el 11 de febrero de 2020.
- ↑ Gorbalenya, A.E.; Baker, S.C.; Baric, R.S.; de Groot, R.J.; Drosten, C.; Gulyaeva, A.A.; Haagmans, B.L.; Lauber, C.; Leontovich, A.M.; Neuman, B.M.; Penzar, D.; Poon, L. L. M.; Samborskiy, D.; Sidorov, I. A.; Sola, I.; Ziebuhr, J. «Severe acute respiratory syndrome-related coronavirus: The species and its viruses – a statement of the Coronavirus Study Group». bioRxiv (en inglés). doi:10.1101/2020.02.07.937862. Archivado desde el original el 11 de febrero de 2020. Consultado el 11 de febrero de 2020.
- ↑ Jiang, Shibo; Shi, Zhengli; Shu, Yuelong; Song, Jingdong; Gao, George F; Tan, Wenjie; Guo, Deyin (de marzo de 2020). «A distinct name is needed for the new coronavirus». The Lancet (en inglés) 395 (10228): 949. PMC 7124603. PMID 32087125. doi:10.1016/S0140-6736(20)30419-0. Consultado el 19 de septiembre de 2020.
- ↑ Wong, Gary; Bi, Yu-Hai; Wang, Qi-Hui; Chen, Xin-Wen; Zhang, Zhi-Gang; Yao, Yong-Gang (18 de mayo de 2020). «Zoonotic origins of human coronavirus 2019 (HCoV-19 / SARS-CoV-2): why is this work important?». Zoological Research (en inglés) 41 (3): 213-219. PMC 7231470. PMID 32314559. doi:10.24272/j.issn.2095-8137.2020.031. Consultado el 19 de septiembre de 2020.
- ↑ Zhou, Peng; Yang, Xing-Lou; Wang, Xian-Guang; Hu, Ben; Zhang, Lei; Zhang, Wei; Si, Hao-Rui; Zhu, Yan; Li, Bei; Huang, Chao-Lin; Chen, Hui-Dong; Chen, Jing; Luo, Yun; Guo, Hua; Jiang, Ren-Di; Liu, Mei-Qin; Chen, Ying; Shen, Xu-Rui; Wang, Xi; Zheng, Xiao-Shuang; Zhao, Kai; Chen, Quan-Jiao; Deng, Fei; Liu, Lin-Lin; Yan, Bing; Zhan, Fa-Xian; Wang, Yan-Yi; Xiao, Gengfu; Shi, Zheng-Li (23 de enero de 2020). «Discovery of a novel coronavirus associated with the recent pneumonia outbreak in humans and its potential bat origin». bioRxiv (en inglés): 2020.01.22.914952. doi:10.1101/2020.01.22.914952. Archivado desde el original el 24 de enero de 2020. Consultado el 5 de febrero de 2020.
- ↑ Rehman, Saif Ur; Shafique, Laiba; Ihsan, Awais; Liu, Qingyou (23 de marzo de 2020). «Evolutionary Trajectory for the Emergence of Novel Coronavirus SARS-CoV-2». Pathogens (Basel, Switzerland) 9 (3). ISSN 2076-0817. PMC 7157669. PMID 32210130. doi:10.3390/pathogens9030240. Consultado el 25 de abril de 2021.
- ↑ «Coronavirus y síndrome respiratorio agudo severo».
- ↑ «A new coronavirus associated with human respiratory disease in China».
- ↑ «Novel coronavirus (2019-nCoV), Wuhan, China». Centers for Disease Control and Prevention. 10 de enero de 2020. Archivado desde el original el 11 de enero de 2020. Consultado el 16 de enero de 2020.
- ↑ Zhang, Y.-Z., et al. (12 de enero de 2020). Centro Nacional para la Información Biotecnológica, EE. UU., ed. Wuhan seafood market pneumonia virus isolate Wuhan-Hu-1, complete genome (en inglés). Bethesda MD. p. GenBank. Archivado desde el original el 13 de enero de 2020. Consultado el 13 de enero de 2020.
- ↑ «中国疾病预防控制中心». www.chinacdc.cn. Archivado desde el original el 6 de febrero de 2020. Consultado el 9 de enero de 2020.
- ↑ Xinhua / huaxia |, ed. (9 de enero de 2020). «New-type coronavirus causes pneumonia in Wuhan: expert». www.xinhuanet.com. Archivado desde el original el 9 de enero de 2020. Consultado el 9 de enero de 2020.
- ↑ «CoV2020». platform.gisaid.org. Consultado el 12 de enero de 2020 (sin acceso público).
- ↑ European Centre for Disease Prevention and Control (ed.). «Q & A on novel coronavirus» (en inglés). Archivado desde el original el 4 de febrero de 2020. Consultado el 11 de febrero de 2020.
- ↑ Parker-Pope, Tara (21 abr. 2020). «¿El virus está en mi ropa? ¿En mis zapatos? ¿En mi pelo? ¿En mi periódico?». New York Times. Consultado el Domingo, 24 de mayo del 20202 – via NYTimes.com.
- ↑ Prompetchara, Eakachai; Ketloy, Chutitorn; Palaga, Tanapat (2020). «Immune responses in COVID-19 and potential vaccines:Lessons learned from SARS and MERS epidemic». Asian Pacific Journal of Allergy and Immunology (en inglés) (38): 1-9. doi:10.12932/AP-200220-0772.
- ↑ Nebehay, Stephanie. «WHO says new China coronavirus could spread, warns hospitals worldwide» (en inglés). Archivado desde el original el 14 de enero de 2020. Consultado el 19 de enero de 2020.
- ↑ «COVID-19, SARS CoV-2». The Sanford Guide to Antimicrobial Therapy (en inglés). Editorial Board. Consultado el 18 de agosto de 2020.
- ↑ «La OMS publica su primera validación para uso en emergencias de una vacuna contra la COVID-19 y hace hincapié en la necesidad de un acceso mundial equitativo». www.who.int. Consultado el 18 de mayo de 2021.
- ↑ «Vacunas contra la COVID-19». www.who.int. Consultado el 18 de mayo de 2021.
- ↑ «Prevalence and genetic diversity analysis of human coronaviruses among cross-border children». Virology Journal (en inglés) 14 (1): 230. November 2017. PMC 5700739. PMID 29166910. doi:10.1186/s12985-017-0896-0.
- ↑ «Healthcare-associated atypical pneumonia». Seminars in Respiratory and Critical Care Medicine 30 (1): 67-85. February 2009. PMID 19199189. doi:10.1055/s-0028-1119811.
- ↑ Murray, Jerry (16 de abril de 2014). «What's New With Ferret FIP-like Disease?» (xls). Archivado desde el original el 24 de abril de 2014. Consultado el 24 de abril de 2014.
- ↑ «Progress and challenges toward the development of vaccines against avian infectious bronchitis». Journal of Immunology Research 2015: 1-12. 2015. PMC 4411447. PMID 25954763. doi:10.1155/2015/424860.
- ↑ «Coronavirus pathogenesis and the emerging pathogen severe acute respiratory syndrome coronavirus». Microbiology and Molecular Biology Reviews 69 (4): 635-664. December 2005. PMC 1306801. PMID 16339739. doi:10.1128/MMBR.69.4.635-664.2005.
- ↑ «Rat Coronavirus—an overview | ScienceDirect Topics». www.sciencedirect.com (en inglés). Consultado el 14 de marzo de 2020.
- ↑ Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin Archivado el 31 de mayo de 2019 en Wayback Machine., Peng Zhou, Hang Fan, Tian Lan, Xing-Lou Yang, Wei-Feng Shi, Wei Zhang, Yan Zhu, Ya-Wei Zhang, Qing-Mei Xie, Shailendra Mani, Xiao-Shuang Zheng, Bei Li, Jin-Man Li, Hua Guo, Guang-Qian Pei, Xiao-Ping An, Jun-Wei Chen, Ling Zhou, Kai-Jie Mai, Zi-Xian Wu, Di Li, Danielle E. Anderson, Li-Biao Zhang, Shi-Yue Li, Zhi-Qiang Mi, Tong-Tong He, Feng Cong, Peng-Ju Guo, Ren Huang, Yun Luo, Xiang-Ling Liu, Jing Chen, Yong Huang, Qiang Sun, Xiang-Li-Lan Zhang, Yuan-Yuan Wang, Shao-Zhen Xing, Yan-Shan Chen, Yuan Sun, Juan Li, Peter Daszak, Lin-Fa Wang, Zheng-Li Shi, Yi-Gang Tong & Jing-Yun Ma, Nature, 5 April 2018.
- ↑ «Cell replacement therapies to promote remyelination in a viral model of demyelination». Journal of Neuroimmunology 224 (1–2): 101-107. July 2010. PMC 2919340. PMID 20627412. doi:10.1016/j.jneuroim.2010.05.013.
- ↑ «Homology-Based Identification of a Mutation in the Coronavirus RNA-Dependent RNA Polymerase That Confers Resistance to Multiple Mutagens». Journal of Virology 90 (16): 7415-7428. August 2016. PMC 4984655. PMID 27279608. doi:10.1128/JVI.00080-16.
- ↑ «Coronavirus gene 7 counteracts host defenses and modulates virus virulence». PLoS Pathogens 7 (6): e1002090. June 2011. PMC 3111541. PMID 21695242. doi:10.1371/journal.ppat.1002090.
- ↑ «Alphacoronavirus protein 7 modulates host innate immune response». Journal of Virology 87 (17): 9754-67. September 2013. PMC 3754097. PMID 23824792. doi:10.1128/JVI.01032-13.
- ↑ «the Merck Veterinary Manual». Archivado desde el original el 9 de octubre de 2013. Consultado el 1 de junio de 2013.
- ↑ «Virus Taxonomy: 2018b Release» (html). International Committee on Taxonomy of Viruses (ICTV) (en inglés). March 2019. Consultado el 24 de enero de 2020.
- ↑ «Taxonomy browser (Betacoronavirus 1)». www.ncbi.nlm.nih.gov. Consultado el 14 de marzo de 2020.
- ↑ «Taxonomy browser (Betacoronavirus)». Centro Nacional para la Información Biotecnológica, Biblioteca Nacional de Medicina de Estados Unidos. Consultado el 14 de marzo de 2020.
Enlaces externos[editar]
- Esta obra contiene una traducción derivada de «Coronavirus» de Wikipedia en inglés, concretamente de esta versión, publicada por sus editores bajo la Licencia de documentación libre de GNU y la Licencia Creative Commons Atribución-CompartirIgual 4.0 Internacional.
- Coronavirus | Acerca de | CDC
- Coronavirus, infecciones por coronavirus
- Coronavirus
- Viralzone : Betacoronavirus
- Base de datos de patógenos de virus y recurso de análisis (ViPR): Coronaviridae
