SMILES
| SMILES | ||
|---|---|---|
| http://www.daylight.com/smiles/ | ||
| Información general | ||
| Extensión de archivo |
.smi | |
| Tipo de MIME | chemical/x-daylight-smiles | |
| Tipo de formato | formato de archivo de química | |
| Formato abierto | ? | |
En química, Simplified Molecular Input Line Entry Specification (especificación de introducción lineal molecular simplificada o SMILES) es una especificación para describir sin ambigüedades la estructura de una molécula usando cadenas ASCII cortas. Las cadenas SMILES pueden ser importadas por la mayoría de editores moleculares para su conversión en dibujos bidimensionales o modelos tridimensionales de las moléculas.
La especificación original de SMILES fue desarrollada por Arthur Weininger y David Weininger a fines de la década de 1980. Ha sido modificada y extendida por otros, destacando Daylight Chemical Information Systems Inc. En 2007 fue desarrollado un estándar abierto llamado "OpenSMILES" por la comunidad de química de código abierto Blue Obelisk. Otras notaciones lineales incluyen a la notación lineal Wiswesser (WLN), ROSDAL y SLN (Tripos Inc.)
En agosto de 2006, la IUPAC introdujo el InChI como un estándar para la representación de estructuras moleculares. Suele considerarse que SMILES tiene la ventaja de ser más inteligible a la lectura humana que el InChI; también tiene una base más amplia de soporte de software fundamentados en teoría de grafos.
Terminología[editar]
El término SMILES se refiere a una notación lineal para codificar estructuras moleculares, mientras que las instancias específicas deberían llamarse cadenas SMILES. Sin embargo, el término SMILES también se usa comúnmente para referirse, tanto a una sola cadena SMILES como a un número de cadenas SMILES, y el significado exacto se hace evidente a partir del contexto. Los términos canónico e isomérico pueden llevar a alguna confusión cuando se aplican a SMILES. Los términos describen atributos diferentes de las cadenas SMILES y no son mutuamente excluyentes.
Generalmente se puede escribir un número de cadenas SMILES igualmente válidas para una molécula. Por ejemplo, CCO, OCC y C(O)C especifican por igual al etanol. Se han desarrollado algoritmos para asegurar que se genera la misma cadena SMILES para una molécula, sin importar el orden de los átomos en la estructura. Esta cadena SMILES es única para cada estructura, aunque es dependiente del algoritmo de "canonicalización" usado para generarlo, y se denomina SMILES canónico. Estos algoritmos convierten primero el SMILES a una representación interna de la estructura molecular y no simplemente manipulan las cadenas como se piensa algunas veces. Los algoritmos para generar SMILES canónicos fueron desarrollados en Daylight Chemical Information Systems, OpenEye Scientific Software y Chemical Computing Group. Una aplicación común de los SMILES canónicos es para el indexado y para asegurar la unicidad de las moléculas en una base de datos.
La notación SMILES permite la especificación de estereoisómeros. Estas características estructuras no pueden ser especificadas solamente mediante conectividad, así que las cadenas SMILES que codifican esta información se denominan SMILES isoméricos. Una característica notable de estas reglas es que permiten la especificación parcial rigurosa de la quiralidad. El término "SMILES isomérico" también se suele aplicar a las cadenas SMILES en las cuales se especifican isótopos.
Ejemplos[editar]

Átomos[editar]
Los átomos están representados por la abreviatura estándar de cada elemento químico, entre corchetes, tales como [Au] para el oro. Los corchetes pueden ser omitidos para "subconjuntos orgánicos" de B, C, N, O, P, S, F, Cl, Br, y I. Todos los demás elementos deben estar encerrados en corchetes. Si los corchetes son omitidos, se asume el número propio de átomos de hidrógeno implícitos; por ejemplo, la cadena SMILES para el agua es simplemente O.
Un átomo que lleva una o varias carga(s) eléctrica(s) es encerrado en corchetes (cualquiera que sea), seguido del símbolo H si es ligado a uno o varios átomo(s) de hidrógeno (estos son entonces seguidos de su número, excepto si no hay que uno: NH4 para el amonio), luego del signo '+' para una carga positiva, o del signo '-' para una carga negativa. El número de las cargas es entonces especificado después del signo (excepto si no hay que uno); sin embargo, es así posible de escribir el signo de la(s) carga(s) tantas veces que el ion tiene de ella(s): en lugar de "Ti+4", pueden muy bien escribir "Ti++++" (Titano IV, Ti4+). Así, el anión hidróxido es representado por [OH-], el catión oxonio por [OH3+], y el catión cobalto III (Co3+) por [Co+3] o por [Co+++].
Enlaces[editar]
Se asume que los enlaces entre átomos de alifáticos son simples, salvo que se especifique lo contrario, y están implicados por adyacencia en las cadenas SMILES. Por ejemplo, la cadena SMILES para el etanol puede ser escrita como CCO. Las etiquetas de cierre de anillos pueden usarse para indicar conectividad entre átomos no adyacentes en la cadena SMILES, lo que para el ciclohexano y el dioxano es C1CCCCC1 y O1CCOCC1 respectivamente. Con un segundo ciclo, la etiqueta será 2 (naftaleno: c1cccc2c1cccc2); más allá de 9, es menester añadir el signo '%' delante la etiqueta, a fin de diferenciarla de dos etiquetas distintas ligados al mismo átomo (~C12~ significará que el átomo de carbono lleva las enlaces de cierre de anillos 1 y 2, aunque ~C%12~ indicará una sola etiqueta, la 12). Los enlaces dobles y triples pueden ser representados por los símbolos '=' y '#' respectivamente, como se ilustra en la cadena SMILES O=C=O (dióxido de carbono) y C#N (cianuro de hidrógeno).
Aromaticidad[editar]
Los átomos aromáticos C, O, S y N se representan por su letra minúscula 'c', 'o', 's' y 'n' respectivamente. El benceno, la piridina y el furano pueden ser representados por las cadenas SMILES c1ccccc1, n1ccccc1 y o1cccc1, respectivamente. Los enlaces entre átomos aromáticos son, por defecto, aromáticos, aunque pueden explicitarse usando el símbolo ':'. Los átomos aromáticos pueden estar unidos por enlace simple a otros, con lo que el bifenilo puede ser representado por c1ccccc1-c2ccccc2. El átomo de nitrógeno aromático unido al hidrógeno, como se encuentra en el pirrol debe ser presentado por [nH], y el imidazol se escribe con la notación SMILES n1c[nH]cc1.
Los algoritmos de Daylight y OpenEye para generar cadenas SMILES canónicas difieren en su tratamiento de la aromaticidad.

Ramificaciones[editar]
Las ramificaciones se describen con paréntesis, como en CCC(=O)O para el ácido propiónico, y C(F)(F)F para el fluoroformo. Los anillos sustituidos pueden ser escritos con el punto de ramificación en el anillo, como se ilustra en las cadenas SMILES COc(c1)cccc1C#N (ver representación) y COc(cc1)ccc1C#N (ver representación), que codifican a los isómeros 3-cianoanisol y 4-cianoanisol. Al escribir las cadenas SMILES para anillos sustituidos en esta forma se pueden hacerlas más legibles.
Estereoquímica[editar]
La configuración de los enlaces dobles se especifica usando los caracteres "/" y "\". Por ejemplo, F/C=C/F (ver representación) es una representación del E-difluoroeteno, en el que los átomos de flúor están en lados opuestos del enlace doble, mientras que F/C=C\F (ver representación) es una representación posible para Z-difluoroeteno, en el que los átomos de flúor están al mismo lado del doble enlace, como se muestra en la figura.
La configuración del átomo de carbono tetraédrico se especifica mediante @ o @@. La L-alanina, el enantiómero más común del aminoácido alanina, puede escribirse como N[C@@H](C)C(=O)O (ver representación). El especificador @@ indica que, cuando se ve desde el átomo de nitrógeno a lo largo del enlace al centro quiral, la secuencia de sustituyentes de hidrógeno (H), metilo (C) y carboxilato (C(=O)O) aparece en sentido horario. La D-alanina puede ser escrita como N[C@H](C)C(=O)O (ver representación). El orden de los sustituyentes en la cadena SMILES es muy importante, y la D-alanina puede ser codificada como N[C@@H](C(=O)O)C (ver representación).
Isótopos[editar]
Los isótopos se especifican con un número igual al número de masa del isótopo precediendo al símbolo del átomos. El benceno, en el que un átomo de carbono estuviera sustituido por carbono-14, se escribe como [14c]1ccccc1, y el deuteriocloroformo es [2H]C(Cl)(Cl)Cl.
Aplicación[editar]
| Molécula | Estructura | Cadena SMILES |
|---|---|---|
| Dinitrógeno | N≡N | N#N |
| Isocianato de metilo (MIC) | CH3–N=C=O | CN=C=O |
| Sulfato de cobre (II) | Cu2+ SO42- | [Cu+2].[O-]S(=O)(=O)[O-] |
| Enantotoxina (C17H22O2) | 
|
CCC[C@@H](O)CC\C=C\C=C\C#CC#C\C=C\CO |
| Piretrina II (C21H28O5) | 
|
COC(=O)C(\C)=C\C1C(C)(C)[C@H]1C(=O)O[C@@H]2C(C)=C(C(=O)C2)CC=CC=C |
| Aflatoxina B1 (C17H12O6) | 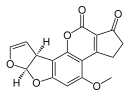
|
O1C=C[C@H]([C@H]1O2)c3c2cc(OC)c4c3OC(=O)C5=C4CCC(=O)5 |
| Glucosa (glucopyranosa) (C6H12O6) | 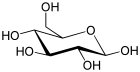
|
OC[C@@H](O1)[C@@H](O)[C@H](O)[C@@H](O)[C@@H](O)1 |
| Cuscutina alias Bergenin (resina) (C14H16O9) | 
|
OC[C@@H](O1)[C@@H](O)[C@H](O)[C@@H]2[C@@H]1c3c(O)c(OC)c(O)cc3C(=O)O2 |
| Una feromona de la cochinilla californiana | 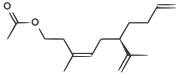
|
CC(=O)OCCC(/C)=C\C[C@H](C(C)=C)CCC=C |
| 2S,5R-Chalcogran: feromona del barrenillo Pityogenes chalcographus[1] | ![(2S,5R)-2-ethyl-1,6-dioxaspiro[4.4]nonane](http://upload.wikimedia.org/wikipedia/commons/thumb/8/8e/2S%2C5R-chalcogran-skeletal.svg/130px-2S%2C5R-chalcogran-skeletal.svg.png)
|
CC[C@H](O1)CC[C@@]12CCCO2 |
| Vanilina | 
|
O=Cc1ccc(O)c(OC)c1 |
| Melatonina (C13H16N2O2) | 
|
CC(=O)NCCC1=CNc2c1cc(OC)cc2 |
| Flavopereirina (C17H15N2) | 
|
CCc(c1)ccc2[n+]1ccc3c2Nc4c3cccc4 |
| Nicotina (C10H14N2) | 
|
CN1CCC[C@H]1c2cccnc2 |
| α-tujona (C10H16O) | 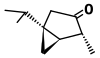
|
CC(C)[C@@]12C[C@@H]1[C@@H](C)C(=O)C2 |
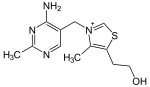
| Tiamina (C12H17N4OS+) (vitamine B1) |
|
OCCc1c(C)[n+](=cs1)Cc2cnc(C)nc(N)2 |
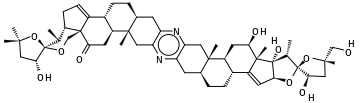
Ilustración con una molécula de más de 9 ciclos, la Cefalostatina-1[2] (molécula esteroidica de fórmula empírica C54H74N2O10 salida de un gusano marino de la familia de las Hydrophiloidea, Cephalodiscus gilchristi):
Dará, partiendo del radical metilo más a la izquierda en la figura:
C[C@@](C)(O1)C[C@@H](O)[C@@]1(O2)[C@@H](C)[C@@H]3CC=C4[C@]3(C2)C(=O)C[C@H]5[C@H]4CC[C@@H](C6)[C@]5(C)Cc(n7)c6nc(C[C@@]89(C))c7C[C@@H]8CC[C@@H]%10[C@@H]9C[C@@H](O)[C@@]%11(C)C%10=C[C@H](O%12)[C@]%11(O)[C@H](C)[C@]%12(O%13)[C@H](O)C[C@@]%13(C)CO
(Notad los '%' delante del índice de las etiquetas de cierre de anillos superior a 9, referirse a la sección "Enlaces", más alto).
Otros ejemplos de SMILES[editar]
La notación SMILES está descrita extensamente en el SMILES theory manual provisto por Daylight Chemical Information Systems y están presentes una cantidad importante de ejemplos ilustrativos. La depict utility de Daylight provee a los usuarios con los medios de comprobar sus propios ejemplos de SMILES y es una valiosa herramienta educacional.
Extensiones[editar]
SMARTS es una notación lineal para la especificación de esquemas subestructurales en moléculas. Aunque hace uso de muchos de los símbolos de SMILES, también permite la especificación de átomos y enlaces comodines, que pueden ser usados para definir consultas subestructurales para búsqueda en una base de datos química. Una mala interpretación común es que la búsqueda subestructural basada en SMARTS involucra comparar cadenas SMILES y SMARTS. En efecto, tanto las cadenas SMILES y SMARTS son convertidas primero a representaciones gráficas internas, a las que se les busca por isomorfismo subgráfico. SMIRKS es una notación lineal para especificar transformaciones en una reacción.
Conversión[editar]
Las cadenas SMILES pueden ser reconvertidas a representaciones bidimensionales usando algoritmos de generación de diagramas de estructura (Helson, 1999). Esta conversión no siempre es inambigua. La conversión a representaciones tridimensionales se logra por aproximaciones de minimización de energía. Hay muchas utilidades de conversión disponibles, basadas en web y descargables.
Véase también[editar]
- Smiles arbitrary target specification lenguaje SMARTS para la especificación de consultas subestructurales.
- SYBYL (otra notación lineal)
- Molecular Query Language - lenguaje de consultas que permite también incluir propiedades numéricas, como valores fisicoquímicos o distancias
- Chemistry Development Kit (conversión y representación bidimensional)
- International Chemical Identifier (InChI), la alternativa gratuita y abierta a SMILES por la IUPAC.
- OpenBabel, JOELib, OELib (conversión)
Referencias[editar]
- Anderson, E.; Veith, G.D; Weininger, D. (1987) SMILES: A line notation and computerized interpreter for chemical structures. Report No. EPA/600/M-87/021. U.S. EPA, Environmental Research Laboratory-Duluth, Duluth, MN 55804
- Weininger, D. (1988), SMILES, a chemical language and information system. 1. Introduction to methodology and encoding rules, J. Chem. Inf. Comput. Sci. 28, 31-36.
- Weininger, D.; Weininger, A.; Weininger, J.L. (1989) SMILES. 2. Algorithm for generation of unique SMILES notation J. Chem. Inf. Comput. Sci. 29, 97-101.
- Helson, H.E. (1999) Structure Diagram Generation In Rev. Comput. Chem. edited by Lipkowitz, K. B. and Boyd, D. B. Wiley-VCH, New York, pages 313-398.
Enlaces externos[editar]
Especificaciones[editar]
- "SMILES - A Simplified Chemical Language"
- Portal de OpenSMILES
- "SMARTS - SMILES Extension"
- Tutorial de SMILES de Daylight SMILES
- Generación de SMILES
Utilidades de software relacionadas con SMILES[editar]
- Representaciones de Daylight
- CACTVS en NCI
- PubChem – editor molecular en línea
- editor molecular JME Archivado el 28 de abril de 2001 en Wayback Machine.
- ACD/ChemSketch
- ChemAxon/Marvin – editor/visor químico en línea y generador/convertidor de SMILES
- ChemAxon/Instant JChem – aplicación de escritorio para almacenamiento/generación/conversión/visualización/búsqueda de estructuras SMILES, particularmente procesamiento por lotes; edición gratuita de uso personal
- Smormo-Ed – editor molecular para Linux que puede leer y escribir SMILES
- InChI.info – sitio web no oficial de InChI, que permite convertir en línea de InChI y SMILES a dibujos moleculares