Reacción en cadena de la polimerasa

La técnica de la reacción en cadena de la polimerasa es una técnica de la biología molecular desarrollada en 1983 por Kary Mullis.[1] Su objetivo es obtener un gran número de copias de un fragmento de ADN particular, partiendo de un mínimo; en teoría basta partir de una sola copia de ese fragmento original, o molde.[2]
La técnica, para mayor comprensión del texto, se puede explicar en los pasos siguientes de una manera simple:
- Juntar una única hebra sintética de solo 20 bases.
- Separar las hebras de ADN a investigar y ver si la hebra sintética encaja en algún lugar de estas.
- Sí lo hace: Se puede «animar» a la pequeña hebra sintética a que se reproduzca, proporcionándole algunas bases más, y las enzimas y condiciones apropiadas para hacer la tarea.
- Si se hace un número suficiente de veces, este fragmento crecerá desde una escala microscópica a una escala macroscópica.
- Después se produce otra corta hebra sintética con una secuencia de bases diferentes y solo se repite el proceso.
- Al final se podrá leer todo el genoma.[3]
Con este procedimiento, resulta mucho más sencillo identificar, con una probabilidad muy alta, virus o bacterias causantes de una enfermedad, identificar personas (cadáveres) o hacer investigación científica sobre el ADN amplificado. Estos usos derivados de la amplificación han hecho que se convierta en una técnica muy extendida, tanto en el campo forense como en los laboratorios químicos, con el consiguiente abaratamiento del equipo necesario para llevar a cabo dicha técnica.
Fundamento e importancia
[editar]
Esta técnica se fundamenta en la propiedad natural de las ADN polimerasas para replicar hebras de ADN, para lo cual se emplean ciclos de altas y bajas temperaturas alternadas para separar las hebras de ADN recién formadas entre sí tras cada fase de replicación y, a continuación, dejar que las hebras de ADN vuelvan a unirse para poder duplicarlas nuevamente. La reacción en cadena de la polimerasa fue perfeccionada por Kary Mullis perteneciente a la Cetus Corporation en California, en la década de 1980. Inicialmente la técnica era lenta, ya que las polimerasas se desnaturalizaban al realizar los cambios de temperatura y era necesario agregar nuevas polimerasas en cada ciclo. Puesto que las temperaturas del ciclo (95 °C en las fases de desnaturalización del ADN) suponen la inmediata desnaturalización de toda proteína, se emplean ADN polimerasas termoestables, extraídas de microorganismos adaptados a vivir a esas temperaturas, restrictivas para la mayoría de los seres vivos. Dichos microorganismos, generalmente arqueas, son: Thermus aquaticus (polimerasa Taq), Pyrococcus furiosus (Pfu), Thermococcus litoralis (Vent) y Thermus thermophilus (Tth). Generalmente se emplean mezclas de polimerasas muy procesivas (Taq) con otras capaces de hacer corrección de errores (Pfu, Vent).
Hoy, todo el proceso de la PCR está automatizado mediante un aparato llamado termociclador, que permite calentar y enfriar los tubos de reacción para controlar la temperatura necesaria para cada etapa de la reacción. Muchos termocicladores modernos hacen uso del efecto Peltier, que permite tanto calentar como enfriar los tubos simplemente invirtiendo la corriente eléctrica. Los tubos usados para PCR tienen una pared muy fina, lo que favorece una buena conductividad térmica, permitiendo que se alcance rápidamente el equilibrio térmico. Casi todos los termocicladores tienen un sistema que calienta la tapa de cierre con el fin de evitar la condensación sobre los tubos de reacción. Los termocicladores más antiguos carecían de este sistema y solucionaban el problema de la condensación con una capa de aceite en la parte superior de la mezcla de reacción o con un poco de cera dentro de los tubos. Actualmente existen algunos termocicladores que utilizan o pueden utilizar aceite mineral en el tubo de PCR como los termocicladores de nueva generación de flujo de aire.
Por lo general, la PCR es una técnica común y normalmente indispensable en laboratorios de investigación médica y biológica para una gran variedad de aplicaciones. Entre ellas se incluyen la clonación de ADN para la secuenciación, la filogenia basada en ADN, el análisis funcional de genes, el diagnóstico de trastornos hereditarios, la identificación de huellas genéticas (usada en técnicas forenses y pruebas de paternidad) y la detección y diagnóstico de enfermedades infecciosas.
Reactivos
[editar]
Para realizar la técnica se necesitan:[4]
- Los 4 desoxirribonucleótidos-trifosfato (dNTP), sustratos para polimerizar nuevo ADN.
- Dos cebadores o iniciadores, oligonucleótidos que son, cada uno, complementarios a una de las dos hebras del ADN. Son secuencias cortas, de entre seis y cuarenta nucleótidos, normalmente de dieciocho a veintidós, que permiten que la polimerasa inicie la reacción. Deben estar enfrentados y no a mucha distancia. Delimitan la zona de ADN a amplificar, es decir, corresponden a los nucleótidos que definen los extremos de la secuencia que se desea replicar. Varios artículos en revistas revisadas por pares brindan recomendaciones sobre elos procesos del diseño de cebadores (o primers, en inglés) [5][6] [7].
- Iones divalentes. Se suele usar magnesio (Mg2+), agregado comúnmente como cloruro de magnesio (MgCl2), o algún otro catión divalente. También se puede emplear manganeso (Mn2+), para mutagénesis de ADN mediante PCR, ya que altas concentraciones de Mn2+ incrementan la tasa de error durante la síntesis de ADN. Actúan como cofactores de la polimerasa.
- Iones monovalentes, como el potasio.
- Una solución tampón o buffer que mantiene el pH adecuado para el funcionamiento de la ADN polimerasa.
- ADN polimerasa o mezcla de distintas polimerasas con temperatura óptima alrededor de 70 °C (la más común es la polimerasa Taq).
- ADN molde, que contiene la región de ADN que se va a amplificar.
- Termociclador, el aparato que mantiene la temperatura necesaria en cada una de las etapas que conforman un ciclo.
Conceptos de la PCR
[editar]- Sensibilidad: se refiere a la cantidad mínima de ADN necesaria para que se produzca la amplificación, es decir, para obtener una banda. Se relaciona con los falsos negativos, ya que puede que una muestra sea positiva, pero sea dada como negativa porque no se ha amplificado por no tener suficiente cantidad de ADN.
- Especificidad: se refiere a la obtención de un solo producto amplificado. Viene determinada por los oligos y la especificidad con la que se unen al ADN molde. De esta forma, si los oligos tienen más de un sitio al que se pueden unir aparecerá más de un producto amplificado. Se relaciona con los falsos positivos, ya que puede que una muestra sea negativa, pero sea dada como positiva porque se ha amplificado una región de ADN no diana o que no se buscaba amplificar.
- Eficiencia: se refiere a la amplificación máxima que se puede obtener en un número determinado de ciclos.
- Fidelidad: se refiere a los errores que comete la ADN polimerasa durante la amplificación. Este concepto es de especial importancia en la secuenciación, pero en otros casos no es tan importante. Una buena fidelidad permite evitar falsos positivos y/o negativos.
Ciclo de amplificación
[editar]

El proceso de PCR por lo general consiste en una serie de 20 a 35 cambios repetidos de temperatura llamados ciclos; cada ciclo suele consistir en 2-3 pasos a diferentes temperaturas. La PCR común se realiza con ciclos que tienen tres pasos de temperatura. Los pasos de ciclos a menudo están precedidos por un choque térmico (llamado "hold") a alta temperatura (> 90 °C), y seguido por otro hold al final del proceso para la extensión de producto final o el breve almacenaje. Las temperaturas usadas y el tiempo aplicado en cada ciclo dependen de gran variedad de parámetros. Estos incluyen la enzima usada para la síntesis de ADN, la concentración de iones divalentes y de los dNTP en la reacción, y la temperatura de unión de los cebadores, así como la longitud del ADN que se desea amplificar.[4]
Actualmente, casi todos los termocicladores dan la opción de realizar la reacción de PCR con la llamada «tapa caliente». Es decir, que el sistema del termociclador aplicará calor a la parte de arriba del vial que contiene la mezcla de PCR. Al comienzo, los laboratorios que empezaron a usar los primeros aparatos que se comercializaron y que no incluían este sistema tenían que poner unas gotas de aceite dentro del vial. El objetivo de este procedimiento, al igual que el de la tapa caliente, es evitar la condensación de la muestra, ya que en el eppendorf se encuentran dos fases:líquido y gas. Al condensarse la muestra, perdemos volumen de la mezcla. Sin embargo, calentando la tapa o poniendo las gotas de aceite evitamos este proceso físico, conservando casi intacto el volumen de la muestra.
Inicio
[editar]Este paso consiste en llevar la reacción hasta una temperatura de 94-96 °C (o 98 °C si se está usando una polimerasa termoestable extrema), que se mantiene durante 1-9 minutos. Esto solo es necesario para ADN polimerasas que requieran activación por calor.
Desnaturalización
[editar]En primer lugar, se desnaturaliza el ADN (se separan las dos cadenas de las cuales está constituido). Este paso puede realizarse de diferentes modos, siendo el calentamiento (94−95 °C) de la muestra la forma más habitual. La temperatura a la cual se decide realizar la desnaturalización depende, por ejemplo, de la proporción de G+C que tenga la cadena, como también del largo de la misma. Otros métodos, raramente empleados en la técnica de la PCR, serían la adición de sales o agentes químicos capaces de realizar la desnaturalización.
Alineamiento o unión del cebador
[editar]A continuación se producirá la hibridación del cebador, es decir, el cebador se unirá a su secuencia complementaria en el ADN molde. Para ello es necesario bajar la temperatura a 40−68 °C durante 20-40 segundos (según el caso), permitiendo así el alineamiento. Los puentes de hidrógeno estables entre las cadenas de ADN (unión ADN-ADN) solo se forman cuando la secuencia del cebador es muy similar a la secuencia del ADN molde. La polimerasa une el híbrido de la cadena molde y el cebador, y empieza a sintetizar ADN. Los cebadores actuarán como límites de la región de la molécula que va a ser amplificada
Extensión o elongación de la cadena
[editar]Actúa la polimerasa, tomando el ADN molde para sintetizar la cadena complementaria y partiendo del cebador como soporte inicial necesario para la síntesis de nuevo ADN. La polimerasa sintetiza una nueva hebra de ADN complementaria a la hebra molde añadiendo los dNTP complementarios en dirección 5'→ 3', uniendo el grupo 5'-fosfato de los dNTP con el grupo 3'-hidroxilo del final de la hebra de ADN creciente (la cual se extiende). La temperatura para este paso depende del ADN polimerasa que usemos. Para la polimerasa Taq, la temperatura de máxima actividad está en 75−80 °C (comúnmente 72 °C). El tiempo de extensión depende tanto del ADN polimerasa usada como de la longitud del fragmento de ADN que se va a amplificar. Hay una regla comúnmente usada: en su temperatura óptima, la polimerasa de ADN polimerizará mil bases en un minuto.
Elongación final
[editar]Etapa única que se lleva a cabo a una temperatura de 70−74 °C durante 5-15 minutos tras el último ciclo de PCR. Con ella se asegura que cualquier ADN de cadena simple restante sea totalmente ampliado.
Conservación
[editar]Este es un paso que se lleva a cabo a 4−15 °C durante un tiempo indefinido para conservar la reacción a corto plazo.
La PCR normalmente se realiza con un volumen de reacción de 15−100 μL, en pequeños tubos de 0,2−0,5 mL que se colocan en el termociclador.
Para verificar que la PCR ha generado el fragmento de ADN previsto, se emplean técnicas de electroforesis, que separan los fragmentos de ADN generados de acuerdo a su carga, esto es, longitud, y, en menor medida y dependiendo de la matriz empleada, a su tamaño: típicamente se emplean la electroforesis en gel de agarosa, para fragmentos grandes; en acrilamida, para los más pequeños; y, de forma más rápida y aplicable a la PCR asociada a marcaje fluorescente, la electroforesis capilar.[8] El/los tamaño/s de los productos de la PCR vienen determinados por un marcador de peso molecular de ADN, el cual contiene fragmentos de ADN de tamaño conocido, y que se corre en el gel junto con los productos de PCR.
Optimización de la PCR
[editar]En la práctica, la PCR puede fallar por varias razones, entre ellas:
- La PCR es una técnica de gran sensibilidad, es decir, necesita una mínima cantidad de ADN para obtener un gran número de copias. Además, puede ser muy propensa a errores si se lleva a cabo en condiciones inadecuadas de esterilidad, que conduzcan a la amplificación de ADN no correspondiente a la muestra a analizar (y por tanto a conclusiones inciertas). Se han de tomar una serie de precauciones para evitar la contaminación con ADN extraño. Ejemplos en los que es especialmente importante evitar la amplificación son la detección de enfermedades (para no diagnosticar erróneamente a los pacientes) o el diagnóstico de identidad y parentesco. Posibles precauciones son:
- Tener especial cuidado durante los pasos previos a la amplificación: recogida, envío, custodia y procesado de muestras.
- Limpieza exhaustiva y esterilización (lejía, etanol, luz UV, psoralenos...) de la superficie de trabajo entre la realización de una PCR y la siguiente.
- En laboratorios de diagnóstico genético, se suelen tener áreas separadas de trabajo: un laboratorio para la preparación de la mezcla madre para la PCR, otro para la adición del ADN a amplificar, otro donde se encuentra la maquinaria para realizar la PCR y otro donde se abre y analiza la muestra ya amplificada.
- Cabinas de seguridad biológica para reducir vapores cargados de amplicones de enfermedades.
- Protección adecuada de los operarios que manipulen las muestras y los instrumentos del laboratorio: guantes, bata, cubrezapatos, gafas de seguridad, pelo recogido... En el diagnóstico molecular es conveniente que se cambien estos elementos al pasar de una zona de trabajo a otra.
- Controles positivos y negativos.
- Tipado de todo el personal del laboratorio. En el caso de encontrar una amplificación no esperada se podrá comprobar si proviene de uno de los operarios.
- Las técnicas de diseño de cebadores son importantes en la mejora de la obtención de productos de PCR y en evitar la formación de productos falsos. Algunas consideraciones al diseñar estos cebadores son:
- Se recomienda usar iniciadores de más de 15 nucleótidos (18−30 nn) Normalmente, se suelen diseñar de 20 nucleótidos. Los cebadores muy cortos hacen que nuestra PCR no sea muy específica, y los que se excedan en longitud harán que perdamos rendimiento en la reacción.
- Los cebadores no deben diferir en más de 3 bases.
- La proporción entre bases púricas y pirimidínicas de los dos oligos sea 1:1 (40-60 % a lo sumo), y que empiecen y terminen con 1 o 2 bases púricas.
- Se requiere una distribución homogénea de los cuatro nucleótidos en la secuencia, evitando los poliT/A/G/C.
- La Tm (melting temperature) de los oligos no puede diferir en más de 5 °C.
- Los cebadores no deben incluir en su secuencia regiones que sean complementarias entre sí, o se formarán dímeros entre ellos.
- Existe una gran variedad de polimerasas disponibles, pudiendo elegir la que más se ajuste a nuestras necesidades. Por ejemplo, algunas tienen actividades inexistentes en otras o las realizan de forma más eficiente, o funcionan en intervalos de temperatura en los cuales otras se desnaturalizan o pierden funcionalidad. Las más habituales son la taq y la pfu.
- Los componentes del tampón de reacción deberán ajustarse a las exigencias de nuestras enzimas.
- El termociclador, por supuesto, debe ser capaz de reproducir correctamente los ciclos de la PCR: para ello, diversas casas comerciales nos ofrecerán termocicladores elaborados con materiales que hagan más eficaces el desarrollo y el transcurso de las etapas, además de diversas facilidades de manejo. Dentro del laboratorio, su manejo, mantenimiento y ubicación será clave.
Aparte de aspectos como la contaminación y algún fallo en la hibridación de cebadores, puede haber otras complejidades que afecten a la PCR, como son:
- Degradación del ADN: esto puede ocurrir cuando se manipula la muestra en condiciones subóptimas o cuando se hace una autopsia, por ejemplo, y resulta en ausencia de amplificación o resultados parciales. Un caso particular es el "allele dropout", o bien pérdida de un alelo, que se puede llegar a confundir con ser homocigótico para dicho alelo.
- Inhibición de la PCR: puede haber agentes que interfieran en el correcto transcurso de la PCR. Requerirá un procesamiento adicional de la muestra para eliminar estos compuestos del medio antes de preparar la mezcla de PCR. Tanto la sangre como el semen contienen este tipo de inhibidores, por lo que es necesario una purificación previa.
- Modificación del ADN: las sustancias como el formol (empleado en conservación de tejidos) o la luz ultravioleta modifican el ADN, alterando así los resultados de la amplificación.
- Artefactos: pueden darse situaciones como las bandas "stutter" o "shadow", que consisten en que la polimerasa amplifica una repetición de más de un microsatélite o repetición en tándem.
- Alelo fuera de patrón: un alelo amplificado puede no coincidir con el patrón de picos de la referencia. Esto es señal de que algo ha ido mal durante la PCR.
- Mezclas: es posible que dos muestran se mezclen de alguna forma y el resultado sea una combinación del patrón de picos que cabe esperar tras la amplificación de cada una de ellas por separado. Esto y el caso anterior dificulta la interpretación de los resultados.
Tipos de PCR
[editar]PCR anidada
[editar]Técnica muy sensible de PCR en la que el producto de una amplificación es utilizado como molde para realizar una segunda amplificación con cebadores que se ubican dentro de la primera secuencia amplificada, es decir, cuando tenemos el primer, como su amplificación se pueden unir los cebadores y se hace de nuevo una amplificación dentro de la amplicón inicial. Este tipo de PCR tiene la ventaja de brindar alta sensibilidad y especificidad. La especificidad aumenta porque como es amplificación de un amplicón obtenido previamente, los cebadores solo van a hibridar en un sitio dentro de la molécula y el resultado será una única banda. Así, evitamos posibles hibridaciones inespecíficas de los cebadores. La desventaja de esta técnica es que no nos permite cuantificar la muestra.
PCR de extensión solapada (Mutagénesis)
[editar]Se introducen cambios de secuencia dentro de fragmentos (clonados) de ADN. Se requieren 2 cebadores mutagénicos y otros 2. Se amplifica un fragmento 5' y un fragmento 3' que se solapan portando ambos la mutación. Se usan los productos en otra reacción para producir el ADN mutado de longitud completa.
PCR in situ
[editar]La PCR in situo consiste en una reacción de PCR en secciones histológicas o células, donde los productos generados pueden visualizarse en el sitio de amplificación. Es realizada sobre preparaciones fijas en un portaobjetos. En la técnica de PCR in situ se realiza una primera amplificación de ADN blanco y luego detección mediante hibridación in situ convencional con sondas de ADN/ARN. De esta manera pueden detectarse cantidades pequeñísimas de genoma. Esta tecnología es de gran alcance en la capacidad de amplificar específicamente una población de secuencias de menor representación.
PCR múltiple
[editar]PCR en la cual se amplifica simultáneamente más de una secuencia. Para ello, se combinan dos o más pares de cebadores en un mismo tubo, junto con el resto de los reactivos de la reacción en cantidades suficientes, para amplificar simultáneamente varios segmentos de ADN. Ventajas: información sobre varios locus en una sola reacción, menor cantidad de molde para el análisis, menor cantidad de reactivos, rápida construcción de bases de datos. Desventajas: para llevarla a cabo adecuadamente y sin errores, se requiere de una cuidadosa optimización del proceso.Un artículo evaluado por pares brinda una revisión general sobre la PCR múltiple [9]
PCR con transcriptasa inversa (RT-PCR)
[editar]Es una variante de la PCR en la que usamos ARN como molde inicial en vez de ADN, y emplea una transcriptasa inversa (como Tth) para realizar la síntesis de un ADN complementario al ARN (ADNc). De esta forma, el desarrollo inicial de una RT-PCR sería:
- 1.er paso: retrotranscripción a partir del ARN.
- 2.º paso: amplificación a partir de la primera hebra de ADNc.
- 3.er paso: PCR estándar.
PCR en tiempo real o PCR cuantitativo (qPCR)
[editar]Reacción de PCR cuya principal característica es que permite cuantificar la cantidad de ADN o ARN presente en la muestra original, o para identificar con una muy alta probabilidad, muestras de ADN específicas a partir de su temperatura de fusión (también denominado valor Tm, del inglés melting temperature).
Se puede dividir en las técnicas basadas en fluorocromos no específicos y en las técnicas basadas en sondas específicas.
En las técnicas basadas en fluorocromos el ADN, que ve multiplicada su cantidad con cada ciclo, se une al fluorocromo (generalmente SYBR Green) produciendo fluorescencia que es medida por el termociclador apto para PCR en tiempo real. Permite cuantificar solo una secuencia por reacción, pero tiene la ventaja de utilizar cebadores normales para su realización. Es mucho más económica que la que usa sondas específicas.
Las técnicas basadas en sondas específicas utilizan una sonda unida a dos fluorocromos que hibrida en la zona intermedia entre el cebador directo (forward) y el inverso (reverse); cuando la sonda está intacta, presenta una transferencia energética de fluorescencia por resonancia (FRET). Dicha FRET no se produce cuando la sonda está dañada y los dos fluorocromos están distantes, producto de la actividad 5'-3' exonucleasa de la ADN polimerasa. Esto permite monitorizar el cambio del patrón de fluorescencia y deducir el nivel de amplificación del gen.
La mayoría de estos inconvenientes se han solucionado con la introducción de la PCR realizada en tiempo real (Q-PCR), que elimina cualquier proceso post-PCR puesto que monitoriza la progresión de la amplificación en el momento en que ocurre. A diferencia de la PCR convencional (en punto final), que mide la acumulación del ADN al final de un número predeterminado de ciclos, con Q-PCR esto se hace durante el proceso de amplificación usando fluorescencia, de forma que su aumento es proporcional a la cantidad de ADN formada. El proceso se puede automatizar fácilmente usando un sistema que realice la amplificación (termociclador) y que a su vez sea capaz de leer fluorescencia. Existe una amplia oferta de aparatos en el mercado. La mayoría pueden trabajar con las diversas opciones de marcado fluorescente y son "abiertos", es decir, permiten programar las condiciones de amplificación y lectura de forma que su uso no queda limitado a unos reactivos determinados. Varios artículos, publicados en revistas revisadas por pares, presentan revisiones generales sobre la PCR en tiempo real [10] [11] [12][13].
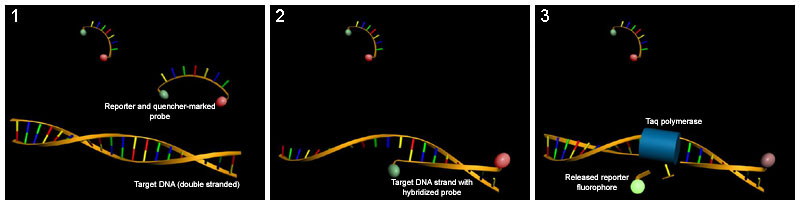
Variaciones de la PCR básica
[editar]- PCR específica de alelo: esta técnica de diagnóstico o clonación es usada para identificar o utilizar los polimorfismos de una sola base (SNPs). Utiliza cebadores específicos para las secuencias normal y mutante. El diseño más habitual es un análisis en dos tubos con dos cebadores: uno normal y otro mutante en reacciones separadas junto con los cebadores control.
- PCR "assembly": consiste en la síntesis artificial de largas secuencias de ADN, realizando para ello la PCR en un fondo de oligonucleótidos largos con secuencias solapantes cortas.
- PCR asimétrica: usada para amplificar preferentemente una cadena del ADN original con respecto a la otra.
- PCR de colonia: mediante esta técnica, colonias de bacterias Escherichia coli pueden ser rápidamente examinadas para construcciones viables de vectores de ADN.
- Amplificación dependiente de helicasa: esta técnica es muy parecida a la PCR convencional, pero en ella se emplea la enzima helicasa y una temperatura constante en lugar de la polimerasa de ADN y los ciclos repetidos de hibridación-elongación.
- PCR hot-start: esta técnica reduce la amplificación inespecífica durante las etapas iniciales de la PCR: mientras que la máquina alcanza la temperatura de la primera etapa (unos 95 °C) puede que ocurra la unión de los cebadores y se produzca amplificación, ya que en el camino para alcanzar estos 95 °C se pasa por la temperatura de anillamiento (que es más baja).
- Para evitar esto, la PCR hot-start se basa en que la reacción comience cuando la máquina ya esté a 95°, debido a que antes no se encuentran presentes la polimerasa o el cloruro de magnesio, lo que podemos conseguir mediante varias técnicas:
- - Añadir la polimerasa o el cloruro de magnesio tras el periodo de calentamiento
- - Separar por una capa de cera los distintos componentes de la reacción. La cera se funde al alcanzar los 95 °C y es entonces cuando los componentes entran en contacto
- - Anticuerpos anti-polimerasa que estén bloqueando a la polimerasa. Al alcanzar los 95 °C estos anticuerpos se inactivan debido a que se desnaturalizan y la polimerasa puede actuar
- PCR específica de intersecuencia (ISSR): se trata de un método de PCR para su uso en huella genética, que amplifica regiones entre repeticiones de secuencia simple para producir una huella genética única de longitudes de fragmento amplificadas.
- PCR inversa: es un método usado para poder realizar la PCR cuando solo es conocida una secuencia interna. Muy útil en la identificación de secuencias que flanquean insertos genómicos.
- PCR mediada por ligación: este método usa pequeños linkers de ADN ligados al ADN de interés y múltiples cebadores hibridando estos linkers.
- PCR específica de metilación (MSP): se usa para detectar metilaciones en islas CpG de ADN genómico.
- Amplificación Múltiple Dependiente de Sonda (Multiplex Ligation-dependent Probe Amplification o MLPA): permite amplificar varias secuencias objetivo con un único par de cebadores, evitando así las limitaciones de resolución de la PCR multiplex.
- PCR cuantitativa: se usa para medir la cantidad de un producto de PCR. En la técnica clásica de PCR, se cuantifica por aproximación con diluciones y amplificación de concentraciones conocidas de la secuencia diana. También se puede cuantificar por método competitivo: se trabaja con concentraciones crecientes conocidas de un fragmento que puede ser amplificado por los mismos oligos que la muestra de estudio, pero de menor tamaño que esta, de forma que con los resultados obtenidos se puede estimar qué concentración del competidor es equivalente a la de la muestra de cantidad desconocida. La cuantificación en PCR está optimizada en la técnica de PCR en tiempo real.
- PCR-TAIL: la PCR termal de entrelazado asimétrico es usada para aislar una secuencia desconocida que flanquea una secuencia conocida.
- PCR touchdown: se trata de una variante de la PCR que se emplea cuando se desconoce la secuencia exacta de los extremos de la secuencia a amplificar, de modo que se asume que puede existir alguna base desapareada en el alineamiento cebador-secuencia. Su finalidad es reducir el fondo no específico bajando gradualmente la temperatura de hibridación a lo largo del progreso de la PCR.
- PAN-AC: este método usa condiciones isotermas para la amplificación, y puede ser usado en células vivas.
- Ciclo térmico rápido para PCR:[14] La amplificación del ADN puede llevarse a cabo rápidamente, como completar 30 ciclos en menos de 30 minutos: ciclo térmico rápido. Normalmente se ha considerado que las etapas de cada ciclo son tres reacciones que ocurren en tres periodos separados y a tres temperaturas diferentes. Este paradigma del equilibrio secuencial no tiene en cuenta que la temperatura de la muestra no cambia instantáneamente, de hecho, durante una PCR la muestra está la mayoría del tiempo en temperaturas de transición. El ciclo térmico rápido aprovecha la ventaja de la instantánea desnaturalización e hibridación, cuyas temperaturas necesitan alcanzarse, pero no mantenerse. Además, para productos cortos, la extensión puede llevarse a cabo durante la transición a la temperatura de extensión, y por lo tanto, tampoco es necesario mantenerla. Un ciclo rápido podría entonces describirse como 94 °C, 0 s; 55 °C, 0 s y 72 °C, 0 s. Como vemos, este modelo no ayuda, porque nos lleva solo a temperaturas extremas y no nos da información sobre lo que pasa entre ellas. En cambio, en el paradigma cinético para PCR de ciclo rápido se describe completamente el historial de temperaturas de la muestra. Se considera que desnaturalización, hibridación y elongación ocurren en un rango de temperaturas y además pueden solapar temporalmente. Hay termocicladores que permiten que un ciclo se complete en 20-60 segundos, por lo que 30 ciclos tardan de 10 a 30 minutos.
Aplicaciones
[editar]La técnica de la PCR tiene multitud de aplicaciones: ya en ciencia básica, como herramienta de detección y/o generación de acervos de fragmentos de ADN de interés; ya en ciencia aplicada, como elemento resolutivo en sí mismo, por ejemplo en diagnóstico clínico.
Investigación
[editar]La PCR convencional, se emplea como base para multitud de técnicas en el laboratorio debido a su robustez y rapidez. De este modo, la PCR de punto final permite controlar y detectar los fragmentos de ADN de interés.

Una aplicación de la PCR de extrema importancia es la clonación de secuencias de ADN en vectores, como pueden ser los plásmidos. Para ello, se emplean cebadores que contienen en su extremo 5' una corta secuencia que permite la interacción posterior con otra complementaria situada en el vector de clonación a emplear. Por ejemplo, se puede incluir una diana de restricción en dichos cebadores, de modo que, y si esta no existía previamente en el fragmento y es única en el vector, pueda efectuarse una ligación mediante la ligasa de T4 tras la digestión con la enzima de restricción apropiada de ambos elementos. Otro método asimilable a esta vía es el empleo de la recombinación dirigida; esto es, se adapta al 5' de los cebadores una secuencia que faculta a una recombinasa la recombinación dirigida con un vector dado.[15]
Medicina
[editar]En medicina, la PCR se emplea fundamentalmente como herramienta de diagnosis (Coleman y Tsongalis, 2006):
- Una de las principales aplicaciones de la PCR es hacer pruebas genéticas para detectar mutaciones en el ADN que provoquen algún tipo de enfermedad.El diagnóstico de enfermedades hereditarias presentes en el genoma es un proceso largo y complicado que puede acortarse significativamente gracias a la PCR. Así, se pueden hacer análisis del ADN de los futuros padres para ver si son portadores, o analizar el ADN de sus hijos por si están afectados por una enfermedad hereditaria. Para el análisis prenatal, las muestras de ADN pueden obtenerse a partir de la amniocentesis, de la muestra de las vellosidades coriónicas o incluso a partir de algunas células fetales que circulan por el torrente sanguíneo de la madre. El uso de la PCR también es esencial para el diagnóstico genético preimplantacional donde se pueden analizar las células del embrión para buscar posibles mutaciones.
- Permite el genotipar la especie o especies que provocan un determinado cuadro infeccioso: para ello, se amplifica una zona del genoma bacteriano cuyo producto de PCR posea unas características de tamaño o temperatura de fusión que permitan identificarlo de forma inequívoca. En el caso de infecciones virales que implican la integración del genoma del patógeno en el ADN del hospedador, como es el de la infección por VIH, la PCR cuantitativa posibilita la determinación de la carga viral existente y por tanto, del estadio de la enfermedad.[16]
- La PCR también se puede usar en revisiones médicas rutinarias, como en los servicios de donantes de sangre; a través de esta técnica se pueden detectar infecciones en el donante (como VIH o Hepatitis B) mientras aún están en el periodo de incubación. Dada la sensibilidad de las pruebas de PCR se pueden tomar muestras colectivas (por ejemplo, 96 pruebas individuales). Si una de estas muestras colectivas da positivo, se toman a partir de ella muestras progresivamente menores hasta que se encuentre el causante.
- También se puede usar la PCR como parte de las pruebas realizadas cuando se hace un trasplante de tejidos. En 2008 se propuso usar esta técnica para reemplazar las pruebas tradicionales con anticuerpos.[17]
- A veces, mediante la PCR se pueden hacer terapias personalizadas para pacientes con ciertos tipos de cáncer que producen mutaciones en los oncogenes a partir de la detección de estas mutaciones.
Paleontología, antropología biológica y ciencias forenses
[editar]Los campos de la paleontología, antropología biológica y la medicina y antropología forense se han visto enormemente beneficiados por esta técnica, puesto que todas ellas construyen con frecuencia el conocimiento de sus correspondientes disciplinas gracias a restos o huellas de seres vivos. Uno de los materiales biológicos que más información puede proporcionar es el ADN.
La relativa estabilidad de este permite que, aunque fragmentado, se conserve durante largos períodos si las condiciones son propicias.[15] En ocasiones las muestras intactas con las que se puede contar son extraordinariamente pequeñas o están deterioradas. La PCR soluciona ambos problemas y proporciona cantidades útiles para posteriores pasos de análisis. En primer lugar aumenta la cantidad de material recuperado a partir de muestras escasas, puesto que como ya se dijo anteriormente, en teoría basta una sola molécula para que el proceso pueda tener lugar. También debido a la naturaleza de la técnica y su propósito de amplificación de fragmentos pequeños, esta fragmentación no impide que este ADN pueda ser empleado como molde para una reacción de PCR.
- En paleontología y Antropología la PCR permite recuperar las escasas cantidades de ADN que aún no se han degradado. Algunos lugares en que el ADN podría preservarse son la brea las cenizas volcánicas, el ámbar, hielos históricos polares o glaciares y ambientes áridos, sedimentos, así como en los cristales de apatita de restos de esqueleto,[18] siendo posible de ese modo caracterizar cadáveres, fósiles u otros restos mediante genotipado por análisis de microsatélites o incluso genomas de taxones extintos, amplificados de este modo, como pueden ser los realizados mediante el ADN genómico del hombre de Neanderthal.[19] El propósito sería utilizar este ADN amplificado para posteriormente realizar estudios filogenéticos o etnográficos o de poblaciones mediante la comparación de secuencias de ADN, o el estudio de las causas de la separación evolutiva de dos especies.
- En las ciencias forenses se emplea para establecer la filiación de una persona o para obtener pruebas a partir de muestras mínimas dejadas por el autor de un crimen como saliva, semen u otros restos de tejidos (Butler, 2005).
Agronomía y diversidad
[editar]Tal y como la PCR multiplex permite producir huellas genéticas de individuos concretos, dentro del marco de la genética forense, existen métodos basados en la PCR que permiten discernir entre grupos infraespecíficos de cultivos de interés agronómico; por ejemplo, de cultivares.[20] Para ello, se emplean oligonucleótidos de un tamaño lo suficientemente pequeño como para que ceben de forma relativamente inespecífica, aunque siempre de tal forma que produzcan un patrón de bandas discreto e interpretable. De este modo, la pauta obtenida tras la electroforesis de los fragmentos tiende a agrupar a los individuos de mayor semejanza, que poseen un comportamiento similar, de los que divergen.
Historia
[editar]
En 1971, un artículo publicado por Kleppe et al. en Journal of Molecular Biology describió por primera vez un método que usaba enzimas para replicar una secuencia pequeña de ADN con cebadores in vitro.[21] Sin embargo, este temprano ejemplo del principio básico de la PCR no recibió mucha atención, y la invención de la reacción en cadena de la polimerasa en 1983 es generalmente atribuida a Kary Mullis.[22][23] Mullis ganó el Premio Nobel por su trabajo en PCR.
Algo muy a tener en cuenta en la PCR es que la ADN polimerasa que se use sea capaz de soportar las altas temperaturas de >90 °C necesarias para la separación de las dos hebras de ADN de la doble hélice tras cada ciclo de replicación. Las ADN polimerasas que se utilizaron originariamente para los experimentos in vitro previos a la PCR no eran capaces de soportar estas altas temperaturas, por lo que los primeros procedimientos para replicar el ADN eran muy ineficientes, largos y requerían grandes cantidades de ADN polimerasa.
El descubrimiento en 1976 de la polimerasa Taq, una polimerasa de ADN extraída de la bacteria termófila Thermus aquaticus que habita medios de muy alta temperatura (50-80 °C), eliminó los grandes inconvenientes del método de la PCR. Este ADN polimerasa es estable a altas temperaturas, permaneciendo activa hasta después de la desnaturalización del ADN, eliminando la necesidad de añadir a la reacción nueva polimerasa tras cada ciclo. Este descubrimiento permitió automatizar el proceso, antes tan tedioso, acoplándolo al uso del termociclador.
Al mismo tiempo que desarrollaba la PCR en 1983, Mullis trabajaba en Emeryville, California (EE. UU.), para una de las primeras empresas biotecnológicas, Cetus Corporation, donde era responsable de sintetizar cadenas cortas de ADN. Mullis afirma que concibió la idea para la PCR una noche mientras cruzaba la Autopista de la Costa Pacífica (EE. UU.) en su coche.[22] Estaba imaginando una nueva forma de analizar mutaciones en el ADN cuando se percató de que, en lugar de eso, había inventado un método para amplificar regiones específicas de ADN mediante ciclos de duplicación repetidos usando ADN polimerasas. Mullis atribuye la invención de esta técnica a los efectos de la droga psicodélica y alucinógena LSD.[24]
En la revista Scientific American, Mullis resumió el procedimiento: "Comenzando con una única molécula del material genético ADN, la PCR puede generar 100 billones de moléculas iguales en una tarde. La reacción es fácil de hacer, no requiere más que un tubo de pruebas, unos pocos reactivos simples y una fuente de calor".[25][26] Fue premiado con el Premio Nobel de Química en 1993 por su invención, y siete años después, él y sus colegas del Cetus llevaron a la práctica su propuesta.
Guerras de patentes
[editar]La técnica de la PCR fue patentada por Cetus Corporation, donde Mullis trabajaba cuando inventó la técnica en 1983. La enzima polimerasa Taq fue también cubierta de patentes. Tuvieron lugar varios pleitos relacionados con la técnica, incluyendo un pleito fracasado generado por DuPont. La compañía farmacéutica Hoffmann-La Roche adquirió los derechos de las patentes en 1992 y actualmente mantiene las que aún están protegidas.[27][28]
Véase también
[editar]- Pruebas de COVID-19
- NASBA (Nucleic acid sequence based amplification o amplificación isotérmica de ácidos nucleicos)
- ADN
- ARN
- Análisis clínicos
- Polimerasa
- TMA
Referencias
[editar]- ↑ «Bartlett & Stirling (2003)—A Short History of the Polymerase Chain Reaction. In: Methods Mol Biol. 226:3-6». Archivado desde el original el 24 de diciembre de 2007. Consultado el 14 de enero de 2008.
- ↑ History of Polymerase Chain Reaction (PCR)
- ↑ Michael., Brooks,. Radicales libres : La anarqu©Øia secreta de la ciencia. ISBN 978-84-344-0035-1. OCLC 924733245. Consultado el 27 de enero de 2022.
- ↑ a b Joseph Sambrook and David W. Russel (2001). Molecular Cloning: A Laboratory Manual (3rd ed. edición). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 0-87969-576-5.
- ↑ Bustin, Stephen A.; Mueller, Reinhold; Nolan, Tania (2020). «Parameters for Successful PCR Primer Design». Methods in Molecular Biology (Clifton, N.J.) 2065: 5-22. ISSN 1940-6029. PMID 31578684. doi:10.1007/978-1-4939-9833-3_2. Consultado el 1 de noviembre de 2024.
- ↑ Singh, Amarjeet; Pandey, Girdhar K. (2015). «Primer design using Primer Express® for SYBR Green-based quantitative PCR». Methods in Molecular Biology (Clifton, N.J.) 1275: 153-164. ISSN 1940-6029. PMID 25697658. doi:10.1007/978-1-4939-2365-6_11. Consultado el 1 de noviembre de 2024.
- ↑ Thornton, Brenda; Basu, Chhandak (2015). «Rapid and simple method of qPCR primer design». Methods in Molecular Biology (Clifton, N.J.) 1275: 173-179. ISSN 1940-6029. PMID 25697660. doi:10.1007/978-1-4939-2365-6_13. Consultado el 1 de noviembre de 2024.
- ↑ Mathews, C. K.; Van Holde, K.E et Ahern, K.G (2003). «6». Bioquímica (3 edición). pp. 204 y ss. ISBN 84-7892-053-2.
- ↑ Markoulatos, P.; Siafakas, N.; Moncany, M. (2002). «Multiplex polymerase chain reaction: a practical approach». Journal of Clinical Laboratory Analysis 16 (1): 47-51. ISSN 0887-8013. PMC 6808141. PMID 11835531. doi:10.1002/jcla.2058. Consultado el 1 de noviembre de 2024.
- ↑ Kubista, Mikael; Andrade, José Manuel; Bengtsson, Martin; Forootan, Amin; Jonák, Jiri; Lind, Kristina; Sindelka, Radek; Sjöback, Robert et al. (2006). «The real-time polymerase chain reaction». Molecular Aspects of Medicine 27 (2-3): 95-125. ISSN 0098-2997. PMID 16460794. doi:10.1016/j.mam.2005.12.007. Consultado el 1 de noviembre de 2024.
- ↑ Schmittgen, Thomas D.; Livak, Kenneth J. (2008). «Analyzing real-time PCR data by the comparative C(T) method». Nature Protocols 3 (6): 1101-1108. ISSN 1750-2799. PMID 18546601. doi:10.1038/nprot.2008.73. Consultado el 1 de noviembre de 2024.
- ↑
- ↑ Bustin, Stephen; Huggett, Jim (2017-12). «qPCR primer design revisited». Biomolecular Detection and Quantification 14: 19-28. ISSN 2214-7535. PMC 5702850. PMID 29201647. doi:10.1016/j.bdq.2017.11.001. Consultado el 1 de noviembre de 2024.
- ↑ PCR Applications. Protocols for functional genomics. Capítulo 14. Edited by Michael A. Innis, David H. Gelfand, John J. Sninsky. ISBN 0-12-372186-5, Academic Press 1999
- ↑ a b Watson, J, D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M. et Losick, R (2004). Molecular Biology of the Gene (Fifth edition edición). San Francisco: Benjamin Cummings. ISBN 0-321-22368-3.
- ↑ Scott L. Butler, Mark S.T. Hansen & Frederic D. Bushman (2007). «A quantitative assay for HIV DNA integration in vivo». Nature Medicine (7): 631-634. doi:10.1038/87979.
- ↑ Quill E "Blood-Matching Goes Genetic" Science Magazine (14 March 2008) pp. 1478-1479.
- ↑ «Museo Victora de Melbourne sobre la preservación del ADN». Archivado desde el original el 24 de abril de 2016. Consultado el 26 de marzo de 2008.
- ↑ James P. Noonan,Graham Coop, Sridhar Kudaravalli, Doug Smith, Johannes Krause, Joe Alessi, Feng Chen, Darren Platt, Svante Pääbo, Jonathan K. Pritchard, Edward M. Rubin (17 de noviembre de 2006). «Sequencing and Analysis of Neanderthal Genomic DNA». Nature Medicine 314 (5802): 1113-1118. DOI: 10.1126/science.1131412.
- ↑ Jinguo Hu1 and Carlos F. Quiros (1991). «Identification of broccoli and cauliflower cultivars with RAPD markers». Plant Cell Reports 10 (10). DOI: 10.1007/BF00234583.
- ↑ Kleppe K, Ohtsuka E, Kleppe R, Molineux I, Khorana HG (1971). «Studies on polynucleotides. XCVI. Repair replications of short synthetic DNA's as catalyzed by DNA polymerases». J. Mol. Biol. 56: 341-361. Archivado desde el original el 7 de septiembre de 2009. Consultado el 31 de enero de 2008.
- ↑ a b Mullis, Kary (1998). Dancing Naked in the Mind Field. Nueva York: Pantheon Books. ISBN 0-679-44255-3.
- ↑ Rabinow, Paul (1996). Making PCR: A Story of Biotechnology. Chicago: University of Chicago Press. ISBN 0-226-70146-8.
- ↑ Mullis, Kary (1998). Dancing Naked in the Mind Field. Nueva York: Pantheon Books. pp. 18. ISBN 0-679-44255-3.
- ↑ Mullis, Kary B. (1990). «Reacción en cadena de la polimerasa». Investigación y ciencia (165).
- ↑ Mullis, Kary (1990). «The unusual origin of the polymerase chain reaction». Scientific American 262 (4): 56-61, 64-5.
- ↑ PR Newswire
- ↑ Consejos sobre cómo sobrevivir a la guerra de las patentes de Taq Archivado el 27 de septiembre de 2007 en Wayback Machine.: GEN Genetic Engineering News Biobusiness Channel: Artículos. 1 de mayo de 2006 (Vol. 26, No. 9).
Bibliografía
[editar]- Saiki RK, Gelfand DH, Stoffel S, Scharf SJ, Higuchi R, Horn GT, Mullis KB, et al.(1988) Primer-directed enzymatic amplification of DNA with a thermostable DNA polymerase. Science 239: 487–491
- Griffiths, J.F. A. et al. (2002). Genética. McGraw-Hill Interamericana. ISBN 84-486-0368-0.
- Coleman, WB y Tsongalis, GJ (2006). Molecular Diagnostics: For the Clinical Laboratorian. Humana Press. ISBN 1-58829-356-4.pgs. 47-56 y 65-74
- Butler, JM (2005). Forensic DNA Typing: Biology, Technology, and Genetics of STR. Academic Press pgs 63-84. ISBN 0-12-147952-8.
Enlaces externos
[editar] Wikimedia Commons alberga una galería multimedia sobre Reacción en cadena de la polimerasa.
Wikimedia Commons alberga una galería multimedia sobre Reacción en cadena de la polimerasa.- Animación PCR
- Artículos sobre PCR
- Nueva técnica más eficiente en PCR
- Please find all details about real-time PCR here: http://www.gene-quantification.info - The reference in real-time PCR
- Simulation en línea de reacciones de PCR frente a procariotas secuenciados.
- Ejercicio en línea en el que se diseñan y simulan experimentos de PCR y PCR-RFLP.
