Haplogrupo E (ADN-Y)
En genética humana, el haplogrupo E es un haplogrupo del cromosoma Y humano formado a partir de muchos polimorfismos, como M96/PF1823, P29/PF1643 y 148 más, y al igual que el haplogrupo D, desciende de DE. Este haplogrupo se habría originado hace unos 70 mil años en África Oriental, es el más característico de toda África y se encuentra también, aunque en menor proporción, en el Cercano Oriente y en Europa, especialmente en el área del Mediterráneo.

Origen[editar]
Probablemente tiene un origen en África Oriental[1] de una población que generó la expansión fuera de África,[2] región donde está la mayor diversidad,[3] originándose en una época en que el Sahara era habitable, hace 69 000[4] aunque otras fuentes calculan unos 65[5] o 73 000 años.[6] Más recientemente, se ha encontrado evidencia adicional que respalda la hipótesis del origen africano.[1]
Origen y antigüedad por subclados[editar]
Los subclados más frecuentes son E1b1a1 y E1b1b1, originados hace unos 20 000 años. E1b1b1 se desarrolla en el norte de África o Asia Occidental, mostrando una relación parcial con las lenguas afroasiáticas, por lo que su presencia fuera de África podría estar relacionada al Neolítico y por la interacción histórica del antiguo Egipto con otras culturas del Mediterráneo.

- E (M96): Con el origen más probable en África Oriental[3] y tiene unos 69 000 años.[4]
- E1 (P147): Hace unos 51 000 años[5] originado en África.
- E1a (M33, M132): 45 000-50 000 años, origen al oeste africano.[7]
- E1b (P177): Unos 50 000 años,[5]
- E1b1 (P2): Originado en África Oriental.[8]
- E1b1a (V38, V100): 40 000 años,[5] en África Oriental.[9]
- E1b1a1 (P1, M2): 39 mil años,[5] en el África centro-occidental.
- E1b1b (M215/Page40): 41 500 años[5] en el África Oriental[3] o Cuerno de África,[10] probablemente Etiopía.
- E1b1b1a1 (M78): 19 mil años,[5] originado el este del Sahara, probablemente en el valle del Nilo al norte de Sudán, o con más probabilidad en Egipto.[11]
- E1b1a (V38, V100): 40 000 años,[5] en África Oriental.[9]
- E1b1 (P2): Originado en África Oriental.[8]
- E2 (M75, P68): 52 000 años,[5] origen africano.
- E1 (P147): Hace unos 51 000 años[5] originado en África.
Distribución[editar]
Se encuentra en todo África, donde es frecuentemente mayoritario. En África del Norte alcanza las frecuencias más altas en algunas etnias de Argelia y Túnez, como en los bereberes, alcanzando más del 90%.[12] En general en Argelia se encontró un 68%,[13] en Libia 92% y en Egipto 47%.[14] En el África subsahariana hay mayores frecuencias, encontrándose en Nigeria 100%,[15] Burkina Faso 99%,[16] en Guinea-Bisáu 97%,[17] Ghana 96%,[18] Senegal 96%,[19] Gambia 91%,[15] Ruanda 89%,[20] Somalia 83%[21] y Etiopía 49%.[22]
El subclado más difundido es E1b1a (V38), que es exclusivamente africano y tiene la mayor frecuencia en África occidental con un 80 %,[23] fue ampliamente disperso debido primero a la expansión bantú y luego por la diáspora africana durante la Edad Moderna.
Se expandió en épocas históricas y prehistóricas hacia el Cercano Oriente y Europa. El subclado E1b1b-M78, aparecido hace unos 18.600 años, se esparce por todo el Mediterráneo, por lo que es común en Europa y Cercano oriente, alcanzando una frecuencia del 27% en Grecia, Portugal (23%), Francia (3%), Irlanda (2%), Gales y Escocia (5%), y en la península ibérica, en Galicia (22%) y Castilla y León (16%). Alcanza la presencia más alta en la comunidad étnica de los pasiegos con 41%.
La distribución de los principales subclados puede resumirse de la siguiente manera:
| E (M96) |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Paragrupo E*[editar]
El paragrupo E* se refiere a E-96(xE1-P147, E2-M75) y se halla en forma relicta y muy disperso en África, encontrándose poco en amharas (Etiopía),[24] pigmeos y bantúes de Gabón y Camerún (África Central),[25] en África austral, Egipto[18] y Arabia Saudita.[24]
Dentro de E*(xE1-P147, E2-M75), se ha identificado al haplogrupo E-V44, el cual sería el clado hermano de E1 (P147). Así pues, E-V44 y E1-P147 descenderían del clado E-M5479 (o E-V3725).
- E-V44: Fue identificado en algunos hombres de África Oriental.[8]
- E-M5479*: Reportado en Etiopía.[5]
Haplogrupo E1[editar]
E1 (P147) es propio de toda África y extendido hacia Arabia y el Mediterráneo.
E1a[editar]
El haplogrupo E1a (M132), antes E1 (M33) está muy extendido en África occidental, especialmente en Malí con 34 %;[26] también en Burkina Faso, norte de Camerún, Senegal y Gambia. Frecuencias importantes se encontraron en los fulani de Camerún con 53 %,[16] los dogón de Mali 45%,[18] en Guinea-Bisáu 16%, especialmente en los diola con 34%.[27]
En menor frecuencia en Norte de África (Marruecos, Sudán, Egipto y saharauis) y en Europa (italianos y albaneses de Calabria).[3]
- E1a1 (M44): Característico de los fulani, especialmente en los de Camerún con 53 %.[16] Presente en Malí. Tiene diversidad en Europa, encontrándose especialmente en Italia, también Lituania, Inglaterra y otros; además del Líbano.[5]
- E1a2 (Z958/CTS10713): Común en los mandinga y fulas de Gambia. Encontrado en yorubas de Nigeria, mendés de Sierra Leona y en afrocaribeños.[5]
- E-Z36289: En Sierra Leona.[28]
E1b[editar]
E1b (P177), tendría dos subclados:
- E1b1 (P2/PF1940/PN2, P179) antes E3, de gran distribución. Presenta una importante dicotomía entre E1b1a (V38) y E1b1b (M215), los cuales están descritos en las secciones subsiguientes.
- E1b2 (P75), sin información reportada, aunque confirmado por Karafet et al. 2008.
E1b1a (V38)[editar]

E1b1a (V38/TSC0077541) es casi exclusivo y mayoritario en el África subsahariana.
- E1b1a1 (P1/PN1, M2/DYS271/SY81, M180, M291 y 261 marcadores más), antes E3a, es predominante en hablantes de lenguas nigero-congoleñas, tiene la mayor diversidad y alta frecuencia en África Occidental (80 %).[23] Es también mayoritario en la población negra de América, encontrándose en afroamericanos de EE. UU. un 62 %[29] y tiene menores frecuencias en Norte de África, Cuerno de África y Cercano Oriente. Alta frecuencia en los bamilekes con 100 %,[30] en los nande 100 %, ewé 97 %, ga 97 %, fante 84 %, ovambo 82 %, baganda 77 %, hereros 71 %,[18] yorubas 93%,[31] en pueblos bantoides del sur de Camerún como bamilekes, ewondo y bakaka promedia 93 %, en fulanis s de Burkina Faso 90 %,[16] en igbos 89 %, en hablantes de lenguas Cross de Nigeria 87%,[32] en mandingas 87 %,[17] en bantúes de Gabón 86%,[33] en Senegal 81 %,[34] en Bijagós 76 %, balantas 73 %, fulanis 73 %, nalúes 71 %,[17] en los luo 66 %,[18] wólof 68 %, zulúes 55 % y xhosas 54 %.[18] En Madagascar es el principal linaje con 35 %,[35] encontrándose en los tandroy de Androy 70 %, antanosy de Anosy 49 %, merina de Antananarivo 44 % y antaisaka de Atsimo-Atsinanana 37.5%.[36] En América es frecuente en Las Bahamas con 59%.[37]
- E-FT183: En Arabia Saudita[5]

- E-Y1705
- E1b1a1a1 (CTS1847, V43)
- E1b1a1a1a (M4732)
- M4732* en el Sahara Occidental[5]
- E-M4895
- E1b1a1a1a1 (M4706) de gran extensión, siendo importante en África Occidental, África Central, África austral, región de los Grandes Lagos de África y moderadamente al norte de África.
- E-BY142058 en Egipto[5]
- E1b1a1a1a1a (M58) En hutus de Ruanda (15 %),[20] Burkina Faso[16] y Sudáfrica[38]
- E1b1a1a1a1b (M10) Raro en bantúes de Tanzania y Centroáfrica. En Adamawa (Camerún)[38]
- E1b1a1a1a1c (L485)
- E-SK595 encontrado en Senegal, Gambia y Marruecos[5]
- E-Z36531 encontrado en Sudáfrica, Marruecos y Cataluña (España)[5]
- E-CTS9883 (Z36539) en los mende (Sierra Leona), wolof, fulas y mandingas de Gambia, fulas de Guinea, en Tamanrasset (Argelia), Senegal, Egipto, Marruecos y España.[5]
- E-L514
- E-L515 en Nigeria, Níger, Ghana, Sierra Leona, Burkina Faso, Camerún, Marruecos y afroamericanos[5]
- (M191, U186, U247) es el subclado de E1b1a más común y es típico del pueblos bantúes. Común en Camerún, Nigeria, Gabón y Congo,[39] en los igbos se encontró 54%, en ibibios 46%, en los anaang 38%,[40] en los gur de Burkina Faso,[41] en Sudáfrica está presente en los joisán, pero especialmente en pueblos bantúes;[42] presente en Kenia, Centroáfrica, disperso en el Cercano Oriente y común en afroamericanos, tanto del Caribe[5] como de Sudamérica[43] y Norteamérica.[44] Importante en los fon de Benín con 57% y en Ruanda 51% en los hutu y 48% en los tutsi.[30]
- E1b1a1a1a2 (M4231)
- E1b1a1a1a2a (U175) extendido en el África Occidental
- E-CTS1036 en Gambia, Sierra Leona y afroamericanos[5]
- E-M4254 extendido en Benín, Costa de Marfil, Nigeria, Sierra Leona, Gabón, Kenia, Etiopía, Camerún, Catar y en sandawes (Tanzania).[5]
- E-U290 en Costa de Marfil, Benín, en Nigeria 12%, en los ewondo de Camerún 58%,[39] y en afroestadounidenses 32.5%.[23]
- E1b1a1a1a2a (U175) extendido en el África Occidental
- E1b1a1a1a1 (M4706) de gran extensión, siendo importante en África Occidental, África Central, África austral, región de los Grandes Lagos de África y moderadamente al norte de África.
- E1b1a1a1a (M4732)
- E1b1a1a1 (CTS1847, V43)
- E-Y1705

- E1b1a2 (M329): Este haplogrupo es frecuente en el suroeste de Etiopía, especialmente entre las poblaciones de habla omótica.[45] Se encontró entre los oromos de Etiopía 2.6%[3] y está disperso en la península arábiga.[5] Se halló en Oromía (Etiopía) en restos de hace 4500 años.[5]
E1b1b (M215)[editar]

El haplogrupo E1b1b (M215/Page40/PF1942), inicialmente llamado E3b, es el haplogrupo más común en hablantes de lenguas afroasiáticas africanas (camíticas)[46] así como en muchos hablantes de lenguas nilo-saharianas y su mayor diversidad se encuentra en Etiopía. Está muy extendido en el cuerno de África, Norte de África, Sudán, bereberes, árabes y judíos askenazíes y sefardíes; también en el Sur de Europa, los Balcanes y gran parte de África subsahariana.
Las mayores frecuencias se encuentran en el Cuerno de África y África del Norte, encontrándose en Somalia 81%,[47] en Marruecos 79%, Túnez 72%,[14] Argelia 55%,[13] Etiopía 49%.[22], Libia 45%[48] y Egipto 44%.[14]
Étnicamente, se ha encontrado alta frecuencia entre bereberes del Sahara, como se puede observar en Túnez, donde se halló un 92.5%[12] y en Argelia 84%.[49] También predomina en los tuareg del Sahel, dando en Mali 91% y en los de Burkina Faso 78%.[50] En el África Oriental destacan los pueblos cushitas como los somalíes, los oromos (Etiopía) con 63%, los beja (Sudán) 52%[19] y los iraqw (Tanzania) con 56%.[18] Entre los pueblos nilo-saharianos se encontró que predomina entre los masalit (Chad) con 72%, los fur (Sudán) 59%[19] y los masái (especialmente en Kenia) 50%.[18] Fuera de África destacan los albaneses con 35%,[51] los griegos con 24%[52] y en general en los pueblos semitas, como por ejemplo los árabes jordanos con 31%.[53]
- E1b1b* poco en amharas de Etiopía y en Yemen
- E1b1b1 (M35.1/PF1944.1, M243)
- E1b1b1a (V68, L539)
- E1b1b1a* se encontró E-V68(xM78) en Cerdeña.[54]
- E1b1b1a1 (M78, L18) (antes E3b1a) común en el cuerno de África, especialmente en Somalia con 78%;[21] presente también en África del Norte, Cercano Oriente y sureste de Europa.
- E1b1b1a1a (Z1902/CTS10890)
- E-V12 principalmente al sur de Egipto (44 %),[10] menor frecuencia en Sudán, coptos y nubios
- E-M224 poco en yemeníes

Distribución de E1b1b-V32 según Cruciani et al. 2007. - E-FGC51357 en la península arábiga
- E-Y2863
- E-V32 típico de hablantes de lenguas cushitas como los borana de Kenia con 71 %,[55] en Somalia (52 a 77 %), en oromos de Etiopía (32 %) y en beyas de Sudán; también en algunos nilosaharianos como los masalit y fur de Darfur (Sudán),[19] en menor frecuencia en Chad y en otras zonas de África del Norte
- E-CTS693 encontrado disperso en Arabia Saudita, Egipto, Italia, Estados Unidos y aislado en Gales, Briansk (Rusia) y otras zonas de Europa y el Mediterráneo.[5]
- E-M224 poco en yemeníes
- E-Z22639
- E-V12 principalmente al sur de Egipto (44 %),[10] menor frecuencia en Sudán, coptos y nubios
- E1b1b1a1b (Z1919/CTS4231)
- E-L618
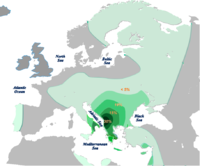
Distribución de E1b1b-V13 según Cruciani et al. 2007. - E-Y182141 encontrado en Argelia, Túnez y Cerdeña.[5]
- E-V13 (L142.1, V36) es típico de Europa y es el haplogrupo E con las mayores frecuencias fuera de África. Es importante en los Balcanes, especialmente en el Peloponeso griego (47 %),[3] en albaneses de Kosovo (46 %),[56] albaneses de Macedonia del Norte (34 %), también en Grecia, Bulgaria, Rumania, Serbia y Kursk; poco en el sur de Italia, en turcochipriotas y druzos. En Europa Occidental promedia 2.5%, en Anatolia 4% y en el Cercano Oriente 2%.[10] Se estima que bajas frecuencias encontradas en Pakistán provienen de antiguos soldados griegos y otras en Gran Bretaña provienen de soldados romanos de origen balcánico. En general, se considera que la presencia de V13 en el Mediterráneo europeo está inflído por la colonización griega.[57]
- E-V22 (CTS9547/L677) principalmente en el cuerno de África y Egipto. En los saho, pueblo cushita de Eritrea central presenta 88%.[8] Importantes frecuencias en los fulani (31 %)[17] y en los pueblos nilóticos chollo (13 %) y dinka (8 %). Común en el Cercano Oriente y norte de África; y menores frecuencias al sur de Europa, Europa Oriental y China.[5] Los levitas samaritanos presentan 100%.[58]
- E-L618
- E1b1b1a1e (M521) poco en Grecia[59]
- E1b1b1a1a (Z1902/CTS10890)
- E1b1b1a2 (CTS1339, V2729) en Sicilia, Cerdeña, Argelia, Marruecos, Camerún y Gambia.[5]
- E1b1b1b (Z827)
- E1b1b1b1 (L19/V257)
- E1b1b1b1* encontrado en bereberes, Sudoeste de Europa y Kenia.

Distribución de E-M81 - E1b1b1b1a (M81) predominante en la región del Magreb (80 %), especialmente en bereberes como los cabilios y otros; también en semitas como árabes y saharauis (76 %);[60] con algo de presencia en Iberia principalmente entre pasiegos (40 %)[55] y algo menos en Galicia y Portugal (12 %), Andalucía (9 %), Castilla y León (10 %), Cantabria (16 %), en Latinoamérica, y baja frecuencia en otras zonas del sur de Europa (6 %, Sicilia) y Cercano Oriente (4 %, Líbano y Turquía).
- E-M183
- E1b1b1b1b (PF2431) especialmente en África Occidental, Magreb y sudoeste de Europa.[5]
- E1b1b1b1* encontrado en bereberes, Sudoeste de Europa y Kenia.
- E1b1b1b2 (Z830/PF1961)
- E1b1b1b2a (PF1962)
- E-PF2025 en Irak, Cerdeña, Lituania y en pueblos eslavos.[5]
- E1b1b1b2a (PF1962)
- E1b1b1b1 (L19/V257)
- E1b1b1a (V68, L539)

- E-M123 común en el Cercano Oriente y sur de Europa.
- E-Y31991 poco en europeos y árabes.[5] Se ha encontrado E-123(xM34) en Omán, en israelíes de origen libio, en albaneses de Calabria (Italia) y menores frecuencias en Iberia, Egipto y Cercano Oriente. Encontrado al norte de Portugal y en Jordania.[53]
- E-M34 importante en semitas. Se halló en Malatya (Turquía) en restos de hace 5200 años, actualmente es común en el Cercano Oriente y está disperso en casi toda Europa.[5] Alta frecuencia en Etiopía y Cercano Oriente, en el área jordana del mar Muerto (31 %),[61] en Omán, Turquía, Israel (10 %), Yemen (8 %), en el sur de Europa (Galicia (10 %), Sicilia (7 %), Portugal) y en Norte de África. En Irán (3.5 %), especialmente en su población kurda con 14 %.[62]
- E-M123 común en el Cercano Oriente y sur de Europa.
- E1b1b1b2b (CTS10880) (antes E-V1515)
- E-Y139640 en la península arábiga.[5]
- E-Y5861
- E-Y5958
- E-V1700
- E1b1b2 (M281, V16) poco en oromos de Etiopía,[55] en Yemen y Arabia Saudita.[5]
Haplogrupo E2[editar]
E2 (M75, P68, CTS20 y otros 167 SNPs) es típico del África subsahariana, especialmente en Sudáfrica y en bantúes kenianos; también se encuentra en Burkina Faso, en hutus, tutsis, malgaches de Madagascar, khoisan, fon de Benín, iraqw de Tanzania, en Sudán, norte de Camerún y Senegal. Hay menores frecuencias fuera de África: en Catar, Omán y el resto de la península arábiga.
- E2* en bajas frecuencias en Namibia, Senegal, Zimbabue y en los mandinga (África Occidental).
- E2a (M41) con alta frecuencia en el pueblo alur con 67 % y en los hema 39%, ambos de R. D. del Congo.[18] En Etiopía 17%.[65] Desde el África Centro-Oriental hasta Sudán, especialmente en la región de los Grandes Lagos de África.
- E2b (M98) Destacan los xhosa de Sudáfrica con 27.5 %,[18] pero está extendido en toda el África Subsahariana, incluyendo Madagascar.
- E2b1 (M54, M90) Encontrado en los xhosa 27.5 %, rimaibe de Burkina Faso 22%, pigmeos mbuti 25%, daba (Camerún) 22%[16] y zulúes 21%.[18] Menores frecuencias (entre 9 y 15%) se encontró en sudafricanos (no joisánidos), bantús de Kenia, shirazíes de las Comoras, nilosaharianos del norte de Camerún y malgaches de Madagascar.
- E2b2 (CTS1441, CTS1307) Encontrado en los yorubas (Nigeria).[5]
Personajes famosos[editar]

E1b1a (V38): E1b1a en Nelson Mandela y en el faraón Ramsés III. En Barack Obama[66] y E1b1a1g en Desmond Tutu[67]
E1b1b (M215): Se ha encontrado el subclado E1b1b1b2* E-Z830 en Albert Einstein,[68] E1b1b1a2 (V13) en los Hermanos Wright, E1b1b1c1* (E-M34*) en Napoleón Bonaparte y William Harvey,[69] E1b1b1b en Adolf Hitler y en Zinedine Zidane.[70][71]
Véase también[editar]
| Adán cromosómico | ||||||||||||||||||||||||
| A | ||||||||||||||||||||||||
| BT | ||||||||||||||||||||||||
| B | CT | |||||||||||||||||||||||
| DE | CF | |||||||||||||||||||||||
| D | E | C | F | |||||||||||||||||||||
| C1 | C2 | G | H | IJK | ||||||||||||||||||||
| IJ | K | |||||||||||||||||||||||
| I | J | LT | K2 | |||||||||||||||||||||
| L | T | MS | P | NO | ||||||||||||||||||||
| M | S | Q | R | N | O | |||||||||||||||||||
| R1 | R2 | |||||||||||||||||||||||
| R1a | R1b | |||||||||||||||||||||||
Enlaces externos[editar]
- Y-DNA Haplogroup E and its Subclades ISOGG
- Learn about Y-DNA Haplogroup E y Y-DNA Phylogenetic Tree - Haplogroup E Genebase 2013 DNA Ancestry Project
- Mapa de E3a (o E1b1a)
- E-M293 (E1b1b1g o E1b1b1b2b), mapa.
- Genética poblacional del África subsahariana en haplogrupos del cromosoma Y
- Haplogroup_E1b1b y The Haplogroup E in Europe en Eupedia forum
Referencias[editar]
- ↑ a b Tyler-Smith, Chris; Xue, Yali; Thomas, Mark G.; Yang, Huanming; Arciero, Elena; Asan; Connell, Bruce A.; Jones, Abigail L. et al. (13 de junio de 2019). «A Rare Deep-Rooting D0 African Y-Chromosomal Haplogroup and Its Implications for the Expansion of Modern Humans out of Africa». Genetics (en inglés): genetics.302368.2019. ISSN 0016-6731. doi:10.1534/genetics.119.302368.
- ↑ Chiaroni J. et al 2009, Y chromosome diversity, human expansion, drift, and cultural evolution
- ↑ a b c d e f Semino Ornella et al 2004, Origin, Diffusion, and Differentiation of Y-Chromosome Haplogroups E and J: Inferences on the Neolithization of Europe and Later Migratory Events in the Mediterranean Area», American Journal of Human Genetics 74 (5): 1023–1034, doi:10.1086/386295, PMID 15069642
- ↑ a b Kamin M, Saag L, Vincente M, et al. (April 2015). «A recent bottleneck of Y chromosome diversity coincides with a global change in culture». Genome Research 25 (4): 459-466. PMC 4381518. PMID 25770088. doi:10.1101/gr.186684.114.
- ↑ a b c d e f g h i j k l m n ñ o p q r s t u v w x y z aa ab ac ad ae af ag ah ai aj ak al am an E Y-full tree Árbol YTree v9.01.00 2021 YFull™
- ↑ Haber M, Jones AL, Connel BA, Asan, Arciero E, Huanming Y, Thomas MG, Xue Y, Tyler-Smith C (June 2019). «A Rare Deep-Rooting D0 African Y-chromosomal Haplogroup and its Implications for the Expansion of Modern Humans Out of Africa». Genetics: genetics.302368.2019. doi:10.1534/genetics.119.302368.
- ↑ Martínez, B., Simão, F., Gomes, V. et al. Searching for the roots of the first free African American community. Sci Rep 10, 20634 (2020). https://doi.org/10.1038/s41598-020-77608-8
- ↑ a b c Trombetta B, D'Atanasio E, Massaia A, et al. Phylogeographic Refinement and Large Scale Genotyping of Human Y Chromosome Haplogroup E Provide New Insights into the Dispersal of Early Pastoralists in the African Continent. Genome Biol Evol. 2015;7(7):1940-1950. Published 2015 Jun 24. doi:10.1093/gbe/evv118
- ↑ a b c Trombetta, Beniamino 2011, A New Topology of the Human Y Chromosome Haplogroup E1b1 (E-P2): Revealed through the Use of Newly Characterized Binary Polymorphisms
- ↑ a b c d Cruciani et al. (2007), "Tracing Past Human Male Movements in Northern/Eastern Africa and Western Eurasia: New Clues from Y-Chromosomal Haplogroups E-M78 and J-M12", Molecular Biology and Evolution 24: 1300-1311, doi:10.1093/molbev/msm049
- ↑ Battaglia, Vincenza et al. “Y-chromosomal evidence of the cultural diffusion of agriculture in Southeast Europe.” European journal of human genetics : EJHG vol. 17,6 (2009): 820-30. doi:10.1038/ejhg.2008.249
- ↑ a b Ennafaa; Fregel; Khodjet-el-khil; Gonzalez (2011), "Mitochondrial DNA and Y-chromosome microstructure in Tunisia", Journal of Human Genetics, 56 (10): 734–41, doi:10.1038/jhg.2011.92, PMID 21833004
- ↑ a b Bekada A, Arauna LR, Deba T, Calafell F, Benhamamouch S, Comas D (2015) Genetic Heterogeneity in Algerian Human Populations. PLoS ONE 10(9): e0138453. https://doi.org/10.1371/journal.pone.0138453
- ↑ a b c Bekada, Asmahan et al. “Introducing the Algerian mitochondrial DNA and Y-chromosome profiles into the North African landscape.” PloS one vol. 8,2 (2013): e56775. doi:10.1371/journal.pone.0056775
- ↑ a b Poznik, G David et al. “Punctuated bursts in human male demography inferred from 1,244 worldwide Y-chromosome sequences.” Nature genetics vol. 48,6 (2016): 593-9. doi:10.1038/ng.3559
- ↑ a b c d e f g h Cruciani, Fulvio et al 2002, A Back Migration from Asia to Sub-Saharan Africa Is Supported by High-Resolution Analysis of Human Y-Chromosome Haplotypes American Journal of Human Genetics 70:1197–1214, 2002.
- ↑ a b c d Rosa et al. 2007 in a study of Guinea Bissau, showed that the Fulani there are about 10% E-M78. Note that this study did not test specifically for V12 or V22, so the E-M78 may have a different exact breakdown of diversity as well as a lower frequency.
- ↑ a b c d e f g h i j k Elizabeth T Wood, Daryn A Stover, Christopher Ehret et al., "Contrasting patterns of Y chromosome and mtDNA variation in Africa: evidence for sex-biased demographic processes," European Journal of Human Genetics (2005) 13, 867–876. (cf. Appendix A: Y Chromosome Haplotype Frequencies)
- ↑ a b c d Hassan, Hisham Y. et al. 2008 Y-Chromosome Variation Among Sudanese: Restricted Gene Flow, Concordance With Language, Geography, and History Archivado el 23 de septiembre de 2016 en Wayback Machine. American Journal of Physical Anthropology 137:316–323 (2008)
- ↑ a b J. R. Luis et al 2004, The Levant versus the Horn of Africa: Evidence for Bidirectional Corridors of Human Migrations
- ↑ a b Sánchez et al. 2005, High frequencies of Y chromosome lineages characterized by E3b1, DYS19-11, DYS392-12 in Somali males European Journal of Human Genetics 13 856–866
- ↑ a b Moran CN, Scott RA, Adams SM, Warrington SJ, Jobling MA, Wilson RH, Goodwin WH, Georgiades E, Wolde B, Pitsiladis YP. Y chromosome haplogroups of elite Ethiopian endurance runners. Hum Genet. 2004 Nov;115(6):492-7. doi: 10.1007/s00439-004-1202-y. Epub 2004 Oct 20. PMID: 15503146.
- ↑ a b c Sims et al. (2007), Sub-Populations Within the Major European and African Derived Haplogroups R1b3 and E3a Are Differentiated by Previously Phylogenetically Undefined Y-SNPs, HUMAN MUTATION Mutation in Brief #940 (2007) Online
- ↑ a b Abu-Amero, Khaled K et al. “Saudi Arabian Y-Chromosome diversity and its relationship with nearby regions.” BMC genetics vol. 10 59. 22 Sep. 2009, doi:10.1186/1471-2156-10-59
- ↑ Gemma Berniell-Lee et al 2009, Genetic and Demographic Implications of the Bantu Expansion: Insights from Human Paternal Lineages
- ↑ Peter A. Underhill, Peidong Shen, Alice A. Lin et al., "Y chromosome sequence variation and the history of human populations," Nature Genetics, Volume 26, November 2000
- ↑ Rosa, Alexandra et al. “Y-chromosomal diversity in the population of Guinea-Bissau: a multiethnic perspective.” BMC evolutionary biology vol. 7 124. 27 Jul. 2007, doi:10.1186/1471-2148-7-124
- ↑ a b Y-DNA Haplogroup E and its Subclades - 2019-2020 ISOGG
- ↑ Hammer, Michael F. et al 2005, Population structure of Y chromosome SNP haplogroups in the United States and forensic implications for constructing Y chromosome STR databases Archivado el 10 de enero de 2017 en Wayback Machine.
- ↑ a b Luis, J.R.; D. J. Rowold (2004 March). "The Levant versus the Horn of Africa: Evidence for Bidirectional Corridors of Human Migrations". The American Society of Human Genetics 74 (3): 532–44. doi:10.1086/382286. PMID 14973781. PMID 14973781. PMC 1182266.
- ↑ The International HapMap Consortium, A haplotype map of the human genome. Vol 437|27 October 2005|doi:10.1038/nature04226
- ↑ Veeramah, Krishna R et al. “Little genetic differentiation as assessed by uniparental markers in the presence of substantial language variation in peoples of the Cross River region of Nigeria.” BMC evolutionary biology vol. 10 92. 31 Mar. 2010, doi:10.1186/1471-2148-10-92
- ↑ Gemma Berniell-Lee, Francesc Calafell, Elena Bosch, Evelyne Heyer, Lucas Sica, Patrick Mouguiama-Daouda, Lolke van der Veen, Jean-Marie Hombert, Lluis Quintana-Murci, David Comas, Genetic and Demographic Implications of the Bantu Expansion: Insights from Human Paternal Lineages, Molecular Biology and Evolution, Volume 26, Issue 7, July 2009, Pages 1581–1589, https://doi.org/10.1093/molbev/msp069
- ↑ Semino et al. (2002) "Ethiopians and Khoisan share the deepest clades of the human Y-chromosome phylogeny". Am J Hum Genet 70:265–268
- ↑ Matthew E. Hurles et al 2005, The Dual Origin of the Malagasy in Island Southeast Asia and East Africa: Evidence from Maternal and Paternal Lineages
- ↑ Sergio Tofanelli, Stefania Bertoncini, Loredana Castrì, Donata Luiselli, Francesc Calafell, Giuseppe Donati, Giorgio Paoli, On the Origins and Admixture of Malagasy: New Evidence from High-Resolution Analyses of Paternal and Maternal Lineages, Molecular Biology and Evolution, Volume 26, Issue 9, September 2009, Pages 2109–2124, https://doi.org/10.1093/molbev/msp120
- ↑ Simms, Tanya M, "The peopling of the Bahamas: A phylogeographical perspective" (2011). ProQuest ETD Collection for FIU. AAI3472063.
- ↑ a b Underhill PA, Passarino G, Lin AA, Shen P, Mirazón Lahr M, Foley RA, Oefner PJ, Cavalli-Sforza LL (January 2001). "The phylogeography of Y chromosome binary haplotypes and the origins of modern human populations". Annals of Human Genetics. 65 (Pt 1): 43–62. doi:10.1046/j.1469-1809.2001.6510043.x. PMID 11415522. S2CID 9441236.
- ↑ a b Montano V, Ferri G, Marcari V, Batini C, Anyaele O, Destro-Bisol G, Comas D. The Bantu expansion revisited: a new analysis of Y chromosome variation in Central Western Africa. Mol Ecol. 2011 Jul;20(13):2693-708. doi: 10.1111/j.1365-294X.2011.05130.x. Epub 2011 Jun 1. PMID: 21627702.
- ↑ Veeramah, Krishna R et al. “Little genetic differentiation as assessed by uniparental markers in the presence of substantial language variation in peoples of the Cross River region of Nigeria.” BMC evolutionary biology vol. 10 92. 31 Mar. 2010, doi:10.1186/1471-2148-10-92
- ↑ de Filippo, Cesare et al. “Y-chromosomal variation in sub-Saharan Africa: insights into the history of Niger-Congo groups.” Molecular biology and evolution vol. 28,3 (2011): 1255-69. doi:10.1093/molbev/msq312
- ↑ Naidoo, Thijessen et al. “Development of a single base extension method to resolve Y chromosome haplogroups in sub-Saharan African populations.” Investigative genetics vol. 1,1 6. 1 Sep. 2010, doi:10.1186/2041-2223-1-6
- ↑ Hünemeier T, Carvalho C, Marrero AR, Salzano FM, Pena SD, Bortolini MC. Niger-Congo speaking populations and the formation of the Brazilian gene pool: mtDNA and Y-chromosome data. Am J Phys Anthropol. 2007 Jun;133(2):854-67. doi: 10.1002/ajpa.20604. PMID: 17427922.
- ↑ Sims LM, Garvey D, Ballantyne J. Sub-populations within the major European and African derived haplogroups R1b3 and E3a are differentiated by previously phylogenetically undefined Y-SNPs. Hum Mutat. 2007 Jan;28(1):97. doi: 10.1002/humu.9469. PMID: 17154278.
- ↑ Christopher Plaster et al. Y-DNA E subclades predicted by Passa
- ↑ Ehret et al. (2004), «The Origins of Afroasiatic», Science 306 (5702): 1680, doi:10.1126/science.306.5702.1680c.
- ↑ Sanchez, J., Hallenberg, C., Børsting, C. et al. High frequencies of Y chromosome lineages characterized by E3b1, DYS19-11, DYS392-12 in Somali males. Eur J Hum Genet 13, 856–866 (2005). https://doi.org/10.1038/sj.ejhg.5201390
- ↑ Triki-Fendri S, Sánchez-Diz P, Rey-González D, Ayadi I, Carracedo Á, Rebai A. Paternal lineages in Libya inferred from Y-chromosome haplogroups. Am J Phys Anthropol. 2015 Jun;157(2):242-51. doi: 10.1002/ajpa.22705. Epub 2015 Feb 11. PMID: 25677690.
- ↑ Abdeli A, Benhassine T. Paternal lineage of the Berbers from Aurès in Algeria: estimate of their genetic variation. Ann Hum Biol. 2019 Mar;46(2):160-168. doi: 10.1080/03014460.2019.1602166. Epub 2019 May 14. PMID: 30939942.
- ↑ Pereira, Luísa et al. “Linking the sub-Saharan and West Eurasian gene pools: maternal and paternal heritage of the Tuareg nomads from the African Sahel.” European journal of human genetics : EJHG vol. 18,8 (2010): 915-23. doi:10.1038/ejhg.2010.21
- ↑ Sarno, Stefania et al. “Shared language, diverging genetic histories: high-resolution analysis of Y-chromosome variability in Calabrian and Sicilian Arbereshe.” European journal of human genetics : EJHG vol. 24,4 (2016): 600-6. doi:10.1038/ejhg.2015.138
- ↑ Di Giacomo F, Luca F, Anagnou N, Ciavarella G, Corbo RM, Cresta M, Cucci F, Di Stasi L, Agostiano V, Giparaki M, Loutradis A, Mammi' C, Michalodimitrakis EN, Papola F, Pedicini G, Plata E, Terrenato L, Tofanelli S, Malaspina P, Novelletto A. Clinal patterns of human Y chromosomal diversity in continental Italy and Greece are dominated by drift and founder effects. Mol Phylogenet Evol. 2003 Sep;28(3):387-95. doi: 10.1016/s1055-7903(03)00016-2. PMID: 12927125.
- ↑ a b Flores, C., Maca-Meyer, N., Larruga, J.M. et al. Isolates in a corridor of migrations: a high-resolution analysis of Y-chromosome variation in Jordan. J Hum Genet 50, 435–441 (2005). https://doi.org/10.1007/s10038-005-0274-4
- ↑ Trombetta, Beniamino et al. “A new topology of the human Y chromosome haplogroup E1b1 (E-P2) revealed through the use of newly characterized binary polymorphisms.” PloS one vol. 6,1 e16073. 6 Jan. 2011, doi:10.1371/journal.pone.0016073
- ↑ a b c d Cruciani et al. 2004, Phylogeographic Analysis of Haplogroup E3b (E-M215) Y Chromosomes Reveals Multiple Migratory Events Within and Out Of Africa. Archivado el 26 de junio de 2008 en Wayback Machine. American Journal of Human Genetics 74 1014–1022
- ↑ Peričic et al. (2005), «High-resolution phylogenetic analysis of southeastern Europe traces major episodes of paternal gene flow among Slavic populations», Mol. Biol. Evol. 22 (10): 1964-75, doi:10.1093/molbev/msi185, PMID 15944443..
- ↑ King, Roy J et al. “The coming of the Greeks to Provence and Corsica: Y-chromosome models of archaic Greek colonization of the western Mediterranean.” BMC evolutionary biology vol. 11 69. 14 Mar. 2011, doi:10.1186/1471-2148-11-69
- ↑ Shen P, Lavi T, Kivisild T, Chou V, Sengun D, Gefel D, Shpirer I, Woolf E, Hillel J, Feldman MW, Oefner PJ. Reconstruction of patrilineages and matrilineages of Samaritans and other Israeli populations from Y-chromosome and mitochondrial DNA sequence variation. Hum Mutat. 2004 Sep;24(3):248-60. doi: 10.1002/humu.20077. PMID: 15300852.
- ↑ Battaglia et al. 2008 "Y-chromosomal evidence of the cultural diffusion of agriculture in southeast Europe" European Journal of Human Genetics.
- ↑ Bosch et al. 2001 High-resolution analysis of human Y-chromosome variation shows a sharp discontinuity and limited gene flow between north-western Africa and the Iberian Peninsula. Am J Hum Genet 68 1019–1029
- ↑ Flores et al. 2005 "Isolates in a corridor of migrations: a high-resolution analysis of Y-chromosome variation in Jordan" J Hum Genet 50 435–441
- ↑ Grugni, Viola et al 2012, Ancient Migratory Events in the Middle East: New Clues from the Y-Chromosome Variation of Modern Iranians PLoS ONE 7(7): e41252. doi:10.1371/journal.pone.0041252
- ↑ Henn et al. 2008 "Y-chromosomal evidence of a pastoralist migration through Tanzania to southern Africa"
- ↑ Cluster Name : E-V42 Haplozone::E-M35 Project Data 2013
- ↑ Underhill, P., Shen, P., Lin, A. et al. Y chromosome sequence variation and the history of human populations. Nat Genet 26, 358–361 (2000).
- ↑ Y-DNA de Barack Obama
- ↑ Schuster et al (2010-02-18). "Bantu genomes from southern Africa". Nature
- ↑ Y Haplogroup Project - Y-DNA SNP
- ↑ What Is Your yDNA Haplogroup?
- ↑ DNA genealogists surprised that Hitler's Y chromsome DNA is African
- ↑ Y-DNA de Zinedine Zidane



