Plásmido
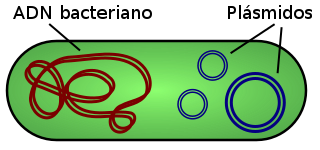
Los plásmidos son moléculas de ADN extracromosómico generalmente circular que se replican de manera autónoma y se transmiten (esto último por un proceso llamado conjugación) independientemente del ADN cromosómico.[1][2] Están presentes principalmente en los procariotas (bacterias y arqueas).[1] En los eucariotas solo se encuentran en las levaduras, pero también se han detectado plásmidos inactivos en las mitocondrias y los cloroplastos, puesto que estos orgánulos son de origen bacteriano.[3] No son infecciosos (patógenos). Son ejemplos de elementos genéticos móviles; junto con los transposones y los virus constituyen vehículos para la transferencia horizontal de genes.
Los plásmidos están físicamente separados del cromosoma del huésped. Tienen un tamaño de 3 a 10 kb y pueden portar un número de 30 a 100 genes. El número de plásmidos por célula puede variar, dependiendo del tipo, desde una sola copia hasta algunos cientos. Los vectores plasmídicos permiten clonar ligandos de ADN exógeno (exterior a la célula) de hasta 4 kb ya que un tamaño mayor a este dificulta la clonación en estos vectores.
Los plásmidos son entidades acelulares, de esta manera son similares a los virus y viroides en tener el mismo comportamiento replicativo, transmisión entre huéspedes y ser vectores de genes. A pesar de que los plásmidos, virus y viroides sean "replicadores" y evolucionen generalmente no se clasifican como seres vivos.[4] En comparación con los virus y viroides que dependen del genoma del huésped para ser replicados, los plásmidos se replican independientemente del genoma, por tanto los plásmidos cumplirían con un atributo de vida: la reproducción. Los plásmidos están filogenéticamente emparentados con los virus de ADN como Monodnaviria y algunos virus satélites de ADN puesto que ambos comparten una proteína única denominada Rep que les permite una replicación en círculo rodante y que no tiene homología con proteínas celulares, lo que sugiere que los plásmidos y los virus pueden tener orígenes anteriores a las células. También es posible según análisis de la proteína Rep que algunos plásmidos sean precursores de los virus de ADN del taxón Monodnaviria con la obtención de proteínas de ciertos virus para fabricar la cápside.[5] Algunos plásmidos también pudieron evolucionar de virus de ADN según análisis filogenéticos.[5]
En contraste con los virus y viroides que son parásitos intracelulares, los plásmidos son endosimbiontes mutualistas. A veces establecen relaciones beneficiosas con su célula huésped pero manteniendo su independencia en la replicación. No obstante en otras ocasiones pueden originar tumores. Portan genes de resistencia a antibióticos. A pesar de que se les considere un elemento genético de sus huéspedes, no siempre suelen estar dentro de ellos.
El término plásmido fue introducido por el biólogo molecular estadounidense Joshua Lederberg en 1952.[6] Hubo que ir afinando la definición con el tiempo, ya que en un principio se había descrito de tal manera que incluía a los bacteriófagos.[7] Los plásmidos solo pueden coexistir como una o más copias en cada bacteria, debido a la división celular pueden perderse en una de las bacterias segregadas.[8]
Pueden ser introducidos en las células bacterianas por un proceso de transformación, por eso se utilizan como vectores de clonación.
Todos los vectores de clonación deben contener, al menos:
- Un origen de replicación para poder tener más de una copia del mismo en la célula infectada.
- Dos genes que confieran resistencia a diferentes antibióticos (cloranfenicol y ampicilina), lo que permite la identificación de las células que portan a dicho vector.
Las moléculas de ADN plasmídico, adoptan una conformación tipo doble hélice al igual que el ADN de los cromosomas, aunque, por definición, se encuentran fuera de los mismos. Se han encontrado plásmidos en casi todas las bacterias. A diferencia del ADN cromosómico, los plásmidos no tienen proteínas asociadas.[9]
En general, no contienen información esencial, sino que confieren ventajas al hospedador en condiciones de crecimiento determinadas. El ejemplo más común es el de los plásmidos que contienen genes de resistencia a un determinado antibiótico, de manera que el plásmido únicamente supondrá una ventaja en presencia de ese antibiótico.[10]
Hay algunos plásmidos integrativos, es decir, plásmidos que tienen la capacidad de insertarse en el cromosoma bacteriano. Estos rompen momentáneamente el cromosoma y se sitúan en su interior, con lo cual la maquinaria celular también reproduce el plásmido automáticamente. Cuando ese plásmido se ha insertado se les da el nombre de episoma.[cita requerida]
Los plásmidos se utilizan como vectores de clonación en ingeniería genética por su capacidad de reproducirse de manera independiente del ADN cromosomal así como también porque es relativamente fácil manipularlos e insertar en ellos nuevas secuencias genéticas.
Los plásmidos usados en ingeniería genética suelen contener uno o dos genes que les confieren resistencia a antibióticos y permiten seleccionar clones recombinantes. Hay otros métodos de selección además de la resistencia a antibióticos, como los basados en fluorescencia o en proteínas que destruyen las células sin uso de antibióticos. Estos nuevos métodos de selección de plásmidos son de uso frecuente en la agrobiotecnología, debido a la fuerte crítica de grupos ecologistas por la posibilidad de que haya antibióticos en los organismos modificados genéticamente.[11]
Algunos plásmidos incluyen un sistema de adición o sistema de muerte postsegregacional (PSK, de postsegregational killing system). Estos producen en conjunto un veneno de larga vida y un antídoto de vida corta.[12][13] Las células hija que retienen una copia del plásmido sobreviven, mientras que una célula hija que falla al integrar el plásmido muere o sufre una reducida tasa de crecimiento debido al veneno que obtuvo de la célula progenitora. Este es un ejemplo de plásmidos como el ADN replicante.[14]
Desde un punto de vista evolutivo, los plásmidos se consideran elementos genéticos egoístas (o parasíticos), ya que en muchos casos no comparten intereses evolutivos con el cromosoma de la célula en la que se encuentran. Los plasmidos contienen genes por su propio mantenimiento y transmisión, los cuales pueden representar una carga fisiológica extra para la célula. A este fenómeno se le conoce como coste en el fitness bacteriano.[15] Sin embargo, los plásmidos también pueden conferir funciones muy beneficiosas para las células en determinadas condiciones, como genes de resistencia a antibióticos en un ambiente de selección con estas moléculas. Además, estudios recientes han demostrado que los genes ubicados en plásmidos evolucionan de una forma distinta a la que lo hacen genes ubicados en los cromosomas. Los plásmidos también favorecen la evolución de sus huéspedes bacterianos gracias a otros elementos móviles que portan, como secuencias de inserción, que pueden provocar mutaciones al saltar y romper genes del cromosoma.[16] Esto sugiere que las implicaciones evolutivas de los plásmidos van mucho más allá de la de ser meros vehículos de transmisión.[17]
Epísomas
[editar]
Un epísoma es un plásmido que puede integrarse por sí mismo al ADN cromosomal del organismo huésped.[18] Por esta razón, puede mantenerse en contacto por un largo tiempo, ser duplicado en cada división celular del huésped y volverse parte básica de su mapa genético. Este término no se usa más en plásmidos, debido a que ahora está claro que una región homóloga con el cromosoma elabora un plásmido dentro de un epísoma.[19]
Los plásmidos usados en ingeniería genética se denominan “vectores”. Se utilizan para transferir genes desde un organismo a otro y típicamente contienen un marcador genético confiriendo un fenotipo el cual puede ser seleccionado a favor o en contra.[20] La mayoría también contienen un polivinculador o sitio de clonado múltiple (MCS),[21] el cual es una pequeña región que contiene los sitios de restricción más comúnmente usados, permitiendo una fácil inserción de fragmentos de ADN en ese lugar.[22]
Tipos
[editar]Una forma de agrupar plásmidos es por su habilidad de transferirse a otra bacteria.[23] Los plásmidos conjugativos contienen “tra-genes”, los cuales ejecutan complejos procesos de conjugación, como la transferencia sexual de plásmidos a otra bacteria. Los plásmidos no-conjugativos, son incapaces de iniciar una conjugación, de allí que ellos pueden transferirse únicamente con la asistencia de los plásmidos conjugativos y lo hacen “por accidente”. Una clase intermedia de plásmidos son los “movilizables” los cuales llevan solo un subtipo de genes requeridos para la transferencia. Ellos pueden “parasitar” un plásmido conjugativo, transfiriéndose a una alta frecuencia solo en su presencia.
Es posible para plásmidos de diferentes tipos el coexistir en una célula simple.
Siete tipos diferentes de plásmidos han sido encontrados en E.Coli. Pero normalmente plásmidos relacionados son incompatibles, en el sentido de que solo uno de ellos sobrevive en la línea celular, debido a la regulación de las funciones vitales de los plásmidos. Por lo tanto, los plásmidos pueden ser diferenciados de acuerdo a grupos de compatibilidad.
Otra forma de clasificar plásmidos es por función. Hay 6 clases principales:
Plásmidos de fertilidad
[editar]También se conocen como factores F[19] los cuales contienen tra-genes, son capaces de conjugarse. Desempeña un importante papel con la conjugación de E. coli. además de haber sido el primero en ser descrito tiene una longitud aproximada de 10 Kb. contiene genes responsables de la unión a la célula, y de la transferencia del plásmido ubicado entre cepas bacterianas específicas en el proceso de conjugación. Gran parte del conjunto de la información para la transferencia de plásmidos se encuentra ubicada en el operón tra, el cual contiene menos de 28 genes. Estos genes dirigen la formación de pili sexuales que unen a una célula donadora, a una receptora, otros genes en cambio colaboran en la transferencia de ADN. También contienen segmentos denominados secuencias de inserción, colaboran en la inserción del plásmido en el cromosoma y en la célula del huésped, por lo que puede existir fuera del cromosoma bacteriano o estar integrado en él.
Plásmidos de resistencia
[editar]
Se conocen como factores R, otorgan resistencia a ciertos antibióticos a los huéspedes,[24] contienen de manera singular genes que codifican enzimas capaces de destruir o modificar antibióticos, normalmente no están integrados en el cromosoma de la bacteria que lo contiene, se han encontrado en los plásmidos genes que codifican la resistencia a antibióticos como la ampicilina, el cloranfenicol y la kanamicina, entre otros, algunos plásmidos R contienen un solo gen de resistencia otros en cambio llegan a tener hasta 8, con frecuencia los genes de resistencia se encuentran en un elemento de transposición de forma que las cepas bacterianas se pueden desarrollar con rapidez plásmido que codifican resistencias múltiples. Como muchos plásmidos de resistencia son a su vez plásmidos de conjugación pueden propagarse por toda una población aunque con menor rapidez que el plásmido de fertilidad. Con frecuencia, los factores R no conjugativos, pasan de una bacteria a otra durante la conjugación promovida por otro plásmido, con este método toda una población puede hacerse resistente a los antibióticos. De hecho el que algunos de estos plásmidos se puedan transferir fácilmente entre especies, promueve aún más la propagación de resistencias. Cuando el huésped consume grandes cantidades de antibióticos se selecciona bacterias con factores R y se hacen más prevalentes, los factores R pueden entonces ser transferidos a géneros más patógenos como Salmonella entre otros producir mayores problemas de salud pública. Un buen ejemplo de la importancia de los plásmidos con genes de resistencia a antibióticos en ambientes clínicos es la alta prevalencia del plásmido pOXA-48 en enterobacterias de pacientes hospitalizados[25].
Col-plásmidos
[editar]Las bacterias también albergan plásmidos con genes que les proporcionan una ventaja competitiva, en el mundo de los microbios, las bacteriocinas son proteínas que destruyen otras bacterias, pueden actuar solamente contra cepas estrechamente relacionadas, o en ocasiones destruyen las células generando poros en la membrana plasmática o degenerando la pared celular, provocando de este modo que se incremente su permeabilidad, otro proceso para destruir la célula es degradando el ADN Y ARN o atacando al peptidoglicano, los plásmidos col[26] contienen genes para la síntesis de bacteriocinas conocidas como colicinas que están dirigidas contra la e coli, existen plásmidos con características parecidas, las cuales contienen genes que codifican bacteriocinas dirigidas contra otras especias por ejemplo producen cloacinas que destruyen especies de enterobacter, el huésped no se ve afectado por las bacteriocinas que produce. Algunos plásmidos Col son conjugativos y contienen genes de resistencia.
Plásmidos degradativos
[editar]Estos plásmidos habilitan la digestión de sustancias inusuales como tolueno o ácido salicílico.[cita requerida]
Plásmidos virulentos
[editar]Estos plásmidos convierten la bacteria en un patógeno. Son capaces de producir dos tipos de toxinas, una toxina termolábil (LT) que es una proteína de gran tamaño muy similar en cuanto a estructura y a mecanismo de acción a la toxina del cólera, y una toxina termoestable (ST),
Plásmidos metabólicos
[editar]Poseen genes para que algunas cepas de rizhobium induzcan a la nodulación de las legumbres y lleven a cabo la fijación del nitrógeno.
Conformaciones
[editar]El ADN plásmido puede aparecer en uno de cinco conformaciones, las cuales (para un tamaño dado) corren a diferentes velocidades en un gel durante electroforesis.[27] Las conformaciones se muestran abajo en orden de movilidad electroforética (velocidad para un voltaje dado), del más lento al más rápido:
Mellado abierto circular
[editar]El ADN tiene un solo corte filamentario.
Lineal
[editar]El ADN tiene terminales libres, ya sea porque los filamentos fueron cortados o porque el ADN era linear in vivo. Usted puede modelar este como un cordón que no se ha conectado a sí mismo.
Circular relajado
[editar]El ADN que interactúa completamente con ambos filamentos sin cortar, pero que ha sido enzimáticamente “relajado”. Usted puede modelar este dejando un cordón relajado y luego conectándolo a sí mismo.
Superespiral desnaturalizado
[editar]El ADN como el ADN superespiral o superenrollado, pero que tiene regiones sin unir que lo hacen ligeramente menos compacto; esto resulta de una excesiva alcalinidad durante la preparación del plásmido.
ADN superespiral
[editar]Es un ADN totalmente intacto con los filamentos sin cortar, y con forma de remolino, resultando en una forma compacta.
La tasa de migración de pequeños fragmentos lineales es directamente proporcional al voltaje aplicado (en el caso de voltajes bajos). En el caso de altos voltajes, grandes fragmentos migran continuamente a diferentes tasas. Por lo tanto, la resolución del gel decrece con el incremento del voltaje.
A un bajo voltaje determinado, la migración de un pequeño fragmento lineal de ADN está en función de su longitud. Fragmentos lineales largos (de 20kb) migran a cierta tasa sin importar la longitud. Esto se debe a que las moléculas “reptan” desde el centro de la molécula siguiendo el sentido de la matriz de gel.
La digestión restrictiva se usa frecuentemente para analizar fragmentos purificados de plásmidos. Estas enzimas rompen específicamente el ADN en ciertas secuencias cortas.
Aplicaciones
[editar]Clonación del ADN
[editar]
La clonación del ADN es una técnica fundamental para la obtención de grandes cantidades de un fragmento de ADN concreto, este fragmento primero debe ser unido a un ADN vector antes de ser clonado. El ADN vector es un vehículo que se utiliza para transportar ADN extraño a una célula huésped, el vector contiene secuencias que le permiten duplicarse dentro de esta célula huésped.
En una técnica el segmento de ADN que va a ser clonado es introducido a un plásmido y luego unido a una célula bacteriana, en ese momento la bacteria capta el plásmido del medio. En otra técnica alternativa el segmento de ADN se une a un fragmento del genoma del virus bacteriano lambda, luego este virus infecta a un cultivo de células bacterianas, obteniendo así una gran cantidad de virus, siendo cada virus contenedor del fragmento de ADN extraño.
En cualquiera de las dos técnicas mencionadas, una vez que el fragmento de ADN extraño se encuentra en el interior de la bacteria será duplicado junto con el ADN bacteriano o viral, y repartido a las células hijas. De este modo el número de moléculas de ADN recombinante aumenta en proporción al número de células que se forman. De modo que si iniciáramos con una sola célula en poco tiempo tendríamos millones de copias de ADN. Cuando se alcanza la cantidad de copias necesarias se puede purificar el ADN recombinante y este podrá ser utilizado en otros procesos. Además de proporcionar un medio para amplificar la cantidad de secuencia de ADN particular la clonación también puede ser utilizada como técnica para aislar en forma pura cualquier fragmento de ADN específico en una población heterogénea de moléculas de ADN.
Clonación de ADN eucariota en plásmidos bacterianos
[editar]El ADN extraño que debe clonarse es introducido en el plásmido para formar una molécula de ADN recombinante, los plásmidos usados para la clonación de ADN son versiones modificadas, de los que se pueden observar en las células bacterianas. De la misma manera sus contrapartes naturales de las cuales derivan, poseen una origen de duplicación y uno o más genes que confieren a la célula receptora resistencia a antibióticos, la resistencia los antibióticos permite selecciona las células que contienen el plásmido recombinante.[28]
Las bacterias que pueden captar el ADN de un medio constituyen una base para la clonación de plásmidos en células bacterianas. En este procedimiento a un cultivo que ha sido pretratado con iones de calcio se le adicionan plásmidos recombinantes. Cuando el plásmido ha sido captado este se duplica de manera autónoma en el interior de la célula receptora. Las células bacterianas que contienen el plásmido se pueden seleccionar porque se desarrollan en presencia del antibiótico contra el cual deben ser resistentes. Cuando se alcanza la cantidad de amplificación deseada se extrae el ADN, el cual puede ser separado con facilidad del plásmido de ADN recombinante. Además pueden tratarse plásmidos recombinantes aislados con la misma enzima restrictiva que se utilizó en su síntesis, la cual libera los segmentos de ADN clonados del resto de ADN que sirvió como Vector. Posteriormente el ADN clonado se puede separar del plásmido.[29]
Uno de los principales beneficios de la clonación del ADN es que además de producir grandes cantidades una parte específica de ADN permite separar diferentes ADN en una mezcla. Inicialmente se notó que las bacterias que poseen plásmidos se pueden separar con tratamientos con antibióticos, cuando se realiza este tratamiento se pueden sedimentar a baja densidad en placas Petri de tal manera que la progenie de una célula permanece separa de la progenie de otra célula. Como existe una gran cantidad de plásmidos recombinantes, las diferentes células sobre el plato el plato de cultivo poseen diversos fragmentos de ADN extraños.
En los platos de cultivos que poseen las colonias bacterianas se investiga la presencia de una secuencia de ADN particular, empleando técnicas combinadas de duplicación de placas e hibridación in situ. Esta duplicación permite la preparación de un gran número de platos de cultivos que contienen colonias representativas de una misma célula bacteriana, y están posicionadas de la misma manera en cada plato. Se utiliza una de las réplicas para la localización de una secuencia de ADN, en este procedimiento se requiere lisar las células y fijar el ADN sobre la superficie, de un filtro cuando el ADN se encuentra fijado el ADN se desnaturaliza para la hibridación in situ, durante el cual el filtro es incubado con una sonda de ADN marcado que posee la secuencia complementaria buscada, posteriormente con la sonda no hibridad se lava y se determina la localización de los híbridos marcados mediante autorradiografía, solo entonces se seleccionaran los representativos identificados vivientes de la clonación.
Pese a que es fácil la búsqueda de un solo gen humano usando este procedimiento, no se trata de un gen práctico debido a que se requiere de cientos de placas de Petri preparadas.
Genética
[editar]
Los plásmidos sirven como una importante herramienta en laboratorios de genética e ingeniería bioquímica, donde son comúnmente usados para multiplicar (hacer muchas copias de) o como genes particulares expresos. Muchos plásmidos están disponibles comercialmente para dichos usos.[30][31]
El gen que va a replicarse se inserta en copias de un plásmido el cual contiene genes que hacen células resistentes a un antibiótico en particular. En el paso siguiente el plásmido es insertado en la bacteria por medio de un proceso llamado transformación. Luego, la bacteria es expuesta a un antibiótico particular. Solo la bacteria que toma copias del plásmido sobrevive al antibiótico debido a que el plásmido lo hace resistente.[31] En particular, los genes protectores son expresados (usados para hacer proteína) y la proteína expresada evita la acción del antibiótico. De esta forma, los antibióticos actúan como un filtro que seleccionan únicamente la bacteria modificada. Ahora, estas bacterias pueden ser cultivadas en largas cantidades, cosechadas y el plásmido de interés puede ser aislado.
Otro uso importante de los plásmidos es fabricar grandes cantidades de proteínas. En este se deja crecer la bacteria que contiene el plásmido que encierra al gen de interés. Solo como la bacteria produce la proteína que le confiere si resistencia a los antibióticos, este también puede ser usado para producir proteínas en grandes cantidades desde el gen insertado. Esta es una forma barata y fácil de producir genes o proteínas que este codifica de forma masiva, como por ejemplo insulina, o inclusive antibióticos.
Extracción del ADN plasmídico
[editar]En el maxiprep se cultivan volúmenes mucho más grandes de bacterias en suspensión. Esencialmente, este es un escalado de la preparación mini-prep, el cual es seguido por una purificación adicional. Esto resulta en una cantidad relativamente grande (0,5 -1 mg) de ADN plásmido muy puro.[32]
En los últimos tiempos muchos kits comerciales han sido creados para realizar la extracción plasmídica a varias escalas, purezas y niveles de automatización.
Resistencia a los antibióticos
[editar]Los plásmidos a menudo contienen genes o paquetes de genes que les confieren una ventaja selectiva, lo que les da la habilidad de hacer a la bacteria resistente a uno o varios antibióticos. Cada plásmido contiene al menos una secuencia de ADN que sirve como un origen de replicación u ORI (un punto inicial para la replicación del ADN), lo cual habilita al ADN para ser duplicado independientemente del ADN cromosomal.
La adquisición de genes necesarios para elaborar estos mecanismos de defensa se ve favorecida por una variedad de sistemas en los que se transfiere genes de manera mezclada, tales como plásmidos conjugativos bacterianos, elementos transponibles y sistemas integrón, que mueven genes de un sistema de ADN a otro y de una célula bacteriana a otra, sin necesidad de que exista una relación con el donante de los genes.[33]
Los plásmidos bacterianos sirven como el andamio sobre el cual están montados arreglos de genes de resistencia a antibióticos, por transposición (elementos transponibles y transposición mediada por ISCR) y mecanismos de recombinación específicos de sitio (casetes de genes integrón).[34]
Véase también
[editar]- bacteria
- conversión lisogénica
- cósmido
- cromosoma artificial bacteriano
- fagémido
- nanobio
- prion
- provirus
- viroide
- virus
Referencias
[editar]- ↑ a b Betancor; Gadea; Flores (2008). «Genética Bacteriana». Temas de Bacteriología y Virología Médica. Consultado el 1 de diciembre de 2016.
- ↑ Betancor; Gadea; Flores (2008). «Genética Bacteriana». Temas de Bacteriología y Virología Médica. Consultado el 1 de diciembre de 2016.
- ↑ Mitochondrial plasmids. Springer Link.
- ↑ Sinkovics J, Horvath J, Horak A (1998). «The origin and evolution of viruses (a review)». Acta Microbiologica et Immunologica Hungarica 45 (3–4): 349-90. PMID 9873943.
- ↑ a b Koonin EV, Dolja VV, Krupovic M, Varsani A, Wolf YI, Yutin N, Zerbini M, Kuhn JH (18 de octubre de 2019). «Create a megataxonomic framework, filling all principal taxonomic ranks, for ssDNA viruses» (docx). International Committee on Taxonomy of Viruses (en inglés). Consultado el 27 de mayo de 2020.
- ↑ Lederberg J (1952). «Cell genetics and hereditary symbiosis». Physiol 32 (4): 403-30.
- ↑ Casali, Nicola.; Preston, Andrew, 1969- (2003). E. coli plasmid vectors : methods and applications. Humana Press. ISBN 978-1-59259-409-2. OCLC 54464053. Consultado el 11 de junio de 2020.
- ↑ Lewin, Benjamin (1 de enero de 1996). Genes. Volumen 1. Reverte. ISBN 9788429118452.
- ↑ Karp, Gerald (9 de marzo de 2011). BIOLOGIA CELULAR Y MOLECULAR. McGraw-Hill Interamericana de España S.L. ISBN 9786071505040.
- ↑ Cirz, Ryan T.; Chin, Jodie K.; Andes, David R.; Crécy-Lagard, Valérie de; Craig, William A.; Romesberg, Floyd E. (10 de mayo de 2005). «Inhibition of Mutation and Combating the Evolution of Antibiotic Resistance». PLOS Biology 3 (6): e176. ISSN 1545-7885. PMC 1088971. PMID 15869329. doi:10.1371/journal.pbio.0030176.
- ↑ Echenique, Viviana (1 de enero de 2004). Biotecnología y mejoramiento vegetal[. Instituto Nacional de Tecnología Agropecuaria. ISBN 9789875211384.
- ↑ Thomas, Christopher M. (2 de septiembre de 2003). Horizontal Gene Pool: Bacterial Plasmids and Gene Spread (en inglés). CRC Press. ISBN 9780203304334.
- ↑ Gerdes, Kenn (19 de octubre de 2012). Prokaryotic Toxin-Antitoxins (en inglés). Springer Science & Business Media. ISBN 9783642332531.
- ↑ Merten, Otto-Wilhelm; Mattanovich, D.; Lang, Christine; Larsson, G.; Neubauer, P.; Porro, D.; Postma, P.; Mattos, J. Teixeira de et al. (17 de abril de 2013). Recombinant Protein Production with Prokaryotic and Eukaryotic Cells. A Comparative View on Host Physiology: Selected articles from the Meeting of the EFB Section on Microbial Physiology, Semmering, Austria, 5th–8th October 2000 (en inglés). Springer Science & Business Media. ISBN 9789401597494.
- ↑ DelaFuente, Javier; Toribio-Celestino, Laura; Santos-Lopez, Alfonso; León-Sampedro, Ricardo; Alonso-del Valle, Aida; Costas, Coloma; Hernández-García, Marta; Cui, Lun et al. (2022-12). «Within-patient evolution of plasmid-mediated antimicrobial resistance». Nature Ecology & Evolution (en inglés) 6 (12): 1980-1991. ISSN 2397-334X. doi:10.1038/s41559-022-01908-7. Consultado el 21 de diciembre de 2022.
- ↑ Sastre-Domínguez, Jorge; DelaFuente, Javier; Toribio-Celestino, Laura; Herencias, Cristina; Herrador-Gomez, Pedro; Costas, Coloma; Hernandez-Garcia, Marta; Canton, Rafael; Rodriguez-Beltran, Jeronimo; Santos-Lopez, Alfonso; San Millan, Alvaro (2024). «Plasmid-encoded insertion sequences promote rapid adaptation in clinical enterobacteria». Nature Ecology & Evolution 8: 2097-2112. doi:10.1038/s41559-024-02523-4.
- ↑ Rodríguez-Beltrán, Jerónimo; DelaFuente, Javier; León-Sampedro, Ricardo; MacLean, R. Craig; San Millán, Álvaro (2021-06). «Beyond horizontal gene transfer: the role of plasmids in bacterial evolution». Nature Reviews Microbiology (en inglés) 19 (6): 347-359. ISSN 1740-1534. doi:10.1038/s41579-020-00497-1. Consultado el 21 de diciembre de 2022.
- ↑ Inc, Encyclopaedia Britannica (1 de junio de 2011). Britannica Enciclopedia Moderna. Encyclopaedia Britannica, Inc. ISBN 9781615355167.
- ↑ a b Pierce, Benjamin A. (20 de enero de 2017). Genética: Un enfoque conceptual. Ed. Médica Panamericana. ISBN 9788498352160.
- ↑ PhD, Y. H. Hui; Khachatourians, George G. (18 de enero de 1995). Food Biotechnology: Microorganisms (en inglés). John Wiley & Sons. ISBN 9780471185703.
- ↑ Schleef, Martin (14 de febrero de 2013). Minicircle and Miniplasmid DNA Vectors: The Future of Non-viral and Viral Gene Transfer (en inglés). John Wiley & Sons. ISBN 9783527670444.
- ↑ Lakshmipathy, Uma; Thyagarajan, Bhaskar (22 de noviembre de 2011). Primary and Stem Cells: Gene Transfer Technologies and Applications (en inglés). John Wiley & Sons. ISBN 9780470610749.
- ↑ Grohmann, Elisabeth; Muth, Günther; Espinosa, Manuel (23 de febrero de 2017). «Conjugative Plasmid Transfer in Gram-Positive Bacteria». Microbiology and Molecular Biology Reviews 67 (2): 277-301. ISSN 1092-2172. PMC 156469. PMID 12794193. doi:10.1128/MMBR.67.2.277-301.2003.
- ↑ Tortora, Gerard J.; Funke, Berdell R.; Case, Christine L. (1 de enero de 2007). Introducción a la microbiología. Ed. Médica Panamericana. ISBN 9789500607407.
- ↑ León-Sampedro, Ricardo; DelaFuente, Javier; Díaz-Agero, Cristina; Crellen, Thomas; Musicha, Patrick; Rodríguez-Beltrán, Jerónimo; de la Vega, Carmen; Hernández-García, Marta et al. (2021-05). «Pervasive transmission of a carbapenem resistance plasmid in the gut microbiota of hospitalized patients». Nature Microbiology (en inglés) 6 (5): 606-616. ISSN 2058-5276. doi:10.1038/s41564-021-00879-y. Consultado el 21 de diciembre de 2022.
- ↑ Ashton, Acton (26 de diciembre de 2012). Advances in Escherichia Research and Application: 2012 Edition (en inglés). ScholarlyEditions. ISBN 9781464991325.
- ↑ Murray, Patrick R.; Rosenthal, Ken S.; Pfaller, Michael A. (21 de enero de 2017). Microbiología médica, 6a ed.. Elsevier España. ISBN 9788480864657.
- ↑ Devlin, Thomas M. (1 de enero de 2000). Bioquímica: Libro de Texto con Aplicaciones Clínicas. Reverte. ISBN 9788429172065.
- ↑ Lodish, Harvey; Berk, Arnold; Zipursky, S. Lawrence; Matsudaira, Paul; Baltimore, David; Darnell, James (1 de enero de 2000). «DNA Cloning with Plasmid Vectors». Book (en inglés).
- ↑ Gonzales, Favian E. Rivera; Lopez, Magdalena I. (1 de enero de 2012). Plasmids: Genetics, Applications and Health (en inglés). Nova Science Publishers, Incorporated. ISBN 9781620813706.
- ↑ a b Scherman, Daniel (10 de diciembre de 2013). Advanced Textbook on Gene Transfer, Gene Therapy and Genetic Pharmacology: Principles, Delivery and Pharmacological and Biomedical Applications of Nucleotide-Based Therapies (en inglés). World Scientific. ISBN 9781783263141.
- ↑ Winstanley, Craig; Rapley, Ralph (1 de enero de 2000). Rapley, Ralph, ed. The Nucleic Acid Protocols Handbook (en inglés). Humana Press. pp. 327-331. ISBN 9780896034594. doi:10.1385/1-59259-038-1:327.
- ↑ Bennett, P M (3 de diciembre de 2016). «Plasmid encoded antibiotic resistance: acquisition and transfer of antibiotic resistance genes in bacteria». British Journal of Pharmacology 153 (Suppl 1): S347-S357. ISSN 0007-1188. PMC 2268074. PMID 18193080. doi:10.1038/sj.bjp.0707607. Consultado el 3 de diciembre de 2016.
- ↑ Svara, Fabian; Rankin, Daniel J. (1 de enero de 2011). «The evolution of plasmid-carried antibiotic resistance». BMC Evolutionary Biology 11: 130. ISSN 1471-2148. PMC 3118148. PMID 21595903. doi:10.1186/1471-2148-11-130. Consultado el 4 de diciembre de 2016.
Bibliografía
[editar]- Klein, Donald W.; Prescott, Lansing M.; Harley, John (1999). Microbiology. Boston: WCB/McGraw-Hill.
Enlaces externos
[editar] Wikcionario tiene definiciones y otra información sobre plásmido.
Wikcionario tiene definiciones y otra información sobre plásmido.
