Corynebacterium
| Corynebacterium | ||
|---|---|---|
 | ||
| Taxonomía | ||
| Dominio: | Bacteria | |
| Filo: | Actinomycetota | |
| Clase: | Actinomycetia | |
| Orden: | Mycobacteriales | |
| Familia: |
Corynebacteriaceae Lehmann & Neumann 1907 | |
| Género: |
Corynebacterium Lehmann & Neumann 1896 | |
| Especies | ||
| ||
Corynebacterium es el único género de la familia Corynebacteriaceae.[3] Son bacilos grampositivos, inmóviles, aerobio facultativos, pertenecientes al filo Actinomycetota. Es uno de los géneros más numerosos de actinobacterias con más de 50 especies, la mayoría no causa enfermedades, sino que son parte de la microbiota saprofita de la piel humana.[1]
Taxonomía[editar]
El género Corynebacterium fue creado por Lehmann y Neumann (1896) para ubicar taxonómicamente a los bacilos de la difteria. El género fue definido basándose en características morfológicas: Corynebacteria proviene del griego corönë (bastón nudoso) y bacterion (bastoncillo). A partir de estudios del gen 16S-rRNA se ha agrupado a las corinebacterias en la subdivisión de eubacterias Gram-positivas de alto contenido en G:C, en estrecha relación filogenética con Arthrobacter, Mycobacterium, Nocardia e incluso Streptomyces.[4]
De acuerdo con el Sistema Integrado de Información Taxonómica, incluye las siguientes especies:[5]
- Corynebacterium accolens Neubauer et al., 1991
- Corynebacterium afermentans Riegel et al., 1993
- Corynebacterium ammoniagenes (Cooke & Keith, 1927) Collins, 1987
- Corynebacterium amycolatum Collins et al., 1988
- Corynebacterium appendicis Yassin et al., 2002
- Corynebacterium aquilae Fernández-Garayzábal et al., 2003
- Corynebacterium argentoratense Riegel et al., 1995
- Corynebacterium atypicum Hall et al., 2003
- Corynebacterium aurimucosum Yassin et al., 2002 emend. Daneshvar et al., 2004
- Corynebacterium auris Funke et al., 1995
- Corynebacterium auriscanis Collins et al., 2000
- Corynebacterium beticola Abdou, 1969
- Corynebacterium bovis Bergey et al., 1923
- Corynebacterium callunae (Lee & Good, 1963) Yamada & Komagata, 1972
- Corynebacterium camporealensis Fernández-Garayzábal et al., 1998
- Corynebacterium canis Funke et al., 2010
- Corynebacterium capitovis Collins et al., 2001
- Corynebacterium casei Brennan et al., 2001
- Corynebacterium caspium Collins et al., 2004
- Corynebacterium ciconiae Fernández-Garayzábal et al., 2004
- Corynebacterium confusum Funke et al., 1998
- Corynebacterium coyleae Funke et al., 1997
- Corynebacterium cystitidis Yanagawa & Honda, 1978
- Corynebacterium deserti Zhou et al., 2012
- Corynebacterium diphtheriae (Kruse, 1886) Lehmann & Neumann, 1896
- Corynebacterium doosanense Lee et al., 2009
- Corynebacterium durum Riegel et al., 1997
- Corynebacterium efficiens Fudou et al., 2002
- Corynebacterium falsenii Sjödén et al., 1998
- Corynebacterium felinum Collins et al., 2001
- Corynebacterium flavescens Barksdale et al., 1979
- Corynebacterium freiburgense Funke et al., 2009
- Corynebacterium freneyi Renaud et al., 2001
- Corynebacterium glaucum Yassin et al., 2003
- Corynebacterium glucuronolyticum Funke et al., 1995
- Corynebacterium glutamicum (Kinoshita et al., 1958) Abe et al., 1967
- Corynebacterium halotolerans Chen et al., 2004
- Corynebacterium hansenii Renaud et al., 2007
- Corynebacterium humireducens Wu et al., 2011
- Corynebacterium ilicis Mandel et al., 1961
- Corynebacterium imitans Funke et al., 1997
- Corynebacterium jeikeium Jackman et al., 1988
- Corynebacterium kroppenstedtii Collins et al., 1998
- Corynebacterium kutscheri (Migula, 1900) Bergey et al., 1925
- Corynebacterium lipophiloflavum Funke et al., 1997
- Corynebacterium lubricantis Kämpfer et al., 2009
- Corynebacterium macginleyi Riegel et al., 1995
- Corynebacterium marinum Du et al., 2010
- Corynebacterium maris Ben-Dov et al., 2009
- Corynebacterium massiliense Merhej et al., 2009
- Corynebacterium mastitidis Fernandez-Garayzabal et al., 1997
- Corynebacterium matruchotii (Mendel, 1919) Collins, 1983
- Corynebacterium minutissimum (ex Sarkany et al., 1962) Collins & Jones, 1983 emend. Yassin et al., 2002
- Corynebacterium mucifaciens Funke et al., 1997
- Corynebacterium mustelae Funke et al., 2010
- Corynebacterium mycetoides (ex Castellani, 1942) Collins, 1983
- Corynebacterium nuruki Shin et al., 2011
- Corynebacterium phocae Pascual et al., 1998
- Corynebacterium pilbarense Aravena-Roman et al., 2010
- Corynebacterium pilosum Yanagawa & Honda, 1978
- Corynebacterium propinquum Riegel et al., 1994
- Corynebacterium pseudodiphtheriticum Lehmann & Neumann, 1896
- Corynebacterium pseudotuberculosis (Buchanan, 1911) Eberson, 1918
- Corynebacterium pyruviciproducens Tong et al., 2010
- Corynebacterium renale (Migula, 1900) Ernst, 1906
- Corynebacterium resistens Otsuka et al., 2005
- Corynebacterium riegelii Funke et al., 1998
- Corynebacterium simulans Wattiau et al., 2000
- Corynebacterium singulare Riegel et al., 1997
- Corynebacterium sphenisci Goyache et al., 2003
- Corynebacterium spheniscorum Goyache et al., 2003
- Corynebacterium sputi Yassin & Siering, 2008
- Corynebacterium stationis (ZoBell & Upham, 1944) Bernard et al., 2010
- Corynebacterium striatum (Chester, 1901) Eberson, 1918
- Corynebacterium suicordis Vela et al., 2003
- Corynebacterium sundsvallense Collins et al., 1999
- Corynebacterium terpenotabidum Takeuchi et al., 1999
- Corynebacterium testudinoris Collins et al., 2001
- Corynebacterium thomssenii Zimmermann et al., 1998
- Corynebacterium timonense Merhej et al., 2009
- Corynebacterium tuberculostearicum Feurer et al., 2004
- Corynebacterium tuscaniense Riegel et al., 2006
- Corynebacterium ulcerans (ex Gilbert & Stewart, 1927) Riegel et al., 1995
- Corynebacterium ulceribovis Yassin, 2009
- Corynebacterium urealyticum Pitcher et al., 1992
- Corynebacterium ureicelerivorans Yassin, 2007
- Corynebacterium variabile (Müller, 1961) Collins, 1987
- Corynebacterium vitaeruminis (Bechdel et al., 1928) Lanéelle et al., 1980
- Corynebacterium xerosis (Lehmann & Neumann, 1896) Lehmann & Neumann, 1899
Características[editar]
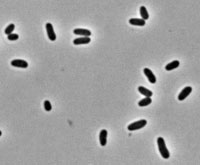
Las características más relevantes del género Corynebacterium fueron descritas por Collins y Cummins (1986).[6] Se trata de bacterias Gram-positivas, catalasa positivas, no esporuladas, que carecen de motilidad, bacilos rectos o ligeramente curvados[7] cuyo tamaño oscila entre 2-6 micrómetros de longitud y 0,5 micrómetros de diámetro, a menudo con la típica forma de V (lo que también se denomina “forma de letras chinas”), aunque también aparecen formas elipsoidales, son aerobias o anaerobias facultativas, quimioorganotrofos, con un contenido en G:C genómico entre 51-65 %. El pleomorfismo en su ciclo de vida se observa en formas bacilares de longitud diversa y frecuentes engrosamientos en los extremos, estando marcadamente influido por las condiciones del cultivo.[8]
Pared celular[editar]
La pared celular es muy característica, presentando un predominio de ácido mesodiaminopimélico en el tetrapéptido de la mureína[7][1] y están presentes motivos repetitivos de arabino-galactano, un heteropolisacárido esencial así como ácidos corinemicólicos (ácidos micólicos de 22 a 36 átomos de carbono), unidos entre sí por enlaces disacáridos específicos denominados bioquímicamente como L-Rhap-(1 → 4)--D-GlcNAc-fosfato. Ello forma un complejo común en las Corynebacterium, el micolil-AG-peptidoglicano (mAGP).[9]
Cultivo[editar]
Con respecto a los requerimientos nutricionales, todos ellos necesitan biotina para su crecimiento y algunas cepas requieren además tiamina y ácido p-aminobenzoico (PABA).[6] Algunas especies de Corynebacterium tienen genomas secuenciados que varían de 2.5-3 Mbp. La bacteria crece en caldo simple, medio de Loeffler, agar sangre y telurito potásico (AST), formando colonias pequeñas grisáceas de aspecto granuloso, traslúcidas con centros opacos, convexas con bordes continuos.[7] El color tiende a ser blanco amarillento en los medios de cultivo de Loeffler. En AST, el organismo puede formar colonias grises con centros negros y bordes dentados dando la apariencia de flores (C. gravis), otras tienen bordes continuos (C. mitis), mientras que otras tienen bordes intermedios entre continuas y dentadas (C. intermedium).
Hábitat[editar]
Las corinebacterias están ampliamente distribuidas en la naturaleza encontrándose en el suelo, el agua, productos alimenticios y también en la mucosa y piel del hombre y animales.[7][1] Estas especies: Corynebacterium bovis, C. mutissium, C. xerosis y C. hoffmani habitan en la piel de todos los seres humanos, especialmente en la zona axilar. Cuando detectan el sudor, se multiplican rápidamente, dando lugar al característico olor de las axilas. Algunas especies son conocidas por sus efectos patógenos en humanos y otros animales. La especie patógena de corinebacterias más conocida es C. diphtheriae, que adquiere la capacidad de producir la toxina diftérica cuando es lisogenizada por el fago beta, siendo inicialmente, es decir, antes de la acción transformadora del fago, no lisogénica y no toxinogénica.[10] Otras especies patógenas del hombre son: C. amicolatum, C. striatum, C. jeikeium, C. urealyticum y C. xerosis (Oteo y col., 2001; Lagrou y col., 1998; Boc y Martone, 1995);[11][12] todas estas especies son patógenos de especial relevancia en pacientes inmunodeprimidos. Entre las especies patógenas de otros animales destacan C. bovis y C. renale.[13]
Patogenia[editar]

Algunas de las especies no-difteroides de Corynebacterium producen enfermedades en determinadas especies animales, y algunas de ellas son también patógenos humanos. Algunas especies atacan hospedadores saludables, mientras que otras atacan hospedadores inmunocomprometidos. Algunos de sus efectos incluyen linfadenitis granulomatosa, neumonitis, faringitis, infecciones de la piel y endocarditis. La endocarditis causada por las especies de Corynebacterium se ven con especial frecuencia en pacientes con dispositivos intravasculares.[14]
En humanos las infecciones por difteroides causan difteria, una enfermedad aguda, contagiosa productora de una pseudomembrana compuesta por células epiteliales muertas, leucocitos, glóbulos rojos y fibrina que se forma alrededor de las amígdalas y la faringe.[15] Es una enfermedad poco común y tiende a ocurrir en personas no vacunadas, en especial niños en edad escolar, en especial en países en desarrollo,[16] ancianos, neutropénicos o pacientes inmunodeficientes, y aquellos con dispositivos prostéticos tales como prótesis valvular cardíaca, shunts o catéteres. En ocasiones puede infectar heridas, la vulva, la conjuntiva y el oído medio, y nosocomiales de un humano a otro.[17]
Las bacterias virulentas y toxigénicas son lisogénicas y producen una exotoxina formada por dos cadenas de polipéptidos, producida por acción transformadora de un gen del profago β.[10]
Uso industrial[editar]
Las especies no patógenas de corinebacterias son utilizadas en procesos industriales de gran relevancia, como la producción de aminoácidos,[18][19] producción de nucleótidos y otros factores nutricionales (Martín, 1989), bioconversión de esteroides,[20] degradación de hidrocarburos,[21] maduración de quesos,[22] producción de enzimas (Khurana y col., 2000) y otros procesos con interés desde el punto de vista aplicado. Algunas especies son productoras de metabolitos semejantes a los antibióticos: bacteriocinas del tipo corinecinas-linocinas,[23][24][17] agentes antitumorales,[25] etc. Una de las especies más estudiadas es C. glutamicum, el término glutamicus se debe a su capacidad de producir ácido glutámico en condiciones aeróbicas[26] usado en la industria alimenticia como glutamato monosódico en la producción de salsa de soja o el yogur.
Véase también[editar]
Referencias[editar]
- ↑ a b c d Collins, Matthew D., Lesley Hoyles, Geoffrey Foster y Enevold Falsen. «Corynebacterium caspium sp. nov., from a Caspian seal (Phoca caspica).» Int J Syst Evol Microbiol 54 (2004), 925-928; Consultado el 30 octubre de 2007.
- ↑ eMedicine.com [1]
- ↑ Parte, A. C. «Family Corynebacteriaceae». List of Prokaryotic names with Standing in Nomenclature (en inglés). Consultado el 6 de junio de 2020.
- ↑ Woese, C. R. (1987) «Bacterial evolution.» Microbiol Rev 51: 221-271. [2]
- ↑ «Corynebacterium (TSN 383)». Sistema Integrado de Información Taxonómica (en inglés).
- ↑ a b Collins, M. D. & Cummins, C. S. (1986). «Genus Corynebacterium Lehmann and Neumann 1896, 350AL.» En Bergey's Manual of Systematic Bacteriology, vol. 2, pp. 1266-1276. Coordinadores: P. H. A. Sneath, N. S. Mair, M. E. Sharpe & J. G. Holt. Baltimore: Williams & Wilkins.
- ↑ a b c d A. F. Yassin, R. M. Kroppenstedt y W. Ludwig. «Corynebacterium glaucum sp. nov.» Int J Syst Evol Microbiol 53 (2003), 705-709; [3] Archivado el 12 de febrero de 2007 en Wayback Machine. Último acceso 30 octubre de 2007.
- ↑ Keddie, R. M. & Cure, G. L. (1977). «The cell wall composition and distribution of free mycolic acids in named strains of coryneform bacteria and in isolates from various natural sources.» J Appl Bacteriol 42, 229-252. [4]
- ↑ Seidel M, Alderwick, L. J., Sahm, H., Besra, G. S., Eggeling, L. «Topology and mutational analysis of the single Emb arabinofuranosyltransferase of Corynebacterium glutamicum as a model of Emb proteins of Mycobacterium tuberculosis.» Glycobiology. 2007 Feb;17(2):210-9. Epub 2006 Nov 6. PMID 17088267. [5] Último acceso 30 octubre de 2007.
- ↑ a b Costa, J. J., Michel, J. L., Rappuoli, R., Murphy, J. R. «Restriction map of corynebacteriophages beta c and beta vir and physical localization of the diphtheria tox operon.» J Bacteriol. 1981 Oct;148(1):124-130. [6] Último acceso 30 octubre de 2007.
- ↑ Kono, M., Sasatsu, M. y Aoki, T. 1983. «R plasmids in Corynebacterium xerosis strains.» Antimicrob. Ag. Chemoter. 23: 506-508. [7]
- ↑ Pitcher, D. G. 1983. «Deoxyribonucleic acid base composition of Corynebacterium diphtheriae and corynebacteria with cell wall type IV.» FEMS Microbiol. Lett. 16: 291−295. [8] (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última). Último acceso 30 octubre de 2007.
- ↑ Watts y col., 2001; Hirsbrunner, G. et al. «Nephrectomy for chronic, unilateral suppurative pyleonephritis in cattle.» Tierarztl Prax, 1996 Feb, 24(1), 17-21; [Nephrectomy_for_chronic_unilateral_suppurative_pyleonephritis_in_cattle]
- ↑ Leóna, Cristóbal y Javier Ariza. «Guías para el tratamiento de las infecciones relacionadas con catéteres intravasculares de corta permanencia en adultos: conferencia de consenso SEIMC-SEMICYUC.» Enferm Infecc Microbiol Clin 2004; 22: 92-101. [9] Archivado el 17 de marzo de 2004 en Wayback Machine.
- ↑ MedlinePlus - Difteria. [10]
- ↑ IIZUKA, Hideyo, FURUTA, Joana Akiko OLIVEIRA, Edison P. Tavares de. «Diphtheria: immunity in an infant population in the city of S. Paulo, SP, Brazil.» Rev. Saúde Pública [online]. 1980, vol. 14, no. 42007-10-29], pp. 462-468. [11]. ISSN 0034-8910
- ↑ a b Kerry-Williams, S. M., Noble, W. C. «Plasmids in group JK coryneform bacteria isolated in a single hospital.» J Hyg (Lond). 1986 Oct;97(2):255-263. [12]
- ↑ Hongo, M., Oki, T. y Ogata, S. 1972. «Phage contamination and control», pp. 63-83. En: The Microbial Production of Amino Acids. K. Yamada, S Kinoshita, T. Tsunoda, K. Aida. (eds.). John Wiley, New York
- ↑ Yamada, K., Kinoshita, S., Tsunoda, T. y Aida, K. 1972. The Microbial Production of Amino Acids. Wiley, New York.
- ↑ Constantinides, A. 1980. «Steroid transformation at high substrate concentrations using immobilized Corynebacterium simplex cells.» Biotechnol. Bioeng. 22: 119-136. [13]
- ↑ Cooper, D. G., Zajic, J. E. y Gracey, D. E. F. 1979. «Analysis of corynomycolic acids and other fatty acids produced by Corynebacterium lepus grown on kerosene.» J. Bacteriol. 137: 795-801. [14]
- ↑ Lee, C.W., Lucas, S. y Desomazeaud, M.J. 1985. «Phenylalanine and tyrosine catabolism in some cheese coryneform bacteria.» FEMS Microbiol. Lett. 26: 201-205. [15] (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última).
- ↑ Kerry-Williams, S. M. y Noble, W. C. 1984. «Plasmid associated bacteriocin production in a JK-type coryneform bacterium.» FEMS Microbiol. Lett. 25: 179-182. [16] (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última). Último acceso 30 octubre de 2007.
- ↑ Suzuki, T., Honda, H. y Katsumata, R. 1972. «Production of antibacterial compounds analogous to chloramphenicol by n-paraffin-grown bacteria.» Agr. Biol. Chem. 36: 2223-2228. [17] Último acceso 30 octubre de 2007.
- ↑ Milas, L. y Scott, M. T. 1978. «Antitumor activity of Corynebacterium parvum.» Adv. Cancer Res. 26: 257-306. [18]
- ↑ Abe, S., Takayama, K. y Kinoshita, S. 1967. «Taxonomical studies on glutamic acid-producing bacteria.» J. Gen. Appl. Microbiol. 13: 279-301. [Abe, S., Takayama, K. y Kinoshita, S. 1967. «Taxonomical studies on glutamic acid-producing bacteria.» J. Gen. Appl. Microbiol. 13: 279-301.]
Bibliografía[editar]
- Ryan, K. J.; Ray, C. G. (coordinadores) (2004). Sherris Medical Microbiology (4th ed. edición). McGraw Hill. ISBN 0-8385-8529-9.
- Database of Corynebacterial Transcription Factors and Regulatory Networks
- Rollins, David M. University of Maryland: Pathogentic Microbiology: Corynebacterium [19]
Enlaces externos[editar]
 Wikimedia Commons alberga una categoría multimedia sobre Corynebacterium.
Wikimedia Commons alberga una categoría multimedia sobre Corynebacterium. Wikispecies tiene un artículo sobre Corynebacterium.
Wikispecies tiene un artículo sobre Corynebacterium.- Tree of Life web project: Corynebacterineae Proyecto Web del Árbol de la Vida.
