Heparán sulfato

El heparán sulfato (HS) o sulfato de heparano es un polisacárido lineal que se encuentra en todos los tejidos animales. Aparece como un proteoglicano en el cual dos o tres cadenas de heparán sulfato se encuentran adosadas en cercana proximidad a las proteínas de la superficie celular o a las de la matriz extracelular.[1][2] Es en esta forma que los heparán sulfatos se unen a una gran variedad de proteínas y regulan una amplia variedad de actividades biológicas, incluyendo procesos de desarrollo, angiogénesis, coagulación de la sangre, abolición de la actividad de desplazamiento mediado por la GrB (Granzima B),[3] y en la metástasis tumoral. Los HS han mostrado fungir como receptores celulares para numerosos virus, incluyendo al virus respiratorio sincicial (Hallak et al. 2000)
Proteoglicanos
[editar]Los mayores heparán sulfato proteoglicanos son los sindecanos transmembrana y los glipicanos anclados a glicosilfosfatidilinositol. Otras formas menores de HSPG de membrana incluyen los betaglicanos[4] y la isoforma V-3 del CD44 presente en los queratinocitos y monocitos activados.[5]
En la matriz extracelular, especialmente en la membrana basal, las proteínas nucleares de los perlecanos multidominio, agrina y colágeno tipo XVIII son las principales especies portadoras de heparán sulfatos.
Posible papel en las enfermedades de Alzheimer y Parkinson
[editar]Los HSPG han sido implicados en la progresión de enfermedades neurodegenerativas. Las fibrillas de la proteína Tau unidas a los HSPG de la superficie celular, desencadenan la agregación intracelular de fibrillas en manera similar a los priones, sugiriendo un mecanismo unificado para las taupatías y sinucleopatías, además de un potencial blanco terapéutico farmacológico.[6]
Estructura de los HS y diferencias con la heparina
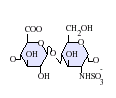
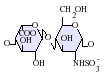
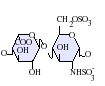
[editar]Los heparán sulfatos son miembros de la familia de carbohidratos de los glicosaminglicanos, y se encuentra muy estrechamente relacionado con la heparina en cuanto a su estructura. Ambos consisten en una repetición de una unidad disacárida con un grado variable de sulfatación. Las principales unidades de disacáridos que aparecen en el heparán sulfato y en la heparina se muestran un poco más abajo.
La unidad disacárida más común que aparece en los heparán sulfatos se encuentra compuesta de un ácido glucurónico (GlcA) unido a una N-acetilglucosamina (GlcNAc), que típicamente forma el 50% del total de unidades de disacárido. En comparación en la heparina el dimero más frecuente es el de Ácido idurónico + N-acetilglucosamina, el cual forma más del 85% de las heparinas provenientes de pulmón de res y aproximadamente un 75% de la heparina proveniente de mucosa intestinal de cerdo. Los problemas aparecen al tratar de definir los GAGs híbridos que contienen ambos tipos de estructuras las similares a heparina y las similares a heparán sulfato. Se ha sugerido que un GAG podría calificar como heparina únicamente si su contenido de grupos N-sulfato exceden ampliamente a los grupos N-acetilo y si la concentración de grupos O-sulfato excede a la de grupos N-sulfato.[7] Sin embargo, estudios demostraron que si los polímeros tienen relación idurónico/glucurónico menor de 50:50 en favor del segundo, no se degradan ante acción de enzimas específicas como condroitinasas y heparinasas y poseen baja actividad anticoagulante, son considerados heparán sulfatos, más allá del nivel de sulfatos que tengan.[8]
Bajo condiciones fisiológicas los grupos ester y amida sulfato se encuentran desprotonados y atraen contraiones cargados positivamente para formar una sal. Se piensa que es en esta forma que los heparán sulfatos se encuentran en la superficie celular.
-
GlcA-GlcNAc -
GlcA-GlcNS -
IdoA-GlcNS -
IdoA(2S)-GlcNS -
IdoA-GlcNS(6S) -
IdoA(2S)-GlcNS(6S)
Abreviaturas
[editar]- GlcA = Ácido β-D-glucurónico
- IdoA = Ácido α-L-idurónico
- IdoA(2S) = Ácido 2-O-sulfo-α-L-idurónico
- GlcNAc = 2-desoxi-2-acetamido-α-D-glucopiranosil
- GlcNS = 2-desoxi-2-sulfamido-α-D-glucopiranosil
- GlcNS(6S) = 2-desoxi-2-sulfamido-α-D-glucopiranosil-6-O-sulfato
Biosíntesis de los HS
[editar]Muchos tipos celulares producen cadenas de heparán sulfatos con muchas estructuras primarias diferentes. Por lo tanto, existe una gran variabilidad en la forma en que se sintetizan los HS. Sin embargo, lo esencial para la síntesis de HS, independientemente de la secuencia primaria de los mismos; es un grupo de enzimas biosintéticas con características similares. Estas enzimas se encuentran formadas por múltiples glicosiltransferasas, sulfotransferasas y una epimerasa. Estas mismas enzimas son las que también sintetizan heparina.
En los años 1980, el Profesor Jeffrey Esko fue el primero en aislar y caracterizar células animales mutantes en las cuales se encontraba alterada el ensamblado de las cadenas de heparán sulfato.[9] Muchas de estas enzimas actualmente han sido purificadas, clonadas a nivel molecular, y se han estudiado sus patrones de expresión. De los trabajos más tempranos y de estos últimos se ha aprendido mucho sobre las etapas fundamentales en la biosíntesis de HS, el orden de reacción de las enzimas y su especificidad, para esto se ha utilizado un sistema libre de células de mastocitoma murino.[10]
Iniciación de la cadena
[editar]La síntesis de heparan sulfatos inicia con la transferencia de una xilosa proveniente de UDP-xilosa por medio de una xilosiltransferasa (XT) a un residuo específico de una serina en una proteína que actúa como núcleo. La adición de dos residuos de galactosas (Gal) por las enzimas galactosiltransferasa I y II (GalTI y GalTII) y una unidad de ácido glucurónico (GlcA) por la glucuroniltransferasa I (GlcATI) completa la formación del tetrasacárido de unión al núcleo proteico.
βGlcA-1,3-βGal-1,3-βGal-1,4-βXyl.
Se cree que la adición de xilosa al núcleo proteico ocurre en el retículo endoplasmático (ER) mientras que el posterior ensamblaje de la región de unión y la cadena restante ocurren en el aparato de Golgi.
Las vías de síntesis de la heparan sulfato, heparina, condroitín sulfato (CS) y dermatán sulfato (DS) divergen luego de la formación de esta estructura de unión común. La siguiente enzima en actuar, GlcNAcT-I o GalNAcTI dirige la síntesis, ya sea hacia la formación de heparán sulfato/heparina o condroitín sulfato/dermatán sulfato respectivamente.
Elongación de la cadena
[editar]Luego de la adición del primer residuo de N-acetilglucosamina (GlcNAc) la elongación del tetrasacárido de unión se continúa por la adición escalonada de residuos de GlcA y GLcNAc. Esto son transferidos desde sus respectivos nucleótidos UDP-azúcar. Esto es llevado a cabo por una o más enzimas relacionadas cuyos genes son miembros de la familia de genes supresores tumorales conocidos como exostosas (EXT).
Las mutaciones del loci de los genes EXT1-3 conduce en humanos a la incapacidad de las células para producir heparán sulfatos y al desarrollo de la enfermedad de las exostosis múltiples hereditarias (MHE). La MHE se caracteriza por tumores recubiertos de cartílago, conocidos como osteocondromas o exostosis, los cuales se desarrollan primeramente en los huesos largos de los individuos afectados, desde la infancia temprana hasta la pubertad.
Modificación de la cadena
[editar]Mientras la cadena se polimeriza, sufre una serie de reacciones de modificación llevada a cabo por cuatro clases de sulfotransferasas y una epimerasa. La disponibilidad del donante de grupos sulfatos, el PAPS es crucial para la actividad de las sulfotransferasas.[11][12]
N-desacetilación/N-sulfatación
[editar]La primera modificación del polímero es la N-desacetilación/N-sulfatación de los residuos de GlcNAc para formar GlcNS. Esto es un prerrequisito para todas las reacciones de modificación subsecuentes, y es llevada a cabo por uno o más miembros de una familia de cuatro enzimas GlcNAc N-desacetilasa/N-sulfotransferasa (NDSTs). En estudios tempranos, de demostró que estas enzimas modificadoras pueden reconocer y actuar sobre cualquier residuo N-acetilado en el polímero en formación.[13] Por lo tanto, la modificación de los residuos de GlcNAc deberían ocurrir aleatoriamente a lo largo de la cadena. Sin embargo, en los heparán sulfatos, los residuos N-sulfatados, se encuentran principalmente agrupados en determinadas regiones y separados por regiones de N-acetilación donde los residuos de GlcNAc permanecen sin modificación.
Generación de GlcNH
2
[editar]Debido a que las actividades N-desacetilasa y N-sulfotransferasa son llevadas a cabo por la misma enzima, la N-sulfatación se encuentra por lo general, estrechamente acoplada a al N-acetilación. Los residuos de GlcNH
2 resultantes de un aparente desacoplamiento entre las dos actividades se han encontrado en las heparinas y en algunas especies de heparán sulfatos.[14]
Epimerización y 2-O-sulfatación
[editar]La epimerización se encuentra catalizada por una enzima, la GlcA C5 epimerasa o también llamada heparosán-N-sulfato-glucouronato 5-epimerasa (EC 5.1.3.17). Esta enzima epimeriza al GlcA para formar ácido idurónico (IdoA). El reconocimiento de sustrato requiere que el residuo de GlcN unido al lado no reductor de un residuo diana potencial de GlcA debe estar N-sulfatado. La uronosil-2-O-sulfotransferasa (2OST) cataliza la sulfatación del residuo de IdoA resultante.
6-O-sulfatación
[editar]Se han identificado tres glucosaminil 6-O-transferasas (6OSTs) que participan en la formación de GlcNS(6S) adyacentes a un IdoA sulfatado o no sulfatado. También se encuentra GlcNAc(6S) en las cadenas de heparán sulfato maduras.
3-O-sulfatación
[editar]Actualmente se conocen siete glucosaminil 3-O-sulfotransferasas (3OSTs) en mamíferos (ocho en los peces cebra).[15][16] Las enzimas 3OST crean un cierto número de disacáridos 3-O-sulfatados, entre los que se incluyen GlcA-GlcNS(3S±6S) (modificados por las 3OST1 y 3OST5), IdoA(2S)-GlcNH
2(3S±6S)(modificados por la 3OST3a, 3OST3b, 3OST5 y 3OST6) y GlcA/IdoA(2S)-GlcNS(3S) (modificado por las 3OST2 y 3OST4).[17][18][19][20] Al igual que otras sulfotransferasas de heparán sulfato, la 3OSTs utiliza 3'-fosfoadenosina-5'-fosfosulfato (PAPS) como donante de grupos sulfato. A pesar de ser la mayor familia de enzimas modificadoras de los heparán sulfato, las 3OSTs producen las modificaciones menos habituales, la 3-O-sulfatación de residuos específicos de glucosamina en el sitio C3-OH.[21]
Las 3OSTs se dividen en dos subcategorías funcionales, aquellas que generan un sitio de unión a la antitrombina III (3OST1 and 3OST5) y aquellas que producen un sitio de unión para la glicoproteína D de HSV 1 (HSV-1 gD) (3OST2, 3OST3a, 3OST3b, 3OST4, 3OST5 y3OST6).[17][18][19][20][22][23][24][25][26][27][28] Debido a que las 3OSTs son la mayor familia de enzimas modificadoras de los heparán sulfatos, y a que sus acciones se encuentran limitadas por el sustrato, y a que producen escasas modificaciones, se ha hipotetizado que las modificaciones llevadas a cabos por las 3OST en los heparán sulfatos desempeñan un importante papel regulatorio en los procesos biológicos.[20][23]
Unión de ligandos
[editar]Los heparán sulfatos se unen a cientos de proteínas extracelulares de las que actúan como pareja. A menudo se las denomina colectivamente como "interactoma de heparina" o "proteínas de unión a la heparina", porque se aíslan por medio de su afinidad cromatográfica al polisacárido relacionado heparina; esto a pesar de que el término "interactoma de heparán sulfato" o "heparanoma" sería más correcto. Las funciones de las proteínas de unión a los heparán sulfato van desde componentes de la matriz extracelular, hasta enzimas y factores de la coagulación, la mayor parte de los factores de crecimiento, citoquinas, quimiocinas y morfógenos.[29]
Interferón-γ
[editar]La región unión al Interferón-γ de la superficie celular, se solapa con la región de unión a los heparán sulfatos, cerca de la región C-terminal de la proteína. La unión de un heparán sulfato bloquea al sitio de unión del receptor, y como resultado los complejos proteína-HS son inactivos.[30]
The HS-binding properties of a number of other proteins are also being studied:
- Antitrombina III
- Factor de crecimiento de fibroblastos
- Factor de crecimiento de hepatocitos
- Interleucina-8
- Factor de crecimiento vascular endotelial
- Wnt/Wingless
- Endostatina
Análogos de heparán sulfato
[editar]Se piensa que los análogos de heparán sulfatos muestran propiedades idénticas a las de los verdaderos heparán sulfatos con la excepción de ser más estables en un ambiente proteolítico, tal como por ejemplo una herida.[31][32] Debido a que los heparán sulfatos se degradan en las heridas crónicas por la acción de la enzima heparanasa, los análogos sólo se unen a sitios naturales donde los heparán sulfatos se encuentran ausentes, y no pueden ser degradados por ninguna heparanasa ni glicanasa conocida.[33] Además la función de los análogos de heparán sulfatos es la misma de los heparán sulfatos, protegiendo a una gran variedad de ligandos porteicos tales como factores de crecimiento y citoquinas. Manteniéndolos en su sitio, el tejido puede utilizar los diferentes ligandos proteicos para proliferación.
Referencias
[editar]- ↑ Gallagher, J.T., Lyon, M. (2000). «Molecular structure of Heparan Sulfate and interactions with growth factors and morphogens». En Iozzo, M, V., ed. Proteoglycans: structure, biology and molecular interactions. Marcel Dekker Inc. New York, New York. pp. 27-59.
- ↑ Iozzo, R. V. (1998). «Matrix proteoglycans: from molecular design to cellular function». Annu. Rev. Biochem. 67: 609-652. PMID 9759499. doi:10.1146/annurev.biochem.67.1.609.
- ↑ Buzza, Marguerite; Zamurs, Laura; Sun, Jiuru; Bird, Catherina (junio de 2005). «Extracellular Matrix Remodeling by Human Granzyme B via Cleavage of Vitronectin, Fibronectin, and Laminin». Journal of Biological Chemistry 280 (25): 23549-23558. doi:10.1074/jbc.M412001200.
- ↑ Andres, J. L.; Noda, M et al. (1992). «Binding of two growth factor families to separate domains of the proteoglycan betaglycan». J. Biol. Chem. 267 (9): 5927-30. PMID 1556106.
- ↑ Jackson, D. G.; Dickinson, R et al. (1995). «Proteoglycan forms of lymphocyte homing receptor CD44 are alternatively spliced variants containing the V-3 exon». J. Cell. Biol 128 (4): 673-685. PMC 2199896. PMID 7532175. doi:10.1083/jcb.128.4.673.
- ↑ Holmes, BB.; Devos, SL.; Kfoury, N.; Li, M.; Jacks, R.; Yanamandra, K.; Ouidja, MO.; Brodsky, FM.; Marasa, J.; Bagchi, D. P.; Kotzbauer, P. T.; Miller, T. M.; Papy-Garcia, D.; Diamond, M. I. (Aug 2013). «Heparan sulfate proteoglycans mediate internalization and propagation of specific proteopathic seeds». Proc Natl Acad Sci U S A 110 (33): E3138-47. PMC 3746848. PMID 23898162. doi:10.1073/pnas.1301440110.
- ↑ Gallagher, J. T. Walker, A. (1985). «Molecular distinctions between Heparan Sulphate and Heparin: Analysis of sulphation patterns indicates Heparan Sulphate and Heparin are separate families of N-sulphated polysaccharides». Biochem. J. 230 (3): 665-74. PMC 1152670. PMID 2933029.
- ↑ Kovensky, José Eduardo (1992). Heparán sulfatos : estudios estructurales y modificaciones químicas (PDF) (Posgrado). Químicas de la Universidad de Buenos Aires.
- ↑ Esko, J.D.; Stewart, T.E.; Taylor, W.H. (1985). «Animal cell mutants defective inglycosaminoglycan biosynthesis». Proc. Natl. Acad. Sci. USA 85: 3197-3201. PMID 3858816.
- ↑ Lindahl, U. et al. (1998). «Regulated diversity of Heparan Sulfate». J. Biol. Chem. 273 (39): 24979-24982. PMID 9737951. doi:10.1074/jbc.273.39.24979.
- ↑ Silbert, J. E. (10 de noviembre de 1967). «Formation of a sulfate glycosaminoglycan with a microsomal preparation from mast cells». J. Biol. Chem. 242 (21): 5146-52. PMID 4228675. Archivado desde el original el 21 de junio de 2008. Consultado el 12 de abril de 2016.
- ↑ Carlsson P; Presto J. et al. (2008). «Heparin/Heparan Sulfate Biosynthesis: Processive formation of N-sulfated domains». J. Biol. Chem. 283 (29): 20008-20014. PMID 18487608. doi:10.1074/jbc.M801652200.
- ↑ Höök, M. et al. (1975). «Biosynthesis of heparin. Studies on the microsomal sulfation process». J. Biol. Chem. 250 (15): 6065-71. PMID 807579.
- ↑ Toida, T. et al. (1 de enero de 1997). «Structural differences and the presence of unsubstituted amino groups in heparan sulphates from different tissues and species». Biochem. J. 322(Pt2) (Pt 2): 499-506. PMC 1218218. PMID 9065769.
- ↑ Cadwallader, AB; Yost, HJ (febrero de 2007). «Combinatorial expression patterns of heparan sulfate sulfotransferases in zebrafish: III. 2-O-sulfotransferase and C5-epimerases». Developmental Dynamics 236 (2): 581-6. PMID 17195182. doi:10.1002/dvdy.21051.
- ↑ Xu, D; Tiwari, V; Xia, G; Clement, C; Shukla, D; Liu, J (Jan 15, 2005). «Characterization of heparan sulphate 3-O-sulphotransferase isoform 6 and its role in assisting the entry of herpes simplex virus type 1». The Biochemical Journal 385 (Pt 2): 451-9. PMC 1134716. PMID 15303968. doi:10.1042/BJ20040908.
- ↑ a b Shukla, D.; Liu, J.; Blaiklock, P.; Shworak, N.W.; Bai, X.; Esko, J.D.; Cohen, G.H.; Eisenberg, R.J.; Rosenberg, R.D.; Spear, P.G. (1999). «A Novel Role for 3-O-Sulfated Heparan Sulfate in Herpes Simplex Virus 1 Entry». Cell 99: 13-22. doi:10.1016/s0092-8674(00)80058-6.
- ↑ a b Xia, G.; Chen, J.; Tiwari, V.; Ju, W.; Li, J.P.; Malmstrom, A.; Shukla, D.; Liu, J. (2002). «Heparan Sulfate 3-O-Sulfotransferase Isoform 5 Generates Both an Antithrombin-binding Site and an Entry Receptor for Herpes Simplex Virus, Type 1». The Journal of Biological Chemistry 277 (40): 37912-37919. doi:10.1074/jbc.m204209200.
- ↑ a b Xu, D.; Tiwari, V.; Xia, G.; Clement, C.; Shukla, D.; Liu, J. (2005). «Characterization of heparan sulphate 3-O-sulphotransferase isoform 6 and its role in assisting the entry of herpes simplex virus type 1». Biochemistry Journal 385: 451-459. doi:10.1042/bj20040908.
- ↑ a b c Lawrence, R.; Yabe, T.; HajMohammadi, S.; Rhodes, J.; McNeely, M.; Liu, J.; Lamperti, E.D.; Toselli, P.A.; Lech, M.; Spear, P.G.; Rosenberg, R.D.; Shworak, N.W. (2007). «The principal neuronal gD-type 3-O-sulfotransferases and their products in the central and peripheral nervous system tissues». Matrix Biology 26: 442-455. doi:10.1016/j.matbio.2007.03.002.
- ↑ Shworak, N.W.; HajMohammadi, S.; de Agostini, A.I.; Rosenberg, R.D. (2003). «Mice deficient in heparan sulfate 3-O-silfotransferase-1: Normal hemostasis with unexpected perinatal phenotypes». Glycoconjugate Journal 19 (4-5): 355-361. doi:10.1023/a:1025377206600.
- ↑ Liu, J.; Shworak, N.W.; Fritze, L.M.S.; Edelberg, J.M.; Rosenberg, R.D. (1996). «Purification of Heparan Sulfate D-Glucosaminyl 3-O-Sulfotransferase». The Journal of Biological Chemistry 271 (43): 27072-27082.
- ↑ a b Shworak, N.W.; Liu, J.; Fritze, L.M.S.; Schwartz, J.J.; Zhang, L.; Logeart, D.; Rosenberg, R.D. (1997). «Molecular Cloning and Expression of Mouse and Human cDNAs Encoding Heparan Sulfate D-Glucosaminyl 3-O-Sulfotransferase». The Journal of Biological Chemistry 272 (44): 28008-28019. doi:10.1074/jbc.272.44.28008.
- ↑ Shworak, N.W.; Liu, J.; Petros, L.M.; Zhang, L.; Kobayashi, M.; Copeland, N.G.; Jenkins, N.A.; Rosenberg, R.D. (1999). «Multiple Isoforms of Heparan Sulfate D-Glucosaminyl 3-O-Sulfotransferase». The Journal of Biological Chemistry 274 (8): 5170-5184. doi:10.1074/jbc.274.8.5170.
- ↑ Chen, J.; Duncan, M.B.; Carrick, K.; Pope, R.M.; Liu, J. (2003). «Biosynthesis of 3-O-sulfated heparan sulfate: unique substrate specificity of heparan sulfate 3-O-sulfotransferase isoform 5». Glycobiology 13 (11): 785-794. doi:10.1093/glycob/cwg101.
- ↑ Duncan, M.B.; Chen, J.; Krise, J.P.; Liu, J. (2004). «The biosynthesis of anticoagulant heparan sulfate by the heparan sulfate 3-O-sulfotransferase isoform 5». Biochimica et Biophysica Acta 1671: 34-43. doi:10.1016/j.bbagen.2003.12.010.
- ↑ Chen, J.; Liu, J. (2005). «Characterization of the structure of antithrombin-binding heparan sulfate generated by heparan sulfate 3-O-sulfotransferase 5». Biochimica et Biophysica Acta 1725: 190-200. doi:10.1016/j.bbagen.2005.06.012.
- ↑ Girardin, E.P.; HajMohammadi, S.; Birmele, B.; Helisch, A.; Shworak, N.W.; de Agostini, A.I. (2005). «Synthesis of Anticoagulantly Active Heparan Sulfate Proteoglycans by Glomerular Epithelial Cells Involves Multiple 3-O-Sulfotransferase isoforms and a Limiting Precursor Pool». The Journal of Biological Chemistry 280 (45): 38059-38070. doi:10.1074/jbc.m507997200.
- ↑ Ori, A. et al. (2008). «The heparanome and regulation of cell function: structures, functions and challenges». Frontiers Biosci. 13: 4309-4338. PMID 18508513. doi:10.2741/3007.
- ↑ Sadir, R. et al. (1998). «The heparan sulphate binding sequence of interferon-γ increased the on rate of the interferon-γ / interferon-γ receptor complex formation». J. Biol. Chem. 273 (18): 10919-10925. PMID 9556569. doi:10.1074/jbc.273.18.10919. Archivado desde el original el 21 de junio de 2008. Consultado el 12 de abril de 2016.
- ↑ Tong et al (2009 Nov-Dec). «Stimulated neovascularization, inflammation resolution and collagen maturation in healing rat cutaneous wounds by a heparan sulfate glycosaminoglycan mimetic, OTR4120.». Wound Repair Regen. 17 (6): 840-52.
- ↑ Tong et al (2011 Jul). «Heparan sulfate glycosaminoglycan mimetic improves pressure ulcer healing in a rat model of cutaneous ischemia-reperfusion injury.». Wound Repair Regen 19 (4): 505-14.
- ↑ Van Neck et al, Heparan sulfate proteoglycan mimetics thrive tissue regeneration: an overview. In Intech book under the working title "Tissue Regeneration", ISBN 978-953-307-876-2 is scheduled for on line publication on Nov 26, 2011”