Paragrupo A (ADN-Y)
El paragrupo A es un conjunto de linajes del cromosoma Y ancestrales de los humanos modernos. Comúnmente conocido como haplogrupo A, se trata en realidad de un paragrupo porque es el grupo parafilético ancestral de los haplogrupos basales del árbol filogenético patrilineal del Homo sapiens y está conformado por los clados A00, A0, A1a y A1b1.
Se encuentra localizado en toda África, en especial en África austral, Sudán del Sur y Etiopía, y está relacionado con los grandes clados que descienden del Adán cromosómico, por lo que tiene gran diversidad y una antigüedad aproximada de 300.000 años. Según ISOGG-2014.[1] y PhyloTree,[2] La relación entre sus clados y su distribución más relevante, se resumen del siguiente modo:
| Adán-Y o A00-T |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||
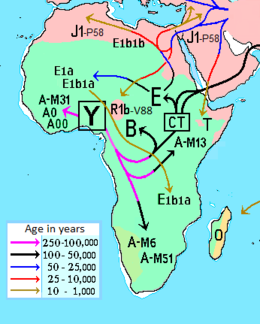
Originalmente se consideraba al paragrupo A como un haplogrupo monofilético, sin embargo, un amplio estudio (tomando 2.204 hombres africanos) de un equipo italiano encabezado por Fulvio Cruciani (2011),[3] descubre que el grupo A es parafilético y mucho más antiguo, redefiniendo y replanteando la genética cromosómica-Y humana. Actualmente (2020), los clados que lo conforman son los siguientes:[1]
Adán-Y
[editar]Adán cromosómico es la raíz o último ancestro varón común más reciente de la población actual, y se le ha denominado también haplogrupo A00-T, a veces Y, y reconocido por el polimorfismo PR2921 principalmente.[4] Luego de descubierto el haplogrupo derivado A00, el más antiguo, se ha estimado unos 275 000 años de antigüedad para el Adán-Y.[5] Dada su gran antigüedad, probablemente no podría considerase un hombre anatómicamente moderno necesariamente.
En ocasiones Adán-Y se refiere al ancestro que incluye otras poblaciones humanas extintas, como neandertales y denisovanos,[4] cuya ADN ha logrado analizarse y por lo tanto el Adán cromosómico sería en este caso mucho más antiguo.
Haplogrupo A00
[editar]A00, definido por muchos marcadores como AF6/L1284, AF4, FGC27705/YP3616 y otros 2362 más, dada su gran antigüedad. Es pues el haplogrupo más antiguo encontrado en la población humana actual y divergió directamente del Adán cromosómico hace unos 235 000 años; encontrándose al menos ocho subclados derivados en Camerún.[6]
Fue descubierto en 2012 en una familia afroamericana de Carolina del Sur de apellido Perry, la cual solicitó el análisis independientemente; por esta razón se le llamó inicialmente "cromosoma Y Perry" o simplemente Y-Perry. El antepasado de esta familia se rastreó hasta Albert Perry, afroamericano nacido entre 1819 y 1827.[7] Se considera que los afroamericanos portadores de este haplogrupo son descendientes del pobladores del África centro-occidental.
Los principales subclados de A00 (AF6) y su distribución son los siguientes:
- A00*: Encontrado en el noroeste de Camerún, en restos de hace unos 8000 años de antigüedad.[6]
- A00-Y125399: Rama principal, con unos 38 000 años de antigüedad.[6]
Comparación con especies humanas extintas
[editar]Dada la antigüedad del haplogrupo africano A00, se le ha comparado con humanos arcaicos y con nuestro pariente simio más cercano, pudiendo concluir que en la actualidad no existe o no se ha encontrado aún ningún hombre que lleve el cromosoma Y neandertal o denisovano, estableciendo las siguientes relaciones:[10][11]
| |||||||||||||||||||||||||
Haplogrupo A0
[editar]A0 (antes A1b) (V148, CTS2809.1/L991.1, V5002/Y9684/Z8911 y otros 954 marcadores): Desciende de A0-T o A0'1'2'3'4 (L1085, AF3), divergiendo hace unos 160 000 años.[12][13] Se ha encontrado a A0 escasamente, especialmente en el África centro-occidental. Reportado en pigmeos bakola al Sur de Camerún con 8.3%, en Ghana y Jamaica.[14]
Son los principales subclados:[15]
- A0a (L979)
- A0a1 (L1070)
- A0a1*: Al oeste de Gambia.
- A0a1a (P114, V151): Identificado en Camerún y Argelia.
- A0a1 (L1070)
- A0b (L92.2, L1035): Encontrado en Barbados.
Haplogrupo A1a
[editar]A1a o A1 (M31, P82, V4, Z11334 y otros 1200 marcadores) desciende de A1 (o A1'2'3'4, antes A1a-T) Tiene una antigüedad aproximada de 133 000 años.[16] Es propio del África Occidental pero en bajas frecuencias, encontrándose por ejemplo en Gambia,[17] Cabo Verde, Senegal, Guinea-Bissau, Mali, Níger y Marruecos.
Este linaje emigró a Europa en algún momento de la historia o de la prehistoria, y fue sorpresivo encontrarlo en Yorkshire, Inglaterra, en hombres de apellido Revis, que a pesar de rastrear a sus antepasados hasta el siglo XVIII, no se ha encontrado información sobre alguna ascendencia africana.[18]
Sus principales clados divergieron hace unos 10 000 años y son los siguientes:[16]
- A1a-Y7969: Disperso en África occidental (Gambia, Mali)
- A1a-Y32411
- A1a-Y32399: En Gambia
- A1a-Y37658: Disperso en parte del norte Europa (Finlandia, Noruega, Irlanda).
Haplogrupo A1b1
[editar]El haplogrupo A1b1, también llamado A2'3 (L419/PF712, M9044/BY864) es el clado de A con mayor dispersión y frecuencia, muy extendido en todo África y llega hasta el Cercano Oriente. Conjuntamente con BT desciende de A1b (P108) y se calcula que posee una antigüedad de 130 000 años.[19]
Distribución
[editar]
África Austral: Es característico del los pueblos khoisán, encontrándose que los haplotipos de estos pueblos se habían separado respecto al resto de África, "una razón de una antigua divergencia con la misma población ancestral." Las frecuencias más altas se encontraron en Namibia entre los san tsumkwe con 66% y los nama con 64%.[20] Es común entre los khoisan frecuencias del 12 al 48%.
África Nor-oriental: Se encuentra muy extendido en Sudán del Sur, especialmente en pueblos cazadores-recolectores; hay alta frecuencia en pueblos nilóticos como los dinka con 62% y en los shilluk 53%.[21] En los nuba (sur del Sudán) da 46%. En Etiopía, se reportó 41% en los falashas,[22] en bantús de Kenia 14%,[23] e iraqw de Tanzania 17%.[24] Semino et al. 2001[25] encontró el haplogrupo A en el 10% de una muestra de los oromo y 14,6% en los amhara.
África Central: En los mandara[20] y fulbe de Camerún con 14 y 12% respectivamente.[22] y pequeñas frecuencias en varias poblaciones de Gabón y en pigmeos baka, bakola y mbuti.
Son sus subclados:
A1b1a o A2
[editar]A1b1a o A2 (L602, V50, V82, V198, V224), posee una antigüedad de unos 120 000 años.[19] Presente principalmente en los pueblos khoisan, encontrándose allí entre 15%[26] y 25% de frecuencia.[20]
- A2* o A-L602*: Encontrado en Camerún.[19]
- A-M14 o A1b1a1 (M14, M23, P3/PN3/M29/L968, M71, M135, M141, M206, M276/P247, M277/P248, MEH1, P4, P5, P36.1, Page71, Page87, Page95)
- A-M6 (M6, M196, Y25057): Presente en África Austral (pueblos khoisan).
A1b1b o A3
[editar]A1b1b o A3 (M32) tiene una antigüedad aproximada de 120 000 años.[19]
- A1b1b1 antes A3a (M28, M59): Típico del Cuerno de África. Se encontró 5% en una muestra mixta de hablantes de lenguas semíticas meridionales en Etiopía.[20] Encontrado en Somalia,[27] Eritrea y Arabia Saudita.[19]
- A1b1b2 antes A3b (L427, M144, Y20629)
- A1b1b2a o A3b1 (M51, P100, P291): Típico de los pueblos joisán. Destacan los nama con 55%.[20] Presente en Angola y Namibia, también en bantúes de Sudáfrica.
- A1b1b2a1 o A3b1a (P71, P102)
- A1b1b2b o A3b2 (M13, M127, M202, M219, M305): Común en Sudán del Sur,[22] Cuerno de África, África Oriental y poco en el Cercano Oriente y Norte de Camerún. Es el principal haplogrupo en los pueblos nilóticos.[21] Hace unos 11 000 años divergió en los siguientes sublados:[19]
- A-YP4751: Disperso en África oriental y resto de África (excepto en África austral).[19]
- A-Y26839: En Kenia, Sudán del Sur, Chad, Arabia Saudita.
- A-Y30506: En África centro-occidental, África del Norte, península arábiga, Italia.
- A-Y23655: Tiene unos 50 000 años, pero diverge en los siguientes dos subclados hace unos 10 000 años.[19]
- A-Y126390: Típico de Arabia Saudita.
- A-Y23865: Importante en el cuerno de África y península arábiga.
- A-YP4751: Disperso en África oriental y resto de África (excepto en África austral).[19]
- A1b1b2a o A3b1 (M51, P100, P291): Típico de los pueblos joisán. Destacan los nama con 55%.[20] Presente en Angola y Namibia, también en bantúes de Sudáfrica.
Filogenia anterior: A como monofilético
[editar]Antes del 2011 se creía que el haplogrupo A era un solo clado monofilético y con una antigüedad mucho menor. La primera rama de A, se designó como mutación M91 (de la cual procedían A1, A2 y A3). Así todos los demás haplogrupos proceden de BT (antes BR, también conocido como YxA).
| Adán cromosómico |
| ||||||||||||
Ediciones anteriores del ISOGG (del 2006 al 2011), consideraban al haplogrupo A del siguiente modo:[28]
- Haplogrupo A (M91, P97)
- A1 (P108)
- A1a (M31, P82)
- A1b (P114)
- A2 (M6, M14, M23, M29/P3/PN3, M49, M71, M135, M141, M196, M206, M212, M276/P247, M277/P248, MEH1, P4, P5, P36.1)
- A3 (M32)
- A3a (M28, M59)
- A3b (M144, M190, M220, P289)
- A3b1 (M51, P100, P291)
- A3b2 (M13, M127, M202, M219, M305)
- A1 (P108)
A partir de Cruciani et al 2011,[3] el grupo A se considera parafilético y como sigue:
|
 |
Véase también
[editar]| Adán cromosómico | ||||||||||||||||||||||||
| A | ||||||||||||||||||||||||
| BT | ||||||||||||||||||||||||
| B | CT | |||||||||||||||||||||||
| DE | CF | |||||||||||||||||||||||
| D | E | C | F | |||||||||||||||||||||
| C1 | C2 | G | H | IJK | ||||||||||||||||||||
| IJ | K | |||||||||||||||||||||||
| I | J | LT | K2 | |||||||||||||||||||||
| L | T | MS | P | NO | ||||||||||||||||||||
| M | S | Q | R | N | O | |||||||||||||||||||
| R1 | R2 | |||||||||||||||||||||||
| R1a | R1b | |||||||||||||||||||||||
Enlaces externos
[editar]- Y-Haplogroup A DNA Project Family Tree DNA
- Y-HAPLOGROUP A The Root of the Tree of our Fathers. Ancient Roots Research.
- Y-DNA Haplogroup A and its Subclades ISOGG
- The New Root – Haplogroup A00 DNAeXplained – Genetic Genealogy
Referencias
[editar]- ↑ a b ISOGG: Y-DNA Haplogroup A and its Subclades (última revisión: May 2014)
- ↑ Oven M. et al 2014. Minimal reference phylogeny for the human Y chromosome (version 9-Apr-2014)
- ↑ a b Fulvio Cruciani, Beniamino Trombetta, Andrea Massaia, Giovanni Destro-Biso, Daniele Sellitto y Rosaria Scozzari 2011, A Revised Root for the Human Y Chromosomal Phylogenetic Tree: The Origin of Patrilineal Diversity in Africa
- ↑ a b Y-DNA Haplogroup Tree 2019-2020 International Society of Genetic Genealogy
- ↑ Fernando L. Mendez, G. David Poznik, Sergi Castellano and Carlos D. Bustamante (2016) The Divergence of Neandertal and Modern Human Y Chromosomes American journal of human genetics, 98(4), 728–734. https://doi.org/10.1016/j.ajhg.2016.02.02
- ↑ a b c A00 Y full.tree 2012-2020 YFull™
- ↑ Y-Haplogroup A Project - Y-DNA Classic Chart Family Tree DNA
- ↑ Bonnie Schrack & Matthew Fomine Forka (2015), How far we've come Experiment.
- ↑ Mendez FL, Krahn T, Schrack B, et al. (March 2013). "An African American paternal lineage adds an extremely ancient root to the human Y chromosome phylogenetic tree". Am. J. Hum. Genet. 92 (3): 454–9. doi:10.1016/j.ajhg.2013.02.002. PMC 3591855. PMID 23453668. PDF Archivado el 24 de septiembre de 2019 en Wayback Machine.
- ↑ Fernando L. Mendez, G. David Poznik, Sergi Castellano y Carlos D. Bustamante 2016, The Divergence of Neandertal and Modern Human Y Chromosomes. AJHG Volume 98, Issue 4, p728–734
- ↑ Martin Petr, Mateja Hajdinjak, Qiaomei Fu, Elena Essel, Hélène Rougie et al. 2020, The evolutionary history of Neanderthal and Denisovan Y chromosomes. Science 25 Sep 2020: Vol. 369, Issue 6511, pp. 1653-1656 DOI: 10.1126/science.abb6460
- ↑ Yfull tree A0-T 2012-2020 YFull™
- ↑ Árbol de cromosoma Y de FTDNA
- ↑ Scozzari R, Massaia A, D’Atanasio E, Myres NM, Perego UA, et al. (2012) Molecular Dissection of the Basal Clades in the Human Y Chromosome Phylogenetic Tree. PLoS ONE 7(11): e49170. doi:10.1371/journal.pone.0049170
- ↑ Yfull Tree A0 2012-2020 YFull™
- ↑ a b Yfull Tree A1a Haplogroup YTree v8.09.00 (08 October 2020) YFull™
- ↑ YFull Experimental YTree v2.21 1000 Genomes Project © HGDP Project. (rev. julio 2014)
- ↑ King, T. E., Parkin, E. J., Swinfield, G., Cruciani, F., Scozzari, R., Rosa, A., Lim, S. K., Xue, Y., Tyler-Smith, C., & Jobling, M. A. (2007). Africans in Yorkshire? The deepest-rooting clade of the Y phylogeny within an English genealogy. European journal of human genetics: EJHG, 15(3), 288–293. https://doi.org/10.1038/sj.ejhg.5201771
- ↑ a b c d e f g h Yfull Tree A1b1 Haplogroup YTree v8.09.00 (08 October 2020) YFull™
- ↑ a b c d e Elizabeth Wood et al. 2005 Contrasting patterns of Y chromosome and mtDNA variation in Africa: evidence for sex-biased demographic processes European Journal of Human Genetics 13, 867–876. doi:10.1038/sj.ejhg.5201408
- ↑ a b Hassan et al. 2008 28/53 (Dinka, Nuer, and Shilluk) Y-Chromosome Variation Among Sudanese:Restricted Gene Flow, Concordance With Language, Geography, and History
- ↑ a b c F. Cruciani et al. 2002 A Back Migration from Asia to Sub-Saharan Africa Is Supported by High-Resolution Analysis of Human Y-Chromosome Haplotypes. Am. J. Hum. Genet. 70:1197–1214, 2002
- ↑ Luis et al. 2004
- ↑ Knight et al. 2003
- ↑ O. Semino et al. "Ethiopians and Khoisan Share the Deepest Clades of the Human Y-Chromosome Phylogeny," (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última)., American Journal of Human Genetics 2002 January; 70(1): 265–268.
- ↑ Underhill PA, Shen P, Lin AA, et al. (November 2000). "Y chromosome sequence variation and the history of human populations". Nat. Genet. 26 (3): 358–61. doi:10.1038/81685. PMID 11062480. S2CID 12893406.
- ↑ Abu-Amero KK, Hellani A, González AM, Larruga JM, Cabrera VM, Underhill PA (2009). "Saudi Arabian Y-Chromosome diversity and its relationship with nearby regions". BMC Genet. 10: 59. doi:10.1186/1471-2156-10-59. PMC 2759955. PMID 19772609.
- ↑ ISOGG 2008
