Exoenzima
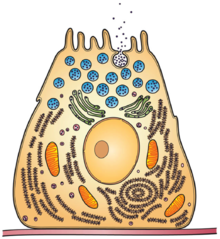
Una exoenzima, o enzima extracelular, es una enzima la cual es secretada por una célula y funciona fuera de esa célula. Las exoenzimas son producidas tanto por células procariotas como por eucariotas y han mostrado ser un componente crucial en muchos procesos biológicos. Comúnmente, estas enzimas están involucradas en el rompimiento de macromoléculas de gran tamaño. El rompimiento de estas macromoléculas es crítico para permitir a sus constituyentes pasar a través de la membrana celular y así entrar a la célula. Para los humanos y otros organismos complejos, este proceso es caracterizado por el sistema digestivo, el cual rompe alimentos sólidos por medio de las exoenzimas.[1] Las moléculas pequeñas, generadas por la actividad exoenzimática, entra a las células y es utilizada para diversas funciones celulares. Bacteria y Fungi también producen exoenzimas para digerir nutrientes provenientes de su ambiente, y estos organismos pueden ser usados para conducir ensayos de laboratorio para identificar la presencia y función de tales exoenzimas.[2] Algunas especies patógenas también utilizan exoenzimas como factores de virulencia para asistir el esparcimiento de estos microorganismos causantes de enfermedades.[3] Además de las funciones integrales en sistemas biológicos, diferentes clases de exoenzimas microbianas han sido utilizados por los seres humanos desde tiempos prehistóricos para diversos propósitos tales como la producción de alimentos, biocombustibles, producción textil y en la industrial del papel.[4] Otro importante rol que las enzimas microbianas cumplen es la ecología natural y la biorremediación de ambientes marinos y terrestres.[5]
Historia
[editar]Muy poca información está disponible acerca del descubrimiento original de las exoenzimas. De acuerdo con el diccionario Merriam-Webster, el término exoenzima fue reconocido por primera vez en el idioma inglés en 1908.[6] El libro "Enzimas Intracelulares: Un Curso de Lecturas Dadas en la Fisiología", por Horace Vernon, se cree que es la primera publicación que utiliza esa palabra en aquel año.[7] Basados en el libro, se puede asumir que las primeras exoenzimas conocidas fueron la pepsina y la tripsina, debido a que ambas fueron mencionadas por Vernon por ser descubiertas por los científicos Briike y Kiihne antes de 1908.[8]
Función
[editar]En Bacteria y Fungi, las exoenzimas juegan un rol integral en permitir a los organismos interactuar de manera efectiva con su ambiente. Muchas bacterias usan enzimas digestivas para romper nutrientes en sus alrededores. Una vez digeridos, estos nutrientes entran en la bacteria, donde éstos son usados para activar vías celulares con la ayuda de las endoenzimas.[9]
Muchas exoenzimas son también usadas como factores de virulencia. Los patógenos, tanto bacteriológicos como fúngicos, puede utilizar exoenzimas como un mecanismo primario con el cual causar enfermedades.[10] La actividad metabólica de las exoenzimas permite a la bacteria invadir a los organismos hospedadores al romper las capas de defensa celulares o necrotizando los tejidos corporales de organismos de gran tamaño.[3] Muchas bacterias gram negativa tienen inyectisomas, o proyecciones del tipo flagelar, para directamente introducir la exoenzima virulenta hacia el interior de la célula hospedadora utilizando un Sistema de Secreción Tipo Tres.[11] Con cualquiera de los procesos, los patógenos pueden atacar la estructura y la función de la célula hospedadora, así como su material genético.[12]
En células eucariotas, las exoenzimas son creadas como cualquier otra enzima por medio de la síntesis de proteínas, y son transportadas vía la ruta secretora. Después de moverlas a través del Retículo Endoplasmático Rugoso, éstas son procesadas a través del Aparato de Golgi, donde son empaquetadas en vesículas y liberadas fuera de la célula.[13] En los seres humanos, la mayoría de estas exoenzimas pueden ser encontradas en el sistema digestivo y son usadas para el rompimiento metabólico de macronutrientes vía hidrólisis. Romper esos nutrientes permite su incorporación hacia otras rutas metabólicas.[14]

Enzimas necrotizantes
[editar]Las enzimas necrotizantes destruyen células y tejidos. Uno de los ejemplos mejor conocidos es una exoenzima producida por Streptococcus pyogenes, la cual causa Fascitis necrotizante en humanos.
Coagulasa
[editar]Por medio de la unión de trombina, la coagulasa facilita la coagulación en una célula convirtiendo finalmente fibrinógeno a fibrina. Bacteras tales como Staphylococcus aureus usan la enzima para formar una capa de fibrina alrededor de su célula para protegerse de los mecanismos de defensa del hospedador.

Cinasas
[editar]Lo opuesto de la coagulasa, la cinasa (también llamadas quinasa) pueden disolver coágulos. Staphyloccocus aureus también puede producir staphyloquinasa, permitiéndoles disolver los coágulos que ellas forman, para rápidamente difundirse en el hospedador en el momento indicado.[15]
Hialuronidasa
[editar]De modo similar a la colagenasa, la hialuronidasa permite a los patógenos penetrar profundamente dentro de los tejidos. Bacterias tales como Clostridium lo hacen usando la enzima para disolver colágeno y ácido hialurónico, proteínas y glúcidos, respectivamente, los cuales mantienen al tejido unido.
Hemolisinas
[editar]Las hemolisinas tienen como objetivo los eritrocitos (o células rojas de la sangre). Atacar y lisar a esas células permite al patógeno dañar al organismo hospedador y a la vez lo provee con una fuente de hierro desde la hemoglobina lisada, como lo hace el fungi Candida albicans.[16]
Ejemplos de exoenzimas digestivas
[editar]Amilasas
[editar]
Las amilasas son un grupo de enzimas extracelulares (glucosidasas) que catalizan la hidrólisis de almidón. Estas enzimas están agrupadas dentro de tres clases basadas en sus secuencias de aminoácidos, mecanismos de reacción, métodos de catálisis y su estructura.[17] Las diferentes clases de amilasas son α-amilasas, β-amilasas y glucoamilasas. Las α-amilasas hidrolizan almidón por medio de rumpuras aleatorias en los enlaces 1,4-a-D-glucosídicos entre unidades de glucosa; las β-amilasas rompen los extremos de cadena no reductoras de los componentes del almidón tales como la amilosa; y las glucoamilasas hidrolizan las moléculas de glucosa desde los extremos de la amilasa y la amilopectina.[18] Las amilasas son sumamente importantes exoenzimas y son encontradas en plantas, animales y microorganismos. En humanos, las amilasas son secretadas tanto por el páncreas como por las glándulas salivales, ambas fuentes de la enzima son requeridas para completar la hidrólisis del almidón.[19]
Lipoproteinlipasa
[editar]La lipoproteinlipasa (LPL) es un tipo de enzima digestiva que ayuda a reglar la admisión de triglicéridos de quilomicrones y otras lipoproteínas de baja densidad desde tejidos grasos en el cuerpo. La función exoenzimática permite romper los triglicéridos en dos ácidos grasos libres y una molécula de monoacilglicerol. La LPL puede ser encontrada en células endoteriales en tejidos ácidos, tales como cardíacos adiposos y músculos. La lipoproteinlipasa es reducida con altos niveles de insulina y aumentada con altos niveles de glucagón y adrenalina.[20]
Pectinasa
[editar]Las pectinasas, también llamadas enzimas pectólicas, son una clase de exoenzimas las cuales están involucradas en el rompimiento de sustancias pécticas, más notablemente la pectina.[21] Las pectinasas pueden ser clasificadas en dos diferentes grupos basados en su acción contra la columna galacturónica de pectina: desterificante y depolimerizante.[22] Estas exoenzimas pueden ser encontradas tanto en plantas como en organismos microbianos, incluyendo fungís y bacterias.[23] Las pectinasas son más comúnmente utilizadas para romper los elementos pécticos encontrados en plantas y en productos derivados de las plantas.
Pepsina
[editar]Descubierta en 1836, la pepsina fue una de las primeras enzimas en ser clasificada como una exoenzima.[8] La enzima es primeramente hecha en su forma inactiva, el pepsinógeno, por las células principales en la cubierta del estómago.[24] Con un impulso del nervio vago, el pepsinógeno es secretado en el estómago, donde se mezcla con ácido clorhídrico para formar pepsina.[25] Una vez activa, la pepsina funciona rompiendo las proteínas en los alimentos tales como carne y huevos.[24] La pepsina funciona mejor si el pH del ácido gástrico está entre 1.5 y 2.5 y es desactivada cuando el ácido es neutralizado a un pH de 7.[24]
Tripsina
[editar]También una de las primeras exoenzimas en ser descubiertas, la tripsina fue nombrada en 1876, cuarenta años después que la pepsina.[26] Esta enzima es responsable del rompimiento de grandes proteínas globulares y su actividad es específica de romper los sitios terminales C de la arginina y los residuos de aminoácidos de la lisina.[26] Es el derivado del tripsinógeno, un precursor inactivo que es producido en el páncreas.[27] Cuando es secretado por el intestino delgado, éste se mezcla con enteroquinasa para formar tripsina activa. Debido a su rol en el intestino delgado, la tripsina trabaja en un pH óptimo de 8.0.[28]
Ensayos bacteriológicos
[editar]La producción de una exoenzima digestiva particular por una célula bacteriana puede ser evaluada usando placas de ensayo. Las bacterias son estriadas a través del agar y son dejadas incubar. La liberación de la enzima hacia los alrededores de la célula causa un rompimiento de la macromolécula en la placa. Si una reacción no ocurre, eso significa que la bacteria no crea una exoenzima capaz de interactuar con los alrededores. Si una reacción ocurre, se vuelve claro que la bacteria sí posee una exoenzima y la determinación de su identidad depende de cuál macromolécula es hidrolizada.[2]
Amilasa
[editar]La amilasa rompe carbohidratos en mono- y disácraridos, así que un agar de almidón debe ser usado para este ensayo. Una vez que la bacteria es estriada en el agar, la placa es inundada con yodo. Debido a que el yodo se enlaza al almidón pero no es digerida por subproductos, un área limpia aparecerá donde la reacción de la amilasa ha ocurrido. Bacillus subtilis es una bacteria que arroja resultados positivos en un ensayo, tal como se muestra en la imagen.[2]
Lipasa
[editar]Los ensayos de lipasa son hechos usando un agar líquido con un colorante azul ópalo. Si la bacteria posee lipasa, un veta muy clara se va a formar en el agar y el colorante rellenará esa veta, creando un halo azul oscuro alrededor del área limpiada. Staphylococcus epidermis resulta positiva en un ensayo de lipasa.[2]
Aplicaciones biotecnológicas e industriales
[editar]Las fuentes microbiológicas de exoenzimas, incluyendo amilasas, proteasas, pectinasas, lipasas, xilanasas, celulasas, entre otras, son usadas por un amplio rango de aplicaciones biotecnológicas e industriales, incluyendo la generación de biocombustibles, la producción de alimentos, la manufactura de papel, la producción de detergentes y textiles, entre otros.[4]
Optimizar la producción de biocombustibles ha sido un foco de atención para investigadores en años recientes y está centrado en el uso de microorganismos para convertir biomasa en etanol. Las exoenzimas que son de particular interés en la producción de etanol son las celubiohidrolasas, las cuales solubilizan la celulosa cristalizada y las xilanasas, las cuales hidrolizan xilano en xilosa.[29] Un modelo de producción de biocombustibles es el uso de una población mezclada de cepas bacterianas o un consorcio que trabaje para facilitar la ruptura de la celulosa en etanol vía secreciones de exoenzimas tales como celulasas y lacasas.[29] En adición al importante rol que juegan en la producción de biocombustibles, la xilanasa es utilizada en otras aplicaciones biotecnológicas e industriales debido a su habilidad de hidrolizar celulosa y hemicelulosa. Estas aplicaciones incluyen la ruptura de desechos agrícolas y forestales, funcionando como aditivos para facilitar un mayor consumo de nutrientes por el ganado, así como un ingrediente en el pan mejorando el volumen y la textura del éste.[30]

Las lipasas son unas de las exoenzimas más utilizadas en biotecnología y aplicaciones industriales. Las lipasas son enzimas ideales para estas aplicaciones debido a que ellas son altamente selectivas en su actividad, éstas son fácilmente producidas y secretadas por bacterias y fungis, su estructura cristalizada está bien caracterizada, no requieren cofactores para su actividad enzimática y ellas no catalizan reacciones secundarias.[31] El rango de usos de las lipasas engloba la producción de biopolímeros, generación de cosméticos, usos como herbicida, usos como solvente.[31] Sin embargo, quizás el uso más conocido para las lipasas en este campo es su uso en la producción de biodiésel. En este ámbito, las lipasas son usadas para convertir aceite vegetal en metil- y otros alcoholes-ésteres de cadena corta por una sola reacción de transesterificación.[32]
Las celulasas, hemicelulasas y pectinasas, son diferentes exoenzimas que están involucradas en una amplia variedad de aplicaciones biotecnológicas e industriales. En la industria alimenticia, estas exoenzimas son utilizadas en la producción de jugos de frutas, néctares de frutas, purés de frutas y en la extracción de aceite de oliva, entre muchos otros.[33] El rol que estas enzimas juegan en estas aplicaciones alimenticias es para parcialmente romper las paredes celulares de las plantas y la pectina. En adición a este rol, las celulasas son usadas en la industria textil para remover excesos de colorantes de la mezclilla, telas de algodón suaves y para restablecer el color de las telas de algodón.[33] Las celulasas y las hemicelulasas (incluyendo las xilanasas) son también utilizadas en industria del papel y de la pulpa para despintar fibras recicladas, modificar gruesas pulpas mecánicas y para la parcial o completa hidrólisis de pulpas de fibra.[33] Las celulasas y las hemicelulasas son usadas en estas aplicaciones industriales debido a su habilidad para hidrolizar los componentes de celulosa y hemicelulosa encontrados en estos materiales.
Aplicaciones en la Biorremediación
[editar]
La biorremediación es el proceso en el cual los contaminantes del medio ambiente son removidos a través del uso de organismos biológicos o de sus productos. La remoción de éstos, usualmente contaminantes peligrosos, es mayormente llevado a cabo por microorganismos presentes naturalmente o introducidos intencionalmente los cuales son capaces de romper o absorber el contaminante designado. Los tipos de contaminantes que comúnmente son objetivos de estrategias de biorremediación son productos del petróleo (incluyendo gasolinas y solventes) y pesticidas.[34] En adición a la habilidad de los microorganismos para digerir y absorber los contaminantes, sus exoenzimas secretadas juegan un importante rol en varias estrategias de biorremediación.[35]
Los agentes fúngicos han mostrado ser organismos viables para llevar a cabo procesos de biorremediación y han sido usados para asistir en la descontaminación de un amplio número de contaminantes, incluyendo hidrocarburos aromáticos policíclicos (PHAs), pesticidas, colorantes sintéticos, clorofenoles, explosivos, petróleo crudo, entre muchos otros.[36] Mientras que los fungís pueden romper muchos de estos contaminantes intracelularmente, ellos también secretan numerosas exoenzimas oxídativas las cuales trabajan extracelularmente. Un aspecto crítico de los fungís en relación con la biorremediación es que ellos secretan estas exoenzimas oxídativas desde sus siempre alargables hifas.[36] Las lacasas son unas importantes exoenzimas oxídativas que los fungís secretan y utilizan oxígeno para oxidar muchos contaminantes. Algunos de los contaminantes en los que las lacasas han sido usadas incluyen efluentes que contienen colorantes provenientes de la industria textil, contaminantes del agua (tales como clorofenoles, PAHs, etc.) y contaminantes con componentes del azufre provenientes del procesamiento del carbón.[36]

Las bacterias son también una fuente viable de exoenzimas capaces de facilitar la biorremediación del medio ambiente. Hay muchos ejemplos del uso de bacterias para este propósito y sus exoenzimas engloban varias clases diferentes de exoenzimas. Son de particular interés para este campo las hidrolasas bacterianas, debido a que ellas tienen intrínsecamente una baja especificidad de sustrato y pueden ser usadas para numerosos contaminantes, incluyendo desechos sólidos.[37] Los desechos plásticos, incluyendo poliuretanos, son particularmente difíciles de degradar, pero una exoenzima ha sido identificada in una bacteria Gram negativa, Comamonas acidovorans, la cual es capaz de degradar los desechos de poliuretano del medio ambiente.[37] El uso de células productoras de exoenzimas microbianas como agentes de biorremediación es también posible a pesar de que su actividad usualmente no es tan robusta, e introducir las enzimas en ciertos ambientes tales como el suelo ha sido desafiante.[37] En adición al tratamiento de suelos vía microorganismos, las bacterias usadas en medios marinos y sus exoenzimas muestran potencial como candidatos en el campo de la biorremediación. Bacterias utilizadas en medios marinos han sido utilizadas para remover metales pesados, en la degradación de petróleo y diésel y en la remoción de policarbonos poliaromáticos, entre otros.[38]
Referencias
[editar]- ↑ Kong F; Singh RP (Jun 2008). «Disintegration of solid foods in human stomach». Journal of Food Science 73 (5): R67-80. PMID 18577009. doi:10.1111/j.1750-3841.2008.00766.x.
- ↑ a b c d Roberts, K. «Exoenzymes». Prince George's Community College. Archivado desde el original el 13 de junio de 2013. Consultado el 8 de diciembre de 2013.
- ↑ a b c Duben-Engelkirk, Paul G. Engelkirk, Janet (2010). Burton's microbiology for the health sciences (9th edición). Philadelphia: Wolters Kluwer Health/Lippincott Williams & Wilkins. pp. 173–174. ISBN 9781605476735.
- ↑ a b Thiel, ed. by Joachim Reitner, Volker (2011). Encyclopedia of geobiology. Dordrecht: Springer. pp. 355–359. ISBN 9781402092121.
- ↑ Arnosti C (15 de enero de 2011). «Microbial extracellular enzymes and the marine carbon cycle». Annual Review of Marine Science 3 (1): 401-25. PMID 21329211. doi:10.1146/annurev-marine-120709-142731.
- ↑ «Merriam-Webster». Consultado el 26 de octubre de 2013.
- ↑ «Lexic.us». Consultado el 26 de octubre de 2013.
- ↑ a b Vernon, Horace. «Intracellular Enzymes: A Course of Lectures Given in the Physiological». Consultado el 26 de octubre de 2013.
- ↑ Kaiser, Gary. «LAB 8: IDENTIFICATION OF BACTERIA THROUGH BIOCHEMICAL TESTING». Biol 230 Lab Manual. Archivado desde el original el 11 de diciembre de 2013. Consultado el 9 de diciembre de 2013.
- ↑ Pakshir, Keyvan (julio de 2013). «FUNGAL EXOENZYMES, CHARACTERISTICS AND DIAGNOSIS». Jundishapur Journal of Microbiology (2).
- ↑ Erhardt M; Namba K; Hughes KT (Nov 2010). «Bacterial nanomachines: the flagellum and type III injectisome». Cold Spring Harbor Perspectives in Biology 2 (11): a000299. PMID 20926516. doi:10.1101/cshperspect.a000299.
- ↑ McGuffie EM; Fraylick JE; Hazen-Martin DJ; Vincent TS; Olson JC (Jul 1999). «Differential sensitivity of human epithelial cells to Pseudomonas aeruginosa exoenzyme S». Infection and Immunity 67 (7): 3494-503. PMID 10377131.
- ↑ Lodish, Harvey (2008). Molecular cell biology (6th ed., [2nd print.]. edición). New York [u.a.]: Freeman. ISBN 0716776014.
- ↑ Andrews, Lary. «Supplemental Enzymes for Digestion». Health and Healing Research. Archivado desde el original el 27 de julio de 2013. Consultado el 9 de diciembre de 2013.
- ↑ Todar, Kenneth. «Mechanisms of Bacterial Pathogenicity». Todar's Online Textbook of Bacteriology. Kenneth Todar, PhD. Consultado el 12 de diciembre de 2013.
- ↑ Favero D; Furlaneto-Maia L; França EJ; Góes HP; Furlaneto MC (Feb 2014). «Hemolytic factor production by clinical isolates of Candida species». Current Microbiology 68 (2): 161-6. PMID 24048697. doi:10.1007/s00284-013-0459-6.
- ↑ Sharma, Archana; Satyanarayana, T. (2013). «Microbial acid-stable alpha-amylases: Characteristics, genetic engineering and applications». Process Biochemistry 48: 201-211. doi:10.1016/j.procbio.2012.12.018.
- ↑ Pandey A; Nigam P; Soccol CR; Soccol VT; Singh D; Mohan R (2000). «Advances in microbial amylases». Biotechnol. Appl. Biochem. 31 (2): 135-52. PMID 10744959. doi:10.1042/ba19990073.
- ↑ Pandol, Stephen. «The Exocrine Pancreas». The Exocrine Pancreas. Morgan & Claypool Life Sciences. Consultado el 25 de noviembre de 2013.
- ↑ Mead JR; Irvine SA; Ramji DP (Dec 2002). «Lipoprotein lipase: structure, function, regulation, and role in disease». Journal of Molecular Medicine 80 (12): 753-69. PMID 12483461. doi:10.1007/s00109-002-0384-9.
- ↑ Jayani, Ranveer Singh; Saxena, Shivalika; Gupta, Reena (1 de septiembre de 2005). «Microbial pectinolytic enzymes: A review». Process Biochemistry 40 (9): 2931-2944. doi:10.1016/j.procbio.2005.03.026.
- ↑ Alimardani-Theuil, Parissa; Gainvors-Claisse, Angélique; Duchiron, Francis (1 de agosto de 2011). «Yeasts: An attractive source of pectinases—From gene expression to potential applications: A review». Process Biochemistry 46 (8): 1525-1537. doi:10.1016/j.procbio.2011.05.010.
- ↑ Gummadi, Sathyanarayana N.; Panda, T. (1 de febrero de 2003). «Purification and biochemical properties of microbial pectinases—a review». Process Biochemistry 38 (7): 987-996. doi:10.1016/S0032-9592(02)00203-0.
- ↑ a b c «Encyclopedia Britannica». Consultado el 14 de noviembre de 2013.
- ↑ Guldvog I; Berstad A (1981). «Physiological stimulation of pepsin secretion. The role of vagal innervation». Scandinavian Journal of Gastroenterology 16 (1): 17-25. PMID 6785873.
- ↑ a b Worthington, Krystal. «Trypsin». Worthington Biochemical Corporation. Consultado el 26 de noviembre de 2013.
- ↑ «Trypsin». Free Dictionary. Consultado el 26 de noviembre de 2013.
- ↑ «Trypsin Product Information». Worthington Biochemical Corporation. Consultado el 26 de noviembre de 2013.
- ↑ a b Alper H; Stephanopoulos G (Oct 2009). «Engineering for biofuels: exploiting innate microbial capacity or importing biosynthetic potential?». Nature Reviews. Microbiology 7 (10): 715-23. PMID 19756010. doi:10.1038/nrmicro2186.
- ↑ Juturu V; Wu JC (1 de noviembre de 2012). «Microbial xylanases: engineering, production and industrial applications». Biotechnology Advances 30 (6): 1219-27. PMID 22138412. doi:10.1016/j.biotechadv.2011.11.006.
- ↑ a b Jaeger, Karl-Erich; Thorsten Eggert (2002). «Lipases for biotechnology». Current Opinion in Biotechnology 13: 390-397. doi:10.1016/s0958-1669(02)00341-5.
- ↑ Fan X; Niehus X; Sandoval G (2012). «Lipases as biocatalyst for biodiesel production». Methods in Molecular Biology 861: 471-83. PMID 22426735. doi:10.1007/978-1-61779-600-5_27.
- ↑ a b c Bhat, M.K. (2000). «Cellulases and related enzymes in biotechnology». Biotechnology Advances 18: 355-383. doi:10.1016/s0734-9750(00)00041-0.
- ↑ «A Citizen’s Guide to Bioremediation». United States Environmental Protection Agency. septiembre de 2012. Consultado el 5 de diciembre de 2013.
- ↑ Karigar CS; Rao SS (2011). «Role of microbial enzymes in the bioremediation of pollutants: a review». Enzyme Research 2011: 805187. PMC 3168789. PMID 21912739. doi:10.4061/2011/805187.
- ↑ a b c Harms H; Schlosser D; Wick LY (Mar 2011). «Untapped potential: exploiting fungi in bioremediation of hazardous chemicals». Nature Reviews. Microbiology 9 (3): 177-92. PMID 21297669. doi:10.1038/nrmicro2519.
- ↑ a b c «Potential of extra cellular enzymes in remediation of polluted soils: a review». Enzyme and Microbial Technology 35 (4): 339-354. septiembre de 2004. doi:10.1016/j.enzmictec.2004.05.006.
- ↑ «Marine bacteria: potential candidates for enhanced bioremediation». Applied Microbiology and Biotechnology 97 (2): 561-71. Jan 2013. PMID 23212672. doi:10.1007/s00253-012-4584-0.

