Transferencia genética horizontal
La transferencia genética horizontal (TGH) es el movimiento de material genético entre organismos unicelulares y/o pluricelulares, que no es a través de la transmisión vertical (la transmisión del ADN de padres a su descendencia).[1] La TGH es sinónimo de transferencia genética lateral (TGL) y ambos términos son intercambiables.[2][3][4] Este movimiento ha demostrado ser un factor importante en la evolución de muchos organismos.[5]
La transferencia genética horizontal es la razón principal de que se propague en las bacterias la resistencia a los antibióticos.[5][6][7][8][9] y juega un papel importante en la evolución de bacterias que pueden degradar compuestos nuevos como los pesticidas creados por los humanos,[10] y en la evolución, el mantenimientos y la transmisión de virulencia.[11] Esta transferencia genética horizontal normalmente involucra el uso de virus, plásmidos y transposones.[12] A su vez, los virus pueden recibir genes especiales de otros agentes más pequeños conocidos como virus satélite. Los plásmidos y transposones pueden recibir genes especiales de los integrones que son casetes de genes con movilidad. Esto demuestra que hay movimiento masivo de genes inclusive entre las partículas vectoras.[13] Los genes que son responsables de la resistencia a antibióticos en una especie de bacteria pueden ser transferidos a otra especie a través de varios mecanismos (e.g. via F-pilus), subsecuentemente armando al recipiente de genes contra antibióticos, lo que se está convirtiendo en un reto médico.
La mayoría de los trabajos en genética se han concentrado en la transferencia vertical, pero hay un incremento en la conciencia que la transferencia genética horizontal es un fenómeno sumamente significativo entre organismos unicelulares, probablemente la forma dominante de transferencia genética.[14][15]
La transferencia genética horizontal artificial es una forma de ingeniería genética.
Historia[editar]
La transferencia genética horizontal fue descrita por primera vez en Seattle en 1951 en una publicación que demostraba que la transferencia de un gen viral a Corynebacterium diphtheriae creó una forma virulenta de una especie no virulenta,[16] resolviendo casi simultáneamente el acertijo de la difteria (que los pacientes podían estar infectados con la bacteria pero sin presentar ningún síntoma, y después cambiaba repentinamente o nunca),[17] y dando el primer ejemplo para la relevancia del ciclo lisogénico.[18] La transferencia genética inter-bacteriana fue descrita por primera vez en Japón en una publicación de 1959 que demostraba la transferencia de resistencia a antibióticos entre diferentes especies de bacteria.[19][20] En los mediados de los 80, Syvanen[21] predijo que la transferencia genética lateral existía, tenía significancia biológica, y estaba involucrada en la formación de la historia evolutiva y del inicio de la vida en la Tierra.
Como mencionan Jian, Rivera y Lake (1999): "Estudios de genes y genomas están indicando que ha ocurrido considerable transferencia genética horizontal en procariotes"[22] (véase también Lake y Rivera, 2007).[23] El fenómenos parece tener algún significado para eucariotas unicelulares también. Como menciona Bapteste et al. (2005), "más evidencia sugiere que la transferencia genética también pueda ser un mecanismo evolutivo importante en la evolución protista."[24][25]
Los injertos a una planta de otra pueden transferir cloroplastos (ADN especializado en plantas que puede conducir la fotosíntesis) ADN mitocondrial y el núcleo celular completo conteniendo el genoma para potencialmente hacer una nueva especie[26] Algunos Lepidoptera (e.g. mariposas monarca y gusanos de seda) han sido modificados genéticamente por transferencia genética horizontal del virus de la avispa.[27] Mordidas del insecto Reduviidae (bicho asesino) pueden, a través de un parásito, infectar humanos con la enfermedad trypanosomal de Chagas, que puede insertar su ADN al genoma humano.[28] Se ha sugerido que la transferencia lateral en humanos puede desempeñar un papel importante para el tratamiento del cáncer.[29]
Richardson y Palmer (2007) argumentan que: "La transferencia genética horizontal ha jugado un papel importante en la evolución bacteriana y es bastante común en algunos eucariotas unicelulares. Sin embargo, la prevalencia y la importancia de la TGH en la evolución de eucariotas pluricelulares permanece incierta.."[30]
Debido al incremento en la cantidad de evidencia sugiriendo la importancia de estos fenómenos para la evolución, los biólogos moleculares como Peter Gogarten han descrito la transferencia genética horizontal como "Un nuevo paradigma para la Biología".[31]
Algunos argumentan que puede representar un peligro oculto en la ingeniería genética, ya que puede permitir que el ADN transgénico se propague entre especies.[25]
Mecanismo[editar]
Hay varios mecanismos para la transferencia genética horizontal:[5][32][33]
- Transformación: la alteración genética de una célula resultando de la introducción, toma y expresión de material genético foráneo (ADN or ARN).[34] Este proceso es relativamente común en las bacterias, pero menos en los eucariotas.[35] La transformación se usa seguido en los laboratorios para insertar nuevos genes en bacterias para experimentos o para aplicaciones industriales o médicas. Véase también biología molecular y biotecnología
- Transducción: el proceso en el que el ADN bacteriano se mueve de una bacteria a otra por un virus (bacteriófago).[34]
- Conjugación bacteriana: un proceso que involucra la transferencia de ADN a través de un plásmido de una célula donadora a una célula receptora recombinante durante el contacto de célula-a-célula.[34]
- Agentes de transferencia genética: elementos como virus codificados por el huésped que se encuentran el orden de alfaproteobacterias, Rhodobacterales.[36]
Un transposón (gen saltarín) es un segmento móvil de ADN que a veces puede recoger un gen resistente e insertarlo en un plásmido o cromosoma, por tanto induciendo la transferencia genética horizontal de resistencia a antibióticos.[34]
Inferencia[editar]
La transferencia genética horizontal es inferida típicamente usando métodos de bioinformática, tanto para identificar firmas de secuencias atípicas (métodos "paramétricos" ) o identificando fuertes discrepancias entre la historia evolutiva de secuencias particulares comparadas a las de sus huéspedes.
Virus[editar]
El virus llamado mimivirus infecta a las amebas. Otro virus, llamado Sputnik, también infecta a las amebas, pero no puede reproducirse a menos que mimivirus ya haya infectado la misma célula.[37] "El genoma de Sputnik revela más visión hacia su biología. Aunque 13 de sus genes muestran poco similitud a cualquier otro gen conocido, tres están cercanamente relacionados con genes de mimivirus y mamavirus, quizás canibalizados por el pequeño virus ya que empaca partículas en alguna parte de su historia. Esto sugiere que el virus satélite puede realizar la transferencia genética horizontal entre virus, de manera paralela a la manera en que los bacteriófagos llevan los genes entre las bacterias."[38] La transferencia genética horizontal también se observa en geminivirus en las plantas de tabaco.[39]
Procariotas[editar]
La transferencia genética horizontal es común entre las bacterias, incluso entre las que su relación está más distanciada. Este proceso se cree es una causa significativa de la aumentada resistencia a fármacos[5][40] cuando una célula bacteriana adquiere resistencia, y los genes de resistencia son transferidos a otras especies.[41][42] Transposición y transferencia genética horizontal, junto con poderosas fuerzas naturales y selectivas han llevado a especies como Staphylococcus aureus a ser resistentes a varios fármacos y otras bacterias patogénicas.[5] Un claro ejemplo que concierne la propagación de exotoxinas es la evolución adaptativa de toxinas Shiga en E. coli a través de la transferencia genética horizontal vía transducción con especies de Shigella.[43] Estrategias para combatir ciertas infecciones bacterianas al marcar estos factores específicos de virulencia y los elementos genéticos móviles han sido propuestos.[11] Por ejemplo, los elementos genéticos transferidos horizontalmente juegan papeles importantes en la virulencia de E. coli, Salmonella, Streptococcus y Clostridium perfringens.[5]
En procariotas, los sistemas de modificación de restricción son conocidos por proporcionar inmunidad contra la transferencia genética horizontal y en estabilizar elementos genéticos móviles. Los genes codificando los sistemas de modificación de restricción han reportado mover entre genomas procariontes dentro de elementos genéticos móviles como plásmidos, profagos, inserción de secuencias/transposones, e integrones. Son una barrera codificada cromosómicamente para los elementos genéticos móviles más que una herramienta de elementos genéticos móviles para infección celular.[44]
Eucariotas[editar]
"Las comparaciones en las secuencias sugiere transferencia horizontal reciente de muchos genes entre diversas especies incluyendo a través de las fronteras de los "dominios" filogenéticos. En consecuencia, determinar la historia filogenética de una especie no se puede hacer de manera concluyente determinando árboles evolucionarios para genes sencillos."[45]
- Los virus durante las infecciones virales también pudieron transferir genes de organismos eucariotas a otros.[46]
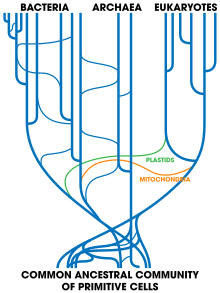
- Los análisis de secuenciación del ADN sugieren que la transferencia genética horizontal ocurrió dentro de los eucariontes del cloroplasto y genomas mitocondriales al genoma nuclear. Como mencionado en la teoría endosimbiótica, los cloroplastos y la mitocondria probablemente se originaron por endosimbiosis bacteriana, dando lugar el progenitor de la célula eucariota.[47]
- La transferencia horizontal ocurre de una bacteria a algún hongo, como la levadura Saccharomyces cerevisiae.[48][49]
- El escarabajo del frijol adzuji ha adquirido material genético de su (no-beneficial) endosimbionte Wolbachia.[50] Nuevos ejemplos han sido reportados demostrando que la bacteria Wolbachia representa un fuente potencial importante de material genético en artrópodos y nematodos .[51][52]
- Striga hermonthica, recibió un gen de sorghum (Sorghum bicolor) en su genoma nuclear.[53]
- El patógeno de la malaria, Plasmodium vivax adquirió material genético de los humanos[54] que le puede facilitar su larga estancia en el cuerpo.[55][56]
- Un mecanismo mediado por bacteriófago transfiere genes entre procariotas y eucariotas. Las señales de localización nuclear en proteínas terminales de bacteriófagos empiezan la replicación del ADN y se juntan con el genoma viral.[57] El papel de los virus y bacteriófagos en TGH en bacteria, sugiere que los genomas que contengan proteínas terminales podrían ser una vehículo entre reinos para transferencias de información genética a través de toda le evolución.[58]
- HhMAN1 es un gen en el genoma de la broca del café (Hypothenemus hampei) que se parece a genes bacterianos, y se cree que se transfirió de una bacteria en el intestino del insecto.[59][60]
- Un gen que permitió que los helechos sobrevivieran en los oscuros bosques vino de los antoceros, que crecen en esteras, arroyos y árboles. El gen neocromo llegó hace alrededor de 180 millones de años.[61]
- Las plantas son capaces de recibir información genética de virus por transferencia genética horizontal.[39]
- Un estudio identificó aproximadamente los 20,000 genes de 100 humanos que probablemente son resultado de transferencia genética horizontal,[62] pero este número ha sido desafiado por varios investigadores que argumentan que estos genes candidatos para TGH es más probable que sean el resultado de una pérdida de genes combinada con diferencias que ocurren en el transcurso de la evolución[63]
- Los rotíferos de la familia Bdelloidea tienen el 'récord' dd TGH en animales con ~8% de sus genes siendo de orígenes bacterianos.[64] Los tardígrados creyeron romper el récord con 17.5% de TGH, pero ese descubrimiento fue un artefacto de contaminación bacteriana,.[65][66]
Transferencia horizontal de transposones (THT)[editar]
La transferencia horizontal de transposones (THT) se refiere al paso de piezas del ADN que están caracterizadas por su habilidad de moverse de un locus a otro entre genomas por otros medios aparte de herencia de padre a descendencia. La transferencia genética horizontal se ha pensando es crucial para la evolución procarionte, pero existe información que sigue aumentando mostrando que la THT es un fenómeno común y bastante esparcido en la evolución eucarionte.[67] Del lado del elemento transponible, a través de transferencia horizontal puede ser visto como una estrategia para escapar una limpieza debido a selección de purificación, decaimiento mutacional y/o mecanicismos de defensa del huésped.[68]
THT puede ocurrir con cualquier tipo de elementos transponibles, pero los transposones de ADN, y los retrotransposones LTR, tienen mayor probabilidad de ser capaces de la THT porque ambos tienen un intermediario de una doble cadena de ADN, estable que se cree es más fuerte que una sola hebra de intermediario de ARN de retrotransposones no-LTR, que pueden ser altamente degradables.[67]
La THT ha sido demostrada que ocurre entre especies y a través de continentes en ambos, plantas y animales.[69] (Ivancevic et al. 2013), aunque algunos elementos transponibles han mostrado mayor éxito en colonizar genomas de ciertas especies sobre otras.[70] Tanto la proximidad espacial como taxonómica ha sido propuesta para favorecer transferencias horizontales de transposones en plantas y animales.[69] No se conoce como la densidad de una población puede afectar la velocidad de los eventos de THT dentro de la población, pero la proximidad cercanas debido a parasitismo y cruza de contaminación debido al acumulamiento han sido propuestos para favorecer THT en plantas y animales.[69] La transferencia exitosa de elementos transponibles requiere la entrega del ADN del donado a la célula huésped (y a la línea de gérmenes para organismos pluricelulares), seguido de la integración al genoma recibidor del huésped.[67] Aunque el mecanismo real del transporte de elementos transponibles de células donadoras a células de huésped no se conoce, está establecido que el ADN desnudo y el ARN pueden circular en fluido corporal.[67] Muchos vectores propuestos incluyen artrópodos, virus, caracol de agua dulce. bacteria endosimbiótica, y bacteria intracelular parasítica.[67] En algunos casos, incluso los elementos transponibles transportan otros elementos transponibles.[70][71]
Transferencia genética horizontal artificial[editar]

La ingeniería genética es esencialmente transferencia genética horizontal, aunque con casetes sintéticos de expresión. El sistema de transposones "Bella durmiente" fue desarrollado como un agente sintético de transferencia genética que fue basado en las habilidades de los transposones Tc1/mariner para invadir genomas de especies extremadamente diversas.[72] Este sistema, ha sido usado para introducir secuencias genéticas en gran variedad de genomas animales.[73][74] (Véase también terapia génica )
Importancia en la evolución[editar]
La transferencia genética horizontal es un factor cofundador en inferir árboles filogenéticos basados en la secuencia de un gen.[75] Por ejemplo, dadas dos bacterias relacionadas distantemente que han intercambiado un gen un árbol filogenético incluyendo esas especies demostrará que están relacionadas cercanamente porque ese gen es el mismo aunque muchos otros genes no son similares. Por esta razón a veces es ideal usar otra información para inferir grande filogenia como la presencia o ausencia de genes, o más común incluir como un gran rango de genes para el análisis filogenético.
Por ejemplo, el gen más común en ser usado para construir relaciones filogenéticas en procariotas es el gen ARNr 16s ya que su secuencia tiende a conservarse entre miembros con distancias filogenéticas cercanas, pero suficiente variables que las diferencias se pueden medir. Sin embargo, en años recientes se ha argumentado que los genes de ARNr 16s también pueden transferirse horizontalmente. Aunque esto puede ser no muy frecuente, la validez de los árboles filogenéticos de ARNr 16s debe ser revaluada.[76]
El biólogo Johann Peter Gogarten sugiere que "la metáfora original de un árbol ya no se ajusta a los datos de las investigaciones recientes sobre el genoma", por lo que "los biólogos deberían utilizar la metáfora de un mosaico para describir las diferentes historias combinadas en los genomas individuales y utilizar la metáfora de una red para visualizar el rico intercambio y los efectos cooperativos del HGT entre los microbios" [31] Existen varios métodos para inferir dichas redes filogenéticas.
Usando genes sencillos como marcadores filogenéticos, es difícil rastrear la filogenía en la presencia de la transferencia genética horizontal. Combinando el simple modelo de cladogenesis con raros eventos de TGH sugiere que no había un simple ancestro en común más reciente que contuviera todos los genes ancestrales a esos compartidos entre tres dominios de la vida. Cada molécula contemporaria tiene su propia historia y se rastrea en la historia a una molécula individual. Sin embargo, estos ancestros moleculares era más probable encontrarlos en diferentes organismos en diferentes momentos."[77]
Genes[editar]
Hay evidencia de transferencia histórica horizontal de los siguientes genes:
- El gen de la enzima licopeno ciclasa para la biosíntesis de carotenoides, transferido entre Chlorobi y Cyanobacteria.[78]
- El gen TetO confiere resistencia a la tetraciclina, entre Campylobacter jejuni.[79]
- Neochrome, gen en algunos helechos que mejora su habilidad de supervivencia en luz tenue. Se cree que se ha adquirido de algas en algún momento durante la era Cretácea.[80][81][82]
Véase también[editar]
- Transducción (genética)
- Retrovirus endógeno
- Línea germinal
- Línea celular HeLa
- Integrón
- Provirus
- Retrotransposón
- Retroposón
- Recombinación genética
- Convergencia evolutiva a nivel molecular
Fuente y notas[editar]
- ↑ Keeling, P. J., & Palmer, J.D. (agosto de 2008). «Horizontal gene transfer in eukaryotic evolution». Nature Reviews Genetics 9 (8): 605-618. PMID 18591983. doi:10.1038/nrg2386.
- ↑ Ochman, H., Lawrence, J. G., & Groisman, E. A. (mayo de 2000). «Lateral gene transfer and the nature of bacterial innovation.». Nature 405 (6784): 299-304. PMID 10830951. doi:10.1038/35012500.
- ↑ Hotopp, J. C. D. (abril de 2011). «Horizontal gene transfer between bacteria and animals». Trends in Genetics 27 (4): 157-163. PMC 3068243. PMID 21334091. doi:10.1016/j.tig.2011.01.005.
- ↑ Robinson, K. M., Sieber, K. B., & Hotopp, J. C. D. (octubre de 2013). «A review of bacteria-animal lateral gene transfer may inform our understanding of diseases like cancer». PLoS Genet 9 (10): e1003877. PMC 3798261. PMID 24146634. doi:10.1371/journal.pgen.1003877.
- ↑ a b c d e f Gyles, C; Boerlin P (marzo de 2014). «Horizontally transferred genetic elements and their role in pathogenesis of bacterial disease». Veterinary Pathology 51 (2): 328-340. PMID 24318976. doi:10.1177/0300985813511131.
- ↑ OECD, Safety Assessment of Transgenic Organisms, Volume 4: OECD Consensus Documents, 2010, pp.171-174
- ↑ Kay E, Vogel TM, Bertolla F, Nalin R, Simonet P (julio de 2002). «In situ transfer of antibiotic resistance genes from transgenic (transplastomic) tobacco plants to bacteria». Appl. Environ. Microbiol. 68 (7): 3345-51. PMC 126776. PMID 12089013. doi:10.1128/aem.68.7.3345-3351.2002.
- ↑ Koonin EV, Makarova KS, Aravind L (2001). «Horizontal gene transfer in prokaryotes: quantification and classification». Annu. Rev. Microbiol. 55 (1): 709-42. PMID 11544372. doi:10.1146/annurev.micro.55.1.709.
- ↑ Nielsen KM (1998). «Barriers to horizontal gene transfer by natural transformation in soil bacteria». APMIS Suppl. 84: 77-84. PMID 9850687.
- ↑ McGowan C, Fulthorpe R, Wright A, Tiedje JM (octubre de 1998). «Evidence for interspecies gene transfer in the evolution of 2,4-dichlorophenoxyacetic acid degraders». Appl. Environ. Microbiol. 64 (10): 4089-92. PMC 106609. PMID 9758850.
- ↑ a b Keen, E. C. (diciembre de 2012). «Paradigms of pathogenesis: Targeting the mobile genetic elements of disease». Frontiers in Cellular and Infection Microbiology 2: 161. PMC 3522046. PMID 23248780. doi:10.3389/fcimb.2012.00161.
- ↑ Naik GA, Bhat LN, Chpoade BA, Lynch JM (1994). «Transfer of broad-host-range antibiotic resistance plasmids in soil microcosms». Curr. Microbiol. 28 (4): 209-215. doi:10.1007/BF01575963.
- ↑ Varga M, Kuntova L, Pantucek R, Maslanova I, Ruzickova V, Doskar J (2012). «Efficient transfer of antibiotic resistance plasmids by transduction within methicillin-resistant Staphylococcus aureus USA300 clone». FEMS Microbiol. Lett. 332 (2): 146-152. PMID 22553940. doi:10.1111/j.1574-6968.2012.02589.x.
- ↑ Lin Edwards (4 de octubre de 2010). «Horizontal gene transfer in microbes much more frequent than previously thought». PhysOrg.com. Consultado el 6 de enero de 2012.
- ↑ Carrie Arnold (18 de abril de 2011). «To Share and Share Alike: Bacteria swap genes with their neighbors more frequently than researchers have realized». Scientific American. Consultado el 6 de enero de 2012.
- ↑ Victor J Freeman (1951). «Studies on the virulence of bacteriophage-infected strains of Corynebacterium Diphtheriae». Journal of Bacteriology 61 (6): 675-688. PMC 386063. PMID 14850426.
- ↑ Phillip Marguilies "Epidemics: Deadly diseases throughout history". Rosen, New York. 2005.
- ↑ André Lwoff (1965). "Interaction among Virus, Cell, and Organism". Nobel Lecture for the Nobel Prize in Physiology or Medicine.
- ↑ Ochiai K, Yamanaka T, Kimura K, Sawada, O (1959). «Inheritance of drug resistance (and its transfer) between Shigella strains and Between Shigella and E. coli strains». Hihon Iji Shimpor (en japonés) 1861: 34.
- ↑ Akiba T, Koyama K, Ishiki Y, Kimura S, Fukushima T (abril de 1960). «On the mechanism of the development of multiple-drug-resistant clones of Shigella». Jpn. J. Microbiol. 4 (2): 219-27. PMID 13681921. doi:10.1111/j.1348-0421.1960.tb00170.x.
- ↑ Syvanen M (enero de 1985). «Cross-species gene transfer; implications for a new theory of evolution» (PDF). J. Theor. Biol. 112 (2): 333-43. PMID 2984477. doi:10.1016/S0022-5193(85)80291-5.
- ↑ Jain R, Rivera MC, Lake JA (marzo de 1999). «Horizontal gene transfer among genomes: The complexity hypothesis». Proc. Natl. Acad. Sci. U.S.A. 96 (7): 3801-6. Bibcode:1999PNAS...96.3801J. PMC 22375. PMID 10097118. doi:10.1073/pnas.96.7.3801.
- ↑ Rivera MC, Lake JA (septiembre de 2004). «The ring of life provides evidence for a genome fusion origin of eukaryotes». Nature 431 (7005): 152-5. Bibcode:2004Natur.431..152R. PMID 15356622. doi:10.1038/nature02848. Archivado desde el original el 27 de septiembre de 2007.
- ↑ Bapteste E, Susko E, Leigh J, MacLeod D, Charlebois RL, Doolittle WF (2005). «Do orthologous gene phylogenies really support tree-thinking?». BMC Evol. Biol. 5 (1): 33. PMC 1156881. PMID 15913459. doi:10.1186/1471-2148-5-33.
- ↑ a b Mae-Wan Ho (1999). «Cauliflower Mosaic Viral Promoter – A Recipe for Disaster?». Microbial Ecology in Health and Disease 11 (4): 194-7. doi:10.3402/mehd.v11i4.7918. Archivado desde el original el 11 de septiembre de 2008. Consultado el 9 de junio de 2008.
- ↑ Le Page, Michael (17 de marzo de 2016). «Farmers may have been accidentally making GMOs for millennia» (en inglés estadounidense). The New Scientist. Consultado el 11 de julio de 2016.
- ↑ Gasmi, Laila; Boulain, Helene; Gauthier, Jeremy; Hua-Van, Aurelie; Musset, Karine; Jakubowska, Agata K.; Aury, Jean-Marc; Volkoff, Anne-Nathalie et al. (17 de septiembre de 2015). «Recurrent Domestication by Lepidoptera of Genes from Their Parasites Mediated by Bracoviruses». PLOS Genet 11 (9): e1005470. ISSN 1553-7404. PMC 4574769. PMID 26379286. doi:10.1371/journal.pgen.1005470.
- ↑ Yong, Ed (14 de febrero de 2010). «Genes from Chagas parasite can transfer to humans and be passed on to children» (en inglés estadounidense). National Geographic. Consultado el 13 de julio de 2016.
- ↑ Riley, DR; Sieber, KB; Robinson, KM; White, JR; Ganesan, A (2013). «Bacteria-Human Somatic Cell Lateral Gene Transfer Is Enriched in Cancer Samples». PLoS Comput Biol 9 (6): e1003107. doi:10.1371/journal.pcbi.1003107.
- ↑ Richardson, Aaron O.; Palmer, Jeffrey D. (enero de 2007). «Horizontal Gene Transfer in Plants» (PDF). Journal of Experimental Botany 58 (1): 1-9. PMID 17030541. doi:10.1093/jxb/erl148. Archivado desde el original el 27 de septiembre de 2007.
- ↑ a b Gogarten, Peter (2000). «Horizontal Gene Transfer: A New Paradigm for Biology». Esalen Center for Theory and Research Conference. Archivado desde el original el 21 de julio de 2012. Consultado el 18 de marzo de 2007.
- ↑ Kenneth Todar. «Bacterial Resistance to Antibiotics». The Microbial World: Lectures in Microbiology, Department of Bacteriology, University of Wisconsin-Madison. Archivado desde el original el 15 de enero de 2012. Consultado el 6 de enero de 2012.
- ↑ Stanley Maloy (15 de julio de 2002). «Horizontal Gene Transfer». San Diego State University. Consultado el 6 de enero de 2012.
- ↑ a b c d Stearns, S. C., & Hoekstra, R. F. (2005). Evolution: An introduction (2nd ed.). Oxford, NY: Oxford Univ. Press. pp. 38-40.
- ↑ R. Bock and V. Knoop (eds.), Genomics of Chloroplasts and Mitochondria, Advances in Photosynthesis and Respiration 35, pp. 223–235 doi 10.1007/978-94-007-2920-9_10, Springer Science+Business Media B.V. 2012
- ↑ Maxmen, A. (2010). «Virus-like particles speed bacterial evolution». Nature. doi:10.1038/news.2010.507.
- ↑ La Scola B, Desnues C, Pagnier I, Robert C, Barrassi L, Fournous G, Merchat M, Suzan-Monti M, Forterre P, Koonin E, Raoult D (septiembre de 2008). «The virophage as a unique parasite of the giant mimivirus». Nature 455 (7209): 100-4. Bibcode:2008Natur.455..100L. PMID 18690211. doi:10.1038/nature07218.
- ↑ Pearson H (agosto de 2008). «'Virophage' suggests viruses are alive». Nature 454 (7205): 677. Bibcode:2008Natur.454..677P. PMID 18685665. doi:10.1038/454677a.
- ↑ a b Bejarano E.R.; Khashoggi A.M.; Witty M.; Lichtenstein C.P. (1994). «Discovery of ancient recombination between geminiviral DNA and the nuclear genome of Nicotiana sp». Proceedings of the National Academy of Sciences 93: 759-764.
- ↑ Barlow M (2009). «What antimicrobial resistance has taught us about horizontal gene transfer». Methods in Molecular Biology (Clifton, N.J.). Methods in Molecular Biology 532: 397-411. ISBN 978-1-60327-852-2. PMID 19271198. doi:10.1007/978-1-60327-853-9_23.
- ↑ Hawkey PM, Jones AM (septiembre de 2009). «The changing epidemiology of resistance». Journal of Antimicrobial Chemotherapy 64 (Suppl 1): i3-10. PMID 19675017. doi:10.1093/jac/dkp256.
- ↑ Francino, MP (editor) (2012). Horizontal Gene Transfer in Microorganisms. Caister Academic Press. ISBN 978-1-908230-10-2.
- ↑ Strauch, Eckhard; Lurz, Rudi; Beutin, Lothar; Characterization (diciembre de 2001). «Shigella sonnei». Infection and Immunity 69 (12): 7588-7595. doi:10.1128/IAI.69.12.7588-7595.2001.
- ↑ Oliveira, PH; Touchon, M; Rocha, EPC (2014). «The interplay of restriction-modification systems with mobile genetic elements and their prokaryotic hosts». Nucleic Acids Res 42 (16): 10618-10631. doi:10.1093/nar/gku734.
- ↑ «Ulrich Melcher (2001) "Molecular genetics: Horizontal gene transfer," Oklahoma State University (Stillwater, Oklahoma USA)». Archivado desde el original el 4 de marzo de 2016. Consultado el 20 de agosto de 2015.
- ↑ Canchaya, C., Fournous, G., Chibani-Chennoufi, S., Dillmann, M. L., Brüssow, H. (Agosto de 2003). «Phage as agents of lateral gene transfer». Curr. Opin. Microbiol. 6 (4): 417-24. PMID 12941415. doi:10.1016/S1369-5274(03)00086-9.
- ↑ Blanchard JL, Lynch M (julio de 2000). «Organellar genes: why do they end up in the nucleus?». Trends Genet. 16 (7): 315-20. PMID 10858662. doi:10.1016/S0168-9525(00)02053-9. Discusses theories on how mitochondria and chloroplast genes are transferred into the nucleus, and also what steps a gene needs to go through in order to complete this process.
- ↑ Hall C, Brachat S, Dietrich FS (junio de 2005). «Contribution of Horizontal Gene Transfer to the Evolution of Saccharomyces cerevisiae». Eukaryotic Cell 4 (6): 1102-15. PMC 1151995. PMID 15947202. doi:10.1128/EC.4.6.1102-1115.2005.
- ↑ Kondo N, Nikoh N, Ijichi N, Shimada M, Fukatsu T (octubre de 2002). «Genome fragment of Wolbachia endosymbiont transferred to X chromosome of host insect». Proc. Natl. Acad. Sci. U.S.A. 99 (22): 14280-5. Bibcode:2002PNAS...9914280K. PMC 137875. PMID 12386340. doi:10.1073/pnas.222228199.
- ↑ Dunning Hotopp JC; Clark ME; Oliveira DC et al. (septiembre de 2007). «Widespread lateral gene transfer from intracellular bacteria to multicellular eukaryotes». Science 317 (5845): 1753-6. Bibcode:2007Sci...317.1753H. PMID 17761848. doi:10.1126/science.1142490.
- ↑ Davis CC, Wurdack KJ (30 de julio de 2004). «Host-to-parasite gene transfer in flowering plants: phylogenetic evidence from Malpighiales». Science 305 (5684): 676-8. Bibcode:2004Sci...305..676D. PMID 15256617. doi:10.1126/science.1100671.
- ↑ Daniel L Nickrent; Albert Blarer; Yin-Long Qiu; Romina Vidal-Russell; Frank E Anderson (2004). «Phylogenetic inference in Rafflesiales: the influence of rate heterogeneity and horizontal gene transfer». BMC Evolutionary Biology 4 (1): 40. PMC 528834. PMID 15496229. doi:10.1186/1471-2148-4-40.
- ↑ Magdalena Woloszynska; Tomasz Bocer; Pawel Mackiewicz; Hanna Janska (noviembre de 2004). «A fragment of chloroplast DNA was transferred horizontally, probably from non-eudicots, to mitochondrial genome of Phaseolus». Plant Molecular Biology 56 (5): 811-20. PMID 15803417. doi:10.1007/s11103-004-5183-y.
- ↑ Yoshida, Satoko; Maruyama, Shinichiro; Nozaki, Hisayoshi; Shirasu, Ken (28 de mayo de 2010). «Horizontal gene transfer by the parasitic plant Striga hermonthica». Science 328 (5982): 1128. Bibcode:2010Sci...328.1128Y. PMID 20508124. doi:10.1126/science.1187145.
- ↑ Nancy A. Moran; Tyler Jarvik (2010). «Lateral Transfer of Genes from Fungi Underlies Carotenoid Production in Aphids». Science 328 (5978): 624-627. Bibcode:2010Sci...328..624M. PMID 20431015. doi:10.1126/science.1187113.
- ↑ Fukatsu T (abril de 2010). «Evolution. A fungal past to insect color». Science 328 (5978): 574-5. Bibcode:2010Sci...328..574F. PMID 20431000. doi:10.1126/science.1190417.
- ↑ Bar D (16 de febrero de 2011). «Evidence of Massive Horizontal Gene Transfer Between Humans and Plasmodium vivax». Nature Precedings. doi:10.1038/npre.2011.5690.1.
- ↑ Redrejo-Rodríguez, M, Muñoz-Espín, D, Holguera, I, Mencía, M, Salas, M, (2012). «Functional eukaryotic nuclear localization signals are widespread in terminal proteins of bacteriophages». Proc. Natl. Acad. Sci. U.S.A. 109 (45): 18482-7. Bibcode:2012PNAS..10918482R. PMID 23091024. doi:10.1073/pnas.1216635109.
- ↑ Lee Phillips, Melissa (2012). «Bacterial gene helps coffee beetle get its fix». Nature. doi:10.1038/nature.2012.10116.
- ↑
- ↑ Carl Zimmer (17 de abril de 2014). «Plants That Practice Genetic Engineering». New York Times.
- ↑ «Human beings’ ancestors have routinely stolen genes from other species». The Economist. 14 de marzo de 2015. Consultado el 17 de marzo de 2015.
- ↑ Salzberg, S.L. (2001). «Microbial Genes in the Human Genome: Lateral Transfer or Gene Loss?». Science 292: 1903-6. PMID 11358996. doi:10.1126/science.1061036.
- ↑ Traci Watson (15 de noviembre de 2012). «Bdelloids Surviving on Borrowed DNA». Science/AAAS News.
- ↑ Koutsovoulos, Georgios; Kumar, Sujai; Laetsch, Dominik R.; Stevens, Lewis; Daub, Jennifer; Conlon, Claire; Maroon, Habib; Thomas, Fran; Aboobaker, Aziz A.; Blaxter, Mark (2016). «No evidence for extensive horizontal gene transfer in the genome of the tardigradeHypsibius dujardini». Proceedings of the National Academy of Sciences 113: 201600338. ISSN 0027-8424. doi:10.1073/pnas.1600338113.
- ↑ Crisp A, Boschetti C, Perry M, Tunnacliffe A, Micklem G (2015). «Expression of multiple horizontally acquired genes is a hallmark of both vertebrate and invertebrate genomes». Genome Biol. 16: 50. PMC 4358723. PMID 25785303. doi:10.1186/s13059-015-0607-3.
- ↑ a b c d e Schaack, Sarah, Gilbert Clement, and Feschotte Cedric. "Promiscuous DNA: Horizontal Transfer of Transposable Elements and Why It Matters for Eukaryotic Evolution." Trends in Ecology and Evolution 25.9 (2010): 537-46. Web of Science. Web. 4 Mar. 2016.
- ↑ Dupeyron, Mathilde et al. "Horizontal Transfer of Transposons between and within Crustaceans and Insects." Mobile DNA 5 (2014): 4. PMC. Web. 4 Mar. 2016.
- ↑ a b c El Baidouri Moaine (2014). «Widespread and Frequent Horizontal Transfers of Transposable Elements in Plants». Genome Research 24 (5): 831-838. doi:10.1101/gr.164400.113.
- ↑ a b Ivancevic A. M.; Walsh A. M.; Kortschak R. D.; Adelson D. L. (2013). «Jumping the fine LINE between species: Horizontal transfer of transposable elements in animals catalyses genome evolution». BioEssays 35: 1071-1082. doi:10.1002/bies.201300072.
- ↑ Wallau, Gabriel Luz, Mauro Freitas Ortiz, and Elgion Lucio Silva Loreto. "Horizontal Transposon Transfer in Eukarya: Detection, Bias, and Perspectives." Genome Biology and Evolution 4.8 (2012): 801–811. PMC. Web. 4 Mar. 2016.
- ↑ Plasterk RH (1996). «The Tc1/mariner transposon family». Curr. Top. Microbiol. Immunol. 204: 125-43. PMID 8556864. doi:10.1007/978-3-642-79795-8_6.
- ↑ Izsvak Z.; Ivics Z.; Plasterk R.H. (2000). «Sleeping Beauty, a wide host-range transposon vector for genetic transformation in vertebrates». J. Mol. Biol. 302 (1): 93-102. PMID 10964563. doi:10.1006/jmbi.2000.4047.
- ↑ Kurtti TJ, Mattila JT, Herron MJ, etal (octubre de 2008). «Transgene expression and silencing in a tick cell line: A model system for functional tick genomics». Insect Biochem. Mol. Biol. 38 (10): 963-8. PMC 2581827. PMID 18722527. doi:10.1016/j.ibmb.2008.07.008.
- ↑ Graham Lawton Why Darwin was wrong about the tree of life New Scientist Magazine issue 2692 21 January 2009 Accessed February 2009
- ↑ «Genomic analysis of Hyphomonas neptunium contradicts 16S rRNA gene-based phylogenetic analysis: implications for the taxonomy of the orders ‘Rhodobacterales’ and Caulobacteral...». Archivado desde el original el 16 de abril de 2013. Consultado el 14 de julio de 2016.
- ↑ Zhaxybayeva, O.; Gogarten, J. (2004). «Cladogenesis, coalescence and the evolution of the three domains of life». Trends in Genetics 20 (4): 182-187. PMID 15041172. doi:10.1016/j.tig.2004.02.004.
- ↑ D.A. Bryant; N.-U. Frigaard (noviembre de 2006). «Prokaryotic photosynthesis and phototrophy illuminated». Trends Microbiol. 14 (11): 488-96. PMID 16997562. doi:10.1016/j.tim.2006.09.001.
- ↑ Avrain L, Vernozy-Rozand C, Kempf I (2004). «Evidence for natural horizontal transfer of tetO gene between Campylobacter jejuni strains in chickens». J. Appl. Microbiol. 97 (1): 134-40. PMID 15186450. doi:10.1111/j.1365-2672.2004.02306.x.
- ↑ Darkened Forests, Ferns Stole Gene From an Unlikely Source — and Then From Each Other by Jennifer Frazer (6 de mayo de 2014). Scientific Am.
- ↑ [https://web.archive.org/web/20160307202422/http://blogs.scientificamerican.com/artful-amoeba/2014/05/06/in-darkened-forests-ferns-stole-gene-from-an-unlikely-source-and-then-each-other/In Archivado el 7 de marzo de 2016 en Wayback Machine. Darkened Forests, Ferns Stole Gene From an Unlikely Source — and Then From Each Other.
- ↑ Li, FW; Rothfels, CJ; Melkonian, M; Villarreal, JC; Stevenson, DW; Graham, SW; Wong, GK; Mathews, S et al. (2015). «The origin and evolution of phototropins.». Frontiers in Plant Science 6: 637. PMC 4532919. PMID 26322073. doi:10.3389/fpls.2015.00637.
Lecturas adicionales[editar]
- Citizendium:Horizontal gene transfer
- Citizendium:Horizontal gene transfer in prokaryotes
- Citizendium:Horizontal gene transfer in plants
- Citizendium:Horizontal gene transfer (History)
- Gyles, C; Boerlin, P (Mar 2014). «Horizontally transferred genetic elements and their role in pathogenesis of bacterial disease». Vet Pathol 51 (2): 328-40. PMID 24318976. doi:10.1177/0300985813511131.
- – Papers by Dr Michael Syvanen on Horizontal Gene Transfer
- Salzberg SL, White O, Peterson J, Eisen JA (junio de 2001). «Microbial genes in the human genome: lateral transfer or gene loss?». Science 292 (5523): 1903-6. Bibcode:2001Sci...292.1903S. PMID 11358996. doi:10.1126/science.1061036. Archivado desde el original el 1 de septiembre de 2006. Consultado el 14 de julio de 2016. «About 40 genes were found to be exclusively shared by humans and bacteria and are candidate examples of horizontal transfer from bacteria to vertebrates. Gene loss combined with sample size effects and evolutionary rate variation provide an alternative, more biologically plausible explanation».
- Qi, Z; Cui, Y; Fang, W; Ling, L; Chen, R (enero de 2004). «Autosomal similarity revealed by eukaryotic genomic comparison.». Journal of biological physics 30 (4): 305-12. PMID 23345874. doi:10.1007/s10867-004-0996-0.
- Woese CR (junio de 2002). «On the evolution of cells». Proc. Natl. Acad. Sci. U.S.A. 99 (13): 8742-7. Bibcode:2002PNAS...99.8742W. PMC 124369. PMID 12077305. doi:10.1073/pnas.132266999. This article seeks to shift the emphasis in early phylogenic adaptation from vertical to horizontal gene transfer. He uses the term "Darwinian Threshold" for the time of major transition of evolutionary mechanisms from mostly horizontal to mostly vertical transfer, and the "origin of speciation".
- Snel B, Bork P, Huynen MA (enero de 1999). «Genome phylogeny based on gene content». Nat. Genet. 21 (1): 108-10. PMID 9916801. doi:10.1038/5052. This article proposes using the presence or absence of a set of genes to infer phylogenies, in order to avoid confounding factors such as horizontal gene transfer.
- Webfocus in Nature with free review articles [1]
- Patil PB, Sonti RV (octubre de 2004). «Variation suggestive of horizontal gene transfer at a lipopolysaccharide (lps) biosynthetic locus in Xanthomonas oryzae pv. oryzae, the bacterial leaf blight pathogen of rice». BMC Microbiol. 4 (1): 40. PMC 524487. PMID 15473911. doi:10.1186/1471-2180-4-40.
- Jin G, Nakhleh L, Snir S, Tuller T (noviembre de 2006). «Maximum likelihood of phylogenetic networks». Bioinformatics 22 (21): 2604-11. PMID 16928736. doi:10.1093/bioinformatics/btl452. for a technique to decrease the impact of HGT events on maximum likelihood cladistical analyses.
- Horizontal Gene Transfer – A New Paradigm for Biology Archivado el 21 de julio de 2012 en Wayback Machine.
- Report on horizontal gene transfer by Mae-Wan Ho, March 22, 1999
- Recent Evidence Confirms Risks of Horizontal Gene Transfer
- Horizontal Gene Transfer at sciences.sdsu.edu
- Jain R, Rivera MC, Lake JA (marzo de 1999). «Horizontal gene transfer among genomes: The complexity hypothesis». Proc. Natl. Acad. Sci. U.S.A. 96 (7): 3801-6. Bibcode:1999PNAS...96.3801J. PMC 22375. PMID 10097118. doi:10.1073/pnas.96.7.3801.
- PDF article on Horizontal Gene Transfer
- The New Yorker, July 12, 1999, pp. 44–61 "Smallpox knows how to make a mouse protein. How did smallpox learn that? 'The poxviruses are promiscuous at capturing genes from their hosts,' Esposito said. 'It tells you that smallpox was once inside a mouse or some other small rodent.'"
- Szpirer C, Top E, Couturier M, Mergeay M (1 de diciembre de 1999). «Retrotransfer or gene capture: a feature of conjugative plasmids, with ecological and evolutionary significance». Microbiology (Reading, Engl.) 145 (Pt 12): 3321-9. PMID 10627031. doi:10.1099/00221287-145-12-3321.
- GMO Safety: Results of research into horizontal gene transfer Can transgenes from genetically modified plants be absorbed by micro-organisms and spread in this way?
- Whitaker JW, McConkey GA, Westhead DR (2009). «The transferome of metabolic genes explored: analysis of the horizontal transfer of enzyme encoding genes in unicellular eukaryotes». Genome Biol. 10 (4): R36. PMC 2688927. PMID 19368726. doi:10.1186/gb-2009-10-4-r36. Archivado desde el original el 17 de junio de 2013. Consultado el 14 de julio de 2016.
