Mutación por desplazamiento del marco de lectura

Una mutación por desplazamiento, desfase, corrimiento o cambio del marco de lectura (también conocida como error de marco o cambio de marco) es un tipo de mutación causado por la inserción o deleción de un número de nucleótidos que no es múltiplo de tres en una secuencia de ADN.
Debido a la naturaleza ternaria del código genético comprendido como una sucesión de codones; la inserción o deleción de un número de nucleótidos no divisible por tres, puede cambiar el marco de lectura del gen, provocando una traducción completamente diferente a la original. Cuanto antes aparezca la inserción o deleción en el gen, mayor es la alteración que sufre la proteína.[1]
Una mutación de marco de lectura no es lo mismo que un polimorfismo de nucleótido simple, en el cual se produce el reemplazo de un único nucleótido, en lugar de ser perdido o ganado. Una mutación de desplazamiento de marco de lectura puede, por lo general, conducir a que la lectura de los codones en la secuencia posterior a la mutación codifique para aminoácidos diferentes. El desplazamiento de marco también puede provocar la aparición o desaparición de un codón de terminación (UAA, UGA, o UAG) en una posición diferente de la secuencia. El polipéptido creado resulta entonces anormalmente corto o demasiado largo, y en la mayor parte de los casos pierde su funcionalidad.
Las mutaciones de marco de lectura aparecen en varias enfermedades genéticas tales como la enfermedad de Tay-Sachs y fibrosis quística; aumentan la susceptibilidad a ciertos tipos de cáncer y a algunos tipos de hipercolesterolemia familiar. En 1997,[2] se consiguió establecer la relación entre una mutación de marco de lectura y la resistencia a la infección por el virus VIH. Se ha propuesto a las mutaciones con cambio de marco de lectura como una posible fuente de diversidad biológica, como en el conocido caso de la aparición de la nylonasa; sin embargo, esta interpretación todavía está sujeta a controversias. Un estudio de Negoro et al (2006)[3] llegó a la conclusión de que esta mutación probablemente no fue un desplazamiento del marco de lectura, sino una sustitución de dos aminoácidos en la hendidura catalítica de una esterasa ancestral que permitió amplificar su actividad hidrolítica.
Antecedentes[editar]
La información contenida en el ADN determina la función de las proteínas en las células de todos los organismos. Los procesos de transcripción y traducción permiten que esta información sea transmitida para dirigir la fabricación de proteínas. Sin embargo, un error en la lectura de estos comunicados puede provocar una función proteica incorrecta y finalmente causar una enfermedad, aun cuando las células poseen una variedad de mecanismos correctores de errores.
Dogma central[editar]
En 1956 Francis Crick describió el flujo de información partiendo desde el ADN hasta llegar a un arreglo específico de aminoácidos para construir una proteína, calificándolo como el dogma central.[1] Para que las células funcionen adecuadamente, las proteínas deben ser producidas con exactitud, asegurando de esta forma sus actividades estructurales o catalíticas. Una proteína incorrectamente fabricada puede tener efectos nocivos en la viabilidad celular, y en la mayoría de los casos, provocar que los organismos superiores se enfermen a causa de una función celular anormal. Para asegurar que un determinado genoma sea capaz de pasar la información que contiene con éxito a las nuevas generaciones, existen mecanismos de detección y reparación de errores incorporados a la replicación del ADN.[1]
Transcripción y traducción[editar]
Luego de la replicación del ADN, la lectura de una sección determinada de la información genética se produce por medio del proceso de transcripción.[1] La secuencia de nucleótidos que contienen la información genética se encuentra ahora en un ácido nucleico de cadena simple que funciona como molde llamado ARN mensajero (ARNm). El ARNm se une a los ribosomas e interactúa con el ARN ribosomal (ARNr). Luego de esto la información contenida en los codones del ARNm es leída (decodificada) por medio de la complementariedad de anticodones del ARN de transferencia (ARNt). A medida que cada codón va siendo decodificado se va uniendo un aminoácido a la cadena en crecimiento hasta la aparición de un codón de terminación (UAG, UGA, UAA). En este punto se produce la liberación de la proteína recién sintetizada.[1] De cada 1000 aminoácidos incorporados a la proteína no más de uno resulta incorrecto. Esta fidelidad en el reconocimiento de los codones, manteniendo la importancia de un adecuado marco de lectura, se consigue por medio de un correcto apareamiento de bases en el sitio A del ribosoma, los procariotas además cuentan con el factor EF-Tu, una GTPasa, que añade estabilidad cinética y un mecanismo de corrección de errores de lectura al proceso.[1]
El desplazamiento del marco de lectura también puede ocurrir durante la traducción, produciendo diferentes proteínas por superposición de diferentes marcos de lectura abiertos, tal como ocurre con las proteínas gag-pol-env de los retrovirus. Esto resulta muy común en virus, y también ocurre en bacterias y levaduras (Farabaugh, 1996). La transcriptasa reversa, al contrario de lo que ocurre con la ARN polimerasa II, se cree que es una de las mayores causas de producción de mutaciones en el marco de lectura. En experimentos controlados, sólo del 3 al 13% de las mutaciones estudiadas se debieron a errores en la ARN polimerasa II. En procariontes, la inducción de mutaciones de marco de lectura se encuentra en el rango de 0,0001 al 0,00001.[4]
Existen varios procesos biológicos que ayudan a prevenir las mutaciones por desfase. Las mutaciones inversas, por ejemplo, ocurren cuando una secuencia mutada recobra su secuencia original (Wild type). Otra posibilidad de corrección de mutaciones es el uso de un mecanismo de mutación supresora. Este tipo de mutaciones revierte el efecto de la mutación original creando un segundo punto de mutación desplazando nuevamente el marco de lectura hasta permitir una correcta lectura de los codones. También se puede hacer uso de un ARN guía par insertar o retirar una uridina en el ARNm luego de la transcripción, esto permite corregir el marco de lectura.[5]
Importancia del codón o triplete[editar]
Un codón es un grupo de tres nucleótidos, o triplete, que codifica para un determinado aminoácido. El primer codón de una secuencia es el que establece el marco de lectura, allí donde un nuevo codón comienza. El esqueleto de una proteína, es decir su secuencia de aminoácidos; se define por una secuencia de codones contiguos.[6] Los codones son una pieza clave para que la información genética se traduzca en la síntesis de proteínas. El marco de lectura se establece cuando comienza la traducción del ARNm, y se mantiene mientras se va leyendo triplete a triplete. La lectura del código genético se encuentra sujeta a tres reglas que aseguran la correcta interpretación de los codones que aparecen en el ARNm:
- Los codones se leen en sentido 5' a 3'
- Los codones no se solapan entre sí, y no hay espacios vacíos entre uno y otro.
- El mensaje se traduce en un marco de lectura fijo.[1]
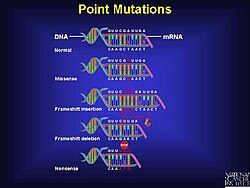
Mecanismo[editar]
Las mutaciones con desfase pueden darse aleatoriamente o pueden deberse a un estímulo externo. La detección de mutaciones de desplazamiento puede llevarse a cabo por varios métodos diferentes. Las mutaciones con desplazamiento son sólo un tipo de mutación que puede provocar la aparición de proteínas incorrectas o incompletas, pero comprenden un significativo porcentaje de los errores que aparecen en el ADN.
Genética o Ambiental[editar]
Este tipo de mutaciones ocurre a nivel de las bases nucleotídicas. El cómo y el porqué de estas mutaciones es objeto de continuo estudio. Por ejemplo se ha llevado a cabo un estudio ambiental, específicamente sobre la producción de mutaciones con desfase inducidas por radiación ultravioleta en una secuencia de aminoácidos al ser replicada con una ADN polimerasa de E. coli deficiente en su actividad 3′ → 5′ exonucleasa. Se cambió la secuencia normal
5′ GTC GTT TTA CAA 3′
por
GTC GTT T TTA CAA (MIDT)
y por
GTC GTT C TTA CAA (MIDC)
Con el objeto de estudiar los desplazamientos en el marco de lectura. Las enzimas de E. coli mutantes pol I variedades Kf y T7 produjeron reversiones causadas por la actividad exonucleasa e inducidas por UV con mayor frecuencia de lo que hicieron sus contrapartes con un dominio exonucleasa funcional. Los datos sugieren que la pérdida de la actividad de corrección de errores aumenta la frecuencia de mutaciones con desfase inducidas por la radiación ultravioleta.[7]
Detección[editar]
Fluorescencia[editar]
Se ha investigado en profundidad los efectos de la vecindad de bases y la estructura secundaria para detectar la frecuencia de las mutaciones con desfase haciendo uso de la fluorescencia. El ADN fluorescentemente marcado por medio de análogos de bases permite el estudio de cambios locales en la secuencia del ADN.[8]
Diferentes estudios sobre los efectos de la longitud de la cadena cebadora o primer, revelan una mezcla en equilibrio de cuatro conformaciones de hibridación cuando las bases de la cadena molde protruyen en forma de bucle, es decir, se observa una estructura flanqueada en ambos lados por ADN dúplex. En contraste, se observa una estructura de doble bucle con una conformación inusual de ADN desapilado aguas abajo, cuando las bases extrudidas se colocan en la unión cebador-molde, mostrando que los desajustes pueden ser modificados por la estructura secundaria del ADN vecino.[9]
Secuenciación[editar]
Dos métodos utilizados para detectar mutaciones en el marco de lectura son la secuenciación de Sanger y la pirosecuenciación, y aunque es probable que los datos generados por estos métodos no sean de la mayor calidad, aun así, han permitido identificar 1,96 millones de indeles que no se solapan con otras bases de datos. Cuando se observa una mutación con desplazamiento de marco, ésta se compara con la Base de Datos de Mutaciones del Genoma Humano (HGMD) para determinar si esta mutación tiene un efecto perjudicial. Esto se hace examinando cuatro características. En primer lugar, la relación entre el ADN afectado y el conservado; en segundo lugar localización de la mutación con respecto al transcrito; tercero la proporción de aminoácidos conservados y afectados y, finalmente; la distancia del indel hasta el final del exón.[10]
La Secuenciación Masiva en Paralelo es un método nuevo que puede ser utilizado para detectar mutaciones. Haciendo uso de este método, es posible secuenciar más de 17 gigabases a la vez, en oposición al limitado rango que tiene la secuenciación de Sanger que es sólo de aproximadamente 1 kilobase. Hay disponibles varias tecnologías para llevar a cabo esta determinación, y se ha estado investigando su aplicación clínica.[11] Al buscar diferentes tipos de carcinomas, los actuales métodos sólo permiten comprobar un gen a la vez. La Secuenciación Masiva en Paralelo puede ensayar simultáneamente una gran cantidad de mutaciones que se sabe provocan cáncer, en contraposición a los ensayos específicos.[12] Un experimento para determinar la exactitud de estos nuevos métodos de secuenciación fue ensayada sobre 21 genes y no produjo falsos positivos para las mutaciones con desplazamiento de marco de lectura.[13]
Diagnóstico[editar]
Una patente de Estados Unidos (N.º 5,958,684) emitida en 1999 para Leeuwen, detalla métodos y reactivos para el diagnóstico de enfermedades causadas o asociadas con un gen que posea una mutación que le cause un desplazamiento de marco de lectura. Entre los métodos se incluye el proporcionar una muestra de tejido o fluido y llevar a cabo un análisis génico para determinar la mutación con desplazamiento, o la proteína que este tipo de mutación produce. La secuencia de nucleótidos del gen sospechoso se obtiene de secuencias génicas ya publicadas o a partir de la clonación y secuenciación del gen sospechoso. Luego se predice la secuencia de aminoácidos a partir del gen.[14]
Frecuencia[editar]
A pesar de que las células poseen varios mecanismos para asegurar una correcta transferencia de la información genética, comenzando por la replicación del ADN, durante la traducción y gracias a las reglas que gobiernan el código genético; las mutaciones ocurren, y las mutaciones con desplazamiento del marco de lectura no son el único tipo. Existen además otros dos tipos de mutaciones puntuales, llamadas mutaciones missense y mutaciones nonsense.[1] Lo que distingue a las mutaciones con desfase es que una mutación con desplazamiento de marco cambia drásticamente el mensaje genético.[1] Las pequeñas inserciones y delecciones (de menos de 20 pares de bases) son responsables de más del 24% de las mutaciones responsables de enfermedades.[10]
Las mutaciones con desplazamiento del marco aparecen con mayor frecuencia en regiones de repetición del ADN. El motivo de esto es que las ADN polimerasas tienden a deslizarse sobre regiones de repetición, permitiendo de esta forma que las mutaciones ingresen en la secuencia.[15] Se pueden llevar a cabo diferentes experimentos para determinar la frecuencia con que aparecen las mutaciones con desplazamiento, ya sea añadiendo o removiendo un número predeterminado de nucleótidos. Se han llevado a cabo experimentos con adición de 4 pares de bases, llamados por lo tanto experimentos +4; y un equipo de la Universidad de Emory ha centrado su atención en las diferencias en las frecuencias que se producen por adición o por remoción de un par de bases. Este equipo demostró que no hay diferencias en las frecuencias de mutaciones observadas por adición o por remoción de un par de bases. Aunque, por supuesto, hay una gran diferencia en la proteína resultante.[15]
La enfermedad de Huntington es una de las enfermedades originadas por una reiteración de nueve codones, entre las que se incluyen la ataxia espinocerebral (ASC) tipos 1, 2, 6, 7 y 3; la atrofia muscular espinobulbar y la palidoluisianatrofia dentatorubal. Esta reiteración es consecuencia de una mutación que expande una región poliglutamina. Podría existir un vínculo entre las enfermedades causadas por las expansiones de regiones poliglutamina y polialanina. Se ha propuesto que un deslizamiento ribosomal durante la traducción de la proteína SCA3 podría ser el mecanismo que causa el desplazamiento del marco codificante de poliglutamina a polialanina. Una deleción de dos nucleótidos o una inserción de nucleótido simple dentro de la región poliglutamina (CAG)n del exón 1 del gen de huntington (gen htt) puede desplazar el marco en +1 (desfase +1) para crear una región (GCA)n, codificante de polialanina, e introducir un nuevo epítopo en el extremo terminal del exón 1 del gen htt (APAAAPAATRPGCG).[16]
Enfermedades relacionadas con mutaciones de desfase[editar]
Varias enfermedades tienen a una mutación con desfase al menos como una parte de su causa. Las mutaciones conocidas más prevalentes pueden, por lo tanto, ayudar en el diagnóstico de estas enfermedades. Actualmente también se han hecho intentos para utilizar a las mutaciones con desfase en forma beneficiosa, como un tratamiento para determinadas enfermedades.
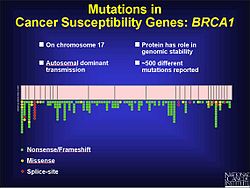

Tipos[editar]
Cáncer[editar]
Se sabe que las mutaciones con desfase son un factor en el cáncer colorrectal, como así también en otros tipos de cáncer asociados a inestabilidad del ADN microsatélite. Como se comentaba anteriormente, es más probable que las mutaciones con desfase ocurran en regiones de repetición de secuencia. Y cuando el mecanismo de corrección de errores del ADN no es capaz de reparar la inserción o deleción de bases, estas mutaciones tienen mayor probabilidad de resultar patogénicas. Esto en parte puede ser debido a que el tumor no recibe el mensaje de dejar de crecer. Experimentos realizados con levaduras y bacterias han ayudado a entender cuales son las características del ADN microsatélite que pueden contribuir a un mecanismo de reparación defectuoso. Entre estas características se incluyen la longitud del microsatélite, el enmascaramiento del material genético y cuan puras sean las repeticiones. Con base en estos experimentos se sabe que los microsatélites de mayor longitud poseen una tasa de mutaciones con desfase más alta. El ADN que flanquea la región también puede contribuir a las mutaciones con desplazamiento del marco de lectura.[17] En el cáncer de próstata una mutación con desfase puede modificar el marco abierto de lectura y evitar que ocurra la apoptosis. Aunque hay factores ambientales que contribuyen a la progresión del cáncer de próstata, también hay una componente genética. Durante un ensayo en el que se investigaron las regiones codificantes para encontrar mutaciones, se descubrieron 116 variantes genéticas, incluyendo 61 mutaciones con desfase.[18] Hay también más de 500 mutaciones en el cromosoma 17 que parecen jugar un papel en el desarrollo de cáncer de mama y ovario asociado al gen BRCA1, muchos de las cuales son mutaciones con desfase.[19]
Enfermedad de Crohn[editar]
La enfermedad de Crohn se encuentra asociada al gen NOD2. Una mutación con desplazamiento dentro de la región codificante del gen podría ser un factor en la aparición de esta enfermedad. La mutación es una inserción de una citosina en la posición 3020. Esto conduce a la aparición de un codón de terminación prematuro, acortando la proteína que debería ser traducida. Cuando la proteína es capaz de adquirir su conformación nativa, responde a los lipopolisacáridos bacterianos, mientras que la inserción 3020inscC evita que la proteína sea capaz de responder.[20]
Fibrosis quística[editar]
La fibrosis quística (FQ) es una mutación que ocurre por mutaciones en el gen CFTR, (gen regulador de la conductancia transmembrana de la fibrosis quística); un gen que codifica para un canal de cloro transmembrana. Hay más de 1500 mutaciones identificadas, pero no todas causan la enfermedad.[21] La mayor parte de los casos de fibrosis quística son el resultado de la mutación ∆F508, la cual elimina el aminoácido completo. Dos mutaciones de desfase son importantes desde el punto de vista diagnóstico para la fibrosis quística, la CF1213delT y la CF1154insTC. Ambas mutaciones por lo general ocurren en tándem con al menos otra mutación. Ambas conducen a una pequeña disminución en la función de los pulmones y ocurren en el 1% de los pacientes testeados. Todas estas mutaciones han sido identificadas por medio de la secuenciación de Sanger.[22]
HIV[editar]
El receptor CCR5 es uno de los cofactores de ingreso implicados en la infección por HIV, frecuentemente involucrado con las cepas no inductoras de sincitios. Se ha identificado una deleción de 32 pares de bases en el gen CCR5 que disminuye la probabilidad de desarrollo del HIV (CCR5 Δ32). Esta región sobre el marco abierto de lectura, contiene una mutación con desfase que provoca un codón de terminación temprano. Esto provoca in vitro la desaparición de la función del correceptor. Se considera que alelo CCR5-1 es el wild type, mientras que el CCR5-2 es el alelo mutante. Aquellos que presentan la mutación en heterocigosis son menos susceptibles al desarrollo del HIV. En un estudio, a pesar de una alta exposición al virus del HIV, no hubo ningún sujeto que presentara la mutación CCR5 que hubiera resultado positivo para HIV.[2]
Enfermedad de Tay-Sachs[editar]
La enfermedad de Tay-Sachs es una dolencia fatal que afecta al sistema nervioso central. Es una enfermedad que se descubre principalmente en infantes y niños pequeños. La progresión de la enfermedad comienza en el útero, pero los síntomas no aparecen hasta aproximadamente los seis meses de edad. No hay cura para esta enfermedad.[23] Se sabe que las mutaciones en el gen de la β-hexosaminidase A (gen Hex A) afectan la aparición de la enfermedad, con 78 mutaciones de diferente tipo descritas hasta el momento, de las cuales se sabe que 67 son causantes de la enfermedad. La mayor parte de las mutaciones observadas (65/78) son mutaciones de nucleótido simple o SNPs, 11 deleciones, 1 de gran tamaño y 10 pequeñas; y 2 inserciones. De las mutaciones observadas 8 son mutaciones con desfase, 6 deleciones y 2 inserciones. En el 80% de la población Ashkenazi afectada por esta enfermedad se observa una inserción de 4 pares de bases en el exón 11 del gen Hex A. Las mutaciones con desfase producen la aparición de un codón de terminación temprano, lo cual se sabe que es causal de aparición de enfermedad en infantes. La enfermedad de aparición tardía, aparentemente podría estar causada por 4 tipos diferentes de mutaciones, una de las cuales es una deleción de 3 pares de bases.[24]
Síndrome Smith-Magenis[editar]
El síndrome Smith-Magenis (SMS) es un síndrome complejo que involucra discapacidades intelectuales, disturbios del sueño, problemas de comportamiento, y una variedad de anormalidades craneofaciales, esqueléticas y viscerales.
La mayoría de los casos de SMS conocidos albergan una deleción común de aproximadamente 3,5 Mpb que involucra al gen inducido por ácido retinoico 1 (RAI1). Otros casos recientemente reportados ilustran la variabilidad en el fenotipo del SMS, casos que no se indicaban anteriormente para la mutación RAI1, incluyendo pérdida de la audición, ausencia de comportamientos autoabusivo, y retrasos globales moderados. La secuenciación del gen RAI1 reveló una mutación en una zona heptamérica poli C (CCCCCCC) en el exón 3 que resulta en una mutación con desfase del marco de lectura. De las siete mutaciones con desfase reportadas, cuatro casos (~57%) se produjeron en este tracto heptamérico poli C. Los resultados parecen indicar que este tracto poli C es un punto caliente para las inserciones y deleciones (indeles), y por lo tanto un objetivo principal a ser estudiado en pacientes de los que se sospecha una mutación en RAI1.[25]
Cardiomiopatía hipertrófica[editar]
La cardiomiopatía hipertrófica es la causa más común de muerte súbita en personas jóvenes, incluyendo a atletas entrenados, y es causada por mutaciones en los genes que codifican para las proteínas del sarcómero cardíaco. Una causa rara de cardiomiopatía hipertrófica son las mutaciones en el gen de la troponina C (TNNC1). Un estudio reciente indica que una mutación con desplazamiento del marco de lectura en el gen de la troponina C (c.363dupG o p.Gln122AlafsX30) fue la causa de cardiomiopatía hipertrófica y muerte súbita de un joven masculino de 19 años de edad.[26]
Curas[editar]
El hallar una cura para las enfermedades causadas por mutaciones con desfase es difícil. La investigación en esta área es evidente. Un ejemplo es la inmunodeficiencia primaria (IDP), una condición hereditaria que provoca un aumento a la susceptibilidad a las infecciones. Hay 120 genes y 150 mutaciones relacionadas con inmunodeficiencias primarias. De momento el tratamiento estándar es la terapia génica, pero es un tratamiento de alto riesgo que a menudo puede conducir a la aparición de otras enfermedades tales como la leucemia. Entre los procedimientos de terapia génica se incluyen la modificación de la proteína de fusión con dedos de cinc, cortando ambos extremos de la mutación, lo que causa su remoción de la secuencia. Otra posibilidad utilizada para tratar la distrofia muscular de Duchenne es la utilización de un salto de exones mediado por oligonucleótidos. Este proceso permite saltar el área mutada de forma que el resto de la secuencia permanece en marco y la función de la proteína no se ve alterada. Sin embargo esto no cura la enfermedad, tan sólo sirve para tratar los síntomas y sólo resulta útil su aplicación en proteínas estructurales o en otros genes repetitivos. Una tercera forma de reparación es el mosaicismo revirtiente, el cual ocurre naturalmente al crearse una mutación que revierta la mutación original, o una segunda mutación en un sitio diferente que corrija el marco de lectura. Esta reversión puede ocurrir por recombinación intragénico, conversión mitótica, un deslizamiento sobre el ADN en un segundo sitio, o por una reversión específica de sitio. Esta es una alternativa posible en varias enfermedades, tales como la inmunodeficiencia combinada severa ligada al X (IDCS), el síndrome de Wiskott-Aldrich, y el síndrome de Bloom. No hay medicinas, ni tratamientos farmacológicos que puedan brindar ayuda a los pacientes con IDP.[27]
Una patente europea del año 2003 a nombre de Bork (EP1369126A1) describe un método para la prevención de algunos tipos de cáncer y para el tratamiento curativo de cánceres y precánceres tales como los causados por la deficiencia en reparación de ADN, tumores esporádicos y tumores asociados a HNPCC. La idea descrita es el uso de inmunoterapia con mezclas de péptidos específicos derivados de mutaciones con desplazamiento del marco de lectura para inducir a los linfocitos T citotóxicos a que respondan específicamente contra las células tumorales.[28]
Véase también[editar]
- Mutación genética
- Transcripción (genética)
- Traducción (genética)
- codón
- proteína
- marco de lectura
- mutación puntual
- Enfermedad de Crohn
- Enfermedad de Tay-Sachs
Lecturas adicionales[editar]
- Farabaugh PJ (1996). «Programmed translational frameshifting». Annu. Rev. Genet. 30 (1): 507-28. PMID 8982463. doi:10.1146/annurev.genet.30.1.507.
- Lewis, Ricki (2005). Human Genetics: Concepts and Applications (6th edición). Boston, Mass: McGraw Hill. pp. 227-228. ISBN 0-07-111156-5.
- Nylonase Enzymes, Abril de 2004, consultado el 2 de junio de 2009.
Enlaces externos[editar]
- MeSH: Frameshift+Mutation (en inglés)
- NCBI dbSNP database — "a central repository for both single base nucleotide substitutions and short deletion and insertion polymorphisms"
- Wise2 - aligns a protein against a DNA sequence allowing frameshifts and introns
- FastY - compare a DNA sequence to a protein sequence database, allowing gaps and frameshifts
- Path - tool that compares two frameshift proteins (back-translation principle)
- HGMD - Human Genome Mutation Database
Referencias[editar]
- ↑ a b c d e f g h i Losick, Richard; Watson, James D.; Tania A. Baker; Bell, Stephen; Gann, Alexander; Levine, Michael W. (2008). Molecular biology of the gene. San Francisco: Pearson/Benjamin Cummings. ISBN 0-8053-9592-X.
- ↑ a b Zimmerman, PA; Alkhatib, G; Spalding, T; Kubofcik, J; Combadiere, C; Weissman, D; Cohen, O; Rubbert, A; Lam, G; Vaccarezza, M; Kennedy, PE; Kumaraswami, V; Giorgi, JV; Detels, R; Hunter, J; Chopek, M; Berger, EA; Fauci, AS; Nutman, TB; Murphy, PM (enero de 1997). «Inherited resistance to HIV-1 conferred by an inactivating mutation in CC chemokine receptor 5: studies in populations with contrasting clinical phenotypes, defined racial background, and quantified risk.». Molecular medicine (Cambridge, Mass.) 3 (1): 23-36. PMID 9132277.
|autor1=y|apellido=redundantes (ayuda); - ↑ http://www.jbc.org/content/280/47/39644.full.pdf+html
- ↑ Zhang, J (agosto de 2004). «Host RNA polymerase II makes minimal contributions to retroviral frame-shift mutations.». The Journal of general virology 85 (Pt 8): 2389-95. PMID 15269381. doi:10.1099/vir.0.80081-0.
- ↑ James D. Watson (et al.) (2007). Molecular biology of the gene (6th ed. edición). San Francisco, Calif.: Benjamin Cummings. ISBN 978-0-8053-9592-1.
- ↑ Cox, Michael; Nelson, David R.; Lehninger, Albert L (2008). Lehninger principles of biochemistry. San Francisco: W.H. Freeman. ISBN 0-7167-7108-X.
- ↑ Sagher, Daphna; Acharya, Sonia; Strauss, Bernard. «Production of UV-induced Frameshift Mutations in Vitro by DNA Polymerases Deficient in 3′ → 5′ Exonuclease Activity». Journal of Molecular Biology 240 (3): 226-242. doi:10.1006/jmbi.1994.1437.
|autor1=y|apellido=redundantes (ayuda) - ↑ Johnson, Neil P.; Walter A. Baase, Peter H. von Hippel, Low-energy circular dichroism of 2-aminopurine dinucleotide as a probe of local conformation of DNA and RNA, doi:10.1073/pnas.0400591101, «PNAS 2004 101:3426-3431; published online before print March 1, 2004» .
- ↑ Baase, Walter A.; Davis Jose , Benjamin C. Ponedel , Peter H. von Hippel , and Neil P. Johnson, «DNA models of trinucleotide frameshift deletions: the formation of loops and bulges at the primer–template junction», Nucleic Acids Research 37 (5): 1682-1689, doi:10.1093/nar/gkn1042, «Nucleic Acids Research Advance Access published on April 1, 2009» .
- ↑ a b Hu, J; Ng, PC (9 de febrero de 2012). «Predicting the effects of frameshifting indels.». Genome Biology 13 (2): R9. PMID 22322200. doi:10.1186/gb-2012-13-2-r9. Error en la cita: Etiqueta
<ref>no válida; el nombre «predicting frameshifts» está definido varias veces con contenidos diferentes - ↑ Tucker, Tracy; Marra, Marco; Friedman, Jan M. (2009). «Massively Parallel Sequencing: The Next Big Thing in Genetic Medicine». The American Journal of Human Genetics 85 (2): 142-154. ISSN 0002-9297. PMC 2725244. PMID 19679224. doi:10.1016/j.ajhg.2009.06.022.
- ↑ Walsh, T.; Casadei, S.; Lee, M. K.; Pennil, C. C.; Nord, A. S.; Thornton, A. M.; Roeb, W.; Agnew, K. J.; Stray, S. M.; Wickramanayake, A.; Norquist, B.; Pennington, K. P.; Garcia, R. L.; King, M.-C.; Swisher, E. M. (2011). «From the Cover: Mutations in 12 genes for inherited ovarian, fallopian tube, and peritoneal carcinoma identified by massively parallel sequencing». Proceedings of the National Academy of Sciences 108 (44): 18032-18037. ISSN 0027-8424. doi:10.1073/pnas.1115052108.
- ↑ Walsh, T.; Lee, M. K.; Casadei, S.; Thornton, A. M.; Stray, S. M.; Pennil, C.; Nord, A. S.; Mandell, J. B.; Swisher, E. M.; King, M.-C. (2010). «Detection of inherited mutations for breast and ovarian cancer using genomic capture and massively parallel sequencing». Proceedings of the National Academy of Sciences 107 (28): 12629-12633. ISSN 0027-8424. doi:10.1073/pnas.1007983107.
- ↑ US Patent 5,958,684 (September 28, 1999) "Diagnosis of Neurodegenerative Disease" by Leeuwen et al
- ↑ a b Harfe, BD; Jinks-Robertson, S (julio de 1999). «Removal of frameshift intermediates by mismatch repair proteins in Saccharomyces cerevisiae.». Molecular and Cellular Biology 19 (7): 4766-73. PMID 10373526. Consultado el 20 de marzo de 2013.
- ↑ Davies, J E; Rubinsztein, D C. «Polyalanine and polyserine frameshift products in Huntington's disease». Journal of Medical Genetics 43 (11): 893-896. doi:10.1136/jmg.2006.044222.
- ↑ Schmoldt, A; Haberland, G (1 de septiembre de 1975). «Digitoxin metabolism by rat liver microsomes.». Biochemical pharmacology 24 (17): 1639-41. doi:10.1093/hmg/ddq151.
|autor1=y|apellido=redundantes (ayuda); - ↑ Xu, XiaoLin; Liu, Feng; Wang, Yue; Shen, JianGuo; Jin, Jizhong; Wang, Zhong; Chen, Lin; Li, Jiadong et al.. «Identification of somatic mutations in human prostate cancer by RNA-Seq». Gene. doi:10.1016/j.gene.2013.01.046.
|autor1=y|apellido=redundantes (ayuda); - ↑ «Cancer Genomics». National Cancer Institute at the National Institute of Health. Archivado desde el original el 18 de marzo de 2013. Consultado el 24 de marzo de 2013.
- ↑ Ogura, Y; Inohara, N; Nicolae, DL; Chen, FF; Ramos, R; Britton, H; Moran, T; Karaliuskas, R; Duerr, RH; Achkar, JP; Brant, SR; Bayless, TM; Kirschner, BS; Hanauer, SB; Nuñez, G; Cho, JH (31 de mayo de 2001). «A frameshift mutation in NOD2 associated with susceptibility to Crohn's disease.». Nature 411 (6837): 603-6. PMID 11385577. doi:10.1038/35079114.
|autor1=y|apellido=redundantes (ayuda) - ↑ Farrell, Philip M.; White, Terry B.; Accurso, Frank J.; Castellani, Carlo; Cutting, Garry R.; Durie, Peter R.; LeGrys, Vicky A.; Massie, John; Parad, Richard B.; Rock, Michael J.; Campbell, Preston W. «Guidelines for Diagnosis of Cystic Fibrosis in Newborns through Older Adults: Cystic Fibrosis Foundation Consensus Report». The Journal of Pediatrics 153 (2): S4-S14. doi:10.1016/j.jpeds.2008.05.005.
|autor1=y|apellido=redundantes (ayuda); - ↑ Iannuzzi, MC; Collins, FS; Hon, CT; Hidaka, N; Strong, T; Becker, L; Drumm, ML; White, MB et al. (febrero de 1991). «Two frameshift mutations in the cystic fibrosis gene.». American Journal of Human Genetics 48 (2): 227-31. PMID 1990834. Consultado el 21 de marzo de 2013.
|autor1=y|apellido=redundantes (ayuda); - ↑ «Learning About Tay-Sachs Disease». National Human Genome Research Institute. Consultado el 24 de marzo de 2013.
- ↑ Myerowitz, R (1997). «Tay-Sachs disease-causing mutations and neutral polymorphisms in the Hex A gene.». Human Mutation 9 (3): 195-208. PMID 9090523. doi:10.1002/(SICI)1098-1004(1997)9:3<195::AID-HUMU1>3.0.CO;2-7.
- ↑ Truong, Hoa T; Blanchard, Christopher L.; Elsea, Sarah H. «Frameshift mutation hotspot identified in Smith-Magenis syndrome: case report and review of literature». BMC Medical Genetics 11 (1): 142. doi:10.1186/1471-2350-11-142.
|autor1=y|apellido=redundantes (ayuda) - ↑ Chung WK, Kitner C, Maron BJ (junio de 2011). «Novel frameshift mutation in Troponin C ( TNNC1) associated with hypertrophic cardiomyopathy and sudden death». Cardiol Young 21 (3): 345-8. PMID 21262074. doi:10.1017/S1047951110001927.
- ↑ Hu, Hailiang; Gatti, Richard A. «New approaches to treatment of primary immunodeficiencies: fixing mutations with chemicals». Current Opinion in Allergy and Clinical Immunology 8 (6): 540-546. doi:10.1097/ACI.0b013e328314b63b.
- ↑ European Patent [1] (December 10, 2003) "Use of coding microsatellite region frameshift mutation-derived peptides for treating cancer" by Bork et al
