Diferencia entre revisiones de «Nucleocytoviricota»
mSin resumen de edición |
|||
| Línea 32: | Línea 32: | ||
En [[2019]] se informó el descubrimiento de virus gigantes de un tamaño comparable al de bacterias comunes o inclusive más grandes que se han identificado en los [[quetognatos]]. Estos virus pueden medir alrededor de (3,1 a 3,9 mpb) de longitud. Se ha detectado la presencia de [[ribosoma]]s, una característica ausente en los demás virus y su morfología es similar a la que presentan las bacterias o los gusanos.<ref>[https://www.asbmb.org/asbmb-today/science/110119/giant-gene-thieves Giant gene thieves]</ref><ref>https://www.hilarispublisher.com/open-access/serendipitous-discovery-in-a-marine-invertebrate-phylum-chaetognatha-of-the-longest-giant-viruses-reported-to-date.pdf</ref><ref>{{cite journal | authors = Shinn GL, Bullard BL | title = Ultrastructure of Meelsvirus: A nuclear virus of arrow worms (phylum Chaetognatha) producing giant "tailed" virions | journal = PLOS ONE | volume = 13 | issue = 9 | pages = e0203282 | date = 19 de septiembre de 2018 | pmid = 30231047 | pmc = 6145532 | doi = 10.1371/journal.pone.0203282 | editor-first = Carmen | editor-last = San Martin | bibcode = 2018PLoSO..1303282S }}</ref> Las especies han sido nombradas como ''[[Megaklothovirus]]'' y ''[[Klothovirus]]'', sin embargo todavía falta más investigación sobre estos virus para conocer más detalles. |
En [[2019]] se informó el descubrimiento de virus gigantes de un tamaño comparable al de bacterias comunes o inclusive más grandes que se han identificado en los [[quetognatos]]. Estos virus pueden medir alrededor de (3,1 a 3,9 mpb) de longitud. Se ha detectado la presencia de [[ribosoma]]s, una característica ausente en los demás virus y su morfología es similar a la que presentan las bacterias o los gusanos.<ref>[https://www.asbmb.org/asbmb-today/science/110119/giant-gene-thieves Giant gene thieves]</ref><ref>https://www.hilarispublisher.com/open-access/serendipitous-discovery-in-a-marine-invertebrate-phylum-chaetognatha-of-the-longest-giant-viruses-reported-to-date.pdf</ref><ref>{{cite journal | authors = Shinn GL, Bullard BL | title = Ultrastructure of Meelsvirus: A nuclear virus of arrow worms (phylum Chaetognatha) producing giant "tailed" virions | journal = PLOS ONE | volume = 13 | issue = 9 | pages = e0203282 | date = 19 de septiembre de 2018 | pmid = 30231047 | pmc = 6145532 | doi = 10.1371/journal.pone.0203282 | editor-first = Carmen | editor-last = San Martin | bibcode = 2018PLoSO..1303282S }}</ref> Las especies han sido nombradas como ''[[Megaklothovirus]]'' y ''[[Klothovirus]]'', sin embargo todavía falta más investigación sobre estos virus para conocer más detalles. |
||
Según algunas teorías los [[virus gigantes]] pudieron haber originado el [[núcleo celular|núcleo]] de las [[célula eucariota|células eucariotas]] al haberse incorporado el virus dentro de la célula donde en lugar de replicarse y destruir la célula huésped, permanecería dentro de la célula originando posteriormente el núcleo y dando lugar a otras innovaciones genómicas. Esta teoría es conocida como la "[[eucariogénesis viral]]".<ref>{{cite journal |doi=10.1007/s002390010215 |year=2001 |author=Philip John Livingstone Bell|journal=Journal of Molecular Evolution |volume=53 |issue=3 |pages=251–256 |title=Viral eukaryogenesis: Was the ancestor of the nucleus a complex DNA virus? |pmid=11523012|bibcode=2001JMolE..53..251L }}</ref> |
|||
== Características y razones para crear esta agrupación == |
== Características y razones para crear esta agrupación == |
||
Revisión del 04:47 22 jun 2020
| Nucleocytoviricota | ||
|---|---|---|
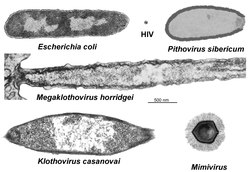 Comparación de dos virus gigantes descubiertos en 2019 con la bacteria Escherichia coli, el Pithovirus, el Mimivirus y el Retrovirus. | ||
| Taxonomía | ||
| Filo: | Nucleocytoviricota | |
| Clasificación de Baltimore | ||
| Grupo: | I (Virus ADN bicatenario) | |
| Clasificación | ||
| ||
Los virus nucleocitoplasmáticos de ADN de gran tamaño (acortado como girus, de virus gigantes), VNCAGT o NCLDV por sus siglas en inglés, (filo Nucleocytoviricota) es un filo al que pertenece actualmente 7 familias de virus ADN. Estos virus son tan grandes como las más pequeñas bacterias (o incluso mayores), tanto en la longitud de su ADN (que va desde 300 Kb a 2,5 Mb) como en diámetro (de 200 nm a 1000 nm).
A modo de comparación, la bacteria de vida libre más pequeña, Mycoplasma genitalium, presenta 450 nm de diámetro y codifica únicamente 482 proteínas. En cambio el Mimivirus codifica 979 proteínas. Otros ejemplo de virus gigante es el Megavirus chilensis, que posee un genoma de alrededor de 1,26 Mb y un diámetro de alrededor de 700 nm. Los más grandes hallados hasta ahora son el Pandoravirus dulcis (con 1,9 mpb), encontrado en un lago en Australia y el Pandoravirus salinus (con 2,5 mpb), que llegan a medir aproximadamente 1 μm de diámetro.[2]
En 2019 se informó el descubrimiento de virus gigantes de un tamaño comparable al de bacterias comunes o inclusive más grandes que se han identificado en los quetognatos. Estos virus pueden medir alrededor de (3,1 a 3,9 mpb) de longitud. Se ha detectado la presencia de ribosomas, una característica ausente en los demás virus y su morfología es similar a la que presentan las bacterias o los gusanos.[3][4][5] Las especies han sido nombradas como Megaklothovirus y Klothovirus, sin embargo todavía falta más investigación sobre estos virus para conocer más detalles.
Según algunas teorías los virus gigantes pudieron haber originado el núcleo de las células eucariotas al haberse incorporado el virus dentro de la célula donde en lugar de replicarse y destruir la célula huésped, permanecería dentro de la célula originando posteriormente el núcleo y dando lugar a otras innovaciones genómicas. Esta teoría es conocida como la "eucariogénesis viral".[6]
Características y razones para crear esta agrupación
Todas estas familias tienen en común el tamaño de su ADN, el poseer genes poco habituales en el resto de virus y la estructura del virión y del ADN, todos bicatenarios. La mayoría de los genes no poseen homólogos en otros tipos celulares.
Una característica que los distingue radicalmente del resto de virus es su abertura en estrella, visible después de liberar su carga genética en el huésped. La salida del genoma del virus se realiza por esta hendidura, mientras que la entrada del ADN, en el ensamblaje intracelular del virus, se realiza exclusivamente por el lado opuesto. Son los únicos virus que poseen un lado de salida y un lado de entrada del ADN determinados.[7]
Se conocen 47 genes del core de los virus gigantes. Se incluyen los 4 genes clave involucrados en la replicación y reparación del ADN: ADN polimerasa B, topoisomerasa II A, Flap endonucleasa y el factor antígeno nuclear de proliferación celular. Otros genes codifican para la ARN polimerasa dependiente de ADN II y el factor de transcripción II B.
Igualmente se ha descubierto que además muchos de ellos son la diana de diferentes tipos de virófagos (virus que parasitan otros virus).
Origen
Su origen, como el de los virus en general, es muy incierto. Es probable que los virus gigantes o girus evolucionaran antes de la separación de eucariotas en grupos extintos. El genoma ancestral era complejo, con al menos 41 genes, incluyendo: (1) la maquinaria de replicación, (2) hasta 4 subunidades de ARN polimerasa, (3) al menos tres factores de transcripción, (4) enzimas de poliadenilación y colocación de la caperuza, (5) el sistema de empaquetamiento de ADN y (6) componentes estructurales de la cápsida icosaédrica y la envoltura vírica.
Entre las diferentes hipótesis sobre las similitudes de las distintas familias de los NCLDV se pueden deber a la recolección de genes de sus hospedadores durante la replicación vírica en el interior celular; o también pueden deberse a que comparten un ancestro común.[8] Este ancestro puede colocarse evolutivamente entre las Archaea y los Eukaryota o sincrónicamente con LUCA (el primer ancestro común de los tres dominios del árbol filogenético de la vida). De esta forma habría que incrementar en uno los dominios del árbol de la vida. Otras hipótesis apuntan a que los virus fueron las primeras formas de vida y los tres dominios (Bacteria, Archaea y Eukarya) surgieron de la evolución de tres de ellos. Hay mucha confusión, entre otras cosas, debido a que los NCLDV poseen homologías génicas con estos tres dominios además de ciertos genes que se pueden considerar propiamente víricos. Todo esto con la transferencia horizontal de genes hace imposible, por ahora, una clasificación fiable de los virus gigantes. Ni siquiera se puede establecer una idea sobre la base de la que incluir los virus gigantes en el árbol de la vida, si es que merecen tal rango.
Relación filogenética con los tres dominios
A pesar de ser virus gigantes, su genoma tiene los componentes necesarios para incluirse en un análisis filogenético. Se ha detectado que estos virus también poseen en sus genomas elementos genéticos móviles como transposones únicos llamados transpoviriones y plásmidos lineales,[9] estos elementos se consideraban propios de los seres vivos. Presencia de ARN polimerasa, pero se clasifican como Virus ADN bicatenario porque usan este método para su replicación. Además de genes relacionados con el funcionamiento del metabolismo lo que respalda que tuvieron ancestros capaces de metabolizarse y reproducirse independientemente. En los recientes virus gigantes de quetognatos se ha reportado la presencia de ribosomas.[10] Se ha propuesto dos filogenias para los NCLDV.
Basada en la secuencia de aminoácidos y genes, esta filogenia propone que basándose en estas secuencias los NCLDV serían el primer dominio divirgente descendiente de LUCA y posiblemente monofilético siendo las relaciones filogenéticas entre subgrupos las siguientes.[11] Las familias marcadas con * no han sido reconocidas por el ICTV:
| Biota |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Otros estudios de los proteomas y los pliegues de las proteínas virales han encontrado a los virus gigantes y los restantes virus como un grupo complejamente parafilético. El análisis ha sugerido que los más probables es que los virus se hallan originado en protobiontes que posteriormente se separaron para dar origen a los procariotas[12] o también es probable que se hayan originado varias veces en el mundo de ARN. La familia Mimiviridae constituyó el grupo hermano de los organismos celulares, mientras que las demás familias de virus gigantes se ramificaban tempranamente en manera parafilética antes de los mimiviridos y los organismos celulares.[13] Lo mismo sucedía con las demás familias de virus ADN y ARN que se bifurcaban en manera parafilética y mezclada antes de los virus gigantes y los organismos celulares.[14][15][16] Puede verse el artículo Biota (taxonomía) para el cladograma verdadero:
| Biota |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Véase también
Referencias
- ↑ Yoshikawa, G., Blanc-Mathieu, R., Song, C., Kayama, Y., Mochizuki, T., Murata, K., ... & Takemura, M. (2019). Medusavirus, a novel large DNA virus discovered from hot spring water. Journal of virology, JVI-02130
- ↑ N. Philippe; M. Legendre; G. Doutre; Y. Couté; O. Poirot; M. Lescot; D. Arslan; V. Seltzer; L. Bertaux; C. Bruley; J. Garin; J.-M. Claverie; C. Abergel N. Pandoraviruses: Amoeba Viruses with Genomes up to 2.5 Mb Reaching That of Parasitic Eukaryotes. Science (2013). DOI: 10.1126/science.1239181.
- ↑ Giant gene thieves
- ↑ https://www.hilarispublisher.com/open-access/serendipitous-discovery-in-a-marine-invertebrate-phylum-chaetognatha-of-the-longest-giant-viruses-reported-to-date.pdf
- ↑ Shinn GL, Bullard BL (19 de septiembre de 2018). «Ultrastructure of Meelsvirus: A nuclear virus of arrow worms (phylum Chaetognatha) producing giant "tailed" virions». En San Martin, Carmen, ed. PLOS ONE 13 (9): e0203282. Bibcode:2018PLoSO..1303282S. PMC 6145532. PMID 30231047. doi:10.1371/journal.pone.0203282.
- ↑ Philip John Livingstone Bell (2001). «Viral eukaryogenesis: Was the ancestor of the nucleus a complex DNA virus?». Journal of Molecular Evolution 53 (3): 251-256. Bibcode:2001JMolE..53..251L. PMID 11523012. doi:10.1007/s002390010215.
- ↑ Virus gigantes L. van Etten, James (2012). Investigación y Ciencia, ed. Virus gigantes.
- ↑ Lakshminarayan M. Iyer, L. Aravind, and Eugene V. Koonin. "Common Origin of Four Diverse Families of Large Eukaryotic DNA Viruses." Journal of Virology, December 2001, p. 11720-11734, Vol. 75, No. 23. Link.
- ↑ Eugene Koonin, Valerian V Doljja (2014). Virus World as an Evolutionary Network of Viruses and Capsidless Selfish Elements. Microbiology and Molecular Biology Reviews.
- ↑ Error en la cita: Etiqueta
<ref>no válida; no se ha definido el contenido de las referencias llamadasbmc1 - ↑ Philippe Colson, Anthony Levasseur, Bernard La Scola, Vikas Sharma, Arshan Nasir, Pierre Pontarotti et Didier Raoult (2018). Ancestrality and Mosaicism of Giant Viruses Supporting the Definition of the Fourth TRUC of Microbes. Frontiers. https://doi.org/10.3389/fmicb.2018.02668
- ↑ Viral evolution: Primordial cellular origins and late adaptation to parasitism. NCBI.
- ↑ Rijja Hussain Bokhari, Nooreen Amirjan, Hyeonsoo Jeong, Kyung Mo Kim, Gustavo Caetano-Anollés, Arshan Nasir (2020). Bacterial Origin and Reductive Evolution of the CPR Group. Oxford Academic. https://doi.org/10.1093/gbe/evaa024
- ↑ The distribution and impact of viral lineages in domains of life. Frontiers.
- ↑ Do Viruses Exchange Genes across Superkingdoms of Life?. Frontiers.
- ↑ Arshan, Nasir; Caetano-Anollés, Gustavo (25 de septiembre de 2015). «A phylogenomic data-driven exploration of viral origins and evolution». Science Advances 1 (8): e1500527. Bibcode:2015SciA....1E0527N. PMC 4643759. PMID 26601271. doi:10.1126/sciadv.1500527.
