Selección de variable
En aprendizaje de máquina y estadística, selección de característica, también conocida como selección de variable, selección de atributo o selección de subconjunto variable, es el proceso de seleccionar un subconjunto de características pertinentes (variables, predictores) para su uso en construcción de modelos. Las técnicas de selección de la característica son utilizadas por cuatro razones:
- Simplificación de modelos con el fin de hacerlas más sencillas de interpretar para los usuarios/investigadores,[1]
- Tiempo de entrenamiento más corto,
- Para evitar la maldición de la dimensión (curse of dimensionality),
- Generalización realzada por reducir overfitting[2] (formalmente, reducción de varianza)
La premisa central cuando se utiliza una técnica de selección de características es que el dato contiene muchas redundantes o irrelevantes, y así pueden ser removidas sin incurrir en mucha pérdida de la información. Las características redundantes o irrelevantes son dos nociones distintas, pues una característica relevante puede ser redundante en la presencia de otra característica relevante con la que está fuertemente correlacionada.
Las técnicas de selección de características deben ser distinguidas de la extracción de característica, la cual crea características nuevas a partir de funciones de las características originales, mientras que la selección de característica devuelve un subconjunto de las características. Las técnicas de selección de la característica son a menudo utilizadas en dominios donde hay muchas características y comparativamente pocas muestras (o puntos de dato). Los casos arquetípicos para la aplicación de la selección de característica incluye el análisis de datos de microarrays de ADN y textos escritos, donde hay varios miles de características, y unas cuantas decenas a centenares de muestras.
Introducción[editar]
Un algoritmo de selección de características puede ser visto como la combinación de una técnica de búsqueda para proponer nuevos subconjuntos de características, junto con una evaluación mide cuál puntúa los diferentes subconjuntos de características. El algoritmo más sencillo prueba cada subconjunto posible de las características encontrando la que minimiza el índice de error, lo que es una búsqueda exhaustiva del espacio, y es computacionalmente intratable excepto para los conjuntos de característica más pequeños. La elección de la evaluación métrica influye fuertemente en el algoritmo, y son estas evaluaciones métricas las que distinguen entre las tres categorías principales de algoritmos de selección de características: envolturas (wrappers), filtros y métodos embebidos.[3]
- Los métodos envolvedores (wrapper methods) utilizan un modelo predictivo para puntuar subconjuntos de característica. Cada subconjunto nuevo suele entrenar un modelo, el cual está probado en un conjunto de resistencia (hold-out set). Contando el número de errores hace en el conjunto de resistencia escogido (el índice de error del modelo) da la puntuación para ese subconjunto. Como los métodos envolvedores, entrena un nuevo modelo para cada subconjunto, es computacionalmente muy intensivo, pero usualmente proporciona el conjunto de características de mejor comportamiento para aquel tipo particular de modelo.
- Los métodos de filtrado utilizan una proxy de medida en vez del índice de error para puntuar un subconjunto de características. Esta medida es escogida para sea rápido de computar, mientras todavía est'a capturando la utilidad del conjunto de características. Las medidas comunes incluyen la información mutua, la información mutua puntual, el Coeficiente de correlación de Pearson, inter/intra distancia de clase o las puntuaciones de pruebas de importancia para cada combinación clase/característica. Los filtros son normalmente menos intensivos computacionalmente que los wrappers, pero producen un conjunto de características que no está relacionado con un tipo específico de modelo predictivo. Esta carencia significa que un conjunto de características resultante de un filtro es más general que el conjunto resultante de un wrapper, normalmente dando rendimiento de predicción más bajo que un wrapper. Aun así el conjunto de característica no contiene las suposiciones de un modelo de predicción, y así es más útil para exponer las relaciones entre las características. Muchos filtros proporcionan una característica ranking en vez de un mejor subconjunto de característica explícito, y el punto de corte en el ranking es escogido vía validación cruzada. Los métodos de filtro también han sido utilizados como un paso de preprocesamiento para métodos wrappers, dejando que un wrapper sea utilizado en problemas más grandes.[4][5][6]
- Los métodos embebidos son un grupo de técnicas atrapa-todo que realizan selección de características como parte del proceso de construcción del modelo. El ejemplar de esta aproximación es el método LASSO para construir un modelo lineal, el cual penaliza los coeficientes de regresión con una penalidad L1, disminuyendo muchos de ellos a cero. Cualquier característica con coeficiente de regresión diferente de cero es elegido por el algoritmo LASSO. Mejoras al LASSO incluyen Bolasso qué potencia las muestras, y FeaLect que puntúa todas las características basándose en análisis combinatorio de los coeficientes de regresión. Otra aproximación popular es el algoritmo de Eliminación de Característica Recursivo, generalmente utilizado con Máquinas de Soporte Vectorial para repetidamente construir un modelo y sacar características con pesos bajos. Estas aproximaciones tienden aser entre filtros y wrappers en términos de complejidad computacional.[7][8]
En estadística tradicional, la forma más popular de seleccionar características es la regresión paso a paso, que es un técnica de wrapper. Es un algoritmo greedy que añade la mejor característica (o elimina la peor) en cada ronda. El problema principal de control es decidir cuándo parar el algoritmo. En aprendizaje de máquina, esto es típicamente hecho por validación cruz. En estadísticas, algunos criterios están optimizados. Esto se debe al problema inherente de anidación. Métodos más robustos han sido explorados, como ramificación y poda y red lineal por partes.
Selección de subconjunto[editar]
La selección de subconjuntos evalúa un subconjunto de características como un grupo por conveniencia. Los algoritmos de selección del subconjunto pueden ser separados en Wrappers, Filtros y Embebidos. Los wrappers utilizan un algoritmo de búsqueda para buscar a través del espacio de características posibles y evaluar cada subconjunto corriendo un modelo en el subconjunto. Los wrappers pueden ser computacionalmente complejos y tener un riesgo de overfitting para el modelo. Los filtros son similares a los wrappers en la aproximación de búsqueda, pero en vez de evaluar contra un modelo, evalúa un filtro más sencillo. Las técnicas embebidas son embebidas para un modelo específico.
Muchos aproximaciones de búsqueda populares utilizan escalada de cerro codicioso, el cual de manera iterativa evalúa un subconjunto de candidato de características, entonces modifica el subconjunto y evalúa si el subconjunto nuevo es una mejora sobre el viejo. La evaluación de los subconjuntos requiere una puntuado métrico que mide un subconjunto de características. La búsqueda exhaustiva es generalmente poco práctica, por lo que si algún implementador (u operador) define un punto de parada, el subconjunto de características con la puntuación más alta descubierta hasta el momento es seleccionado como el subconjunto de característica satisfactorio. El criterio de parada varía para cada algoritmo; los criterios posibles incluyen: una puntuación de subconjunto supera un umbral, el máximo permitido de tiempo de ejecución del programa fue superado, etc.
Las técnicas de búsqueda alternativas están basadas en la búsqueda de proyección de un objetivo que encuentra proyecciones de baja dimensión del dato con mayor puntuación: las características que tiene las proyecciones más grandes en el espacio de menor dimensión son las elegidas.
Aproximaciones de búsqueda incluyen:
- Exhaustivo
- Mejor primero
- Recocido Simulado
- Algoritmo genético
- Selección hacia adelante codicioso
- Eliminación hacia atrás codicioso[9][10][11]
- Optimización de enjambre de la partícula[12]
- Búsqueda de proyección apuntada
- Búsqueda Dispersa[13]
- Búsqueda por Entornos Variables[14][15]
Dos métricas de filtros populares para problemas de clasificación son correlación e información mutua, a pesar de que tampoco es son métricas exactas o 'medidas de distancia' en el sentido matemático, como fallan para cumplir la desigualdad triangular y así no computar cualquier 'distancia' real – tendrían que ser considerados 'puntuaciones'. Estas puntuaciones están computadas entre un candidato a característica (o conjunto de características) y la categoría de producción deseada. Aun así, hay ciertas métricas que son simplemente una función de la información mutua; ve aquí.[16]
Otras selecciones de filtros son:
- Separación de clases
- Probabilidad de error
- Distancia entre clases
- Distancia probabilista
- Entropía
- Selección de característica basada en consitencia
- Selección de característica basada en correlación
Criterios de optimalidad[editar]
El criterio de elección de optimalidad es difícil cuando hay objetivos múltiples en una tarea de selección de características. Muchos de los similares incorporan una medida de exactitud, penalizada por el número de características seleccionadas (ej. el Criterio de información bayesiano). El más viejo es el Cp de Mallows estadístico y criterio de información de Akaike (AIC). Estos añaden variables si el t-statidístico es más grande que
Aprendizaje de estructura[editar]
Selección de característica del filtro es un caso concreto de un paradigma más general llamada Aprendizaje de Estructura. La selección de características encuentra para característica específica una variable de objetivo concreta mientras que aprendizaje de estructura encuentra las relaciones entre todas las variables, normalmente expresando estas relaciones como un grafo. Los algoritmos más comunes de aprendizaje de estructura suponen que el dato está generado por una Red bayesiana, por lo que la estructura es un modelo de un grafo dirigido. La solución óptima al problema de selección de característica de filtro es la Manta de Markov del nodo objetivo, y en una Red bayesiana, hay un Manta de Markov única para cada nodo.[17]
Árboles regularizados[editar]
Las características de un árbol de decisión o un árbol ensamblado han demostrado ser redundantes. Un método reciente llamado árbol regularizado puede ser utilizado para la selección del subconjunto de características.[18] Los árboles regularizados penalizan usando una variable similar a las variables seleccionadas en nodos de previos del árbol separando el nodo actual. Árboles regularizados solo necesitan construir un modelo de árbol (o un árbol ensemble modelo) y así éste es computacionalmente eficaz.
Visión general en metaheuristics métodos[editar]
Un metaheurística es una descripción general de un algoritmo dedicado a solucionar difíciles (típicamente problemas NP-duros) problemas de optimización para los que no hay soluciones clásicas. Generalmente, un metaheurística es un algoritmo estocástico que tiende a encontrar un óptimo global.
Principios principales[editar]
Los métodos de selección de la característica son típicamente presentados en tres clases basados en cómo combinan el algoritmo de selección y la construcción del modelo.
Método de filtro[editar]

Los tipos de métodos de filtro seleccionan variables a toda costa del modelo. Están basados sólo en características generales como la correlación con la variable a pronosticar. Los métodos de filtro suprimen las variables menos interesantes. Las otras variables serán parte de una clasificación o un modelo de regresión usado para clasificar o para pronosticar datos. Estos métodos son particularmente eficaces en y resistentes al overfitting.[19]
Sin embargo, los métodos de filtrado tienden a seleccionar variables redundantes porque no consideran las relaciones entre variables. Por tanto, son principalmente utilizados como método de preprocesamiento.
Método Wrapper[editar]
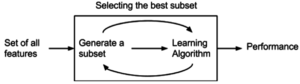
Los métodos wrapper evalúan subconjuntos de variables que lo permiten, aproximaciones de filtro diferente, para detectar las interacciones posibles entre variables. Las dos desventajas principales de estos métodos son :[20]
- El riesgo creciente de overfitting cuando el número de observaciones es insuficiente.
- El tiempo de computación significativo cuándo el número de variables es grande.
Métodos embebidos[editar]

Los métodos embebidos han sido recientemente propuestos para intentar combinar las ventajas de los métodos anteriores. Un algoritmo de aprendizaje aprovecha su proceso de selección variable propio y realiza la selección de selección y clasificación simultáneamente.
Aplicación de selección de característica metaheuristics[editar]
Esto es una encuesta de la aplicación de las metaheurísticas de selección de característica utilizados recientemente en la literatura. Esta encuesta fue realizada por J. Hammon en su tesis.
| Aplicación | Algoritmo | Aproximación | classifier | Función de evaluación | Ref |
|---|---|---|---|---|---|
| SNPs | Selección de característica que utiliza Semejanza de Característica | Filtro | r2 | Phuong 2005 | |
| SNPs | Algoritmo genético | Wrapper | Árbol de decisión | Exactitud de clasificación (10-pliegue) | Shah 2004[21] |
| SNPs | HillClimbing | Filtro + Wrapper | Naive Bayesiano | Suma residual pronosticada de plazas | Mucho tiempo 2007[22] |
| SNPs | Simulado Annealing | Naive bayesian | Exactitud de clasificación (5-pliegue) | Ustunkar 2011[23] | |
| Segmentos parole | Colonia de hormigas | Wrapper | Red Neuronal artificial | MSE | Al-ani 2005[cita requerida] |
| Marketing | Simulado Annealing | Wrapper | Regresión | AIC, r2 | Meiri 2006[24] |
| Economía | Simulado Annealing, Algoritmo Genético | Wrapper | Regresión | BIC | Kapetanios 2005[25] |
| Masa espectral | Algoritmo genético | Wrapper | Regresión Lineal múltiple, Parcial Menos Plazas | Raíz-malo-error cuadrado de predicción | Broadhurst 2007[26] |
| Spam | Binario PSO + Mutación | Wrapper | Árbol de decisión | weighted Costó | Zhang 2014 |
| Microarray | Tabu Búsqueda + PSO | Wrapper | Máquina de Vector del soporte, K Vecinos más Cercanos | Distancia euclidiana | Chuang 2009[27] |
| Microarray | PSO + Algoritmo genético | Wrapper | Máquina de Vector del soporte | Exactitud de clasificación (10-pliegue) | Alba 2007[28] |
| Microarray | El algoritmo genético + Iteró Búsqueda Local | Embedded | Máquina de Vector del soporte | Exactitud de clasificación (10-pliegue) | Duval 2009[29] |
| Microarray | Búsqueda Local iterada | Wrapper | Regresión | Probabilidad posterior | Hans 2007[30] |
| Microarray | Algoritmo genético | Wrapper | K Vecinos más cercanos | Exactitud de clasificación (Deja-un-fuera cruz-validación) | Jirapech-Umpai 2005[31] |
| Microarray | Algoritmo Genético híbrido | Wrapper | K Vecinos más cercanos | Exactitud de clasificación (Deja-un-fuera cruz-validación) | Oh 2004[32] |
| Microarray | Algoritmo genético | Wrapper | Máquina de Vector del soporte | Sensibilidad y especificidad | Xuan 2011[33] |
| Microarray | Algoritmo genético | Wrapper | Todo paired Máquina de Vector del Soporte | Exactitud de clasificación (Deja-un-fuera cruz-validación) | Peng 2003[34] |
| Microarray | Algoritmo genético | Embedded | Máquina de Vector del soporte | Exactitud de clasificación (10-pliegue) | Hernandez 2007[35] |
| Microarray | Algoritmo genético | Híbrido | Máquina de Vector del soporte | Exactitud de clasificación (Deja-un-fuera cruz-validación) | Huerta 2006[36] |
| Microarray | Algoritmo genético | Máquina de Vector del soporte | Exactitud de clasificación (10-pliegue) | Muni 2006[37] | |
| Microarray | Algoritmo genético | Wrapper | Máquina de Vector del soporte | EH-DIALL, CLUMP | Jourdan 2004[38] |
| La enfermedad de Alzheimer | Welch t-prueba | Filtro | kernel Máquina de vector del soporte | Exactitud de clasificación (10-pliegue) | Zhang 2015[39] |
| Visión de ordenador | Selección de Característica infinita | Filtro | Independiente | Precisión mediana, ROC AUC | Roffo 2015[40] |
| Microarrays | Eigenvector Centrality FS | Filtro | Independiente | Precisión mediana, Exactitud, ROC AUC | Roffo & Melzi 2016[41] |
| XML | Simétrico Tau (ST) | Filtro | Estructural Associative Classificatión | Exactitud, Cobertura | Shaharanee & Hadzic 2014 |
Selección de característica embebidas en algoritmos de aprendizaje[editar]
Algunos algoritmos de aprendizaje realizan la selección de características como parte de su operación global. Estos incluyen:
- Árboles regularizados, p. ej. regularized el bosque aleatorio implementado en el RRF paquete
- Árbol de decisión[42]
- Memetic Algoritmo
- Aleatorio multinomial logit (RMNL)
- Redes que codifican coche con un bottleneck-capa
- Submodular Selección de característica[43][44][45]
- Aprendizaje local basado en selección de características. Comparado con métodos tradicionales, no implica cualquier búsqueda heurística, puede fácilmente controlar problemas multi-clase, y trabaja paraproblemas lineales y no lineales . Está apoyado por una fundación teórica fuerte. Los experimentos numéricos mostraron que el método puede conseguir una solución cercana al óptimo incluso cuándo el dato contiene >1M características irrelevantes.[46]
Ve también[editar]
- Análisis de clusters
- Minería de datos
- Reducción de dimensionalidad
- Extracción de característica
- Ingeniería de características
- Optimización de Hiperparámetros
Referencias[editar]
- ↑ An Introduction to Statistical Learning. Springer. 2013. p. 204. Archivado desde el original el 23 de junio de 2019. Consultado el 16 de noviembre de 2017.
- ↑ Bermingham, Mairead L.; Pong-Wong, Ricardo; Spiliopoulou, Athina; Hayward, Caroline; Rudan, Igor; Campbell, Harry; Wright, Alan F.; Wilson, James F. et al. (2015). «Application of high-dimensional feature selection: evaluation for genomic prediction in man». Sci. Rep. 5. Se sugiere usar
|número-autores=(ayuda) - ↑ Guyon, Isabelle; Elisseeff, André (2003). «An Introduction to Variable and Feature Selection». JMLR 3.
- ↑ Referencia vacía (ayuda)
- ↑ Forman, George (2003). «An extensive empirical study of feature selection metrics for text classification». Journal of Machine Learning Research 3: 1289-1305.
- ↑ «Divergence-based feature selection for separate classes» (PDF). Neurocomputing (ELSEVIER) 101 (4): 32-42. 2013.
- ↑ Bach, Francis R (2008). «Bolasso: model consistent lasso estimation through the bootstrap». Proceedings of the 25th international conference on Machine learning: 33-40. doi:10.1145/1390156.1390161.
- ↑ Zare, Habil (2013). «Scoring relevancy of features based on combinatorial analysis of Lasso with application to lymphoma diagnosis». BMC Genomics 14: S14. PMC 3549810. PMID 23369194. doi:10.1186/1471-2164-14-S1-S14.
- ↑ Figueroa, Alejandro (2015). «Exploring effective features for recognizing the user intent behind web queries». Computers in Industry 68: 162-169. doi:10.1016/j.compind.2015.01.005.
- ↑ https://www.researchgate.net/publication/259174469_Learning_to_Rank_Effective_Paraphrases_from_Query_Logs_for_Community_Question_Answering. Falta el
|título=(ayuda) - ↑ «Category-specific models for ranking effective paraphrases in community Question Answering». Expert Systems with Applications 41: 4730-4742. 2014. doi:10.1016/j.eswa.2014.02.004.
- ↑ Zhang, Y.; Wang, S.; Phillips, P. (2014). «Binary PSO with Mutation Operator for Feature Selection using Decision Tree applied to Spam Detection». Knowledge-Based Systems 64: 22-31. doi:10.1016/j.knosys.2014.03.015.
- ↑ F.C. Garcia-Lopez, M. Garcia-Torres, B. Melian, J.A. Moreno-Perez, J.M. Moreno-Vega. Solving feature subset selection problem by a Parallel Scatter Search, European Journal of Operational Research, vol. 169, no. 2, pp. 477–489, 2006.
- ↑ F.C. Garcia-Lopez, M. Garcia-Torres, B. Melian, J.A. Moreno-Perez, J.M. Moreno-Vega. Solving Feature Subset Selection Problem by a Hybrid Metaheuristic. In First International Workshop on Hybrid Metaheuristics, pp. 59–68, 2004.
- ↑ M. Garcia-Torres, F. Gomez-Vela, B. Melian, J.M. Moreno-Vega. High-dimensional feature selection via feature grouping: A Variable Neighborhood Search approach, Information Sciences, vol. 326, pp. 102-118, 2016.
- ↑ Alexander Kraskov, Harald Stögbauer, Ralph G. Andrzejak, and Peter Grassberger, "Hierarchical Clustering Based on Mutual Information", (2003) ArXiv q-bio/0311039
- ↑ Aliferis, Constantin (2010). «Local causal and markov blanket induction for causal discovery and feature selection for classification part I: Algorithms and empirical evaluation». Journal of Machine Learning Research 11: 171-234.
- ↑ H. Deng, G. Runger, "Feature Selection via Regularized Trees Archivado el 12 de septiembre de 2015 en Wayback Machine.", Proceedings of the 2012 International Joint Conference on Neural Networks (IJCNN), IEEE, 2012
- ↑ J. Hammon. Optimisation combinatoire pour la sélection de variables en régression en grande dimension : Application en génétique animale. November 2013 (en francés)
- ↑ T. M. Phuong, Z. Lin et R. B. Altman. Choosing SNPs using feature selection. Archivado el 13 de septiembre de 2016 en Wayback Machine. Proceedings / IEEE Computational Systems Bioinformatics Conference, CSB. IEEE Computational Systems Bioinformatics Conference, pages 301-309, 2005. PubMed.
- ↑ Shah, S. C.; Kusiak, A. (2004). «Data mining and genetic algorithm based gene/SNP selection». Artificial intelligence in medicine 31 (3): 183-196. PMID 15302085. doi:10.1016/j.artmed.2004.04.002.
- ↑ Long, N.; Gianola, D.; Weigel, K. A (2011). «Dimension reduction and variable selection for genomic selection : application to predicting milk yield in Holsteins». Journal of Animal Breeding and Genetics 128 (4): 247-257. doi:10.1111/j.1439-0388.2011.00917.x.
- ↑ G. Ustunkar, S. Ozogur-Akyuz, G. W. Weber, C. M. Friedrich et Yesim Aydin Son. Selection of representative SNP sets for genome-wide association studies : a metaheuristic approach. Optimization Letters, November 2011.
- ↑ R. Meiri et J. Zahavi. Using simulated annealing to optimize the feature selection problem in marketing applications. European Journal of Operational Research, vol. 171, no. 3, pages 842-858, Juin 2006
- ↑ G. Kapetanios. Variable Selection using Non-Standard Optimisation of Information Criteria. Working Paper 533, Queen Mary, University of London, School of Economics and Finance, 2005.
- ↑ D. Broadhurst, R. Goodacre, A. Jones, J. J. Rowland et D. B. Kell. Genetic algorithms as a method for variable selection in multiple linear regression and partial least squares regression, with applications to pyrolysis mass spectrometry. Analytica Chimica Acta, vol. 348, no. 1-3, pages 71-86, August 1997.
- ↑ Chuang, L.-Y.; Yang, C.-H. (2009). «Tabu search and binary particle swarm optimization for feature selection using microarray data». Journal of computational biology 16 (12): 1689-1703. PMID 20047491. doi:10.1089/cmb.2007.0211.
- ↑ E. Alba, J. Garia-Nieto, L. Jourdan et E.-G. Talbi. Gene Selection in Cancer Classification using PSO-SVM and GA-SVM Hybrid Algorithms. Archivado el 18 de agosto de 2016 en Wayback Machine. Congress on Evolutionary Computation, Singapor : Singapore (2007), 2007
- ↑ B. Duval, J.-K. Hao et J. C. Hernandez Hernandez. A memetic algorithm for gene selection and molecular classification of an cancer. In Proceedings of the 11th Annual conference on Genetic and evolutionary computation, GECCO '09, pages 201-208, New York, NY, USA, 2009. ACM.
- ↑ C. Hans, A. Dobra et M. West. Shotgun stochastic search for 'large p' regression. Journal of the American Statistical Association, 2007.
- ↑ Aitken, S. (2005). «Feature selection and classification for microarray data analysis : Evolutionary methods for identifying predictive genes». BMC Bioinformatics 6 (1): 148. doi:10.1186/1471-2105-6-148.
- ↑ Oh, I. S.; Moon, B. R. (2004). «Hybrid genetic algorithms for feature selection». IEEE Transactions on Pattern Analysis and Machine Intelligence 26 (11): 1424-1437. doi:10.1109/tpami.2004.105.
- ↑ Xuan, P.; Guo, M. Z.; Wang, J.; Liu, X. Y.; Liu, Y. (2011). «Genetic algorithm-based efficient feature selection for classification of pre-miRNAs». Genetics and Molecular Research 10 (2): 588-603. PMID 21491369. doi:10.4238/vol10-2gmr969.
- ↑ Peng, S. (2003). «Molecular classification of cancer types from microarray data using the combination of genetic algorithms and support vector machines». FEBS Letters 555 (2): 358-362. doi:10.1016/s0014-5793(03)01275-4.
- ↑ J. C. H. Hernandez, B. Duval et J.-K. Hao. A genetic embedded approach for gene selection and classification of microarray data. In Proceedings of the 5th European conference on Evolutionary computation, machine learning and data mining in bioinformatics, EvoBIO'07, pages 90-101, Berlin, Heidelberg, 2007. SpringerVerlag.
- ↑ E. B. Huerta, B. Duval et J.-K. Hao. A hybrid GA/SVM approach for gene selection and classification of microarray data. evoworkshops 2006, LNCS, vol. 3907, pages 34-44, 2006.
- ↑ D. P. Muni, N. R. Pal et J. Das. Genetic programming for simultaneous feature selection and classifier design. IEEE Transactions on Systems, Man, and Cybernetics, Part B : Cybernetics, vol. 36, no. 1, pages 106-117, February 2006.
- ↑ L. Jourdan, C. Dhaenens et E.-G. Talbi. Linkage disequilibrium study with a parallel adaptive GA. International Journal of Foundations of Computer Science, 2004.
- ↑ Zhang, Y.; Dong, Z.; Phillips, P.; Wang, S. (2015). «Detection of subjects and brain regions related to Alzheimer's disease using 3D MRI scans based on eigenbrain and machine learning». Frontiers in Computational Neuroscience 9: 66. doi:10.3389/fncom.2015.00066.
- ↑ «Infinite Feature Selection». 2015 IEEE International Conference on Computer Vision (ICCV): 4202-4210. 1 de diciembre de 2015. doi:10.1109/ICCV.2015.478.
- ↑ Roffo, Giorgio (September 2016). «Features Selection via Eigenvector Centrality». NFmcp2016. Consultado el 12 de noviembre de 2016.
- ↑ R. Kohavi and G. John Wrappers for feature subset selection, Artificial intelligence 97.1-2 (1997): 273-324
- ↑ Das el al, Submodular meets Spectral: Greedy Algorithms for Subset Selection, Sparse Approximation and Dictionary Selection
- ↑ Liu et al, Submodular feature selection for high-dimensional acoustic score spaces Archivado el 17 de octubre de 2015 en Wayback Machine.
- ↑ Zheng et al, Submodular Attribute Selection for Action Recognition in Video Archivado el 18 de noviembre de 2015 en Wayback Machine.
- ↑ Y. Sun, S. Todorovic, S. Goodison (2010) Local-Learning-Based Feature Selection for High-Dimensional Data Analysis, IEEE Transactions on Pattern Analysis and Machine Intelligence, 32(9): 1610-1626
Enlaces externos[editar]
- Paquete de Selección de la característica, Arizona Universidad Estatal (Matlab Código)
- NIPS Reto 2003
- Naive Bayes Implementación con selección de característica en Visual Básico Archivado el 14 de febrero de 2009 en Wayback Machine. (incluye ejecutable y código de fuente)
- Mínimo-redundancia-máximo-pertinencia (mRMR) programa de selección de la característica
- FEAST (algoritmos de Selección de Característica de Código abierto en C y MATLAB)
