Endonucleasa
Las endonucleasas son enzimas que catalizan la ruptura de enlaces fosfodiéster en diferentes regiones ubicadas en el interior de una cadena polinucleotídica. Esto las diferencia de las exonucleasas, que catalizan la escisión de enlaces fosfodiéster en los extremos de las cadenas.
Algunas endonucleasas, tales como la Desoxirribonucleasa I, cortan al ADN en forma relativamente inespecífica (esto es sin consideraciones en cuanto a la secuencia de bases), mientras que muchas, llamadas típicamente enzimas de restricción, o endonucleasas de restricción, provocan rupturas únicamente en determinadas secuencias de nucleótidos muy específicas.[1]
Enzimas de restricción
Las enzimas de restricción son endonucleasas provenientes de eubacterias y arqueas que reconocen secuencias de ADN muy específicas.[2] La secuencia de nucleótidos reconocida para escisión por una enzima de restricción se denomina sitio de restricción. Típicamente un sitio de restricción es una secuencia palindrómica de entre cuatro a seis nucleótidos de extensión. La mayor parte de las endonucleasas de restricción escinden a la molécula de ADN bicatenario en forma desigual, dejando extremos complementarios de cadena simple. Estos extremos pueden luego reconectarse a través de un proceso de hibridación, por lo que son denominados "extremos adhesivos". Una vez apareados, los enlaces fosfodiéster de los fragmentos pueden ser unidos por medio de una ADN ligasa.
Existen cientos de endonucleasas de restricción conocidas, cada una de las cuales ataca un sitio de restricción diferente. Los extremos de ADN escindidos por una misma endonucleasa de restricción pueden ser emparejados juntos sin importar el origen del ADN. Estas moléculas de ADN formadas por trozos de orígenes diferentes se denominan ADN recombinante; es decir ADN formado por la unión de diferentes genes para crear nuevas combinaciones.[1] Las endonucleasas de restricción (enzimas de restricción) se dividen en tres categorías, Tipo I, Tipo II, y Tipo III, de acuerdo a su mecanismo de acción.
Estas enzimas son frecuentemente utilizadas en ingeniería genética para producir ADN recombinante para su posterior introducción en células bacterianas, vegetales o animales; como así también en biología sintética.[3]
Categorías
En última instancia, hay tres categorías de endonucleasas de restricción que contribuyen relativamente a la escisión de secuencias específicas. Los tipos I y III son complejos multisubunidades de gran tamaño que incluyen tanto la actividad endonucleasa como la actividad metilasa.
Las endonucleasas de tipo I pueden escindir al ADN en ubicaciones aleatorias, a 1000 o más pares de bases de distancia de la secuencia de reconocimiento y requieren de ATP como fuente de energía.
Las endonucleaasas de tipo II se comportan de forma ligeramente diferente, fueron aisladas por Hamilton Smith en 1970, se trata de las versiones más simples de endonucleasas y no requieren de ATP para su función. Algunos ejemplos de endonucleasas de tipo II son la BamHI, EcoRI, EcoRV y HaeIII.
Las endonucleasas de tipo III, escinden el ADN a no más de 25 pares de bases de distancia de la secuencia de reconocimiento y también requieren de ATP en el proceso.[1]
Notaciones
La notación más comúnmente utilizada para las endonucleasas de restricción es de la forma "VwxYZ", donde "Vwx" corresponde a las iniciales de la nomenclatura binomial para la forma de vida de donde proviene (bacteria), "Y" es la denominación de la cepa (y es opcional); y "Z" (en números romanos) indica diferentes enzimas de restricción pertenecientes a una misma forma de vida. Así por ejemplo, "EcoRI" significa que la endonucleasa pertenece a Escherichia coli ("Eco"); la cepa es la RY13 ("R"), y que fue la primera descubierta en esa forma de vida: número "I".
Otro ejemplo: "HaeII" y "HaeIII" se refieren a la bacteria Haemophilus aegyptius, mientras que las enzimas son la número II y número III, respectivamente.[1]: 64–64 Las enzimas de restricción que se utilizan en biología molecular, por lo general reconocen una secuencia diana corta, de aproximadamente 4 - 8 pares de bases. Por ejemplo la EcoRI reconoce y escinde la secuencia 5'– G·AATTC –3'.[4]
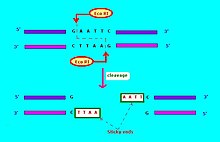
Una endonucleasa típicamente requiere de un sitio de reconocimiento, y una secuencia de escisión (típicamente de bases nucleotídicas: A, C, G, T). Si el sitio de reconocimiento se encuentra fuera de la región de escisión, entonces se dice que la endonucleasa es de tipo I. Si la secuencia de reconocimiento se superpone con la secuencia de escisión, entonces la endonucleasa se considera una enzima de restricción y es de tipo II.
Consideraciones adicionales
Las endonucleasas pueden escindir ADN de cadena doble (ADNcd), o de cadena simple (ADNcs), o incluso ARN. El presente artículo se encuentra centrado en las endonucleasas que actúan sobre ADNcd, sin embargo, lo explicado en este artículo puede aplicarse a:
- ADNcd estándar
- ADN no estándar
- Uniones de Holliday
- ADN triple cadena, ADN cuádruple cadena (G-quadruplex)
- Híbridos doble cadena de ADN y ARN (una cadena es de ADN, mientras que la otra es de ARN)[1]: 72–73
- ADN sintético o artificial (por ejemplo, ADN que contiene bases diferentes a las canónicas, C, G, T).[1]: chapter 3
Adicionalmente, las investigaciones actuales están orientadas al desarrollo de endonucleasas de restricción sintéticas o artificiales, específicamente que posean un sitio de reconocimiento que sea único dentro de un genoma.
Las endonucleasas de restricción o enzimas de restricción, típicamente escinden las cadenas de ácidos nucleicos de dos formas diferentes: con extremos romos, o con extremos adhesivos.[1]: 64
Reparación del ADN
Las endonucleasas desempeñan un papel importante en la reparación del ADN. Específicamente las endonucleasas AP, catalizan la ruptura del ADN exclusivamente en sitios AP (sitios apurínicos-apirimidínicos, es decir sitios en los cuales se ha perdido la base nitrogenada, pero se conserva la columna de azúcar-fosfato), y por lo tanto preparan al ADN para la subsecuente escisión, síntesis reparadora y ligado. Por ejemplo, cuando se produce una despurinización, esta lesión deja al azúcar desoxirribosa sin su base nitrogenada.[5] La endonucleasa AP reconoce este azúcar y esencialmente corta el ADN en ese sitio, permitiendo que luego continúe la reparación del ADN.[6] Las células de E. coli contienen dos endonucleasas AP: la endonucleasa IV (endoIV) y la exonucleasa III (exoIII). Mientras que en eucariotas, solo hay un tipo de endonucleasa AP.[7]

Endonucleasas comunes
Debajo se presentan tablas de las endonucleasas procariotas y eucariotas más comunes.[8]
| Enzima procariota | Origen | Comentarios |
|---|---|---|
| Endonucleasa RecBCD | E. coli | Parcialmente dependiente de ATP; actúa además como exonucleasa; funciona en recombinación y reparación |
| Endonucleasa T7 | Codificada en fago T7 (gen 3) | Esencial para la replicación del fago; preferencia sobre ADN simple cadena. |
| T4 endonucleasa IV | Codificada en fago T4 (denA) | Rompe la secuencia -TpC- para producir oligonucleótidos terminados en 5'-dCMP-; la longitud de la cadena del producto varía dependiendo de las condiciones |
| Endonucleasa Bal 31 | 'Alteromonas espejiana | También funciona como exonucleasa; escinde los extremos 3' y 5' de ADN dúplex |
| Endonucleasa I (endo I) | E. coli (endA) | Localización periplasmática; la longitud promedio del producto es 7; inhibida por ARNt; produce rupturas en ADN doble cadena; cuando está acomplejado con ARNt produce muescas en una sola cadena; las mutantes endo I crecen normalmente |
| Nucleasa microcóccica | Staphylococcus | Produce extremos 3'-P; requiere Ca2+ ; también actúa sobre ARN; prefiere ADN de cadena simple y regiones ricas en AT |
| Endonucleasa II (endo VI, exo III) | E. coli (xth) | Ruptura a continuación de un sitio AP; también actúa como exonucleasa 3'-->5'; actividad fosfomonoesterasa en el extremo 3'-P |
| Enzima eucariota | Origen | Comentarios |
|---|---|---|
| Endonucleasa de Neurospora | Neurospora crassa | También actúa sobre ARN |
| Nucleasa-S1 | Aspergillus oryzae | También actúa sobre ARN |
| Nucleasa-P1 | Penicillium citrinum | También actúa sobre ARN |
| Nucleasa de judía mungo | Brotes de Vigna radiata | También actúa sobre ARN |
| Nucleasa de ustilago (Dnasa I) | Ustilago maydis | También actúa sobre ARN |
| Dnasa I | Páncreas bovino | La longitud de cadena del producto es de 4 pb; produce cortes en las dos cadenas en presencia de Mn2+ |
| Endonucleasa AP | Núcleo, mitocondria | Enzima involucrada en la vía de reparación de ADN por escisión. |
| Endo R | Células HeLa | Enzima específica para sitios GC |
Mutaciones
La xerodermia pigmentosa es una enfermedad genética autosómica recesiva, rara causada por una endonucleasa UV-específica defectuosa. Los pacientes con esta mutación son incapaces de reparar el daño al ADN causado por la luz del sol.[9]
La anemia de células falciformes es una enfermedad causada por una mutación puntual. La secuencia alterada por la mutación elimina el sitio de reconocimiento para la endonucleasa de restricción MstII.[10]
Las mutaciones en las endonucleasas de splicing del ARNt causan hipoplasia pontocerebelar. Las hipoplasias pontocerebelares (HPC) representan a un grupo de enfermedades genéticas autosómicas recesivas que son causadas por mutaciones en tres de las cuatro subunidades de la ARNt endonucleasa que forman el complejo de splicing.[11]
Véase también
Referencias
- ↑ a b c d e f g Cox M, Nelson DR, Lehninger AL (2005). Lehninger principles of biochemistry. San Francisco: W.H. Freeman. pp. 952. ISBN 0-7167-4339-6.
- ↑ Stephen T. Kilpatrick; Jocelyn E. Krebs; Lewin, Benjamin; Goldstein, Elliott (2011). Lewin's genes X. Boston: Jones and Bartlett. ISBN 0-7637-6632-1.
- ↑ Simon M (2010). Emergent computation: Emphasizing Bioinformatics. Nueva York: Springer. p. 437. ISBN 1441919635.
- ↑ Losick R, Watson JD, Baker TA, Bell S, Gann S, Levine MW (2008). Molecular biology of the gene. San Francisco: Pearson/Benjamin Cummings. ISBN 0-8053-9592-X.
- ↑ Ellenberger T, Friedberg EC, Walker GS, Wolfram S, Wood RJ, Schultz R (2006). DNA repair and mutagenesis. Washington, D.C: ASM Press. ISBN 1-55581-319-4.
- ↑ Alberts B (2002). Molecular biology of the cell. Nueva York: Garland Science. ISBN 0-8153-3218-1.
- ↑ Nishino T, Morikawa K (diciembre de 2002). «Structure and function of nucleases in DNA repair: shape, grip and blade of the DNA scissors». Oncogene 21 (58): 9022-32. PMID 12483517. doi:10.1038/sj.onc.1206135.
- ↑ Tania A. Baker; Kornberg, Arthur (2005). DNA replication. University Science. ISBN 1-891389-44-0.
- ↑ Medical Biochemistry at a Glance. Nueva York: Wiley. 2012. ISBN 0-470-65451-1.
- ↑ Ferrier DR, Champe PC, Harvey RP (2008). Biochemistry. Philadelphia: Wolters Kluwer/Lippincott Williams & Wilkins. ISBN 0-7817-6960-4.
- ↑ Budde BS, Namavar Y, Barth PG, Poll-The BT, Nürnberg G, Becker C, van Ruissen F, Weterman MA, Fluiter K, te Beek ET, Aronica E, van der Knaap MS, Höhne W, Toliat MR, Crow YJ, Steinling M, Voit T, Roelenso F, Brussel W, Brockmann K, Kyllerman M, Boltshauser E, Hammersen G, Willemsen M, Basel-Vanagaite L, Krägeloh-Mann I, de Vries LS, Sztriha L, Muntoni F, Ferrie CD, Battini R, Hennekam RC, Grillo E, Beemer FA, Stoets LM, Wollnik B, Nürnberg P, Baas F (septiembre de 2008). «tRNA splicing endonuclease mutations cause pontocerebellar hypoplasia». Nat. Genet. 40 (9): 1113-8. PMID 18711368. doi:10.1038/ng.204.
