Maltasa
| Maltasa-glucoamilasa (alfa-glucosidasa) | ||||
|---|---|---|---|---|
| Estructuras disponibles | ||||
| PDB | ||||
| Identificadores | ||||
| Símbolos | 7043 MGA | |||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 3.2.1.20 | |||
| Locus | Cr. 7 q34 | |||
| Ortólogos | ||||
| Especies |
| |||
| Entrez |
| |||
| UniProt |
| |||
| RefSeq (ARNm) |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
La maltasa es una enzima que cataliza la hidrólisis de la maltosa (disacárido) en las dos glucosas simples que la componen. En los seres humanos se encuentra presente en el intestino delgado y juega un papel fundamental en la digestión. Pertenece a la familia de las disacaridasas, enzimas encargadas de hidrolizar los disacáridos en los monosacáridos que los forman.
La digestión del almidón requiere seis enzimas intestinales, de las cuales dos son α-amilasas. Las cuatro restantes se han identificado como maltasas, exoglucosidasas unidas a la superficie luminal de los enterocitos.[1]

En la mayoría de los casos es equivalente decir alfa-gluosidasa a maltasa, pese a que el término "maltasa" pone énfasis en la naturaleza del disacárido del sustrato del cual se esconde la glucosa, mientras que "alfa-glucosidasa" enfatiza en el enlace sin importar el tamaño del glúcido.
Nomenclatura y clasificación
[editar]La maltasa pertenece a la familia de las α-glucosidasas (esenciales para descomponer el glucógeno en glucosa),[2] y se encuentra en el borde en cepillo de las microvellosidades intestinales del intestino delgado. Puesto que la maltasa cataliza la digestión de oligosacáridos de almidón lineales (sin ramificaciones α(1→6)) para obtener glucosa, también se las conoce como α-glucosidasas; aunque también hay ciertas maltasas como la sacarasa-isomaltasa que son capaces de romper estos enlaces α(1→6) para hacer más lineales las moléculas y permitir la degradación de las maltasas-glucoamilasas.
Maltasa-glucoamilasa
[editar]Juega un papel crucial en la digestión del almidón. Gracias a esta enzima el ser humano es capaz de digerir almidones de origen vegetal, ya que degrada los enlaces o glicosídicos α(1→4), que son los más lineales y de esta manera a partir de grandes polisacáridos se obtienen unidades de glucosa que son vitales en el metabolismo humano.[3]
Sacarasa-glucoamilasa
[editar]Es esencial para la digestión de carbohidratos, incluidos el almidón, la sacarosa y la isomaltosa. Trabaja junto con la maltasa-glucoamilasa para degradar polisacáridos grandes, concretamente se encarga en catalizar la hidrólisis de los enlaces α(1→6) que puedan haber.
Alfa-amilasa 1
[editar]Su función es escindir los enlaces α-glucosidasa en los oligosacáridos y polisacáridos que forman el compuesto para que el resto de enzimas puedan catalizar otras reacciones. Así como las demás enzimas actúan sobre disacáridos o oligosacáridos, en primer lugar es vital la escisión de las macromoléculas en moléculas más pequeñas que puedan ser degradadas por las otras enzimas.[4]
Estructura
[editar]

La maltasa o α-glucosidasa pertenece al familia GH13 de enzimas intestinales que actúan sobre sustratos con enlaces alfa-glicosídicos. Los siguientes genes pueden codificar para la maltasa:
- La maltasa-glucoamilasa (codificada en humanos por el gen MGAM) es una enzima digestiva compuesta por un dominio citosólico, un dominio transmembrana, un enlace O-glicosídico y dos unidades enzimáticas:
- Extremo N-terminal: compuesto por cinco dominios proteicos (un primer dominio de trébol P rico en cisteína, un segundo, cuarto y quinto dominio formado por dos láminas betas antiparalelas y el tercer dominio que es el más grande y el que le otorga la actividad catalítica). A este extremo N-terminal no se le pueden acoplar substratos demasiado grandes y por eso demuestra afinidad hacia la maltosa, la maltotriosa, la maltotetrosa y la maltopentosa.[5]
- Extremo C-terminal: contiene sitios de unión adicionales y por ende puede degradar sustratos más grandes y tiene unos 21 aminoácidos más que el extremo N-terminal (explicando así su función más amplia).
- El ácido α-glucosidasa, codificado por el gen GAA y cuya función principal es la lisis de azúcares complejos. Este ácido contiene aproximadamente 952 aminoácidos y es procesado post-traduccionalmente mediante la glicosilación y supresión de péptidos en los extremos N- y C- terminales.
- La sacarasa-isomaltasa (codificada por el gen SI) es un complejo enzimático que se encuentra en los márgenes de las vellosidades del intestino delgado. Está formada por 1827 aminoácidos y contiene más de 19 sitios de glicosilación. Tiene un dominio citosólico, un dominio transmembrana, un enlace O-glicosídico y dos subunidades homólogas, la sacarasa y la isomaltasa, de 120 kDa y 140 kDa respectivamente. La subunidad isomaltasa es la responsable del anclaje de la enzima con células intestinales ya que dispone de un segmento hidrofóbico de aminoácidos especializado en la asociación con la membrana de dichas células.[6]
- La Α-Amilasa es una enzima (gen AMY1A) con un peso molecular de 50kDa y tres dominio estructurales:
- El dominio A, también conocido como dominio central, está formado por 8 láminas paralelas β-plegadas rodeadas por 8 hélices α.
- El dominio B, de menor tamaño, se encuentra entre la tercera hélice α y la lámina β que la precede.
- El dominio C, cuya función no está bien definida aún, se encuentra unido al dominio A. Tiene una estructura β antiparalela.[7]
- En organismos concretos, existen además dominios adicionales (C, D, F, G, H e I) unidos al dominio A. Su función no está bien definida aún.

Topología de la alfa-amilasa 1 del páncreas humano.
Síntesis
[editar]En mamíferos
[editar]La maltasa de las microvellosidades intestinales de los mamíferos se sintetiza como una única cadena polipeptídica que no sufre ningún tipo de escisión proteolítica, ni intracelular ni extracelular. A esta se le añaden carbohidratos ricos en manosa por glicosilación cotraduccional en aquellos dominios de la membrana en los que precipita Ca2+.[8]
En levaduras
[editar]Las levaduras presentan, en cinco loci diferentes, el grupo de genes MAL (MAL1, MAL2, MAL3, MAL4 y MAL6). La presencia de como mínimo uno de estos genes es indispensable para que se lleve a cabo la síntesis de la maltasa, ya que sugiere que los genes MAL codifican por proteínas reguladoras de este proceso. En consecuencia, el déficit de estos genes en levaduras implicaría la incapacidad de estas para fermentar la maltosa.[9]
Gen
[editar]El gen que codifica la maltasa se sitúa en el cromosoma 7 del cariotipo y es la siguiente secuencia de aminoácidos:
M-D-L-H-L-H-L-P-A-S-S-S-T-V-S-I-L-T-S-F-T-N-K-S-A-M-I-V-Y-Y-C-V-I-F-F-D-L-F-S-S-R-G-I-S-I-K-L-P-E-N-P-N-P-Q-P-L-K-W-W-Q-S-E-I-I-Y-Q-V-Y-P-R-S-F-M-D-S-D-G-D-G-V-G-D-L-K-G-I-E-S-K-V-D-Y-I-A-D-L-G-A-G-A-V-W-L-S-P-I-Y-E-S-P-M-K-D-F-G-Y-D-I-S-N-F-T-N-I-D-P-I-F-G-T-M-D-D-F-L-S-L-A-T-K-L-R-E-N-G-L-K-L-V-M-D-F-V-P-N-H-S-S-D-L-H-V-W-F-N-L-S-V-H-R-Q-E-K-Y-E-D-F-Y-V-W-K-N-A-S-G-Y-D-S-E-E-N-P-I-V-P-N-N-W-V-S-F-F-G-G-S-A-W-E-W-N-E-I-R-E-Q-F-Y-F-H-Q-F-V-A-G-Q-P-D-L-N-F-E-N-P-E-V-L-H-E-M-L-E-I-M-K-F-W-L-D-M-G-V-D-G-F-R-M-D-T-V-P-S-M-F-E-D-Q-R-F-L-D-E-A-E-D-P-N-R-P-P-N-S-I-P-T-D-Y-S-Y-W-S-H-I-Y-T-Y-D-L-P-A-T-R-D-S-W-Q-I-S-E-N-Y-L-I-S-D-G-K-E-R-C-M-M-I-E-A-Y-V-G-S-L-D-K-L-F-L-Y-Y-G-T-K-T-K-PI-A-H-F-P-E-N-F-E-F-I-T-R-S-N-Q-V-D-F-N-M-L-I-L-L-L-P-G-S-A-V-T-Y-Y-G-E-E-I-G-M-T-N-T-N-L-T-W-E-E-T-V-D-P-P-G-C-Q-A-G-P-E-R-Y-Q-L-F-S-R-D-P-E-R-T-P-M-Q-W-N-Y-D-K-N-S-G-F-S-T-G-D-S-T-W-L-P-I-N-P-N-Y-K-E-L-N-V-Q-A-Q-N-ET-S-G-D-S-H-L-K-V-Y-K-Q-I-L-S-L-R-K-T-D-T-W-K-Y-G-SL-E-A-Y-A-L-E-N-G-Q-G-F-G-F-A-R-I-Y-K-G-S-G-F-I-V-L-A-N-Y-L-D-G-M-I-I-N-V-G-E-H-F-K-D-I-P-D-S-A-M-V-Y-T-T-S-V-N-F-Q-P-E-M-S-V-G-S-F-I-S-T-K-S-V-T-I-R-G-K-H-S-I-I-L-T-F-S-K-S-E-N-E-K-K-N-D-T-W-I-W-T-I-V-N-W-I-L-L-S-K-A-N-F-Y-L-F-T-I-I-S-D-L-N-N-G-H-I-V-K-P-V-W-N-L-Q-N-N-Y-N-T-V[10][11]
Esta secuencia de aminoácidos está expresada con el código de una letra, y cabe destacar que puede variar ligeramente en cada especie y cada tipo de enzima, así como por mutaciones que pueden o no inutilizar la enzima.
| Enzima | Gen que la codifica |
|---|---|
| Alfa-glucosidasa | GAA |
| Maltasa-glucoamilasa | MGAM |
| Sacarasa-isomaltasa | SI |
| Alfa-amilasa 1 | AMY1A |
Dos genes principales de α-glucosidasa (maltasa), sacarasa-isomaltasa (SI) y maltasa-glucoamilasa (MGAM), respectivamente, se expresan en el intestino delgado.
El gen MGAM codifica una glucosidasa terminal de almidón con una alta actividad. Esta es inhibida por los oligómeros productos de la hidrólisis del almidón por la α-amilasa.
SI es una glucosidasa terminal del almidón con baja actividad. Esta es inhibida por los productos oligoméricos de α-amilasa. Su actividad se ve amplificada de sobremanera por la actividad interna de α-amilasa.[2]
Mecanismo enzimático
[editar]
La insuficiencia de Α-Amilasa en el cuerpo humano es contrarrestada mediante la digestión del almidón por parte de maltasa de la mucosa.
Se sabe de 4 actividades distintas de la mucosa duodenal en las que interviene la maltasa. Dos de estas actividades de maltasa están asociadas con sacarasa-isomaltasa y las dos restantes son maltasa-glucoamilasas (maltasas II y III) sin características distintivas.[1]
Como se trata de una enzima perteneciente a la familia GH13, su mecanismo se basa en la escisión del enlace α-glucosídico mediante hidrólisis. La maltasa centra su actividad en escindir el enlace α-(1→4) entre las dos unidades de glucosa que conforman la maltosa. La tasa de hidrólisis está controlada por el sustrato (número de carbohidratos).

Regulación molecular
[editar]Se cree que la maltasa es sintetizada por las células de la membrana mucosa que recubre la pared duodenal.
La flexibilidad de las enzimas digestivas en respuesta a los cambios en el nivel de macronutrientes de la dieta es algo común en mamíferos y aves. Se ha demostrado que en roedores un cambio de dieta a una más alta en carbohidratos provoca un aumento del control transcripcional del SI intestinal y el MGAM en un periodo de seis a doce horas. Lo mismo sucedió en polluelos de gorriones domésticos. A su vez, un cambio inverso de regreso a las dietas habituales provoca la disminución del ARNm de SI y su actividad. No obstante, esta flexibilidad enzimática no es universal pues existen animales incapaces de adaptarse de tal forma.[12]
Información reciente sugiere que la señal provocada por la existencia de glucosa en la mucosa duodenal está asociada directamente con la expresión de los genes de sacarasa-isomaltasa (SI) y maltasa-glucoamilasa (MGAM) mediante un código de histonas como la acetilación y las dimetilaciones/trimetil, y que las estructuras promotoras de los genes MGAM y SI son similares. Las regulaciones de estas expresiones génicas están relacionadas tanto con la cantidad como con la calidad de los carbohidratos alimentados. Por lo tanto, un cambio en la expresión génica viene acompañado de cambios drásticos en la cromatina provocados por modificaciones de las histonas, y en consecuencia influye directamente en la regulación de la transcripción de los genes.
Se sabe que la histona H3 es la responsable de accionar el promotor y las regiones transcritas de SI y MGAM, y que las acetilaciones de las histonas H3 y H4, así como el control general de la síntesis de aminoácidos y los factores de transcripción de las regiones transcritas de los genes SI y MGAM aumentan en presencia de una dieta rica en almidón.[2]
Las modulaciones de los mensajes de MGAM y SI están mediadas por acetilaciones y metilaciones de histonas del promotor y las regiones transcritas. Asimismo, la lisina y la arginina son modificadas postraduccionalmente en relación con los distintos estados de transcripción inducidos por la presencia de almidones.
Fructosa
[editar]La fructosa modula activamente los mensajes de SI.
Almidón
[editar]Los almidones modulan la expresión de los mensajes de MGAM y SI.
Estos hallazgos indican que la regulación de los genes intestinales mediada por carbohidratos involucra dos mecanismos: uno relacionado con la cantidad de carbohidratos en la dieta, y otro específicamente provocado por la fructosa.[2]
Distribución en los tejidos
[editar]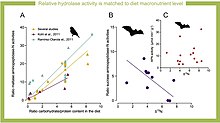

Tanto la maltasa glucoamilasa (MGAM) como la sacarosa isomaltasa (SI) actúan de forma conjunta en el borde de cepillo de las microvellosidades del epitelio del intestino delgado. La localización de dichas enzimas en estos tejidos intestinales se debe al paso continuado de nutrientes y de carbohidratos sobre los cuales pueden actuar y catalizar su escisión mediante la hidrólisis del enlace glicosídico.[13]
Presencia en animales
[editar]Los macronutrientes presentes en la dieta de los animales regulan la presencia de maltasa. Asimismo, los mamíferos voladores tienen una menor presencia menos presencia de estas enzimas en el intestino delgado ya que este tiene una mayor permeabilidad a los solutos orgánicos hidrofílicos, como la glucosa, que se absorben pasivamente. Esto explica la mayor presencia de microvellosidades y de tight-junctions tanto en mamíferos voladores como en aves y la peor adaptación del mecanismo de regulación moléculas de enzimas con un cambio de dieta.[12]
También se encuentra en plantas, bacterias, vertebrados y levaduras. La familia Desmodontinae parece ser el único vertebrado que no muestra actividad intestinal de maltasa.[14]
Historia
[editar]La historia del descubrimiento de la maltasa está ligada al uso de la maltosa. Este se remonta al 1806, cuando debido al "Decreto de Berlín" impuesto por Napoleón Bonaparte provocó un bloqueo continental de azúcar a los países invadidos que derivó en el inicio de la búsqueda de fuentes de azúcar alternativas.[15] Esto conllevó el descubrimiento de la remolacha azucarera como fuente alterna de azúcar y más tarde el descubrimiento de la α-amilasa como enzima de digestión del almidón en el año 1833 por Payen.
En 1856, Claude Bernard descubrió la actividad de la sacarasa en el intestino delgado, denominada por aquel entonces como invertasa.[16] Encontró dicha actividad en una gran diversidad de mamíferos, incluidos los Homo sapiens sapiens. En el siglo XX, el grupo de Borkström en Suecia concluyó que la concentración luminal de invertasa era pequeña en comparación con la de α-amilasa.[15]
En el 1880 la actividad de la maltasa intestinal fue descubierta y discernida de la amilasa G.T. Brown puesto que la amilasa hidrolizaba almidón en maltosa, y la maltasa hidrolizaba maltosa en glucosa.[17]
Para la década del 1960, los avances en bioquímica habilitaron a Dahlqvist y Semenza para fraccionar y caracterizar 2 estructuras peptídicas principales en las que encontraban intrínsecas 4 enzimas responsables de la actividad de la maltasa. Además, un péptido de maltasa era activo en la hidrólisis de la sacarosa y substratos derivados de la isomaltasa. La estructura peptídica restante fue denominada como maltasa-glucoamilasa.[16]
Gracias a los avances actuales es posible la observación bioquímica y por tanto la comprensión de estas fracciones de maltasa. Entre 1992[18] y 1998,[19] gracias a la clonación y secuenciación de las enzimas hidrolasas de almidón de la mucosa se confirmó que hay 2 actividades de la maltasa expresadas por la maltasa-glucoamilasa y dos por la sacarosa-glucoamilasa.
La actividad científica actual se centra en detallar la especificidad estructural de esta enzima, la modulación de la actividad, la selección de substrato y patologías relacionadas con la escasez o absencia de actividad de la maltasa.
Aplicaciones industriales
[editar]Fermentación
[editar]La enzima maltasa juega un rol crucial en la fermentación, proceso mediante el cual la levadura consume los azúcares que se encuentran presentes en la masa y que proceden de la degradación del almidón de la harina: glucosa, sacarosa, fructosa y maltosa. El último disacárido en consumirse es la maltosa, y esto supone un problema pues el proceso de la fabricación del pan se hace más lento y menos eficiente en cuanto a las cualidades físicas de la materia.
Por ello, la compañía Brist-Brocades desarrolló un método para solucionar el problema del empleo tardío de la maltosa. En primer lugar, confirmaron que para que la levadura pudiese metabolizar la maltosa, es imprescindible que las enzimas maltosa permeasa y la maltasa estén activas. La primera enzima se encarga de utilizar maltosa del medio e introducirla en la levadura, y la segunda, degrada la maltosa. Cuando hay niveles altos de glucosa, se produce un bloqueo en las regiones promotoras, que son las que se encargan de la expresión génica de los genes que sintetizan ambas enzimas, en el caso de la maltasa, el gen GAA. Este bloqueo se solucionó mediante la modificación genética de las levaduras para que consumieran primero la maltosa: la levadura transgénica conocida como levadura MAL. El resultado obtenido fue muy positivo: se aumentó la producción de gas carbónico y la capacidad fermentativa, por lo que, de esta manera se redujo el tiempo empleado en el proceso.
La levadura MAL fue la primera levadura transgénica en obtener el permiso de comercialización.
En un futuro próximo, la necesidad de este tipo de producciones enzimáticas sobre los productos lácteos aumentará de forma significativa, debido a la necesidad de productos lácteos de gran valor nutricional en el país para superar la malnutrición y la obesidad y cambiar hacia alimentos bajos en grasa y saludables.[20]

Industria cervecera
[editar]El primer paso para la producción de la cerveza es el malteado, donde los almidones se transforman en maltosa y dextrinas. La maltosa, se trata de un azúcar utilizable para la fermentación alcohólica, y las dextrinas están relacionadas con el sabor.
El papel de la enzima digestiva maltasa-glucoamilasa es catalizar la hidrólisis del almidón. Tiene la capacidad de descomposición del almidón en glucosa, y posteriormente en pequeñas unidades de glucosa.
Esta enzima es una fuente eficaz de fermentación, y se utiliza tanto para elaborar cerveza, como para elaborar sake.
Deficiencia de la maltasa
[editar]La importancia clínica de la maltasa e isomaltasa se debe a su crucial actividad en la digestión de los almidones de los alimentos y de glucosa libre absorbible. Enfermedades relacionadas con la maltasa son la enfermedad de Pompe, el Alzheimer y la intolerancia a la enzima; todas debidas a su deficiencia. Los resultados sobre la falta de disacáridos relacionados con la deficiencia de actividad enzimática de la maltasa son reportados por numerosos laboratorios clínicos mediante el uso de ensayos de Dahlqvist modificados.
La enfermedad de Pompe
[editar]
En 1932, el patólogo holandés Johannes Cassinus Pompe realizó una autopsia a una niña de 7 meses que había muerto sin causa conocida. Al realizarla, pudo observar una acumulación masiva de glucógeno en sus tejidos corporales. Más tarde descubrió que la causa de la aglutinación del glucógeno era la deficiencia de la enzima α-glucosidasa ácida o maltasa ácida cuya función es hidrolizar el glucógeno produciendo glucosa dentro del lisosoma celular. Fue de esta manera que se identificó el primer caso de lo que se conoce hoy como la enfermedad de Pompe.
Esta anomalía afecta gravemente al metabolismo normal del glucógeno, un polisacárido indispensable ya que se trata de la fuente de energía de los animales. Tras su hidrólisis se obtiene maltosa, isomaltosa y glucosa. Esta última molécula es la molécula básica para la vida, y su insuficiencia acarrea numerosos problemas. La incapacidad para hidrolizar el glucógeno resulta en el almacenamiento de glucógeno en los lisosomas.
La deficiencia de la maltasa es una anomalía que está determinada genéticamente y causadas por mutaciones en el gen GAA. La enfermedad de Pompe se hereda de forma autosómica recesiva, lo que quiere decir que los progenitores son portadores de mutaciones en el gen GAA, pero no sufren la enfermedad.
La enfermedad de Pompe afecta a 1 de cada 40.000 nacimientos vivos en todo el mundo. Por ende es considerada una enfermedad rara.
Durante mucho tiempo, esta enfermedad fue considerada como incurable, pero la aprobación de la terapia de sustitución enzimática[21] (TSE) ha abierto la investigación de nuevas curas para la enfermedad. Gracias a este desarrollo, la enfermedad de Pompe se ha sumado a los pocos trastornos por almacenamiento lisosómico, para los que el tratamiento ya es una realidad.
Intolerancia a la maltasa
[editar]La intolerancia a la maltasa, que requiere la ausencia simultánea de 4 enzimas autónomas suele ir acompañada por la intolerancia de la sacarosa e isomaltosa.[22] La intolerancia a la maltasa causa un problema de malabsorción intestinal, ya que el organismo se encuentra incapaz de absorber determinados nutrientes sin las proteínas catalíticas necesarias.
Según un estudio realizado por la Unidad de Gastroenterología de la Facultad de Medicina Clínica de Santiago de Chile, la malabsorción intestinal de los hidratos de carbono es causada por una carencia de las enzimas encargadas de metabolizar los azúcares, que puede ser o bien por una falta de este tipo de enzimas o bien por una disminución de la actividad de las mismas. Por lo tanto, los individuos que tengan un mal funcionamiento de la maltasa, al igual que de otras enzimas vitales tales como la amilasa o la lactasa, tendrán una mayor probabilidad de desarrollar un síndrome de malabsorción de hidratos de carbono.
Alzheimer
[editar]Se ha demostrado que proporciones reducidas del gen AMY1A en el cerebro aumentan el riesgo de padecer Alzheimer.[23] De esta manera, se cree que la alfa-amilasa 1 está relacionada con esta afección. La enzima alfa-amilasa 1 se encuentra principalmente en la saliva, donde descompone los polisacáridos de los alimentos, como el almidón. La actividad de la enzima corresponde al número de copias del gen de la alfa-amilasa salival (AMYA1), que varía mucho entre los individuos. Además, esta enzima se encuentra en otros órganos, como los pulmones, el corazón, los ovarios o el intestino, y ha sido recientemente descubierto en el hipocampo.
Así mismo, se ha demostrado la presencia de glucógeno en la sinapsis neuronal, por lo tanto la enzima alfa-amilasa juega un papel importante en la degradación del glucógeno sináptico, y consecuentemente en la formación de memorias. A pesar de que esta idea aún no esté confirmada, se encontró apoyo para la hipótesis cuando se observó una reducción, o incluso pérdida completa de la inmunoreactividad de la α-amilasa sináptica del hipocampo en pacientes con demencia de Alzheimer.
Véase también
[editar]Referencias
[editar]- ↑ a b Nichols, Buford L.; Quezada‐Calvillo, Roberto; Ao, Zihua; Hamaker, Bruce R; Marini, Juan; Jahoor, Farook (2008-03). «Contribution of Mucosal Maltase‐Glucoamylase to Mouse Small Intestinal Starch α‐Glucogenesis and Total Glucose Metabolism». The FASEB Journal 22 (S1). ISSN 0892-6638. doi:10.1096/fasebj.22.1_supplement.686.2. Consultado el 12 de octubre de 2022.
- ↑ a b c d Goda, Toshinao; Honma, Kazue (2018-06). «Molecular Regulations of Mucosal Maltase Expressions». Journal of Pediatric Gastroenterology & Nutrition (en inglés) 66 (3): S14-S17. ISSN 0277-2116. doi:10.1097/MPG.0000000000001980. Consultado el 11 de octubre de 2022.
- ↑ Nichols, Buford L.; Avery, Stephen; Sen, Partha; Swallow, Dallas M.; Hahn, Dagmar; Sterchi, Erwin (4 de febrero de 2003). «The maltase-glucoamylase gene: Common ancestry to sucrase-isomaltase with complementary starch digestion activities». Proceedings of the National Academy of Sciences (en inglés) 100 (3): 1432-1437. ISSN 0027-8424. PMC 298790. PMID 12547908. doi:10.1073/pnas.0237170100. Consultado el 30 de octubre de 2022.
- ↑ Stedman, Thomas Lathrop (2000). Stedman's medical dictionary. (27th ed edición). Lippincott Williams & Wilkins. ISBN 0-683-40007-X. OCLC 42772946. Consultado el 30 de octubre de 2022.
- ↑ Otto, B.; Wright, N. (1 de septiembre de 1994). «Trefoil peptides. Coming up clover». Current biology: CB 4 (9): 835-838. ISSN 0960-9822. PMID 7820556. doi:10.1016/s0960-9822(00)00186-x. Consultado el 30 de octubre de 2022.
- ↑ «PDB101: Global Health: Diabetes Mellitus: Drugs: Alpha glucosidase inhibitors: Alpha glucosidase». RCSB: PDB-101. Consultado el 29 de octubre de 2022.
- ↑ Raquel Parada Puig. «Alfa amilasa: características, estructura, funciones». Lifeder. Consultado el 25 de octubre de 2022.
- ↑ Danielsen, E M; Sjöström, H; Norén, O (15 de febrero de 1983). «Biosynthesis of intestinal microvillar proteins. Pulse-chase labelling studies on maltase-glucoamylase, aminopeptidase A and dipeptidyl peptidase IV». Biochemical Journal 210 (2): 389-393. ISSN 0306-3283. doi:10.1042/bj2100389. Consultado el 30 de octubre de 2022.
- ↑ Needleman, R. (1991-09). «Control of maltase synthesis in yeast». Molecular Microbiology 5 (9): 2079-2084. ISSN 0950-382X. doi:10.1111/j.1365-2958.1991.tb02136.x. Consultado el 30 de octubre de 2022.
- ↑ Bujnicki, Janusz (15 de agosto de 2021). «Faculty Opinions recommendation of Highly accurate protein structure prediction with AlphaFold.». Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. Consultado el 24 de octubre de 2022.
- ↑ Citovsky, Vitaly (6 de mayo de 2022). «Faculty Opinions recommendation of AlphaFold Protein Structure Database: massively expanding the structural coverage of protein-sequence space with high-accuracy models.». Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. Consultado el 24 de octubre de 2022.
- ↑ a b Karasov, William H.; Caviedes-Vidal, Enrique (2021-03). «Adaptation of intestinal epithelial hydrolysis and absorption of dietary carbohydrate and protein in mammals and birds». Comparative Biochemistry and Physiology Part A: Molecular & Integrative Physiology (en inglés) 253: 110860. doi:10.1016/j.cbpa.2020.110860. Consultado el 30 de octubre de 2022.
- ↑ Sim, Lyann; Quezada-Calvillo, Roberto; Sterchi, Erwin E.; Nichols, Buford L.; Rose, David R. (18 de enero de 2008). «Human Intestinal Maltase–Glucoamylase: Crystal Structure of the N-Terminal Catalytic Subunit and Basis of Inhibition and Substrate Specificity». Journal of Molecular Biology (en inglés) 375 (3): 782-792. ISSN 0022-2836. doi:10.1016/j.jmb.2007.10.069. Consultado el 30 de octubre de 2022.
- ↑ Schondube, Jorge E.; Herrera-M, L. Gerardo; Martínez del Rio, Carlos (2001-01). «Diet and the evolution of digestion and renal function in phyllostomid bats». Zoology 104 (1): 59-73. ISSN 0944-2006. doi:10.1078/0944-2006-00007. Consultado el 12 de octubre de 2022.
- ↑ a b Lentze, Michael J. (2018-06). «The History of Maltose-active Disaccharidases». Journal of Pediatric Gastroenterology and Nutrition (en inglés estadounidense) 66: S4. ISSN 0277-2116. doi:10.1097/MPG.0000000000001960. Consultado el 12 de octubre de 2022.
- ↑ a b Nichols, Buford L.; Baker, Susan S.; Quezada-Calvillo, Roberto (2018-06). «Metabolic Impacts of Maltase Deficiencies». Journal of Pediatric Gastroenterology & Nutrition (en inglés) 66 (3): S24-S29. ISSN 0277-2116. doi:10.1097/MPG.0000000000001955. Consultado el 7 de octubre de 2022.
- ↑ Brown, Horace Tabberer; Heron, John; Roberts, William (1 de enero de 1880). «IV. Some observations upon the hydrolytic ferments of the pancreas and small intestine». Proceedings of the Royal Society of London 30 (200-205): 393-407. doi:10.1098/rspl.1879.0137. Consultado el 10 de octubre de 2022.
- ↑ Chantret, I; Lacasa, M; Chevalier, G; Ruf, J; Islam, I; Mantei, N; Edwards, Y; Swallow, D et al. (1 de agosto de 1992). «Sequence of the complete cDNA and the 5′ structure of the human sucrase-isomaltase gene. Possible homology with a yeast glucoamylase». Biochemical Journal (en inglés) 285 (3): 915-923. ISSN 0264-6021. PMC 1132882. PMID 1353958. doi:10.1042/bj2850915. Consultado el 10 de octubre de 2022.
- ↑ Nichols, Buford L.; Eldering, Joyce; Avery, Stephen; Hahn, Dagmar; Quaroni, Andrea; Sterchi, Erwin (1998-01). «Human Small Intestinal Maltase-glucoamylase cDNA Cloning». Journal of Biological Chemistry (en inglés) 273 (5): 3076-3081. doi:10.1074/jbc.273.5.3076. Consultado el 10 de octubre de 2022.
- ↑ «Use of Enzimes on Dairy Products».
- ↑ van der Ploeg, Ans T.; Reuser, Arnold J. J. (11 de octubre de 2008). «Pompe's disease». Lancet (London, England) 372 (9646): 1342-1353. ISSN 1474-547X. PMID 18929906. doi:10.1016/S0140-6736(08)61555-X. Consultado el 12 de octubre de 2022.
- ↑ Dahlqvist, Arne (1 de marzo de 1962). «SPECIFICITY OF THE HUMAN INTESTINAL DISACCHARIDASES AND IMPLICATIONS FOR HEREDITARY DISACCHARIDE INTOLERANCE*». Journal of Clinical Investigation (en inglés) 41 (3): 463-470. ISSN 0021-9738. PMC 290939. PMID 13883118. doi:10.1172/JCI104499. Consultado el 10 de octubre de 2022.
- ↑ Byman, Elin; Nägga, Katarina; Gustavsson, Anna-Märta; Andersson-Assarsson, Johanna; Hansson, Oskar; Sonestedt, Emily; Wennström, Malin; The Netherlands Brain Bank (21 de noviembre de 2020). «Alpha-amylase 1A copy number variants and the association with memory performance and Alzheimer’s dementia». Alzheimer's Research & Therapy 12 (1): 158. ISSN 1758-9193. PMC 7680592. PMID 33220711. doi:10.1186/s13195-020-00726-y. Consultado el 15 de octubre de 2022.
Bibliografía
[editar]- Citovsky, Vitaly (6 de mayo de 2022). «Faculty Opinions recommendation of AlphaFold Protein Structure Database: massively expanding the structural coverage of protein-sequence space with high-accuracy models.». Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. Consultado el 10 de octubre de 2022.
- Maxwell, Anthony (28 de julio de 2021). «Faculty Opinions recommendation of Highly accurate protein structure prediction with AlphaFold.». Faculty Opinions – Post-Publication Peer Review of the Biomedical Literature. Consultado el 10 de octubre de 2022.

