Electroforesis proteica

La electroforesis proteica es un método de separación de proteínas mediante la aplicación de un campo eléctrico.
La electroforesis es un proceso en el que un gradiente de potencial produce el transporte de las partículas cargadas. En el transporte electroforético, a la fuerza del campo se opone la resistencia viscosa del medio, lo que produce, cuando se igualan, una velocidad constante de las partículas.
Así pues, la movilidad electroforética de una molécula depende directamente de su carga e inversamente del coeficiente de rozamiento, que a su vez depende directamente del tamaño y la forma de la molécula y la viscosidad del medio. La migración de las partículas depende del pH del medio en que estén, ya que su carga neta depende del pH. En la electroforesis, el amortiguador ejerce dos funciones: por un lado, lleva la carga eléctrica y, por otro, determina la carga neta de las moléculas que se separan y, por ende, la dirección de la migración electroforética. La fuerza iónica del amortiguador determina la anchura de la nube iónica que rodea las moléculas cargadas. Cuanto mayor es la fuerza iónica, más estrechas son las bandas de separación. Sin embargo, mucha fuerza iónica produce un calentamiento mayor y tal vez la desnaturalización de las sustancias que van a separarse, por lo que hay que llegar a una solución de compromiso. Y también del mismo .
Existen diferentes tipos en función del tipo de separación empleado: electroforesis de zona (separación en función de la carga), isoelectroenfoque y separación por tamaño en tamiz molecular (también aplicable a ácidos nucleicos).
Técnicas[editar]
Electroforesis de zona: las proteínas son moléculas anfóteras: su carga neta depende del pH del medio. Normalmente, la separación electroforética de proteínas se hace a pH alcalino, en el que la mayoría de las proteínas presentan una carga global negativa. También, se puede trabajar a pH ácidos, pero no demasiado bajos, ya que las proteínas precipitan en medio ácido (básicamente se usa en la detección de variantes de la hemoglobina).
Como medio de soporte se puede usar (de más antiguo a más reciente): papel, acetato de celulosa, agarosa, poliacrilamida y electroforesis capilar. La muestra cuyas proteínas se quieren separar se inserta en un medio de soporte y se aplica una diferencia de potencial durante un tiempo determinado para separar las proteínas. Cada proteína migrará más o menos en función de su carga (que también determina hacia qué polo se dirigirá la proteína, ánodo (+) o cátodo (-) y su tamaño. A mayor carga y menor tamaño, más velocidad de migración.
Isoelectroenfoque: en lugar de separar las proteínas en función de su carga a un pH dado, se separan en función de su punto isoeléctrico (pI): el pI es el pH en el que la carga neta de la proteína es nula, y depende de la composición aminoacídica de la proteína. Se crea un gradiente de pH mediante anfolitos (estabilizan el pH a lo largo del gel). Cada proteína migrará hasta alcanzar su pI, punto en el cual precipitará al acumularse (de ahí el nombre, isoelectroenfoque).
También se suelen utilizar acoplados a zimogramas si deseamos determinar el pI de una enzima o en electroforesis bidimensional como primera dimensión.
Separación por tamaño: permite separar proteínas y ácidos nucleicos. En el caso de las proteínas, deben ser tratadas con SDS (sodio dodecil sulfato) para que su carga sea negativa y todas migren hacia el ánodo (no es necesario hacer eso con los ácidos nucleicos, ya que tienen carga negativa); la separación se hace en medios de soporte en el que se ha creado un tamiz molecular (matriz), que hace que las proteínas más pequeñas corran más que las más grandes.
Visualización[editar]
Una vez separadas las proteínas, deben fijarse y teñirse para poder ser visualizadas. Hay diferentes protocolos en función de lo que se quiere estudiar: fijación por calor o química y tinción en caso de estudios no específicos (proteinograma y electroforesis Hb, por ejemplo) o fijación mediante anticuerpos previa a la tinción en caso de estudiar proteínas específicas (inmunofijación).
Por lo general para la tinción de geles de poliacrilamida se utiliza tinción con plata, tinción de Coomassie o tinción fluorescente. Dependiendo del fin de nuestro análisis. La tinción con plata no es utilizada si se desean hacer análisis por espectrometría de masas (MS) pero es más sensible que la tinción con Azul de Coomassie, por otro lado, la tinción fluorescente es muy sensible y compatible con MS pero los costos de tinción son elevados.
Una tinción sensible y barata es la denominada "Blue Silver" o Coomassie G250.[1] Además, esta tinción es compatible con MS. Está compuesta por azul de coomassie diluido en una solución coloidal y se utiliza agua destilada para destiñir el gel de poliacrilamida.
Aplicaciones[editar]
Pueden analizarse las proteínas contenidas en diferentes líquidos biológicos: sangre, plasma (el líquido sanguíneo sin células), suero (plasma sin fibrinógeno), orina, LCR, líquido sinovial, saliva, lágrimas. Así como alimentos, especialmente lácteos y cereales.
Este tipo de análisis electroforético tiene aplicaciones en investigación y en clínica, tanto humana como animal. Además es una técnica muy empleada para el análisis de proteínas alimentarias y últimamente se está empleando para realizar genotipado y detección de OMG (organismos modificados genéticamente).
Valoración del proteinograma[editar]
Hay que tener presente que el proteinograma proporciona la información correspondiente a las proteínas mayoritarias de cada banda y que, por tanto, la información que parece aportar sobre una proteína específica puede estar distorsionada en presencia de concentraciones aumentadas de las otras proteínas que comparten la zona de migración. La valoración cuantitativa de cada una de las principales proteínas plasmáticas se realizan por otros procedimientos analíticos.
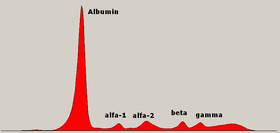
En el proteinograma realizado por electroforesis capilar se definen las siguientes bandas:
Banda de la prealbúmina[editar]
Es una banda difusa, por delante de la albúmina,[2] poco visible y que está constituida por dos proteínas con un gran interés para la valoración nutricional como son la prealbúmina y la proteína fijadora del retinol.[3] Por sus bajas concentraciones, se requieren técnicas inmunológicas para valorar su cuantía.
Banda de la albúmina[editar]
Es la banda más importante del proteinograma, representando más del 50% del mismo en condiciones normales. Está integrada por la proteína más abundante del plasma.[4] Su interés se centra en las hepatopatías (cirrosis) y nefropatías (síndrome nefrótico), donde está disminuida. Se eleva en situaciones de deshidratación y por prolongación del tiempo del torniquete a la hora de la extracción de la muestra. Una alteración cualitativa sin significado patológico que se puede presentar es la bisalbuminemia, que puede tener un origen genético o adquirido (generalmente debido a la toma de medicamentos).
Banda de la α1-globulina(alfa1)[editar]
Esta banda está formada por un conjunto de proteínas como la α1-antitripsina, la α1-glucoproteína, la transcortina,[5] la α1-lipoproteína,[6] y la α-fetoproteína, siendo las dos primeras las que representan el mayor porcentaje de la misma. Esta banda se eleva en los procesos inflamatorios agudos, por lo que las proteínas que la integran se denominan reactantes de fase aguda.
Mayor interés tiene su disminución, ya que indica un déficit de α1-antitripsina, sobre todo en la deficiencia homocigota (PiZZ) en donde la concentración plasmática de esta proteína está por debajo del 10-15% de su concentración en los sujetos sanos (MM). En ausencia de proceso inflamatorio agudo, los sujetos homocigotos SS y los heterocigotos MS, MZ y SZ tienen alrededor del 50% de las concentraciones encontradas en los sujetos con el fenotipo MM.
Banda de la α2-globulina(alfa2)[editar]
Las tres principales proteínas que la integran son la α2-macroglobulina,[7] la haptoglobina[8] y la ceruloplasmina.[9] Se comportan también como reactantes de la fase aguda positivos; la α2-macroglobulina tiene una función de antiproteasa sérica; la haptoglobina se une a la hemoglobina en los procesos hemolíticos intravasculares donde está disminuida y la ceruloplasmina tiene una misión transportadora del cobre, siendo sus concentraciones bajas en la enfermedad de Wilson.
Bandas de las β-globulinas(beta)[editar]
Las proteínas más importantes son la transferrina,[10] la hemopexina,[11] la β-lipoproteína y el c3 nativo.
La transferrina se eleva en situaciones de déficit de hierro y desciende en el síndrome nefrótico y las hepatopatías; la hemopexina desciende en las anemias hemolíticas y se eleva en las reacciones agudas; el c3 desciende en el LES activo y se eleva como reactante de fase aguda positivo.
En esta banda pueden aparecer otras extras correspondientes a la hemoglobina de los sueros hemolizados, el fibrinógeno[12] (cuando el proteinograma se realiza con plasma) y las gammapatías monoclonales, sobre todo IgA.
Banda de la γ-globulina(gamma)[editar]
Está formada por las tres principales inmunoglobulinas:[13] IgG,[14] IgA[15] e IgM;[16] en ausencia de una gammapatía monoclonal, su estimación electroforética solamente aportará información sobre la concentración sérica, sobre todo de la IgG. Su elevación puede ser policlonal, donde todas las inmunoglobulinas se elevan dando lugar a una curva simétrica, o bien monoclonal donde aparece un pico correspondiente a la síntesis de una sola cadena de inmunoglobulinas. La elevación policlonal de la IgA[15] da lugar a lo que se llama puente βδ-globulina.
Las elevaciones policlonales suelen deberse a una activación de la inmunidad humoral, inducida por múltiples mecanismos patológicos, como las infecciones, las enfermedades autoinmunes, las hepatopatías y los procesos inflamatorios agudos y crónicos.
Las elevaciones monoclonales se deben, sobre todo, al mieloma múltiple,[17] la enfermedad de Waldeströn, la enfermedad de las cadenas pesadas y, con mucha frecuencia, a las denominadas gammapatías monoclonales de significado incierto.
Véase también[editar]
Referencias[editar]
- ↑ Candiano, G., Bruschi, M., Musante, L., Santucci, L., Ghiggeri, G.M., Carnemolla, B., Orecchia, P., Zardi, L. and Righetti, P.G. (2004). «Blue silver: A very sensitive colloidal Coomassie G-250 staining for proteome analysis.». Electrophoresis, 25 (9): 1327 - 1333.
- ↑ «Albúmina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 7 de octubre de 2017. Consultado el 8 de octubre de 2017. - ↑ «Vitamina A»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 12 de septiembre de 2017. Consultado el 8 de octubre de 2017. - ↑ «Plasma (sangre)»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 17 de julio de 2017. Consultado el 8 de octubre de 2017. - ↑ «Transcortina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 4 de noviembre de 2015. Consultado el 8 de octubre de 2017. - ↑ «Alfalipoproteina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 25 de febrero de 2014. Consultado el 8 de octubre de 2017. - ↑ «Alfa-2 macroglobulina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 16 de julio de 2017. Consultado el 8 de octubre de 2017. - ↑ «Haptoglobina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 17 de febrero de 2016. Consultado el 8 de octubre de 2017. - ↑ «Ceruloplasmina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 26 de abril de 2017. Consultado el 8 de octubre de 2017. - ↑ «Transferrina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 20 de enero de 2017. Consultado el 8 de octubre de 2017. - ↑ «Hemopexina»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 29 de agosto de 2013. Consultado el 8 de octubre de 2017. - ↑ «Fibrinógeno»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 15 de mayo de 2017. Consultado el 8 de octubre de 2017. - ↑ «Anticuerpo»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 25 de septiembre de 2017. Consultado el 8 de octubre de 2017. - ↑ «Inmunoglobulina G»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 4 de mayo de 2017. Consultado el 8 de octubre de 2017. - ↑ a b «Inmunoglobulina A»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 28 de noviembre de 2016. Consultado el 8 de octubre de 2017. - ↑ «Inmunoglobulina M»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 29 de marzo de 2017. Consultado el 8 de octubre de 2017. - ↑ «Mieloma múltiple»
|url=incorrecta con autorreferencia (ayuda). Wikipedia, la enciclopedia libre. 24 de mayo de 2017. Consultado el 8 de octubre de 2017.
Enlaces externos[editar]
- Laboratorio Espinosa: Electroforesis de proteínas
- BIOtk - Análisis de proteinograma electroforético: BIOtk (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última).
