Receptor de tipo Toll

Los receptores tipo Toll (o Toll-like receptor TLRs) constituyen una familia de proteínas que forman parte del sistema inmunitario innato. Estos receptores son transmembranosos y reconocen patrones moleculares expresados por un amplio espectro de agentes infecciosos, y estimulan una variedad de respuestas inflamatorias. Además, la señalización mediada por los TLRs en las células presentadoras de antígeno (CPAs) representa una parte importante en el vínculo entre la respuesta inmune innata y la adaptativa. Proteínas de esta familia se encuentran en plantas, invertebrados, y vertebrados, ya que desempeñan un papel clave en unas vías de señalización bien conservadas.[1] Después de las defensinas, pueden ser el componente del sistema inmune más antiguo. Existen 11 TLRs en el ser humano, cada TLRs está codificado por un gen diferente.
Estructura
[editar]Todos los receptores de esta familia son glicoproteínas transmembranas de tipo I.[2] Comparten un dominio en el citoplasma llamado dominio TIR (Toll/IL-1 Receptor) que inicia la vía de señalización por la cual los receptores afectan la actividad celular en la presencia de sus ligandos.[3] Este dominio se une a otros dominios TIR en proteínas adaptoras como MyD88, permitiendo que la señal siga transmitiéndose.[3] Sus dominios N-terminales extracelulares (compuestos de 550 a 980 aminoácidos) contienen una secuencia repetida rica en leucina (LRRs) y una secuencia flanqueadora rica en cisteína.[4] A pesar de esta homología, los dominios extracelulares son más variables que los citoplásmicos entre distintos TLRs, reflejando las diferencias entre sus ligandos.[1] Sin embargo, estos receptores no tienen tanta diversidad como los receptores de linfocitos T ni como los anticuerpos, porque se codifican en genes somáticos no recombinados. Después de unirse a sus ligandos, los TLRs forman heterodímeros o homodímeros, un paso esencial en su activación.[1]
Historia de su descubrimiento
[editar]En 1980 unos investigadores alemanes vieron que en ausencia de una proteína las moscas no desarrollaban su eje dorsoventral. Por cómo quedaron anatómicamente las moscas con mutación, se llamó a la proteína Toll (del alemán coloquial "increíble"). Vieron que se trataba de un receptor transmembranal de señales; y más tarde se fueron descubriendo moléculas relacionadas, que fueron llamadas en conjunto receptores tipo Toll.
Más tarde, en 1996, Hoffman y Lemaitre vieron que la mutación en Toll además de influir en el desarrollo de la mosca también la hacía susceptible a la infección letal por el hongo Aspergillus fumigatus, que no supone ningún peligro para moscas de tipo silvestre.
En 1997, Medzhitov y Janeway descubrieron lo que hoy es el TLR-4, una proteína que vieron que activaba la expresión de genes de inmunoreacción al transfectarse a una línea experimental de células humanas.
En 1998 ya se demostró que los TLR son parte de la inmunidad innata en humanos y ratones, y hasta la fecha se han descubierto 11 en humanos y 12 en ratones.
Función
[editar]Los TLRs reconocen y se unen a patrones moleculares asociados a patógenos (PAMPs, por su nombre en inglés) que son grupos de características químicas comunes a ciertos tipos de patógenos. Patrones detectados por TLRs incluyen lipopolisacárido, un compuesto encontrado en las superficies de bacterias Gram negativas, y ARN de doble cadena, que es parte integral de los ciclos de vida de muchos virus. Estos PAMPs generalmente son importantes para la supervivencia del patógeno, así que se conservan bien. La activación de estos receptores induce respuestas inflamatorias en leucocitos de linaje mieloide, señalando por la vía NF-κB. TLRs activados también aumentan la producción de moléculas co-estimuladoras, como la CD80, CD86 y CD40. Estas proteínas, expresadas en la superficie de células presentadoras de antígeno, son necesarias para la activación de linfocitos T por células dentríticas y macrófagos ya mostrando antígenos en sus moléculas MHC tipo II.[3]
Señalización
[editar]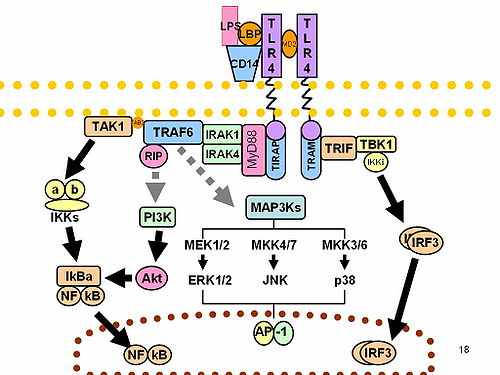
Hay dos vías distintas de señalización asociadas con los TLRs: la que requiere la proteína adaptora MyD88, y la MyD88-independiente. Todos los TLRs menos TLR-3 señalan por la vía dependiente de MyD88. MyD88 activa NF-κB, induciendo la producción de citocinas inflamatorias como IL-1, IL-8, TNF-alfa, e IL-12.[1] MyD88 tiene un dominio TIF que se une al dominio TIF en el receptor activado. Al unirse al TLR, MyD88 recluta cinasas de la familia IRAK a través de su dominio muerte (death domain (DD)).[1] Las proteínas IRAK interactúan con TRAF-6, permitiendo que TRAF-6 active al MAPKKK TAK1. TAK1 fosforila el complejo IKK, activándolo. El complejo IKK activado destruye el inhibidor de NF-κB (IκB) dejando NF-κB libre que puede entrar el núcleo y aumentar la transcripción de ciertos genes.[3] También, TAK1 puede estimular la vía AP-1 mediante activación de la vía de cinasas MAP.[2] MyD88 unido al TLR-7 o TLR-9 también puede asociarse con IRF-7 e inducir la producción de interferón (IFN) de tipo I.[2]
La vía MyD88-independiente, empleada en la señalización de TLR-3 y TLR-4, involucra el adaptador TRIF, que también contiene un dominio TIR.[4][3] Como MyD88, TRIF activa TRAF-6 y así promueve la producción del factor de transcripción NFκB.[4] TRIF también activa IRF-3, que induce la transcripción de IFN-β. Señalización a través de TLR-3, que reconoce ARN de doble cadena, siempre produce IFN, una proteína específicamente dirigida contra los virus, ya que TLR-3 requiere TRIF para señalar.[5]
TLRs conocidos
[editar]| Receptor | Ligando(s) | [6] Ubicación de ligando | [6] Adaptador(es) | Ubicación del receptor | Tipos de células[6] | |
|---|---|---|---|---|---|---|
| TLR 1 | lipopéptidos triaciles | bacterias | MyD88/MAL | superficie celular | ||
| TLR 2 | glicolípidos | bacterias | MyD88/MAL | superficie celular |
| |
| lipopeptidos múltiples | bacterias | |||||
| lipoproteínas múltiples | bacterias | |||||
| ácido lipoteicoico | bacterias Gram-positivas | |||||
| HSP70 | células de hospedero | |||||
| zymosan (Beta-glucan) | fungi | |||||
| varios otros | ||||||
| TLR 3 | ARN de doble candena, poly I:C | virus | TRIF | compartimento celular |
| |
| TLR 4 | lipopolisacárido | bacterias Gram-negativas | MyD88/MAL/TRIF/TRAM | superficie celular |
| |
| proteínas de choque térmico | bacterias y células de hospedero | |||||
| fibrinógeno | células de hospedero | |||||
| fragmentos de heparán sulfato | células de hospedero | |||||
| fragmentos de ácido hialurónico | células de hospedero | |||||
| níquel | ||||||
| varias opioides | ||||||
| TLR 5 | flagelina | bacterias | MyD88 | superficie celular |
| |
| TLR 6 | lipopéptidos diaciles | Mycoplasma | MyD88/MAL | superficie celular |
| |
| TLR 7 | imidazoquinolina | compuestas sintéticas pequeñas | MyD88 | compartimento celular |
| |
| loxoribina (un análogo de guanosina) | ||||||
| bropirimina | ||||||
| ARN de cadena simple | ||||||
| TLR 8 | compuestas sintéticas pequeñas; ARN de cadena simple | MyD88 | compartimento celular |
| ||
| TLR 9 | oligonucleótido ADN CpG no metilado | bacterias | MyD88 | compartimento celular |
| |
| TLR 10 | Hongos | desconocido | MyD88 | superficie celular |
| |
| TLR 11 | Ácido hialurónico | Toxoplasma gondii | MyD88 | compartimento celular[9] | ||
| TLR 12 | desconocido | desconocido | ? | |||
| TLR 13 | rRNA[11] | virus | MyD88, TAK-1 | compartimento celular |
Descubrimiento
[editar]Primero, el receptor Toll se identificó en Drosophila como una proteína de señalización en vías importantes al desarrollo del embrión. Luego, su rol en la respuesta inmune innata se descubrió cuando se notó que el receptor Toll era necesaria para luchar contra infecciones fúngicas.[1] Unos receptores relacionados que respondieron a otros tipos de patógenos se encontraron posteriormente en Drosophila, y luego se descubrieron sus homólogos en mamíferos.
Mutaciones en cáncer
[editar]En 2013, DUlak y colaboradores publicaron un estudio genómico donde se trataban 149 muestras de tejido tumoral de adenocarcinoma de esófago. El gen correspondiente al que codifica para el receptor de tipo toll TRL4 se encontró mutado hasta en el 6% de las muestras sujetas a análisis, los autores sugieren que estas mutaciones activan la respuesta inmune innata ante la exposición de patógenos como Helicobacter pylori. Polimorfismos en este gen como p.D379, p.F487 y p.E439 sugieren una incapacidad para la correcta formación del complejo TLR4/MD-2 y potencialmente podría conducir la progresión tumoral. [12]
Véase también
[editar]Referencias
[editar]- ↑ a b c d e f Moreno, C.; Sánchez-Ibarrola, A. (2003). «Receptores tipo Toll: bases moleculares de la relación entre respuestas innatas y adaptativas del sistema inmunitario» (pdf). Rev Med Univ Navarra 47 (3): 29-33. Archivado desde el original el 17 de enero de 2012. Consultado el 16 de mayo de 2012.
- ↑ a b c Kumar, Himanshu; Kawai, Taro; Akira, Shizuo (2009). «Toll-like receptors and innate immunity» (pdf). Biochemical and Biophysical Research Communications (388): 621-625. doi:10.1016/j.bbrc.2009.08.062. Consultado el 17 de mayo de 2012.
- ↑ a b c d e Murphy, Kenneth; Paul Travers, Mark Walport (2008). Janeway's Immunobiology (en inglés) (7a edición). Garland. ISBN 0815341237.
- ↑ a b c Guzmán Masias, Karol (2010). La inmunidad innata y los receptores tipo Toll (TLR'S). Sistema de Revisiones en Investigación Veterinaria de San Marcos.
- ↑ Yamamoto, Masahiro; Sato, Shintaro; Hemmil, Hiroaki; Hoshino, Katsuaki; Kaisho, Tsuneyasu; Sanjo, Hideki; Takeuchi, Osamu; Sugiyama, Masanaka; Okabe, Masaru; Takeda, Kiyoshi; Akira, Shizuo (2003). «Role of Adaptor TRIF in the MyD88-Independent Toll-Like Receptor Signaling Pathway» (html). Science 301 (5633): 640-643. doi:10.1126/science.1087262. Consultado el 17 de mayo de 2012.
- ↑ a b c Si no se especifica, ref es: Waltenbaugh C, Doan T, Melvold R, Viselli S (2008). Immunology. Lippincott's Illustrated reviews. Philadelphia: Wolters Kluwer Health/Lippincott Williams & Wilkins. p. 17. ISBN 0-7817-9543-5.
- ↑ a b c d Sallusto F, Lanzavecchia A (2002). «The instructive role of dendritic cells on T-cell responses». Arthritis Res. 4 Suppl 3: S127-32. PMID 12110131. doi:10.1186/ar567. Archivado desde el original el 15 de septiembre de 2019. Consultado el 17 de mayo de 2012.
- ↑ Gerondakis, Steve; Grumont RJ; Banerjee A (2007). «Regulating B-cell activation and survival in response to TLR signals». Immunology and Cell Biology 85 (6): 471-475. PMID 17637697. doi:10.1038/sj.icb.7100097. Consultado el 21 de agosto de 2011.
- ↑ Pifer R, Benson A, Sturge CR and Yarovinsky F (November 2010). "UNC93B1 is essential for TLR11 activation and IL-12 dependent host resistance to Toxoplasma Gondii". Journal of Biological Chemistry. doi 10.1074/jbc.M110.171025
- ↑ Mishra BB, Gundra UM, Teale JM (2008). «Expression and distribution of Toll-like receptors 11-13 in the brain during murine neurocysticercosis». Journal of Neuroinflammation 5: 53. PMC 2631477. PMID 19077284. doi:10.1186/1742-2094-5-53.
- ↑ Shi Z, Cai Z, Sanchez A, et al. (febrero de 2011). A novel Toll-like receptor that recognizes vesicular stomatitis virus 286 (6). pp. 4517-24. PMID 21131352. doi:10.1074/jbc.M110.159590.
- ↑ Dulak, A. M., Stojanov, P., Peng, S., Lawrence, M. S., = Fox, C., Stewart, C., Bandla, S., Imamura, Y., Schumacher, S. E., Shefler, E., McKenna, A., Cibulskis, K., Sivachenko, A., Carter, S. L., Saksena, G., Voet, D., Ramos, A. H., Auclair, D., Thompson, K., Sougnez, C., Onofrio, R. C., Guiducci, C., Beroukhim, R., Zhou, D., Lin, L., Lin, J., Reddy, R., Chang, A., Luketich, J. D., Pennathur, A., Ogino, S., Golub, T. R., Gabriel, S. B., Lander, E. S., Beer, D. G., Godfrey, T. E., Getz, G. & Bass, A. J. (2013). «Exome and whole genome sequencing of esophageal adenocarcinoma identifies recurrent driver events and mutational complexity.». Nature Genet. 45 (5).
