Gen superpuesto
Un gen superpuesto (OLG, por sus siglas en inglés)[1][2] es un gen cuya secuencia de nucleótidos expresable se superpone parcialmente con la secuencia de nucleótidos expresable de otro gen.[3] De esta forma, una secuencia de nucleótidos puede contribuir a la función de uno o más productos génicos . Los genes superpuestos están presentes y son una característica fundamental tanto en los genomas celulares como virales.[2] La definición actual de un gen superpuesto varía significativamente entre eucariotas, procariotas y virus.[2] En procariotas y virus, la superposición debe ser entre secuencias codificantes, pero no entre transcritos de ARNm, y se define cuando estas secuencias codificantes comparten uno o más nucleótidos en la misma cadena o en cadenas opuestas. En eucariotas, la superposición de genes casi siempre se define como superposición de transcritos de ARNm. Específicamente, una superposición de genes en eucariotas se da cuando al menos un nucleótido se comparte entre los límites de los transcritos primarios de ARNm de dos o más genes, de modo que una mutación de una base del ADN en cualquier punto de la región superpuesta afectaría los transcritos de todos los genes involucrados. Esta definición incluye tanto las regiones no traducidas (UTR) 5′ y 3′ como los intrones.
La sobreimpresión se refiere a un tipo de superposición en la que toda o parte de la secuencia de un gen se lee en un marco de lectura alterno de otro gen en el mismo locus.[4] Se cree que los marcos de lectura abiertos (ORF, por sus siglas en inglés) alternos se crean mediante sustituciones de nucleótidos críticas dentro de un gen expresable preexistente, el cual se puede inducir para que exprese una proteína nueva conservando la función del gen original.[5] Se ha planteado la hipótesis de que la sobreimpresión es un mecanismo para el surgimiento <i id="mwKw">de novo</i> de nuevos genes a partir de secuencias existentes, ya sean genes más antiguos o regiones del genoma que antes no codificaban.[6] Se cree que la mayoría de los genes superpuestos, o genes cuyas secuencias de nucleótidos expresables se superponen parcialmente entre sí, evolucionaron en parte debido a este mecanismo, lo que sugiere que cada superposición se compone de un gen ancestral y un gen nuevo.[7] En consecuencia, también se cree que la sobreimpresión es una fuente de nuevas proteínas, ya que las proteínas de novo codificadas por estos nuevos genes normalmente carecen de homólogos remotos en las bases de datos.[8] Los genes sobreimpresos son características particularmente comunes de la organización genómica de los virus, y es probable que aumente en gran medida la cantidad de genes expresables potenciales a partir de un pequeño conjunto de información genética viral.[9] Es probable que la sobreimpresión sea responsable de la generación de numerosas proteínas nuevas por parte de los virus a lo largo de su historia evolutiva.
Clasificación[editar]
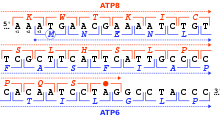
Los genes pueden superponerse de varias formas y pueden clasificarse por sus posiciones relativas entre sí.[3][11][12][13][14]
Referencias[editar]
- ↑ Nelson, Chase W (1 de octubre de 2020). «Dynamically evolving novel overlapping gene as a factor in the SARS-CoV-2 pandemic». eLife 9. PMC 7655111. PMID 33001029. doi:10.7554/eLife.59633. Consultado el 11 de noviembre de 2020.
- ↑ a b c Wright, Bradley W.; Molloy, Mark P.; Jaschke, Paul R. (5 de octubre de 2021). «Overlapping genes in natural and engineered genomes». Nature Reviews Genetics (en inglés) 23 (3): 154-168. ISSN 1471-0064. PMC 8490965. PMID 34611352. doi:10.1038/s41576-021-00417-w.
- ↑ a b Y. Fukuda, M. Tomita et T. Washio (1999). «Comparative study of overlapping genes in the genomes of Mycoplasma genitalium and Mycoplasma pneumoniae». Nucleic Acids Res. 27 (8): 1847-1853. PMC 148392. PMID 10101192. doi:10.1093/nar/27.8.1847.
- ↑ Pavesi, Angelo (26 de mayo de 2021). «Origin, Evolution and Stability of Overlapping Genes in Viruses: A Systematic Review». Genes (en inglés) 12 (6): 809. ISSN 2073-4425. PMC 8227390. PMID 34073395. doi:10.3390/genes12060809.
- ↑ Normark, Staffan; Bergström, Sven; Edlund, Thomas; Grundström, Thomas; Jaurin, Bengtake; Lindberg, Frederik P.; Olsson, Olof (December 1983). «Overlapping Genes». Annual Review of Genetics 17 (1): 499-525. ISSN 0066-4197. PMID 6198955. doi:10.1146/annurev.ge.17.120183.002435.
- ↑ Keese, PK; Gibbs, A (15 de octubre de 1992). «Origins of genes: "big bang" or continuous creation?». Proceedings of the National Academy of Sciences of the United States of America 89 (20): 9489-93. Bibcode:1992PNAS...89.9489K. PMC 50157. PMID 1329098. doi:10.1073/pnas.89.20.9489.
- ↑ Keese, P. K.; Gibbs, A. (15 de octubre de 1992). «Origins of genes: "big bang" or continuous creation?». Proceedings of the National Academy of Sciences 89 (20): 9489-9493. Bibcode:1992PNAS...89.9489K. ISSN 0027-8424. PMC 50157. PMID 1329098. doi:10.1073/pnas.89.20.9489.
- ↑ Gibbs, Adrian; Keese, Paul K. (19 de octubre de 1995), «In search of the origins of viral genes», Molecular Basis of Virus Evolution (Cambridge University Press): 76-90, ISBN 9780521455336, doi:10.1017/cbo9780511661686.008, consultado el 3 de diciembre de 2021.
- ↑ Pavesi, Angelo; Magiorkinis, Gkikas; Karlin, David G.; Wilke, Claus O. (15 de agosto de 2013). «Viral Proteins Originated De Novo by Overprinting Can Be Identified by Codon Usage: Application to the "Gene Nursery" of Deltaretroviruses». PLOS Computational Biology 9 (8): e1003162. Bibcode:2013PLSCB...9E3162P. PMC 3744397. PMID 23966842. doi:10.1371/journal.pcbi.1003162.
- ↑ Anderson S, Bankier AT, Barrell BG, de Bruijn MH, Coulson AR, Drouin J, Eperon IC, Nierlich DP, Roe BA, Sanger F, Schreier PH, Smith AJ, Staden R, Young IG (April 1981). «Sequence and organization of the human mitochondrial genome». Nature 290 (5806): 457-465. Bibcode:1981Natur.290..457A. PMID 7219534. doi:10.1038/290457a0.
- ↑ Fukuda, Yoko; Nakayama, Yoichi; Tomita, Masaru (December 2003). «On dynamics of overlapping genes in bacterial genomes». Gene 323: 181-187. PMID 14659892. doi:10.1016/j.gene.2003.09.021.
- ↑ Johnson Z, Chisholm S (2004). «Properties of overlapping genes are conserved across microbial genomes». Genome Res 14 (11): 2268-72. PMC 525685. PMID 15520290. doi:10.1101/gr.2433104.
- ↑ Normark S.; Bergstrom S.; Edlund T.; Grundstrom T.; Jaurin B.; Lindberg F.P.; Olsson O. (1983). «Overlapping genes». Annual Review of Genetics 17: 499-525. PMID 6198955. doi:10.1146/annurev.ge.17.120183.002435.
- ↑ Rogozin, Igor B.; Spiridonov, Alexey N.; Sorokin, Alexander V.; Wolf, Yuri I.; Jordan, I.King; Tatusov, Roman L.; Koonin, Eugene V. (May 2002). «Purifying and directional selection in overlapping prokaryotic genes». Trends in Genetics 18 (5): 228-232. PMID 12047938. doi:10.1016/S0168-9525(02)02649-5.
