Diferencia entre revisiones de «Centrómero»
| Línea 293: | Línea 293: | ||
===ADN satélite=== |
===ADN satélite=== |
||
En ''[[Drosophila melanogaster]]'' la secuencia AATAACATAG está repetida en tándem en regiones próximas al centrómero. Dado que que estas secuencias cortas de 10 pb no son representativas del genoma de una especie, suele suceder que su contenido en G+C es diferente al contenido en G+C del resto del [[genoma]]. Esto hace que cuando el ADN de una especie eucarionte se centrifuga en gradiente de densidad de cloruro de cesio, aparezca una banda principal que contiene la mayor parte del ADN de la especie y una banda satélite (minoritaria) que está formada por una secuencia corta de ADN repetida en tándem. El ADN satélite en algunas especies tiene mayor densidad que el ADN principal (mayor contenido en G+C) y en otras especies tiene menor densidad y, por tanto, menor contenido en G+C que el ADN principal. Cuando el ADN satélite de ratón se marca radiactivamente y se realiza una [[hibridación in situ|hibridación ''in situ'']] con el ADN de cromosomas metafásicos mitóticos, se observa que el marcaje radiactivo (hibridación) se produce en regiones próximas al centrómero. El ADN satélite también se denomina α-satélite. |
En ''[[Drosophila melanogaster]]'' la secuencia AATAACATAG está repetida en tándem en regiones próximas al centrómero. Dado que que estas secuencias cortas de 10 pb no son representativas del genoma de una especie, suele suceder que su contenido en G+C es diferente al contenido en G+C del resto del [[genoma]]. Esto hace que cuando el ADN de una especie eucarionte se centrifuga en gradiente de densidad de cloruro de cesio, aparezca una banda principal que contiene la mayor parte del ADN de la especie y una banda satélite (minoritaria) que está formada por una secuencia corta de ADN repetida en tándem. El ADN satélite en algunas especies tiene mayor densidad que el ADN principal (mayor contenido en G+C) y en otras especies tiene menor densidad y, por tanto, menor contenido en G+C que el ADN principal. Cuando el ADN satélite de ratón se marca radiactivamente y se realiza una [[hibridación in situ|hibridación ''in situ'']] con el ADN de cromosomas metafásicos mitóticos, se observa que el marcaje radiactivo (hibridación) se produce en regiones próximas al centrómero. El ADN satélite también se denomina α-satélite. |
||
A pesar de que en la mayor parte de los casos no se han detectado motivos especialmente definidos, una secuencia candidata en el ADN satélite tendría que estar repetida en cientos de kilobases, ya que éste es el tamaño mínimo de un centrómero funcional que se ha identificado en diferentes organismos. Por ejemplo, en ''Drosophila'' la unidad minima necesaria de repeticiones en tándem es de 420 kb, en maíz se necesitan 500 kb y en humanos la unidad mínima consiste de 100 kb.<ref name=Henikoff2001>{{citation |
|||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
}}</ref> Por tanto en animales y plantas se presentan centrómeros "regionales", frente a los centrómeros "puntuales" que se encuentran en levaduras. |
|||
Una característica interesante de la mayor parte del ADN satélite es su unidad de longitud, ya que aunque no se han detectado secuencias con motivos conservados, la longitud de la unidad que se repite es muy parecida entre organismos. En primates, por ejemplo, la unidad básica que se repite tiene 171 pb, en el pez ''[[Sparus aurata]]'' la repetición centromérica tiene 186 pb, en el insecto ''[[Chironomus pallidivittatus]]'' tiene 155 pb, en ''[[Arabidopsis thaliana]]'' y en el maíz tiene 180 pb, y en el arroz 168 pb.<ref name=Henikoff2001/> La estrecha variación en longitud de la unidad que se repite en el ADN satélite corresponde aproximadamente al rango de longitud del ADN que rodea a un [[nucleosoma]], y repeticiones más largas, como las que se encuentran en los centrómeros del cerdo (340 pb) corresponden aproximadamente a la longitud de dos nucleosomas. Hay excepciones notorias, como las repeticiones pentaméricas que se encuentran en ''[[Drosophila melanogaster]]''. En general, la selección de la longitud de un nucleosoma podría limitar la evolución del ADN centromérico, de acuerdo con su función estructural en el [[genoma]]. |
|||
El ADN satélite forma parte de la [[heterocromatina]] constitutiva, que permanece condensada en casi todas las células somáticas de un organismo. Estas regiones son pobres en [[gen]]es y pueden inducir la represión de la [[expresión génica]] de las regiones adyacentes de manera [[epigenética]]. Este fenómeno se denomina "variegación por efecto de la posición" (PEV, por ''Position Effect Variegation'').<ref name=Perrod2003>{{citation |
|||
| last1 = Perrod | first1 = S. |
|||
| last2 = Gasser | first2 = S.M. |
|||
| year = 2003 |
|||
| title = Long-range silencing and position effects at telomeres and centromeres: parallels and differences |
|||
| journal = Cellular and Molecular Life Sciences (CMLS) |
|||
| volume = 60 |
|||
| issue = 11 |
|||
| pages = 2303–2318 |
|||
| doi = 10.1007/s00018-003-3246-x |
|||
| url = http://www.fmi.ch/html/research/research_groups/epigenetics/susan_gasser/gasser/images/perrod%20review.pdf |
|||
| ⚫ | |||
Sin embargo, a pesar de que el ADN satélite se encuentra en los centrómeros humanos nativos, se han detectado centrómeros humanos generados ''de novo'' (neocentrómeros) que carecen de α-satélite u otras repeticiones en tándem,<ref name=Lo2001>{{citation |
Sin embargo, a pesar de que el ADN satélite se encuentra en los centrómeros humanos nativos, se han detectado centrómeros humanos generados ''de novo'' (neocentrómeros) que carecen de α-satélite u otras repeticiones en tándem,<ref name=Lo2001>{{citation |
||
| Línea 313: | Línea 343: | ||
===ADN CEN y evolución=== |
===ADN CEN y evolución=== |
||
El proceso de segregación cromosómica está sometida a una fuerte presión evolutiva, dado que la pérdida o ganancia de cromosomas (una situación denominada [[aneuploidía]]) puede producir importantes alteraciones fenotípicas, como el síndrome de Down en humanos, por ejemplo. Por ello, la maquinaria encargada de distribuir los cromosomas entre las células hijas durante la [[división celular]] presenta una gran sofisticación y está sometida a un estricto control (véase [[checkpoint de mitosis]]). Los centrómeros son las regiones cromosómicas sobre las que se ensamblan los [[cinetocoro]]s, que son las estructuras proteicas responsables del anclaje de los cromosomas al [[huso mitótico]], y por ello la zona responsable del movimiento cromosómico y su regulación. Sin embargo, a pesar de ello las secuencias de ADN que definen las secuencias centroméricas están muy poco conservadas y evolucionan rápidamente incluso entre [[especie]]s muy relacionadas. Esto no quiere decir que las secuencias de ADN del centrómero son hipermutables, sino que las variantes de las secuencias se fijan por expansión y contracción, y pueden aparecer ''de novo'' en sitios nuevos (neocentrómeros). Estos cambios en el ADN centromérico tiene lugar debido a la existencia de diferentes procesos [[mutación|mutacionales]], como errores en la [[replicación del ADN]], intercambio desequilibrado, transposición y excisión. Las proteínas centroméricas también presentan signos inesperados de una rápida evolución. Por todo ello, se ha sugerido que en el núcleo de esta rápida evolución existe un conflicto genético en funcionamiento.<ref name=Henikoff2001> |
El proceso de segregación cromosómica está sometida a una fuerte presión evolutiva, dado que la pérdida o ganancia de cromosomas (una situación denominada [[aneuploidía]]) puede producir importantes alteraciones fenotípicas, como el síndrome de Down en humanos, por ejemplo. Por ello, la maquinaria encargada de distribuir los cromosomas entre las células hijas durante la [[división celular]] presenta una gran sofisticación y está sometida a un estricto control (véase [[checkpoint de mitosis]]). Los centrómeros son las regiones cromosómicas sobre las que se ensamblan los [[cinetocoro]]s, que son las estructuras proteicas responsables del anclaje de los cromosomas al [[huso mitótico]], y por ello la zona responsable del movimiento cromosómico y su regulación. Sin embargo, a pesar de ello las secuencias de ADN que definen las secuencias centroméricas están muy poco conservadas y evolucionan rápidamente incluso entre [[especie]]s muy relacionadas. Esto no quiere decir que las secuencias de ADN del centrómero son hipermutables, sino que las variantes de las secuencias se fijan por expansión y contracción, y pueden aparecer ''de novo'' en sitios nuevos (neocentrómeros). Estos cambios en el ADN centromérico tiene lugar debido a la existencia de diferentes procesos [[mutación|mutacionales]], como errores en la [[replicación del ADN]], intercambio desequilibrado, transposición y excisión. Las proteínas centroméricas también presentan signos inesperados de una rápida evolución. Por todo ello, se ha sugerido que en el núcleo de esta rápida evolución existe un conflicto genético en funcionamiento.<ref name=Henikoff2001/> |
||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
| ⚫ | |||
==Referencias== |
==Referencias== |
||
Revisión del 08:59 5 dic 2008
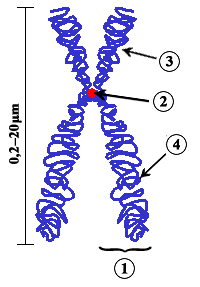
En Biología, el centrómero es la constricción primaria que, utilizando tinciones tradicionales, aparece menos teñida que el resto del cromosoma. Es la zona por la que el cromosoma interacciona con las fibras del huso acromático desde profase hasta anafase, tanto en mitosis como en meiosis, y es responsable de realizar y regular los movimientos cromosómicos que tienen lugar durante estas fases. Las estructuras centroméricas que interaccionan con las fibras del huso se denominan cinetocoros. Además del ADN centromérico, que consta fundamentalmente de heterocromatina constitutiva, en la estructura del centrómero también intervienen proteínas centroméricas.
En la levadura de gemación (Saccharomyces cerevisiae) el ADN centromérico consta únicamente de 125 pb y está conservado entre los diferentes cromosomas.[1] Sin embargo, el ADN centromérico en metazoos puede constar de megabases, y no contiene secuencias consenso fácilmente identificables (ver la revisión de Choo en 1997[2]). A pesar de las diferencias entre el ADN centromérico de levaduras y metazoos, el cinetocoro se ensambla en ambos casos sobre nucleosomas centroméricos que contienen una forma especializada de histona H3 (Cse4p en levaduras [3] o su homólogo CENP-A en metazoos).
El centrómero tiene un comportamiento diferente durante la anafase mitótica y la anafase-I de la meiosis, de manera que durante la anafase mitótica las cromátidas hermanas se separan a polos opuestos (segregación anfitélica) mientras que en la anafase-I de la meiosis lo que se separa a polos opuestos son los cromosomas homólogos completos, cada uno constituido por dos cromátidas (segregación sintélica).
El centrómero en Saccharomyces cerevisiae
Los análisis llevados a cabo en S. cerevisiae para aislar el ADN centromérico (ADN CEN) de todos sus cromosomas, han establecido que en todos los centrómeros de levaduras estudiados existen tres regiones muy conservadas:
- La región I (CDEI), secuencia de 8-9 bp (PuTCACPuTG) que se encuentra en el límite izquierdo de todos los centrómeros.
- La región II (CDEII), secuencia de 76-86 pb rica en pares AT (87-95%) que separa las regiones I y II.
- La región III (CDEIII), secuencia de 25 bp altamente conservada en el extremo derecho de los centrómeros. Presenta una secuencia palindrómica TGTTT(T/A)TGNTTTCCGAAANNNAAAA.
En la cromatina en su forma nativa, el ADN CEN es un segmento de 220-250 pb protegido de la acción de las nucleasas y flanqueado en ambos extremos por sitios hipersensibles al corte y un conjunto de nucleosomas altamente organizados, que contienen la histona especializada Cse4p (el homólogo en levaduras de CENP-A) en lugar de H3.
Las mutaciones en las regiones I y II reducen pero no inactivan la función del centrómero, mientras que las que ocurren en la región III lo inactivan completamente. En los mutantes por deleción de la región CDEII, la función centromérica puede restablecerse casi completamente insertando una secuencia de ADN compuesta sólo de A-T al azar y de tamaño equivalente.[4] Por tanto, en esta región los factores críticos para una segregación cromosómica correcta son el contenido en A-T y la longitud del ADN, más que la secuencia de nucleótidos, quizás porque influyen en la conformación del ADN centromérico.
CDEIII es la zona de unión del complejo proteico CBF3 (por Centromere Binding Factor 3), compuesto por las proteínas Ndc10p, Cep3p, Ctf13p, y Skp1p [5] (esta proteína - también denominada p23SKP1- es además parte del complejo SCULCDC4 implicado en los procesos de degradación mediados por ubiquitina necesarios para la progresión a través del ciclo celular). En ausencia de CBF3, el cinetocoro no es funcional, tanto in vivo como in vitro,[6][7] y presentan alteración de la asociación con el centrómero de todas las proteínas del cinetocoro conocidas, incluida Cse4p.[8] Sin embargo, la unión de CBF3 al ADN centromérico in vivo no requiere Cse4p.[9] Por tanto, la unión específica de CBF3 a la región CDEIII participa en la definición de la localización del cinetocoro en levaduras. Otra proteína esencial para la viabilidad de las células de levaduras es CBF5p, que co-purifica con el complejo CBF3 en condiciones de baja astringencia.[10] Esta proteína también se une a microtúbulos in vitro, y parece ser importante en la transición G1/S del ciclo celular, ya que la eliminación de CBF5p bloquea la división celular antes de que ocurra la replicación del ADN.
Por otro lado, CDEI es el sitio de unión de un homodímero de Cbf1p.[11] Cbf1p no es esencial para la función del cinetocoro, pero induce el plegado del ADN[12] y por tanto puede contribuir a la estructura de nivel superior del centrómero. Cbf1p tiene similitud estructural e identidad de secuencia limitada a CENP-B, que se une al ADN centromérico de metazoos y también induce el plegado del ADN.[13]
Mif2p es una proteína esencial en levaduras, similar a CENP-C de metazoos, cuya eliminación produce fallos en la segregación cromosómica, retraso en mitosis y microtúbulos con morfología aberrante.[14] MIF2 interacciona genéticamente con tres de los genes que codifican proteínas centroméricas: CBF1, CBF3a y CBF3b. Aunque la unión de Mif2 al centrómero depende de Ndc10,[15] su localización en el ADN CEN está muy disminuida en mutantes para Cse4.[16] Mif2 presenta un dominio acídico y otro dominio rico en prolina, lo que se denomina un "gancho AT" (AT hook), un motivo que es común a las proteínas que se unen a secuencias de ADN ricas en AT (como las proteínas HMGI(Y) de mamíferos), lo que sugiere que Mif2 se une a la región CDEII.[17]
En S. cerevisiae, el ADN centromérico se replica en una etapa temprana de la fase S,[18] tal vez porque es necesario replicar el ADN centromérico para iniciar el ensamblaje de los cinetocoros hermanos.
El centrómero en la levadura de fisión
El ADN centromérico de Schizosaccharomyces pombe es considerablemente más complejo que el de S. cerevisiae y comparte algunas propiedades con los centrómeros regionales de los eucariotas superiores.[19] El centrómero de S. pombe está constituido por 40-100 kb de ADN organizado en distintos tipos de repeticiones específicas de los centrómeros (las repeticiones tipo K), que están a su vez organizadas en un gran repetición invertida.[20] Como ocurre en eucariotas superiores, la organización de los centrómeros en la levadura de fisión varía considerablemente entre diferentes cromosomas y entre cepas muy próximas.[21] El centro de la repetición invertida, el núcleo central, contiene una secuencia de 4-7 kb que es fundamental para la función centromérica. Las regiones centrales del centrómero de S. pombe se organizan en una estructura inusual, que depende de la existencia de un elemento dentro de las repeticiones tipo K (denominado enhancer centromérico) y que es fundamental para la función del centrómero.[22] Las regiones K y el núcleo central son las dianas de los mecanismos epigenéticos que afectan a la función del centrómero in vivo. Por otro lado, existe redundancia funcional tanto entre las repeticiones tipo K como en el núcleo central.
Centrómeros en metazoos
La determinación de los centrómeros de metazoos constituye una tarea difícil y esquiva. En animales y plantas, los centrómeros están incluidos en regiones de ADN satélite altamente repetido, que resulta difícil de analizar incluso con los métodos de mapeo más potentes.
ADN satélite
En Drosophila melanogaster la secuencia AATAACATAG está repetida en tándem en regiones próximas al centrómero. Dado que que estas secuencias cortas de 10 pb no son representativas del genoma de una especie, suele suceder que su contenido en G+C es diferente al contenido en G+C del resto del genoma. Esto hace que cuando el ADN de una especie eucarionte se centrifuga en gradiente de densidad de cloruro de cesio, aparezca una banda principal que contiene la mayor parte del ADN de la especie y una banda satélite (minoritaria) que está formada por una secuencia corta de ADN repetida en tándem. El ADN satélite en algunas especies tiene mayor densidad que el ADN principal (mayor contenido en G+C) y en otras especies tiene menor densidad y, por tanto, menor contenido en G+C que el ADN principal. Cuando el ADN satélite de ratón se marca radiactivamente y se realiza una hibridación in situ con el ADN de cromosomas metafásicos mitóticos, se observa que el marcaje radiactivo (hibridación) se produce en regiones próximas al centrómero. El ADN satélite también se denomina α-satélite.
A pesar de que en la mayor parte de los casos no se han detectado motivos especialmente definidos, una secuencia candidata en el ADN satélite tendría que estar repetida en cientos de kilobases, ya que éste es el tamaño mínimo de un centrómero funcional que se ha identificado en diferentes organismos. Por ejemplo, en Drosophila la unidad minima necesaria de repeticiones en tándem es de 420 kb, en maíz se necesitan 500 kb y en humanos la unidad mínima consiste de 100 kb.[23] Por tanto en animales y plantas se presentan centrómeros "regionales", frente a los centrómeros "puntuales" que se encuentran en levaduras.
Una característica interesante de la mayor parte del ADN satélite es su unidad de longitud, ya que aunque no se han detectado secuencias con motivos conservados, la longitud de la unidad que se repite es muy parecida entre organismos. En primates, por ejemplo, la unidad básica que se repite tiene 171 pb, en el pez Sparus aurata la repetición centromérica tiene 186 pb, en el insecto Chironomus pallidivittatus tiene 155 pb, en Arabidopsis thaliana y en el maíz tiene 180 pb, y en el arroz 168 pb.[23] La estrecha variación en longitud de la unidad que se repite en el ADN satélite corresponde aproximadamente al rango de longitud del ADN que rodea a un nucleosoma, y repeticiones más largas, como las que se encuentran en los centrómeros del cerdo (340 pb) corresponden aproximadamente a la longitud de dos nucleosomas. Hay excepciones notorias, como las repeticiones pentaméricas que se encuentran en Drosophila melanogaster. En general, la selección de la longitud de un nucleosoma podría limitar la evolución del ADN centromérico, de acuerdo con su función estructural en el genoma.
El ADN satélite forma parte de la heterocromatina constitutiva, que permanece condensada en casi todas las células somáticas de un organismo. Estas regiones son pobres en genes y pueden inducir la represión de la expresión génica de las regiones adyacentes de manera epigenética. Este fenómeno se denomina "variegación por efecto de la posición" (PEV, por Position Effect Variegation).[24]
Sin embargo, a pesar de que el ADN satélite se encuentra en los centrómeros humanos nativos, se han detectado centrómeros humanos generados de novo (neocentrómeros) que carecen de α-satélite u otras repeticiones en tándem,[25] lo que indica que el ADN satélite no es fundamental para definir un centrómero funcional.
ADN CEN y evolución
El proceso de segregación cromosómica está sometida a una fuerte presión evolutiva, dado que la pérdida o ganancia de cromosomas (una situación denominada aneuploidía) puede producir importantes alteraciones fenotípicas, como el síndrome de Down en humanos, por ejemplo. Por ello, la maquinaria encargada de distribuir los cromosomas entre las células hijas durante la división celular presenta una gran sofisticación y está sometida a un estricto control (véase checkpoint de mitosis). Los centrómeros son las regiones cromosómicas sobre las que se ensamblan los cinetocoros, que son las estructuras proteicas responsables del anclaje de los cromosomas al huso mitótico, y por ello la zona responsable del movimiento cromosómico y su regulación. Sin embargo, a pesar de ello las secuencias de ADN que definen las secuencias centroméricas están muy poco conservadas y evolucionan rápidamente incluso entre especies muy relacionadas. Esto no quiere decir que las secuencias de ADN del centrómero son hipermutables, sino que las variantes de las secuencias se fijan por expansión y contracción, y pueden aparecer de novo en sitios nuevos (neocentrómeros). Estos cambios en el ADN centromérico tiene lugar debido a la existencia de diferentes procesos mutacionales, como errores en la replicación del ADN, intercambio desequilibrado, transposición y excisión. Las proteínas centroméricas también presentan signos inesperados de una rápida evolución. Por todo ello, se ha sugerido que en el núcleo de esta rápida evolución existe un conflicto genético en funcionamiento.[23]
Referencias
- ↑ Fitzgerald-hayes, M.; Clarke, L.; Carbon, J. (1982), «Nucleotide sequence comparisons and functional analysis of yeast centromere DNAs», Cell 29 (1): 235-44, doi:10.1016/0092-8674(82)90108-8.
- ↑ Choo, K.H.A. (1997), The centromere.
- ↑ Meluh, P.B.; Yang, P.; Glowczewski, L.; Koshland, D.; Smith, M.M. (1998), «Cse 4 P is a Component of the Core Centromere of Saccharomyces Cerevisiae», Cell(Cambridge) 94 (5): 607-613.
- ↑ Gaudet, A.M.; Fitzgerald-hayes, M. (1990), The eukaryotic nucleus. Molecular biochemistry and macromolecular assemblies. Telford Press, Caldwell, New Jersey: 845-881.
- ↑ Lechner, J.; Carbon, J. (1991), «A 240 Kd Multisubunit Protein Complex, Cbf 3, is a Major Component of the Budding Yeast Centromere», Cell(Cambridge) 64 (4): 717-725.
- ↑ Goh, P.Y.; Kilmartin, J.V. (1993), «NDC10: a gene involved in chromosome segregation in Saccharomyces cerevisiae», J Cell Biol 121 (3): 503-12, doi:10.1083/jcb.121.3.503.
- ↑ Sorger, P.K.; Doheny, K.F.; Hieter, P.; Kopski, K.M.; Huffaker, T.C.; Hyman, A.A. (1995), «Two Genes Required for the Binding of an Essential Saccharomyces cerevisiae Kinetochore Complex to …», Proceedings of the National Academy of Sciences 92 (26): 12026-12030.
- ↑ Ortiz, J.; Stemmann, O.; Rank, S.; Lechner, J. (1999), «A putative protein complex consisting of Ctf19, Mcm21, and Okp1 represents a missing link in the …», Genes & Development 13 (9): 1140-1155, doi:10.1101/gad.13.9.1140.
- ↑ Measday, V.; Hailey, D.W.; Pot, I.; Givan, S.A.; Hyland, K.M.; Cagney, G.; Fields, S.; Davis, T.N. et al. (2002), «Ctf3p, the Mis6 budding yeast homolog, interacts with Mcm22p and Mcm16p at the yeast outer …», Genes & Development 16 (1): 101-113, doi:10.1101/gad.949302 .
- ↑ Jiang, W.; Middleton, K.; Yoon, H.J.; Fouquet, C.; Carbon, J. (1993), «An essential yeast protein, CBF5p, binds in vitro to centromeres and microtubules», Molecular and Cellular Biology 13 (8): 4884-4893.
- ↑ Mellor, J.; Jiang, W.; Funk, M.; Rathjen, J.; Barnes, C.A.; Hinz, T.; Hegemann, J.H.; Philippsen, P. (1990), «CPF1, a yeast protein which functions in centromeres and promoters», The EMBO Journal 9 (12): 4017.
- ↑ Niedenthal, Rainer K.; Sen-gupta, Mark; Andreas, Wilmen; Hegemann, Johannes H. (1993), «Cpf1 protein induced bending of yeast centromere DNA element I», Nucleic Acids Research 21 (20): 4726-4726, PMID 8233820, doi:10.1093/nar/21.20.4726.
- ↑ Tanaka, Y.; Nureki, O.; Kurumizaka, H.; Fukai, S.; Kawaguchi, S.; Ikuta, M.; Iwahara, J.; Okazaki, T. et al. (2001), «… of the CENP-B protein–DNA complex: the DNA-binding domains of CENP-B induce kinks in the CENP-B …», The EMBO Journal 20: 6612-6618, doi:10.1093/emboj/20.23.6612 .
- ↑ Brown, M.T. (1993), «MIF2 is required for mitotic spindle integrity during anaphase spindle elongation in Saccharomyces …», The Journal of Cell Biology 123 (2): 387-403.
- ↑ Meluh, P.B.; Koshland, D. (1997), «Budding yeast centromere composition and assembly as revealed by in vivo cross-linking», Genes & Development 11 (24): 3401-3412, doi:10.1101/gad.11.24.3401.
- ↑ Westermann, S.; Cheeseman, I.M.; Anderson, S.; Yates Iii, J.R.; Drubin, D.G.; Barnes, G. (2003), The Journal of Cell Biology 163 (2): 215 http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=2173538
|url=sin título (ayuda). - ↑ Meluh, P.B.; Koshland, D. (1995), «Evidence that the MIF2 gene of Saccharomyces cerevisiae encodes a centromere protein with homology …», Molecular Biology of the Cell 6 (7): 793-807.
- ↑ McCarroll, R.M.; Fangman, W.L. (1988), «Time of replication of yeast centromeres and telomeres», Cell(Cambridge) 54 (4): 505-513.
- ↑ Clarke, L.; Baum, M.; Marschall, L.G.; Ngan, V.K.; Steiner, N.C. (1993), «Structure and Function of Schizosaccharomyces pombe Centromeres», Cold Spring Harbor Symposia on Quantitative Biology 58: 687-687.
- ↑ Allshire, R.C. (1997), «Centromeres, checkpoints and chromatid cohesion», Current Opinion in Genetics & Development 7 (2): 264-273, doi:10.1016/S0959-437X(97)80137-2.
- ↑ Steiner, N.C.; Hahnenberger, K.M.; Clarke, L. (1993), «Centromeres of the fission yeast Schizosaccharomyces pombe are highly variable genetic loci», Molecular and Cellular Biology 13 (8): 4578-4587.
- ↑ Ngan, V.K.; Clarke, L. (1997), «The centromere enhancer mediates centromere activation in Schizosaccharomyces pombe», Molecular and Cellular Biology 17 (6): 3305-3314.
- ↑ a b c Henikoff, Steven; Ahmad, Kami; Malik, Harmit S. (2001), «The Centromere Paradox: Stable Inheritance with Rapidly Evolving DNA», Science 293 (5532): 1098-1102, PMID 11498581, doi:10.1126/science.1062939.
- ↑ Perrod, S.; Gasser, S.M. (2003), «Long-range silencing and position effects at telomeres and centromeres: parallels and differences», Cellular and Molecular Life Sciences (CMLS) 60 (11): 2303-2318, doi:10.1007/s00018-003-3246-x.
- ↑ Lo, A.W.I.; Craig, J.M.; Saffery, R.; Kalitsis, P.; Irvine, D.V.; Earle, E.; Magliano, D.J.; Choo, K.H.A. (2001), «A 330 kb CENP-A binding domain and altered replication timing at a human neocentromere», The EMBO Journal 20: 2087-2096, doi:10.1093/emboj/20.8.2087.
Bibliografía
- Alberts, Bruce, et al. Molecular Biology of the Cell, 4th ed. New York: Garland Publishing, 2000.
- Atherly, Alan G., Jack R. Girton, and John F. McDonald. The Science of Genetics. Philadelphia, PA: Saunders College Publishing, 1998.
- Cooper, D. N. Human Gene Mutation. Bios Scientific Publishers Ltd., 1997.
- Departamento de Genética. Facultad de Genética. Universidad Complutense de Madrid. Centrómero. [1]
