Replicación en círculo rodante

La replicación en círculo rodante es un proceso de replicación unidireccional de ácido nucleico que puede sintetizar rápidamente múltiples copias de las moléculas circulares de ADN o ARN tales como plásmidos, algunos transposones, genomas de virus de ADN monocatenario, viroides, virusoides y algunos virus de ADN bicatenario.
La replicación en círculo rodante ha encontrado una amplia utilidad en la investigación académica y biotecnología, y ha sido usada exitosamente para la amplificación de ADN a partir de una pequeña cantidad inicial.
Mecanismo de replicación de ADN[editar]
La replicación en círculo rodante de ADN es inducida por una proteína iniciadora codificada por el ADN del plásmido o del bacteriófago, la cual muesca una hebra circular de la molécula de ADN bicatenaria en un sitio llamado origen de doble cadena (DSO, double-strand origin). La proteína continúa unida al extremo 5' fosfato de la cadena muescada y el extremo libre 3' hidróxilo sirve como partidor para la síntesis de ADN por la ADN polimerasa III. Usando la hebra continua como modelo, la replicación ocurre alrededor de la molécula circular de ADN, desplazando la hebra muescada como una molécula de ADN monocatenario gracias a la helicasa PcrA (abreviación de plasmid copy reduced), enzima codificada por la célula huésped y que actúa en presencia de la proteína iniciadora de la replicación del plásmido.
Tras una continua repetición en tándem, la replicación produce copias múltiples en una molécula llamada concatémero. Estas copias lineales monocatenarias pueden ser convertidas en moléculas circulares bicatenarias a través de los siguientes procesos:
Primero, la proteína iniciadora hace una otra muesca al terminar la síntesis de la hebra principal. Luego la ARN polimerasa y la ADN polimerasa III replican el ADN de origen monocatenario (SSO, single-stranded origin) para hacerlo circular y bicatenario. La ADN polimerasa I remueve el partidor y lo reemplaza con ADN, y la ADN ligasa se une en los extremos para cerrar el ADN bicatenario circular.
Virología[editar]
Algunos virus replican su ADN en las células huésped por medio de la replicación en círculo rodante. Por ejemplo, el herpesvirus humano 6 expresa un conjunto de "genes tempranos" que se cree que están involucrados en este proceso.[1] Luego de formados los grandes concatémeros, estos son cortados por ribozimas entre las regiones del pac-1 y pac-2 del genoma del HHV-6 para la síntesis de nuevos viriones.[2] Otro virus que también hace uso de esta forma de replicación es el fago P22.
Amplificación por círculo rodante (ACR)[editar]
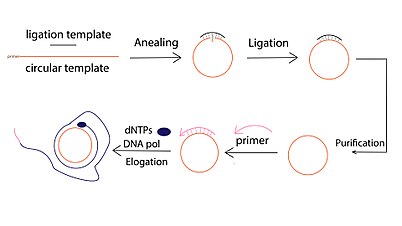
La técnica de amplificación por círculo rodante ha sido exitosamente utilizada para amplificación de ADN de cantidades muy pequeñas de material genético.[3] Esta técnica de amplificación se conoce como Rolling Circle Amplification (RCA) en inglés. Diferente de técnicas de amplificación de ADN convencionales como reacción en cadena de la polimerasa (PCR), la ACR es una técnica de amplificación de ácido nucleico isoterma donde la polimerasa añade nucleótidos solos a un cebador hibridado a una secuencia circular qué acaba generando una molécula de ADN monocatenaria con decenas o cientos de repeticiones de la secuencia complementaria a la secuencia circular.[4]
Hay cinco componentes importantes necesarios para la reacción de ACR:
- Una polimerasa de ADN
- Un buffer
- Un cebador o primer
- Una secuencia circular de ADN
- Nucleótidos trifosfato
Las polimerasas usadas para ACR comúnmente son Phi29, Bst, y Vent exo-ADN polimerasa para amplificación de AD, y T7 ARN polimerasa para amplificar ARN. La polimerasa Phi29 es la más procesiva de estas y por lo tanto la más usada comúnmente. Estas polimerasas son isotérmicas, y, a diferencia de la PCR, ACR se realiza a una temperatura constante entre temperatura ambiente y 65C.
La ACR tiene generalmente 3 partes:
- Circularización de la secuencia: Generalmente se empieza con una secuencia linear que se circulariza al hibridar con otra secuencia especifica. Esta secuencia puede entonces ser ligada para convertirse en la secuencia circular que será amplificada
- Amplificación inducida por cebador. un primer o cebador, o otra secuencia que hibridase con la secuencia circular puede ser amplificada por la polimerasa.
- Detección de los productos de la amplificación, lo cual puede hacerse de varias maneras. Una muy común es el uso de nucleótidos trifosfato unidos a fluorocromos, pero hay una gran variedad de técnicas.
La ACR produce una amplificación lineal, ya que la polimerasa da vueltas constantemente a cada secuencia circular, y sus productos complementarios crecen a velocidad constante. Para incrementar la cantidad de productos se han desarrollado varias técnicas. Una es la llamada ACR Ramificada o Hyperbranched RCA o HRCA en inglés, en la cual se añaden cebadores complementarios a los productos del ACR, que son a su vez amplificados incrementando exponencialmente la amplificación.[5] Otro ejemplo es la amplificación de círculo a círculo, o C2CA en inglés. En esta técnica se realiza un ACR, y los productos son digeridos con una enzima de restricción. Esto genera fragmentos lineales que pueden ser circularizados usando un oligonucleótido de restricción, lo cual general una gran cantidad de nuevas secuencias circulares que pueden ser usadas para otra ronda de ACR.[6]
Referencias[editar]
- ↑ Arbuckle, Jesse. «The molecular biology of human herpesvirus-6 latency and telomere integration». Microbes and infection 13 (8-9). PMC 3130849. PMID 21458587. doi:10.1016/j.micinf.2011.03.006.
- ↑ Cloning human herpes virus 6A genome into bacterial artificial chromosomes and study of DNA replication intermediates.
- ↑ Ali, M. Monsur; Li, Feng; Zhang, Zhiqing; Zhang, Kaixiang; Kang, Dong-Ku; Ankrum, James A.; Le, X. Chris; Zhao, Weian (2014). «Rolling circle amplification: a versatile tool for chemical biology, materials science and medicine». Chemical Society Reviews 43 (10): 3324-41. PMID 24643375. doi:10.1039/C3CS60439J.
- ↑ Ali, M. Monsur; Li, Feng; Zhang, Zhiqing; Zhang, Kaixiang; Kang, Dong-Ku; Ankrum, James A.; Le, X. Chris; Zhao, Weian (21 de mayo de 2014). «Rolling circle amplification: a versatile tool for chemical biology, materials science and medicine». Chemical Society Reviews 43 (10): 3324-3341. ISSN 1460-4744. PMID 24643375. doi:10.1039/c3cs60439j.
- ↑ Lizardi, Paul M.; Huang, Xiaohua; Zhu, Zhengrong; Bray-Ward, Patricia; Thomas, David C.; Ward, David C. (1998-07). «Mutation detection and single-molecule counting using isothermal rolling-circle amplification». Nature Genetics (en inglés) 19 (3): 225-232. ISSN 1546-1718. doi:10.1038/898.
- ↑ Dahl, Fredrik; Banér, Johan; Gullberg, Mats; Mendel-Hartvig, Maritha; Landegren, Ulf; Nilsson, Mats (30 de marzo de 2004). «Circle-to-circle amplification for precise and sensitive DNA analysis». Proceedings of the National Academy of Sciences (en inglés) 101 (13): 4548-4553. ISSN 0027-8424. PMID 15070755. doi:10.1073/pnas.0400834101.
Enlaces externos[editar]
- Sistemas de replicación del ADN con una pequeña cantidad de moléculas de ADN circular Genomes 2, T. Brown et al., en NCBI Books (en inglés)
- MicrobiologyBytes: Viroides y virusoides (en inglés)
