Nucleósido monofosfato quinasa
{{Ficha de enzima | #EC 2.7.4, fosfotransferasas siendo el aceptor un grupo fosfato |EC 2.7.4.4]])[1] es una enzima que cataliza la reacción química[2]
ATP + nucleósido fosfato ⇌ ADP + nucleósido difosfato
Así, los dos sustratos de esta enzima son ATP y nucleósido monofosfato, mientras que sus dos productos son ADP y nucleósido difosfato.[3][4]
Esta enzima pertenece a la familia de las transferasas, concretamente a las que transfieren grupos que contienen fósforo (fosfotransferasas) con un grupo fosfato como aceptor.[5] El nombre sistemático de esta clase de enzimas es ATP:nucleósido-fosfato fosfotransferasa. Esta enzima también se denomina NMP-quinasa o nucleósido-monofosfato quinasa.
Estructura
[editar]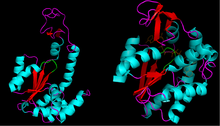
Se han resuelto varias estructuras cristalinas de esta clase de enzimas, revelando que comparten un dominio común de unión al ATP. Esta sección de la enzima se conoce comúnmente como bucle P,[7] en referencia a su interacción con los grupos fosforilo del ATP. Este dominio de unión también consiste en una lámina β flanqueada por hélices α.
El [bucle P] suele tener la secuencia de aminoácidos Gly-X-X-X-X-Gly-Lys.[8] Secuencias similares se encuentran en muchas otras proteínas de unión a nucleótidos.
Mecanismo
[editar]Interacción con iones metálicos
[editar]Para permitir la interacción con esta clase de enzimas, el ATP debe unirse primero a un ion metálico como el magnesio o el manganeso.[9] El ion metálico forma un complejo con el grupo fosforilo, así como con varias moléculas de agua.[10] Estas moléculas de agua forman entonces enlaces de hidrógeno con un residuo de aspartato conservado en la enzima.[11]
La interacción del ion metálico facilita la unión al mantener la molécula de ATP en una posición que permite la unión específica al sitio activo y al proporcionar puntos adicionales para la unión entre el sustrato y la enzima. Esto aumenta la energía de unión.
Cambios conformacionales
[editar]La unión del ATP hace que el bucle P se mueva, lo que a su vez hace que el dominio de la tapa baje y fije el ATP en su sitio[12][13] La unión del nucleósido monofosfato induce más cambios que hacen que la enzima sea catalíticamente capaz de facilitar una transferencia del grupo fosforilo del ATP al nucleósido monofosfato.[14]
La necesidad de estos cambios conformacionales evita la hidrólisis del ATP.
Este mecanismo enzimático es un ejemplo de catálisis por aproximación: la nucleósido-fosfato quinasa une los sustratos para juntarlos en la posición correcta para que se transfiera el grupo fosforilo.
Función biológica
[editar]Dominios catalíticos similares están presentes en varias proteínas, incluyendo:
- ATP sintasa
- Miosina y otras proteínas motoras moleculares
- Proteína G y otras proteínas implicadas en la transducción de señales
- Helicasas para desenrollar ADN y ARN
- Metabolismo de la pirimidina
Evolución
[editar]Cuando se realizó un árbol filogenético compuesto por miembros de la familia de las nucleósido-fosfato cinasas,[15] se demostró que estas enzimas habían divergido originalmente a partir de un ancestro común en variedades largas y cortas. Este primer cambio fue drástico: la estructura tridimensional del dominio de la tapa cambió significativamente.
Tras la evolución de las variedades larga y corta de las NMP-cinasas, cambios menores en las secuencias de aminoácidos dieron lugar a la diferenciación de la localización subcelular.
Referencias
[editar]- ↑ «Nucleoside-phosphate kinase». enzyme.expasy.
- ↑ The Enzymes 6 (2nd edición). New York: Academic Press. 1962. pp. 139-149.
- ↑ Ayengar P, Gibson DM, Sanadi DR (July 1956). «Transphosphorylations between nucleoside phosphates». Biochimica et Biophysica Acta 21 (1): 86-91. PMID 13363863. doi:10.1016/0006-3002(56)90096-8.
- ↑ Lieberman I, Kornberg A, Simms ES (July 1955). «Enzymatic synthesis of nucleoside diphosphates and triphosphates». The Journal of Biological Chemistry 215 (1): 429-40. PMID 14392176. doi:10.1016/S0021-9258(18)66050-8.
- ↑ Heppel LA, Strominger JL, Maxwell ES (April 1959). «Nucleoside monophosphate kinases. II. Transphosphorylation between adenosine monophosphate and nucleoside triphosphates». Biochimica et Biophysica Acta 32: 422-30. PMID 14401179. doi:10.1016/0006-3002(59)90615-8.
- ↑ Müller CW, Schlauderer GJ, Reinstein J, Schulz GE (February 1996). «Adenylate kinase motions during catalysis: an energetic counterweight balancing substrate binding». Structure 4 (2): 147-56. PMID 8805521. doi:10.2210/pdb4ake/pdb.
- ↑ Dreusicke D, Schulz GE (November 1986). «The glycine-rich loop of adenylate kinase forms a giant anion hole». FEBS Letters 208 (2): 301-4. PMID 3023140. S2CID 11786335. doi:10.1016/0014-5793(86)81037-7.
- ↑ Byeon L, Shi Z, Tsai MD (March 1995). «Mechanism of adenylate kinase. The "essential lysine" helps to orient the phosphates and the active site residues to proper conformations». Biochemistry 34 (10): 3172-82. PMID 7880812. doi:10.1021/bi00010a006.
- ↑ Berg JM, Tymoczko JL, Stryer L (2002). Biochemistry. New York: W H Freeman. ISBN 0-7167-3051-0. Consultado el 8 de enero de 2016. (requiere registro).
- ↑ Krishnamurthy H, Lou H, Kimple A, Vieille C, Cukier RI (January 2005). «Associative mechanism for phosphoryl transfer: a molecular dynamics simulation of Escherichia coli adenylate kinase complexed with its substrates». Proteins 58 (1): 88-100. PMID 15521058. S2CID 20874015. doi:10.1002/prot.20301.
- ↑ Pai EF, Sachsenheimer W, Schirmer RH, Schulz GE (July 1977). «Substrate positions and induced-fit in crystalline adenylate kinase». Journal of Molecular Biology 114 (1): 37-45. PMID 198550. doi:10.1016/0022-2836(77)90281-9.
- ↑ Müller CW, Schulz GE (March 1992). «Structure of the complex between adenylate kinase from Escherichia coli and the inhibitor Ap5A refined at 1.9 A resolution. A model for a catalytic transition state». Journal of Molecular Biology 224 (1): 159-77. PMID 1548697. doi:10.2210/pdb1ake/pdb.
- ↑ Schlauderer GJ, Proba K, Schulz GE (February 1996). «Structure of a mutant adenylate kinase ligated with an ATP-analogue showing domain closure over ATP». Journal of Molecular Biology 256 (2): 223-7. PMID 8594191. doi:10.1006/jmbi.1996.0080.
- ↑ Vonrhein C, Schlauderer GJ, Schulz GE (May 1995). «Movie of the structural changes during a catalytic cycle of nucleoside monophosphate kinases». Structure 3 (5): 483-90. PMID 7663945. doi:10.1016/s0969-2126(01)00181-2.
- ↑ Fukami-Kobayashi K, Nosaka M, Nakazawa A, Go M (May 1996). «Ancient divergence of long and short isoforms of adenylate kinase: molecular evolution of the nucleoside monophosphate kinase family». FEBS Letters 385 (3): 214-20. PMID 8647254. S2CID 24934783. doi:10.1016/0014-5793(96)00367-5.
Enlaces externos
[editar]- Esta obra contiene una traducción derivada de «Nucleoside-phosphate kinase» de Wikipedia en inglés, concretamente de esta versión, publicada por sus editores bajo la Licencia de documentación libre de GNU y la Licencia Creative Commons Atribución-CompartirIgual 4.0 Internacional.
