Estructura de comunidades
En el estudio de redes complejas, se dice que una red tiene estructura de comunidades si los nodos de la red pueden ser fácilmente agrupados en grupos de nodos, potencialmente superpuestos. En el caso particular de buscar comunidades no superpuestas, la red se divide de forma natural en grupos de nodos densamente conectados internamente y con pocas conexiones entre grupos. La definición más general está basada en el principio de que un par de nodos tiene mayor probabilidad de estar conectado si ambos son miembros de la(s) misma(s) comunidad(es), y menor probabilidad de estar conectado si no comparten comunidades.
Propiedades[editar]
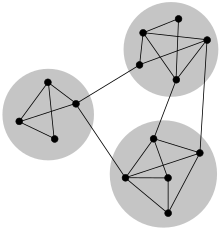
En el estudio de redes, tales como redes de computadoras e información, redes sociales y redes biológicas, se han observado múltiples características que ocurren comúnmente, incluyendo la propiedad del mundo pequeño, larga cola de la distribución de grado y agrupamiento, entre otros. Otra característica común es estructura de comunidades[1] [2] [3] .[4] En el contexto de redes, la estructura de comunidades se refiere a la ocurrencia de grupos de nodos en una red que son más densamente conectados internamente que el resto de la red, como se muestra en la imagen de ejemplo a la derecha. Esta heterogeneidad de las conexiones sugiere que la red tiene ciertas divisiones naturales.
Las comunidades son a menudo definidas en términos de la partición del conjunto de vértices, es decir, cada nodo pertenece a una y solo a una comunidad, como en la figura. Esto es una simplificación útil y la mayor parte de los métodos de detección de comunidades encuentran este tipo de estructura de comunidades. Aun así en algunos casos una representación mejor podría ser una dónde los vértices pertenezcan a más de una comunidad. Esto podría pasar en una red social donde cada vértice represente una persona, y las comunidades representen diferentes grupos de amigos: una para familia, otra para compañeros de trabajo, una para amigos en el mismo club deportivo, y así sucesivamente. El uso de cliques para detectar comunidades, explicado más adelante, es un ejemplo de cómo pueden ser encontradas estas comunidades con superposición.
Existen redes que no tienen ninguna estructura de comunidades significativa. Muchos modelos de red básicos, como por ejemplo grafos aleatorios y el modelo Barabási–Albert, no muestran estructura de comunidades.
Aplicaciones[editar]
La estructura de comunidades es bastante común en redes reales. Las redes sociales incluyen grupos comunitarios (el origen del término, de hecho) basados en ubicaciones comunes, intereses comunes, ocupaciones comunes, etc.[5] Las redes metabólicas tienen comunidades basadas en grupos funcionales. En las redes de citaciones se forman comunidades por temas de investigación.[1] Ser capaz de identificar estas sub-estructuras dentro de una red puede ayudar a entender de forma intuitiva cómo el funcionamiento de la red y su topología se afectan el uno al otro. Tal intuición puede ser útil en mejorar algunos algoritmos sobre grafos tales como agrupamiento espectral.[6]
Algoritmos para encontrar comunidades[editar]
Encontrar comunidades en una red aleatoria puede ser una tarea computacionalmente difícil. El número de comunidades en la red (si hay alguna) es usualmente desconocido y, a menudo, las comunidades tienen diferente tamaño y/o densidad. A pesar de estas dificultades, múltiples métodos de detección de comunidades han sido desarrollados y empleados con diferentes niveles de éxito.[4]
Método de corte mínimo[editar]
Uno de los algoritmos más antiguos para dividir redes es el método de corte mínimo (y variantes como corte de proporción y corte normalizado). Este método se usa, por ejemplo, para equilibrar la carga del cálculo en paralelo con el fin de minimizar la comunicación entre los nodos del procesador.
En el método de corte mínimo, la red está dividida en un número predeterminado de partes, normalmente de aproximadamente el mismo tamaño, que se escogen de forma tal que el número de las aristas entre grupos sea mínimo. El método funciona bien en muchas aplicaciones para las cuales fue inicialmente concebido pero es menos adecuado para encontrar estructura de comunidades en redes generales pues encontrará comunidades sin importar si están implícitas en la estructura, y solo encontrará un número fijo de comunidades.[7]
Agrupamiento jerárquico[editar]
Otro método para detectar estructuras de comunidades es el agrupamiento jerárquico. En el cual se define una medida de similitud que cuantifica algún tipo (usualmente topológico) de similitud entre pares de nodos. Algunas de las medidas más usadas son similitud coseno, el índice de Jaccard, y la distancia Hamming entre filas de la matriz de adyacencia. Luego se agrupan los nodos similares en comunidades de acuerdo a dicha medida. Existen varios esquemas comunes para realizar el agrupamiento, los dos más sencillos sonː agrupamiento de enlace-simple, en el cual se considera que dos grupos son comunidades separadas si y solo si para todo par de nodos en diferentes grupos la similitud es menor que un umbral, y agrupamiento de enlace-comleto, en el cual se cumple que dentro de cada grupo todo par de nodos tiene una similitud mayor que un umbral. Un acercamiento novedoso en esta dirección es el uso de varias medidas de similitud y disimilitud, combinadas a partir de sumas convexas, sumas convexas, lo cual ha mejorado en gran medida el funcionamiento de este tipo de metodología.[8]
Algoritmo Girvan–Newman[editar]
Otro algoritmo comúnmente utilizado para detectar comunidades es el algoritmo Girvan–Newman.[1] El cual identifica las aristas que se encuentran entre comunidades y las elimina, dejando atrás las comunidades. La identificación de estas aristas se realiza empleando la medida de centralidad en teoría de grafos: intermediación, la cual asigna un número a cada arista que es mayor si la arista se encuentra entre muchos pares de nodos.
El algoritmo Girvan–Newman obtiene resultados de una calidad razonable y es popular porque ha sido implementado en múltiples paquetes estándar de software. Pero tiene un costo temporal alto (O(m2n)) en una red de n vértices y m aristas, por lo que es poco práctico para redes de más de unos cuantos miles de nodos.[9]
Maximización de modularidad[editar]
A pesar de sus conocidas deficiencias, uno de los métodos más usados para detección de comunidades es maximización de modularidad.[9] Modularidad es una función de beneficio que mide la calidad de una partición de la red en comunidades. El método de maximización de modularidad detecta comunidades buscando entre posibles particiones de la red la que tenga un valor particularmente alto de modularidad. Dado que una búsqueda exhaustiva por todas las posibles particiones de la red es normalmente impracticable, los algoritmos prácticos se basan en métodos aproximados de optimización tales con algoritmos golosos, recosido simulado, u optimización espectral, con diferentes enfoques basados en velocidad y exactitud.[10][11]
Un enfoque popular es el método Louvain, que iterativamente optimiza comunidades locales hasta que la modularidad global no puede ser mejorada a través de modificaciones a la distribución de comunidades actual.[12] El mejor algoritmo de maximización de modularidad en la actualidad (ganador del décimo Concurso de Implementación CIMACS) es un algoritmo iterativo de ensamblaje.[13]
La utilidad de la optimización de modularidad es cuestionable, dado que se ha sido mostrado que falla en detectar comunidades menores que cierta escala, dependiendo del tamaño de la red (límite de resolución[14]); por otra parte los valores de modularidad se caracterizan por una gran degeneración de particiones con alta modularidad, cercana al máximo absoluto, que pueden ser muy diferentes entre ellas.[15]
Inferencia estadística[editar]
Los métodos basados en inferencia estadística intentan ajustar un modelo generador a los datos de la red, el cual codifica la estructura de comunidades. La ventaja general de esta enfoque con respecto a sus alternativas es su naturaleza de principios, y la capacidad de dirigirse inherentemente a asuntos de importancia estadística. La mayor parte de los métodos en la literatura están basados en el modelo de bloque estocástico[16] así como variantes que incluyen afiliación mixta,[17][18] grado-corrección,[19] y estructuras jerárquicas.[20] La selección del modelo puede realizarse utilizando enfoques de principios como longitud de descripción mínima[21] y selección de modelo bayesiana.[22] Actualmente existen muchos algoritmos para realizar una inferencia eficaz de modelos de bloque estocástico, incluyendo propagación de creencia[23][24] y Monte Carlo aglomerativo.[25]
A diferencia de los enfoques que intentan agrupar la red a partir de una función de calidad ad hoc, esta clase de método está basado en modelos generativos que no solo sirven como una descripción a gran escala de la estructurad de la red, sino que también puede ser utilizado para generalizar los datos, y predecir la ocurrencia de aristas faltantes o falsas en la red.[26][27]
Métodos basados en cliques[editar]
Los cliques son subgrafos en los cuales todos los nodos están conectados al resto de los nodos del clique. No es sorprendente que existan múltiples enfoques para detectar comunidades en una red basados la detección de cliques en un grafo y en el análisis de como estos se superponen dado que los nodos no pueden estar más densamente conectados que en un clique. Note que como un nodo puede ser miembro de más de un clique, en estos métodos un nodo puede ser miembro de más de una comunidad formado una estructura de comunidades con superposición.
Un enfoque es encontrar los cliques maximales, es decir los cliques que no son subgrafos de otros cliques. El algoritmo clásico para encontrarlos es el algoritmo Bron-Kerbosch. Es posible utilizar la superposición de los cliques maximales para definir comunidades de múltiples formas. La más sencilla es considerar solo los cliques maximales que sean más grandes que un tamaño mínimo (número de nodos). La unión de estos cliques forma un grafo cuyas componentes conexas definen comunidades.[28] Estos enfoques a menudo son implementadas en softwares de análisis de red social tales como UCInet.
El enfoque alternativo es utilizar cliques de tamaño fijo, k. La superposición de los cuales puede definir un tipo de hipergrafo k-regular o una estructura que es una generalización del grafo línea (el caso cuando k=2) conocido como Grafo Clique.[29] En el cual los vértices representan cliques en el grafo original y existe una arista entre dos nodos si los cliques que representan tienen algún nodo en común. El Grafo clique puede ser utilizado para detectar una estructura de comunidades aplicando cualquiera de los métodos de detección de comunidades anteriores (los cuales asignan cada nodo a una comunidad) al grafo clique, asignando cada clique a una comunidad. Luego como un nodo puede pertenecer a varios cliques puede pertenecer a varias comunidades. Por ejemplo el método de Percolación de Cliques[30] define comunidades como grupos de percolación de k-cliques. Para hacer esto encuentra todos los k-cliques en una red, es decir todos los sub-grafos completos de k-nodos. Luego define que dos k-cliques son adyacentes si comparten k − 1 nodos, lo cual es utilizado para definir aristas en un grafo clique. Entonces se define una comunidad como la unión máxima de k-cliques en los que podemos alcanzar cualquier k-clique en ella desde cualquier otro de sus k-cliques a través de una serie de k-cliques adyacentes. Es decir comunidades que son las componentes conexas del grafo clique. Como un nodo puede pertenecer a múltiples grupos de percolación de k-cliques al mismo tiempo las comunidades se pueden superponer.
Métodos de prueba para algoritmos de detección de comunidades[editar]
La evaluación de algoritmos para determinar cuáles son mejores para detectar estructura de comunidades es todavía una cuestión abierta. Un ejemplo típico es la «prueba de los cuatro grupos», en la cual una red se divide en cuatro grupos del mismo tamaño grupos (normalmente de 32 nodos cada uno) y las probabilidades de conexión dentro y entre los grupos varían para crear estructuras más o menos desafiantes para el algoritmo de detección. Esta prueba es un caso especial del modelo plantado de l-partición[31] de Condon y Karp o, de forma más general, de «modelos de bloque estocástico», una clase general de modelos de red aleatoria que contienen estructura de comunidades. Se han propuesto otras pruebas más flexibles que permiten variar el tamaño de los grupos y distribuciones de grado no triviales, tales como LFR benchmark propuesto por Lancichinetti et al.,[32] que son extensiones de la prueba de los cuatro grupos que incluye distribuciones de grados heterogéneas y un tamaño de comunidad variable, lo cual los hacen más fiables para los algoritmos de detección de comunidades.
Las pruebas generadas por computadora comúnmente utilizadas comienzan con una red con comunidades bien definidas. Luego esta estructura se degrada agregando o eliminando aristas de forma que cada vez es más difícil detectar las comunidades originales. Al final, la red llega a un punto donde es esencialmente aleatoria. Este tipo de prueba es denominada «abierta». Su rendimiento se evalúa por medidas tales como información mutua normalizada o variación de información. Esto algoritmos comparan la solución obtenida por un algoritmo con la estructura comunitaria original, evaluando la semejanza de ambas particiones.
Véase también[editar]
Enlaces externos[editar]
- Community detection in graphs - una introducción
- Are there implementations of algorithms for community detection in graphs? - Stack Overflow
- What are the differences between community detection algorithms in igraph? - Stack Overflow
Referencias[editar]
- ↑ a b c M. Girvan and M. E. J. Newman (2002).
- ↑ S. Fortunato (2010).
- ↑ F. D. Malliaros and M. Vazirgiannis (2013).
- ↑ a b M. A. Porter, J.-P. Onnela and P. J. Mucha (2009).
- ↑ Hamdaqa, Mohammad; Tahvildari, Ladan; LaChapelle, Neil; Campbell, Brian (2014).
- ↑ Zare, Habil; P. Shooshtari; A. Gupta; R. Brinkman (2010).
- ↑ M. E. J. Newman (2004).
- ↑ Alvarez, Alejandro J.; Sanz-Rodríguez, Carlos E.; Cabrera, Juan Luis (2015-12-13).
- ↑ a b M. E. J. Newman (2004).
- ↑ L. Danon, J. Duch, A. Díaz-Guilera and A. Arenas (2005).
- ↑ R. Guimera, L. A. N. Amaral (2004).
- ↑ V.D. Blondel, J.-L. Guillaume, R. Lambiotte and E. Lefebvre (2008).
- ↑ Michael Ovelgönne and Andreas Geyer-Schulz (2013).
- ↑ S. Fortunato and M. Barthelemy (2007).
- ↑ B. H. Good, Y.-A. de Montjoye and A. Clauset (2010).
- ↑ Holland, Paul W.; Kathryn Blackmond Laskey; Samuel Leinhardt (1983-06).
- ↑ Airoldi, Edoardo M.; David M. Blei; Stephen E. Fienberg; Eric P. Xing (2008-06).
- ↑ Ball, Brian; Brian Karrer; M. E. J. Newman (2011).
- ↑ Karrer, Brian; M. E. J. Newman (2011-01-21).
- ↑ Peixoto, Tiago P. (2014-03-24).
- ↑ Martin Rosvall and Carl T. Bergstrom (2007).
- ↑ Yan, Xiaoran; Jacob E. Jensen; Florent Krzakala; Cristopher Moore; Cosma Rohilla Shalizi; Lenka Zdeborova; Pan Zhang; Yaojia Zhu (2012-07-17).
- ↑ Gopalan, Prem K.; David M. Blei (2013-09-03).
- ↑ Decelle, Aurelien; Florent Krzakala; Cristopher Moore; Lenka Zdeborová (2011-12-12).
- ↑ Peixoto, Tiago P. (2014-01-13).
- ↑ Guimerà, Roger; Marta Sales-Pardo (2009-12-29).
- ↑ Clauset, Aaron; Cristopher Moore; M. E. J. Newman (2008-05-01).
- ↑ M.G. Everett and S.P. Borgatti (1998).
- ↑ T.S. Evans (2010).
- ↑ G. Palla, I. Derényi, I. Farkas and T. Vicsek (2005).
- ↑ Condon, A.; Karp, R. M. (2001).
- ↑ A. Lancichinetti, S. Fortunato and F. Radicchi (2008).
