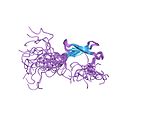
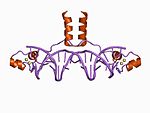
Dedo de zinc


Los dedos de zinc son pequeños motivos estructurales de proteínas que pueden coordinar uno o más iones de zinc para ayudar a estabilizar sus pliegues. Normalmente funcionan como módulos de interacción que unen el ADN, ARN, proteínas y moléculas pequeñas. Estas proteínas tienen un rol estructural esencialmente. Este rol puede consistir en bajar la entalpía de plegamiento de una proteína e inducir una conformación activa, o bien, estabilizar una estructura cuaternaria en particular. El zinc también tiene un rol regulativo, es decir, no participa en los varios pasos catalíticos, pero su presencia sí incrementa la rapidez catalítica.[1] El nombre de "dedo de zinc" fue acuñado para describir la hipótesis de la estructura de la unidad repetida del factor de transcripción IIIA en Xenopus laevis.
Los dedos de zinc coordinan iones de zinc con una combinación de residuos de cisteína e histidina. Pueden ser clasificados por el tipo y orden de estos residuos de coordinación con el cinc (por ejemplo, Cys2His2, Cys4, y Cys6). Un método más sistemático los clasifica en distintos "grupos de pliegues" basado en la forma de la columna de la proteína en el dominio plegado. El "grupo de pliegues" más común de dedos de zinc es el de tipo Cys2His2 (el "clásico dedo de zinc"), clave triple y cinta de cinc.[2]
Clasificación[editar]
Dedos de zinc Cis2His2[editar]
El grupo de plegamiento tipo Cis2His2 es la clase de dedos de zinc mejor caracterizados y son muy comunes en factores de transcripción de mamíferos. Estos dominios adoptan un pliegue ββα y tienen el siguiente motivo de secuencia aminoacídica: X2-Cis-X2,4-Cis-X12-His-X3,4,5-His [3] Esta clase de dedos de zinc puede tener una variedad de funciones tales como la unión a ARN y las interacciones proteína-proteína, pero es conocido por su papel en la unión a secuencia específica de ADN, en proteínas como Zif268. En estas, los dominios de dedos de zinc se suceden típicamente en repeticiones en tándem con dos, tres o más dedos por el dominio de unión a ADN por proteína. Estos arreglos en tándem se pueden unir al surco mayor del ADN y están normalmente espaciados a intervalos de 3 pares de bases. La α-hélice de cada dominio (a menudo llamada la "hélice de reconocimiento") puede hacer contactos con secuencias específica de bases de ADN; los residuos de una sola hélice de reconocimiento puede contactarse con 4 o más bases para producir un patrón de superposición de contactos con los dedos de zinc adyacente.
Dedos de zinc Zn2/Cis6[editar]
Los miembros de esta clase contiene complejos de cinc binuclear en donde dos iones de zinc están unidos a seis residuos de cisteína. Estos dedos de zinc se pueden encontrar en varios factores de transcripción incluyendo la proteína Gal4 de la levadura.
Química inorgánica de los dedos de zinc[editar]
Debido a su química de coordinación básica, el zinc no interactúa directamente con el ácido nucleico. En vez de eso, el ion de cinc debe servir un rol estructural, definiendo el plegado y la estructura tridimensional del andamiaje de la proteína en cuestión. Esta estructura, definida por un metal en su centro, es capaz de reconocer su estructura complementaria en el polímero de ácido nucleico.[4]
Técnicas de estudio[editar]
Según los estudios basados en bases de datos cristalográficas para metaloproteínas y un detallado examen de la secuencia emergente para dedos de zinc, Berg,[4] propuso una estructura tridimensional. Estudios de EXAFS también respaldaron el sitio tetrahédrico del zinc. Asimismo, estudios de RMN coinciden.
El RMN ha sido la técnica más recurrentemente usada para el estudio de los dedos de zinc. La espectroscopía de resonancia magnética nuclear (RMN) es una técnica de investigación que explota las propiedades magnéticas de ciertos núcleos atómicos. Este tipo de espectroscopía nos permite determinar las características físicas y químicas de los átomos o de las moléculas que los contienen. Se apoya en el fenómeno de resonancia magnética nuclear en el que el campo magnético intramolecular alrededor de un átomo en una molécula cambia la frecuencia de resonancia, dándonos así acceso a detalles de la estructura electrónica de una molécula y de sus grupos funcionales individuales. Da información acerca de la estructura, la dinámica, estado de reacción y ambiente químico de las moléculas.
Un reto que presenta el estudio de ciertas proteínas, incluyendo dedos de cinc, es que la formación de cuerpos de exclusión, la toxicidad de proteínas exógenas, la modificación de cadenas laterales, etcétera, influencian la expresión de la proteína y pueden complicar la purificación de la misma. Por lo tanto, ha sido difícil expresar suficientes proteínas para estudiarlas in vitro. En los últimos años se han desarrollado sistemas de expresión fusionados como GST y etiquetamiento con histidina (His-tag por su abreviación en inglés) para facilitar la expresión de proteínas y simplificar la purificación de proteínas.[5]
Los sistemas de expresión His-tag son ampliamente usados debido a su bajo peso molecular y que no afectan la estructura de la proteína ni sus funciones. Esto significa que no es necesario separar el His-tag de la proteína buscada. Por otra parte, las proteínas fusionadas His-tag pueden ser fácilmente purificadas por una resina de afinidad Ni-NTA. En estudios recientes, los dedos de cinc han sido expresados, separados y purificados usando un sistema His-tag/Ni-NTA. A partir de los efectos del His-tag en las proteínas de dedos de cinc, se pueden evaluar sus propiedades y su estructura. Para esto se pueden usar técnicas de espectrometría como espectroscopía de absorción UV-Vis, espectroscopía de dicroísmo circular (CD), prueba de titulación de ion de Cinc, prueba de degeneración de urea y prueba de actividad de hidrolasa.
Matrices de dedos de zinc prediseñadas[editar]
Diversas estrategias se han desarrollado para el diseño de dedos de zinc Cys2His2 que se unan a las secuencias deseadas. Estas incluyen "ensamblaje modular" y estrategias de selección que emplean, ya sea expresión de fagos o sistemas de selección celular. Tales arreglos de dedos de zinc prediseñados pueden ser utilizado en numerosas aplicaciones, tales como factores de transcripción artificiales, metilasas de dedo de zinc, recombinasas de dedo de zinc y nucleasas con dedos de zinc.[6] Factores de transcripción artificiales con matrices de dedos de cinc prediseñados se han utilizado en numerosos estudios científicos y se evalúa un factor de transcripción artificial que activa la expresión de VEGF en seres humanos. Las nucleasas de dedos de zinc se han convertido en reactivos útiles para la manipulación de los genomas de muchos organismos superiores incluyendo Drosophila melanogaster, Caenorhabditis elegans, tabaco, y diversos tipos de células de mamíferos,[7] y pez cebra.[8] Un ensayo clínico en curso es la evaluación de las nucleasas de dedos de zinc que interrumpen el gen CCR5 en células T CD4+ humanas como un posible tratamiento para el VIH/SIDA.
Ensamblaje modular[editar]
El método más sencillo para generar nuevas matrices de dedos de cinc es combinando pequeños "módulos" de dedos de cinc de especificidad conocida. EL proceso de ensamblaje modular más común combina tres dedos de cinc de los cuales cada uno puede reconocer una secuencia de ADN de 3 pares de bases para generar una matriz de 3 dedos que tenga un sitio de reconocimiento de 9 pares de bases. Otros procedimientos pueden utilizar ya sea módulos de uno o dos dedos para generar matrices de dedos de cinc con seis o más dedos. El principal inconveniente con este procedimiento es que la especificidad de cada uno de los dedos de cinc puede superponerse y puede depender del contexto de los dedos de cinc circundantes y el ADN. Sin métodos para poder tener en cuenta esta "dependencia del contexto", el procedimiento estándar de ensamblaje modular a veces falla a menos que se use para reconocer secuencias de la forma (GNN)N.
Métodos de selección[editar]
Numerosos métodos de selección se han utilizado para generar matrices de dedos de cinc capaces de apuntar a las secuencias deseadas. Los esfuerzos iniciales de selección se utilizaron para seleccionar proteínas de unión a ADN expresadas en fagos de un gran conjunto de arreglos de dedos de cinc aleatorios. Los esfuerzos más recientes han utilizado una sistemas de uno y dos híbridos de levadura, bacterias y células de mamífero. Un método aún más reciente para seleccionar los nuevos arreglos de dedos de cinc de bacterias utiliza un sistema de dos híbrido y ha sido denominado "OPEN" por sus creadores.[9] Este sistema combina conjuntos preseleccionados de dedos de cinc individuales que fueron seleccionados para unirse a un triplete y, a continuación, se utiliza una segunda ronda de selección para obtener matrices de 3 dedos capaces de unirse a la secuencia de 9 pares de bases deseado. Este sistema fue desarrollado por el Zinc Finger Consortium como una alternativa a las fuentes comerciales de matrices de dedos de cinc prediseñados.
Función biológica[editar]
Además de ser útiles en lectura, escritura y transcripción de ADN, existen hormonas que usan dedos de zinc. Son estas las mismas hormonas que están fuertemente asociadas con la homeostasis de crecimiento de todo el organismo. Se listan en la siguiente tabla las conexiones de dichas hormonas con la homeostasis de otros elementos (M).[4]
| Conexiones de los elementos con los dedos de zinc | |
| Sistema efector | Elemento conectado (M) |
| Glucocorticoides (esterol) | Na, niveles de K (riñón) |
| Vitamina D (esterol) | Proteínas de transporte de Ca, osteocalcina e incluso metalotioneína (Zn), fosfatasa alcalina (P). |
| Tiroxina | Metabolismo del yodo (Fe), (I), Ca-ATPasa (Ca), tejido conector/Ca. |
| Ácido retinoico | Cinc deshidrogenasa (Zn) |
Posible aplicación en terapia molecular[editar]
Los dedos de cinc tienen también una posible aplicación clínica para la solución de enfermedades. La estrategia se basa en utilizar los dedos de cinc unidos a nucleasas (finger zinc nucleases, ZFN). De esa manera, este complejo introducido en la célula sería capaz de unirse al ADN en unas zonas específicas cercanas a la mutación gracias al reconocimiento de los dedos de cinc, y después las nucleasas actuarían cortando la doble hebra de ADN. Si ahora introducimos una cadena de ADN silvestre (sin la mutación), entonces por recombinación homóloga podríamos arreglar esa mutación. Las ventajas de esta estrategia son: no hay integración de ninguna secuencia (no estamos manipulando el genoma), se realiza una reparación de la secuencia del gen, la eficiencia es muy alta y además no requiere mantener la expresión de algún gen mucho tiempo. Como inconvenientes a esta estrategia encontramos que es probablemente solo válida para terapias ex-vivo, y que las nucleasas tienen alto poder inmunogénico, por lo que aún falta por demostrar su inocuidad.
Referencias[editar]
- ↑ Silva,, J.J.R, et al (1994). The biological chemistry of the elements: the inorganic chemistry of life (en inglés). Oxford: Clarendon Press.
- ↑ S.E. Krishna; I.Majumdar; N.V. Grishin (enero de 2003). «SURVEY AND SUMMARY: Structural classification of zinc fingers». Nucleic Acids Res. 31: 532-550.
- ↑ C.O. Pabo; E.Peisach; R.A. Grant (2001). «Design and Selection of Novel Cys2His2 Zinc Finger Proteins». Annu. Rev. Biochem. 70: 313-40.
- ↑ a b c Bertini, I (1994). Bioniorganic Chemistry (en inglés). University Science Books.
- ↑ Zhao, Dongxin; Huang, Zhongxian (20 de julio de 2016). «Effect of His-Tag on Expression, Purification, and Structure of Zinc Finger Protein, ZNF191(243-368)». Bioinorganic Chemistry and Applications (en inglés) 2016: 1-6. ISSN 1565-3633. PMC 4971304. PMID 27524954. doi:10.1155/2016/8206854. Consultado el 6 de mayo de 2017.
- ↑ A.C. Jamieson; J.C. Miller; C.O. Pabo (mayo de 2003). «Drug Discovery with Engineered zinc-finger proteins». Nat. Rev. Drug Discov. 2: 361-8.
- ↑ D. Carroll (2008). «Progress and prospects: Zinc-finger nucleases as gene therapy agents». Gene Therapy 15: 1463-1468.
- ↑ S.C. Ekker (2008). «Zinc finger-based knockout punches for zebrafish genes». Zebrafish 5: 1121-3.
- ↑ M.L. Maeder et al. (septiembre de 2008). «Rapid "Open-Source" Engineering of Customized Zinc-Finger Nucleases for Highly Efficient Gene Modification». Mol. Cell 31: 294-301.
Enlaces externos[editar]
- ,,.,-.-.--.-.
- McDowall J. European Molecular Biology Laboratory - European Bioinformatics Institute (EMBL-EBI), ed. «Protein of the Month: Zinc Fingers».
- Goodsell DS. Research Collaboratory for Structural Bioinformatics (RCSB) Protein Data Bank (PDB), ed. «Molecule of the Month: Zinc Fingers». Archivado desde el original el 31 de mayo de 2008. Consultado el 21 de marzo de 2021.