Cas9
| Cas9 | |
|---|---|

| |
| Estructura cristalina de Cas9 S pyogenes en el complejo con sgRNA y su ADN objetivo en la resolución 2,6 A ˚. (Nishimasu, et al. 2015) | |
| Identificadores | |
| Organismo | |
| Símbolo | cas9 |
| Alt. symbols | SPy_1046 |
| RefSeq (Prot) | NP_269215.1 |
| UniProt | Q99ZW2 |
| Otros datos | |
| Número EC | 3.1.-.- |
| Cromosoma | Genomico: 0.85 - 0.86 Mb |
Cas9 (CRISPR associated protein 9) es una enzima endonucleasa de ADN dirigida por un ARN guía que se encuentra asociada con el sistema CRISPR (Repeticiones Palindrómicas Cortas Agrupadas y Regularmente Interespaciadas). Este se trata de un sistema de inmunidad que puede encontrarse en numerosas bacterias, como el Streptococcus pyogenes. Cas9 es capaz de memorizar secuencias de ADN de bacteriófagos invasores o de plásmidos que ataquen a la bacteria para, más tarde, reconocer este ADN, y actuar sobre él, inactivándolo.[1][2] Cas9 realiza este proceso desenrollando el ADN exógeno (actuando como lo haría una helicasa) y comprobando si es complementario a alguna región espaciadora de CRISPR. Si el substrato de ADN es reconocido, Cas9 se adhiere al ADN invasor y lo corta en una determinada región de forma que queda inactivado. En este sentido, el sistema CRISPR-Cas9 es un mecanismo muy similar al mecanismo de interferencia de ARN (ARNi) de eucariotas.
La proteína Cas9 se ha utilizado en gran medida como una herramienta de ingeniería genómica para inducir la rotura dirigida del ADN de doble cadena. Estas rupturas pueden conducir a la inactivación génica o a la introducción de genes heterólogos a través de unión no homóloga y recombinación homóloga respectivamente en muchos organismos de modelo de laboratorio. Junto con las nucleasas de zinc y las proteínas TALEN, Cas9 se está convirtiendo en una herramienta destacada en el campo de la edición del genoma. Cas9 ha ganado la tracción en los últimos años, ya que puede escindir casi cualquier secuencia complementaria a la guía RNA.[2] Debido a que la especificidad de destino de Cas9 proviene de la ARN guía: la complementariedad de ADN y no modificaciones de la propia proteína (como TALENs y dedos de zinc), la ingeniería Cas9 para dirigir el nuevo ADN es sencillo[3][4] Las versiones de Cas9 que se unen, pero no escinden el ADN cognado puede ser utilizado para localizar transcripcional activador o represores a secuencias de ADN específico con el fin de controlar la activación transcripcional y la represión[5][6] La orientación de Cas9 se ha simplificado mediante la ingeniería de un solo ARN guía quimérico. Los científicos han sugerido que los impulsos genéticos basados en Cas9 pueden ser capaces de editar los genomas de poblaciones enteras de organismos.[7] En 2015, los científicos utilizaron Cas9 para modificar el genoma de embriones humanos por primera vez.[8]
Estudios estructurales de Cas9
[editar]Visión de conjunto
[editar]
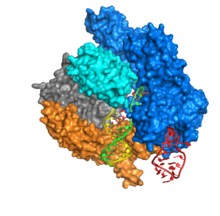

La estructura cristalina de Cas9 se unió al ADN, según lo resuelto por Anders et al en su papel de la naturaleza 2014. Las interpretaciones estructurales se realizaron utilizando el software Quimera de UCSF.
Cas9 presenta una arquitectura bi-lobulada con el ARN guía situado entre el lóbulo alfa-helicoidal (azul) y el lóbulo de la nucleasa (cian, naranja y gris). Estos dos lóbulos están conectados a través de una sola hélice de puente. Existen dos dominios de nucleasa situados en el lóbulo de nucleasa multidominio, el RuvC (gris) que escinde la cadena de ADN no diana y el dominio de nucleasa HNH (cian) que escinde la cadena diana de ADN. De manera interesante, el dominio RuvC está codificado por sitios secuencialmente dispares que interactúan en la estructura terciaria para formar el dominio de escisión RuvC.
Una característica clave del ADN diana es que debe contener un motivo adyacente protospacer (PAM) que consiste en la secuencia de tres nucleótidos-NGG. Este PAM es reconocido por el dominio de interacción PAM (dominio PI, naranja) situado cerca del extremo C-terminal de Cas9. Cas9 experimenta cambios conformacionales distintos entre los Estados Unidos de apo, de ARN de guía y de ARN de guía: enlazados de ADN, que se detallan a continuación. PAM es reconocido por Arg 1333 y Arg 1335 en la ranura principal por una β-horquilla, donde se unen a dG2 y dG3 de PAM.[9]
Estructuras de cristal en detalle
[editar]Cas9 reconoce la arquitectura de tallo-bucle inherente en el locus CRISPR, que media la maduración del CRRNA-tracrRNA ribonucleoprotein complejo.[10] Cas9 en complejo con CRISPR ARN (CRRNA) y trans-activación crRNA (tracrRNA), además, reconoce y degrada la dsDNA objetivo.[11] En la estructura de co-cristal que se muestra aquí, el complejo de CRRNA-tracrRNA se reemplaza por un ARN de guía única quimérico (sgRNA, en rojo) que se ha demostrado que tiene la misma función que el complejo de ARN natural.[12] La base de sgRNA emparejado con ssDNA objetivo está anclado por Cas9 como una arquitectura en forma de T. Esta estructura cristalina de la enzima Cas9 unida a ADN revela cambios conformacionales distintos en el lóbulo alfa-helicoidal con respecto al lóbulo de nucleasa, así como la localización en el dominio HNH. La proteína consiste en un lóbulo de reconocimiento (REC) y un lóbulo de nucleasa (NUC). Debe observarse que todas las regiones excepto el HNH forman estrechas interacciones entre sí y el complejo sgRNA-ssDNA, mientras que el dominio HNH forma pocos contactos con el resto de la proteína. En otra conformación del complejo Cas9 observada en el cristal, el dominio HNH no es visible. Estas estructuras sugieren la flexibilidad conformacional del dominio HNH.
Se han publicado varias estructuras de cristal, incluyendo:
- Jinek et al. Las estructuras de las endonucleasas Cas9 revelan la activación conformacional mediada por ARN. Science, Feb 2014[13]
- Anders et al. Bases estructurales del reconocimiento de ADN diana dependiente de PAM por la endonucleasa Cas9. Nature Sept 2014.[14]
- Nishimasu et al. Estructura cristalina de Cas9 en complejo con ARN guía y ADN objetivo. Cell Feb 2014[15]
- Jiang et al. Un complejo de ARN de guía Cas9 preorganizado para el reconocimiento de ADN diana. Ciencia, junio de 2015[16]
- Jiang et al. Estructuras de un complejo CRISPR-Cas9 R-loop cebado para la escisión del ADN. Ciencia, Feb 2016[17]
Interacciones entre sgRNA y Cas9
[editar]En el complejo sgRNA-Cas9, basado en la estructura cristalina, los dominios REC1, BH y PI tienen contactos importantes con la columna vertebral o las bases en la región de repetición y espaciadora.[18][14] Se han probado varios mutantes Cas9 que incluyen deleción de dominios REC1 o REC2 y mutaciones de residuos en BH. Los mutantes relacionados con REC1 y BH muestran una actividad menor o ninguna en comparación con el tipo salvaje, lo que indica que estos dos dominios son cruciales para el reconocimiento de sgRNA en la secuencia repetida y la estabilización de todo el complejo. Aunque las interacciones entre la secuencia espaciadora y Cas9, así como el dominio PI y región de repetición necesitan estudios adicionales, el co-cristal demuestra clara interfaz entre Cas9 y sgRNA. De hecho, la estructura cristalina reciente de Cas9 unido a ARN de guía única revela que la secuencia de "semilla" de ARN de 10 nucleótidos se preordena en una conformación de forma A para el reconocimiento de ADN diana. Además de las secuencias de semillas preordenadas, la comparación del complejo Cas9-sgRNA con la estructura unida al ADN diana (PDB 4UN3) revela que los sitios que interaccionan con PAM (R1333 y R1335) responsables de la 5'-NGG-3 'PAM Reconocimiento se colocan previamente antes de unirse al ADN diana. En conjunto, estas observaciones estructurales muestran que la región espaciadora del sgRNA, especialmente la región de la semilla, es esencial para desencadenar Cas9 para formar una estructura de reconocimiento de ADN competente que está listo para acoplar secuencias diana de ADN de doble hebra.[16]
Digestión del objetivo
[editar]El análisis de secuencias anteriores y los estudios bioquímicos han sugerido que Cas9 contiene dominios homólogos de endonucleasa RNasa H y HNH que son responsables de escisiones de dos hebras de ADN diana, respectivamente. Estos resultados son finalmente probados en la estructura. Aunque la baja similitud de secuencias, la secuencia similar a RNasa H tiene un pliegue RuvC (un miembro de la familia RNasa H) y la región HNH se pliega como T4 Endo VII (un miembro de la familia de endonucleasas HNH). Los trabajos anteriores sobre Cas9 han demostrado que el dominio HNH es responsable de la escisión de la secuencia complementaria del ADN diana y RuvC es responsable de la secuencia no complementaria (Westra, et al., 2012; Wiedenheft, et al., 2014).
Véase también
[editar]- Edición génica CRISPR
- Máquina molecular
- CRISPR
- CRISPR/Cpf1
- Nucleasas con dedos de zinc
- FokI
- Transdiferenciación con mediador de CRISPR-activador
Referencias
[editar]- ↑ Heler R; Samai P; Modell JW; Weiner C; Goldberg GW; Bikard D; Marraffini LA (Mar 2015). «Cas9 specifies functional viral targets during CRISPR-Cas adaptation». Nature 519 (7542): 199-202. Bibcode:2015Natur.519..199H. PMC 4385744. PMID 25707807. doi:10.1038/nature14245.
- ↑ a b Jinek M; Chylinski K; Fonfara I; Hauer M; Doudna JA; Charpentier E (Aug 2012). «A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity». Science 337 (6096): 816-21. Bibcode:2012Sci...337..816J. PMID 22745249. doi:10.1126/science.1225829.
- ↑ Cong, L.; Ran, F. A.; Cox, D.; Lin, S.; Barretto, R.; Habib, N.; Hsu, P. D.; Wu, X.; Jiang, W.; Marraffini, L. A.; Zhang, F. (3 de enero de 2013). «Multiplex Genome Engineering Using CRISPR/Cas Systems». Science 339 (6121): 819-823. PMC 3795411. PMID 23287718. doi:10.1126/science.1231143.
- ↑ Mali P; Esvelt KM; Church GM (Oct 2013). «Cas9 as a versatile tool for engineering biology». Nature Methods 10 (10): 957-63. PMC 4051438. PMID 24076990. doi:10.1038/nmeth.2649.
- ↑ Mali P; Aach J; Stranges PB; Esvelt KM; Moosburner M; Kosuri S; Yang L; Church GM (Sep 2013). «CAS9 transcriptional activators for target specificity screening and paired nickases for cooperative genome engineering». Nature Biotechnology 31 (9): 833-8. PMC 3818127. PMID 23907171. doi:10.1038/nbt.2675.
- ↑ Gilbert LA; Larson MH; Morsut L; Liu Z; Brar GA; Torres SE; Stern-Ginossar N; Brandman O; Whitehead EH; Doudna JA; Lim WA; Weissman JS; Qi LS (Jul 2013). «CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes». Cell 154 (2): 442-51. PMC 3770145. PMID 23849981. doi:10.1016/j.cell.2013.06.044.
- ↑ Esvelt KM; Smidler AL; Catteruccia F; Church GM (Jul 2014). «Concerning RNA-guided gene drives for the alteration of wild populations». eLife 3: e03401. PMID 25035423. doi:10.7554/eLife.03401.
- ↑ Cyranoski, David; Reardon, Sara (22 de abril de 2015). «Chinese scientists genetically modify human embryos». Nature. doi:10.1038/nature.2015.17378.
- ↑ Anders, Carolin; Niewoehner, Ole; Duerst, Alessia; Jinek, Martin. «Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease». Nature 513 (7519): 569-573. PMC 4176945. PMID 25079318. doi:10.1038/nature13579.
- ↑ Wiedenheft B; Sternberg SH; Doudna JA (Feb 2012). «RNA-guided genetic silencing systems in bacteria and archaea». Nature 482 (7385): 331-8. Bibcode:2012Natur.482..331W. PMID 22337052. doi:10.1038/nature10886.
- ↑ Ran FA; Hsu PD; Wright J; Agarwala V; Scott DA; Zhang F (2013). «Genome engineering using the CRISPR-Cas9 system». Nat Protoc 8 (11): 2281-308. PMC 3969860. PMID 24157548. doi:10.1038/nprot.2013.143.
- ↑ Jinek M; Chylinski K; Fonfara I; Hauer M; Doudna JA; Charpentier E (2012). «A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity». Science 337 (6096): 816-21. Bibcode:2012Sci...337..816J. PMID 22745249. doi:10.1126/science.1225829.
- ↑ Jinek M; Jiang F; Taylor DW; Sternberg SH; Kaya E; Ma E; Anders C; Hauer M; Zhou K; Lin S; Kaplan M; Iavarone AT; Charpentier E; Nogales E; Doudna JA (Mar 2014). «Structures of Cas9 endonucleases reveal RNA-mediated conformational activation». Science 343 (6176): 1247997. PMC 4184034. PMID 24505130. doi:10.1126/science.1247997.
- ↑ a b Anders C; Niewoehner O; Duerst A; Jinek M (Sep 2014). «Structural basis of PAM-dependent target DNA recognition by the Cas9 endonuclease». Nature 513 (7519): 569-73. Bibcode:2014Natur.513..569A. PMC 4176945. PMID 25079318. doi:10.1038/nature13579.
- ↑ Nishimasu H; Ran FA; Hsu PD; Konermann S; Shehata SI; Dohmae N; Ishitani R; Zhang F et al. (Feb 2014). «Crystal structure of Cas9 in complex with guide RNA and target DNA». Cell 156 (5): 935-49. PMC 4139937. PMID 24529477. doi:10.1016/j.cell.2014.02.001.
- ↑ a b Jiang F; Zhou K; Ma L; Gressel S; Doudna JA (Jun 2015). «A Cas9-guide RNA complex preorganized for target DNA recognition». Science 348 (6242): 1477-81. PMID 26113724. doi:10.1126/science.aab1452.
- ↑ Jiang F; Taylor DW; Chen JS; Kornfeld JE; Zhou K; Thompson AJ; Nogales E; Doudna JA (Feb 2016). «Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage». Science 351 (6275): 867-71. PMC 5111852. PMID 26841432. doi:10.1126/science.aad8282.
- ↑ Nishimasu H; Ran FA; Hsu PD; Konermann S; Shehata SI; Dohmae N; Ishitani R; Zhang F et al. (Feb 2014). «Crystal structure of Cas9 in complex with guide RNA and target DNA». Cell 156 (5): 935-49. PMC 4139937. PMID 24529477. doi:10.1016/j.cell.2014.02.001.
Lectura adicional
[editar]- Kennedy EM; Cullen BR (2015). «Bacterial CRISPR/Cas DNA endonucleases: A revolutionary technology that could dramatically impact viral research and treatment». Virology. 479-480: 213-20. PMID 25759096. doi:10.1016/j.virol.2015.02.024."Bacterial CRISPR/Cas ADN endonucleases: Una tecnología revolucionaria que dramáticamente podría impactar viral búsqueda y tratamiento". Virología. 479-480: 213@–20. doi:10.1016/j.virol.2015.02.024. PMID 25759096.
- Ian M. Slaymaker; Linyi Gao; Bernd Zetsche; David Un. Scott; Winston X. Yan; Feng Zhang (2015). Ian M. Slaymaker; Linyi Gao; Bernd Zetsche; David A. Scott; Winston X. Yan; Feng Zhang (2015). «Rationally engineered Cas9 nucleases with improved specificity». Science: Published online. doi:10.1126/science.aad5227.Ciencia: Publicado en línea. doi:10.1126/ciencia.aad5227.
- CRISPR-Cas: Un Manual de Laboratorio Editado por Jennifer Doudna, Universidad de California, Berkeley; Prashant Malí, Universidad de California, San Diego
- : Novel CRISPR/Cas9 Monoclonal Anticuerpo Diagenode lanza la industria primer monoclonal el anticuerpo que apunta CRISPR/Cas9
