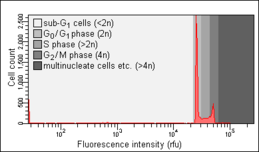
Análisis del ciclo celular
El análisis del ciclo celular mediante la medición del contenido de ADN es un método que emplea con mayor frecuencia la citometría de flujo para distinguir células en diferentes fases del ciclo celular. Antes del análisis, las células suelen permeabilizarse y tratarse con un tinte fluorescente que tiñe cuantitativamente el ADN, como yoduro de propidio (PI) o 4,6-diamidino-2-fenilindol (DAPI). La intensidad de la fluorescencia de las células teñidas se correlaciona con la cantidad de ADN que contienen. A medida que el contenido de ADN se duplica durante la fase S, el contenido de ADN (y, por lo tanto, la intensidad de la fluorescencia) de las células en la fase G0 y la fase G1 (antes de S), en la fase S y en la fase G2 y la fase M (después de S) identifica la posición de la fase del ciclo celular en las fases principales (fase G0/G1 contra S contra G2 /M) del ciclo celular. El contenido de ADN celular de las células individuales a menudo se representa como su histograma de frecuencia para proporcionar información sobre la frecuencia relativa (porcentaje) de las células en las fases principales del ciclo celular.
Las anomalías del ciclo celular reveladas en el histograma de frecuencia del contenido de ADN a menudo se observan después de diferentes tipos de daño celular, por ejemplo, el daño del ADN que interrumpe la progresión del ciclo celular en determinados puntos de control. Esta detención de la progresión del ciclo celular puede conducir a una reparación eficaz del ADN, que puede evitar la transformación de células normales en células cancerosas (carcinogénesis), o a la muerte celular, a menudo por apoptosis. A menudo se observa una detención de células en G0 o G1 como resultado de la falta de nutrientes (factores de crecimiento), por ejemplo, después de la privación de suero. El análisis del ciclo celular fue descrito por primera vez en 1969 en el Laboratorio Científico de Los Álamos por un grupo de la Universidad de California utilizando la técnica de tinción de Feulgen.[1] El primer protocolo para el análisis del ciclo celular mediante la tinción con yoduro de propidio fue presentado en 1975 por Awtar Krishan de la Escuela de Medicina de Harvard, y todavía se cita ampliamente en la actualidad.[2]
El análisis del ciclo celular incluye, además de la medición del contenido de ADN celular, otros componentes relacionados con el ciclo celular. La medición simultánea del contenido de ADN y ARN celular, o la susceptibilidad del ADN a la desnaturalización a bajo pH, utilizando el colorante metacromático naranja de acridina, revela los compartimentos del ciclo celular G1Q, G1A y G1B y también permite discriminar entre S, G2 y células mitóticas.[3] Las células en G1Q están inactivas, retiradas temporalmente del ciclo celular (también identificables como G0 ), las G1A están en la fase de crecimiento mientras que las G1B son las células justo antes de entrar en S, con su crecimiento (contenido de ARN y proteínas, tamaño) similar al de las células que inician la replicación del ADN. Los compartimentos similares del ciclo celular también se reconocen mediante un análisis multiparamétrico que incluye la medición de la expresión de ciclina D1, ciclina E, ciclina A y ciclina B1, cada una en relación con el contenido de ADN[4]. La medición simultánea del contenido de ADN y de la incorporación del precursor de ADN 5- bromo-2'-desoxiuridina (BrdU) por citometría de flujo es un ensayo especialmente útil, que ha sido ampliamente utilizado en el análisis del ciclo celular in vitro e in vivo.[5] Sin embargo, la incorporación de 5-etinil-2'-desoxiuridina (EdU), el precursor cuya detección ofrece ciertas ventajas sobre BrdU, ahora se ha convertido en la metodología preferida para detectar células replicantes de ADN (fase S).[6]
Procedimiento experimental[editar]
A menos que la tinción se realice con Hoechst 33342, el primer paso en la preparación de células para el análisis del ciclo celular es la permeabilización de las membranas plasmáticas de las células. Esto generalmente se hace incubándolos en una solución tampón que contiene un detergente suave[7] como Triton X-100 o NP-40, o fijándolos en etanol. La mayoría de los tintes fluorescentes de ADN (una de las excepciones es Hoechst 33342) no son permeables a la membrana plasmática, es decir, no pueden atravesar una membrana celular intacta. Por lo tanto, la permeabilización es crucial para el éxito del siguiente paso, la tinción de las células.
Antes (o durante el paso de tinción), las células a menudo se tratan con ARNasa A para eliminar los ARN. Esto es importante porque ciertos tintes que tiñen el ADN también tiñen el ARN, creando así artefactos que distorsionarían los resultados. Una excepción es el fluorocromo metacromático naranja de acridina, que bajo el protocolo de tinción específico puede teñir diferencialmente tanto el ARN (generando luminiscencia roja) como el ADN (fluorescencia verde), o en otro protocolo, después de la eliminación del ARN y la desnaturalización parcial del ADN, para teñir diferencialmente ADN de doble cadena (fluorescencia verde) frente a ADN de cadena sencilla (luminiscencia roja) [3]. Además del yoduro de propidio y la naranja de acridina, los tintes cuantificables que se usan con frecuencia incluyen (pero no se limitan a) DRAQ5, 7-aminoactinomicina D, DAPI y Hoechst 33342.
Discriminación de doblete[editar]
Dado que las células, y especialmente las células fijas, tienden a pegarse, los agregados de células deben excluirse del análisis mediante un proceso denominado discriminación de dobletes. Esto es importante porque un doblete de dos células G0 /G1 tiene el mismo contenido total de ADN y, por lo tanto, la misma intensidad de fluorescencia que una sola célula G2 /M.[8][9] A menos que se reconozcan como tales, los dobletes G0 /G1 contribuirían a la identificación y recuento de falsos positivos de células G2 /M.
Métodos relacionados[editar]
Ensayo de Nicoletti[editar]
El ensayo Nicoletti, llamado así por su inventor, el médico italiano Ildo Nicoletti, es una forma modificada de análisis del ciclo celular. Es utilizado para detectar y cuantificar la apoptosis, una forma de muerte celular programada, mediante el análisis de células con un contenido de ADN inferior a 2n ("células sub-G0/G1"). Estas células suelen ser el resultado de la fragmentación apoptótica del ADN: durante la apoptosis, las endonucleasas celulares degradan el ADN. Por lo tanto, los núcleos de las células apoptóticas contienen menos ADN que los núcleos de las células G0 /G1 sanas, lo que da como resultado un pico sub-G0 /G1 en el histograma de fluorescencia que se puede usar para determinar la cantidad relativa de células apoptóticas en una muestra. Este método fue desarrollado y descrito por primera vez en 1991 por Nicoletti y colaboradores en la Facultad de Medicina de la Universidad de Perugia.[10] En 2006 se publicó un protocolo optimizado desarrollado por dos de los autores de la publicación original.[11] Los objetos medidos dentro del pico sub-G0G1, con un contenido de ADN inferior al 5% del pico G0G1, con toda probabilidad son cuerpos apoptóticos y, por lo tanto, no representan células apoptóticas individuales.[12]
Referencias[editar]
- ↑ Van Dilla MA, Trujillo TT, Mullaney PF, Coulter JR (14 de marzo de 1969). «Cell Microfluorometry: A Method for Rapid Fluorescence Measurement». Science 163 (3872): 1213-1214. Bibcode:1969Sci...163.1213V. PMID 5812751. doi:10.1126/science.163.3872.1213.
- ↑ Krishan A. (July 1975). «Rapid flow cytofluorometric analysis of mammalian cell cycle by propidium iodide staining». The Journal of Cell Biology 66 (1): 188-193. PMC 2109516. PMID 49354. doi:10.1083/jcb.66.1.188.
- ↑ Darzynkiewicz Z, Traganos F, Melamed MR (1980). «New cell cycle compartments identified by multiparameter flow cytometry». Cytometry 1 (2): 98-108. PMID 6170495. doi:10.1002/cyto.990010203.
- ↑ Darzynkiewicz Z, Gong JP, Juan G, Ardelt B, Traganos F (1996). «Cytometry of cyclin proteins». Cytometry 25 (1): 1-13. PMID 8875049. doi:10.1002/(SICI)1097-0320(19960901)25:1<1::AID-CYTO1>3.0.CO;2-N.
- ↑ Gray JW, Dolbeare F, Pallavicini MG, Beisker W, Waldman F (1986). «Cell cycle analysis using flow cytometry». Int J Radiat Biol Relat Stud Phys Chem Med. 49 (2): 237-55. PMID 3510993. doi:10.1080/09553008514552531.
- ↑ Buck SB, Bradford J, Gee KR, Agnew BJ, Clarke ST, Salic A (2008). «Detection of S-phase cell cycle progression using 5-ethynyl-2'-deoxyuridine incorporation with click chemistry, an alternative to using 5-bromo-2'-deoxyuridine antibodies». BioTechniques 44 (7): 927-9. PMID 18533904. doi:10.2144/000112812.
- ↑ Vindeløv LL, Christensen IJ, Nissen NI (March 1983). «A detergent-trypsin method for the preparation of nuclei for flow cytometric DNA analysis». Cytometry 3 (5): 323-327. PMID 6188586. doi:10.1002/cyto.990030503.
- ↑ Sharpless T, Traganos F, Darzynkiewicz Z, Melamed MR (1975). «Flow cytofluorimetry: discrimination between single cells and cell aggregates by direct size measurements». Acta Cytol 19 (6): 577-81. PMID 1108568.
- ↑ Wersto RP, Chrest FJ, Leary JF, Morris C, Stetler-Stevenson MA, Gabrielson E (15 de octubre de 2001). «Doublet discrimination in DNA cell-cycle analysis». Cytometry 46 (5): 296-306. PMID 11746105. doi:10.1002/cyto.1171.
- ↑ Nicoletti I, Migliorati G, Pagliacci MC, Grignani F, Riccardi C (3 de junio de 1991). «A rapid and simple method for measuring thymocyte apoptosis by propidium iodide staining and flow cytometry». Journal of Immunological Methods 139 (2): 271-279. PMID 1710634. doi:10.1016/0022-1759(91)90198-O.
- ↑ Riccardi C, Nicoletti I (9 de noviembre de 2006). «Analysis of apoptosis by propidium iodide staining and flow cytometry». Nature Protocols 1 (3): 1458-1461. PMID 17406435. doi:10.1038/nprot.2006.238.
- ↑ Darzynkiewicz Z, Bedner E, Traganos F (2001). «Difficulties and pitfalls in analysis of apoptosis». Methods Cell Biol 63: 527-559. PMID 11060857. doi:10.1016/s0091-679x(01)63028-0.
Otras lecturas[editar]
- «Cell Cycle Basics» (PDF, 0.1 MB). University College London. Archivado desde el original el 6 de junio de 2011. Consultado el 20 de mayo de 2010.
- Rabinovitch, Peter. «Introduction to Cell Cycle Analysis» (PDF, 0.5 MB). Phoenix Flow Systems, Inc. Consultado el 20 de mayo de 2010.