Pirosecuenciación
La pirosecuenciación es una tecnología que permite determinar el orden de una secuencia de ADN mediante luminiscencia. Se basa en el principio de "secuenciación por síntesis" en el que se detecta la incorporación de nucleótidos al ADN por acción de la ADN polimerasa. La particularidad de este método es que detecta la liberación de pirofosfato cuando los nucleótidos son incorporados. A partir del pirofosfato se generará luz por medio de diversas reacciones enzimáticas en cadena.
Fundamentos - Luminiscencia[editar]
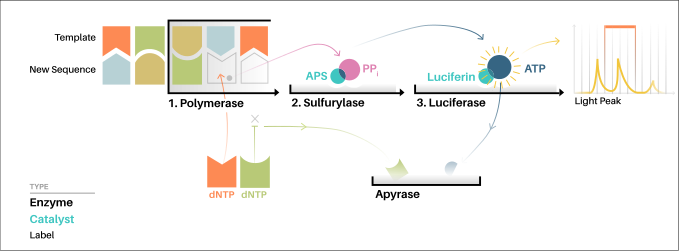
El ADN se desnaturaliza para obtener ADN monocatenario y se hibridará con un cebador. En el tubo de reacción se encontrarán las enzimas luciferasa, ATP sulfurilasa, ADN polimerasa y apirasa además de los sustratos luciferina y adenosín-5-fosfosulfato (APS).
A continuación se añaden los nucleótidos uno después del otro, al contrario que en una reacción de secuenciación normal donde se añaden todos a la vez.
1. Si el nucleótido añadido al medio de reacción corresponde al que necesita la polimerasa, se incorpora a la cadena que se está sintetizando (elongamiento) y se libera un pirofosfato.
2. Posteriormente, una ATP sulfurilasa en presencia del APS transforma este pirofosfato (PPi) en ATP , que se acoplará a la luciferina, formando un complejo el cual será utilizado por una luciferasa.
3. Como resultado se produce oxiluciferina y una señal luminosa de intensidad proporcional al número de nucleótidos incorporados.Es el secuenciador o más precisamente el sensor CCD, el que captura esta señal lumínica y lo reproduce como un pico en un pirograma.
Finalmente, una apirasa se encarga de degradar aquellos nucleótidos que no han sido incorporados y quedan sueltos con el fin de que al añadir un nuevo dNTP, no existan restos de reacciones anteriores.
De esta forma se puede deducir la secuencia partiendo del tamaño de los picos obtenidos. Si hay un polimorfismo en una secuencia no habría problemas para detectarlo pues el tamaño de los picos también es proporcional a las cadenas portadoras de un nucleótido u otro.
Secuenciación 454[editar]
Es una variante de la pirosecuenciación descrita por Jonathan Rothberg y colaboradores de la empresa 454 Life Sciences en 2005.[1] Esta técnica permite secuenciar de forma paralela y masiva muchos fragmentos de ADN a la vez. De este modo la secuenciación 454 se convirtió en la primera tecnología de la next-generation sequencing suponiendo una gran reducción en los precios de la secuenciación de genomas completos. En 2013 la plataforma de secuenciación 454 dejó de ser competitiva y la compañía Roche, que había adquirido 454 Life Science en 2007, decidió dejar de comercializarla.[2]
Procedimiento:
- Fragmentar el ADN de doble cadena a secuenciar mediante un proceso conocido como nebulización para así poder marcar los extremos con dos primers. Uno de ellos se va adherir a las microesferas y el otro se usará para comenzar la amplificación. Finalmente, se separan los fragmentos del ADN de doble cadena en su forma monocatenaria.
- Mediante una emulsión de aceite se generan microesferas, dónde se unirá un único fragmento de ADN monocatenario. Luego se añade una emulsión que contiene los microrreactores. En cada microrreactor se encuentran los elementos necesarios para la reacción de PCR: oligonucleótidos, dNTPs, polimerasa y un único fragmento de ADN molde.
- Mediante la amplificación por PCR, se generan muchas copias del mismo fragmento de ADN original y los fragmentos amplificados resultantes seguirán inmovilizados en las microesferas.
- Después se liberan las cuentas de la emulsión y se colocan en placas donde ocurrirá la secuenciación. Primero se vierte la emulsión sobre una fase sólida con celdillas en el cual solo cabe una microesfera. Luego añadimos las enzimas sulfurilasa y luciferasa.
- En cada una de estas celdas se produce una pirosecuenciación. La secuenciación completa se lleva a cabo añadiendo uno de los cuatro nucleótidos y observando qué celdilla se ilumina. Después de cada adición de nucleótidos se lava y se añade el siguiente nucleótido. Este proceso se repite hasta completar la secuencia.
- Los resultados se compararán con un genoma de referencia.
Aplicaciones[editar]

Es una técnica que se utiliza mucho en el diagnóstico molecular, sobre todo cuando ya se sabe de antemano la secuencia que se desea obtener, aunque también se utiliza para obtener la secuencia de un ADN problema. En el caso de no conocer la secuencia en particular, la máquina va liberando por orden las bases A, T, G, C. El resultado nos otorgará un patrón de picos que tendremos que interpretar. En el caso en que queramos detectar una mutación puntual conociendo de antemano la secuencia, lo que haremos será liberar las bases que sabemos que debe tener nuestra secuencia por orden, y veremos dónde se encuentra la mutación en nuestro paciente. Esto nos permitirá ver si el paciente es homocigoto sano o enfermo, o si es heterocigoto.
Implicancias[editar]
Su bajo coste y velocidad, que permitió a la compañía secuenciar el código de un adenovirus en 2003 en un solo día,[3] plantea la posibilidad de secuenciar genomas completos de forma rutinaria, lo cual es de especial interés en biomedicina. De hecho, el genoma de James Watson, codescubridor de la estructura del ADN, fue descrito y publicado mediante esta técnica.[4]
Además, esta técnica se emplea para realizar tipajes de organismos infecciosos y para tipajes HLA.[5]
Ventajas y limitaciones[editar]
Algunas de las ventajas de la pirosecuenciación son que no es necesario usar geles, marcadores fluorescentes o didesoxinucleótidos lo que supone una mayor rapidez y un menor coste que la secuenciación de Sanger. No obstante, solo permite lecturas de entre 200-400 pares de bases lo que puede complicar el ensamblaje de secuencias de ADN repetitivo para la secuenciación de genomas completos.
Referencias[editar]
- ↑ Marguiles et al (2005) “Genome sequencing in microfabricated high-density picolitre reactors” Nature 437, 376-380. https://doi.org/doi:10.1038/nature03959;
- ↑ Hollmer, Mark (17 de octubre de 2013). «Roche to close 454 Life Sciences as it reduces gene sequencing focus». Fierce Biotech.
- ↑ Cristina de Martos. «Más cerca de la secuenciación personalizada del ADN». Consultado el 15 de diciembre de 2007.
- ↑ «James Watson's Personal Genome Sequence» (en inglés). Archivado desde el original el 3 de agosto de 2012.
- ↑ Ramon, Daniel et al. “Pyrosequencing trade mark : A one-step method for high resolution HLA typing.” Journal of translational medicine vol. 1,1 9. 26 Nov. 2003, doi:10.1186/1479-5876-1-9
Enlaces externos[editar]
