Microbiota pulmonar
La microbiota pulmonar es la comunidad microbiana pulmonar que consiste en una compleja variedad de microorganismos que se encuentran en el tracto respiratorio inferior, particularmente en la capa mucosa y las superficies epiteliales. Estos microorganismos incluyen bacterias, hongos, virus y bacteriófagos. La parte bacteriana de la microbiota se ha estudiado más de cerca. Se compone de un núcleo de nueve géneros: Prevotella, Sphingomonas, Pseudomonas, Acinetobacter, Fusobacterium, Megasphaera, Veillonella, Staphylococcus y Streptococcus.[1][2][3] Son aerobios, así como anaerobios y bacterias aerotolerantes. Las comunidades microbianas son muy variables en individuos particulares y se componen de alrededor de 140 familias distintas. El árbol bronquial, por ejemplo, contiene una media de 2000 genomas bacterianos por cm² de superficie. Las bacterias dañinas o potencialmente dañinas también se detectan rutinariamente en muestras respiratorias. Los más significativos son Moraxella catarrhalis, Haemophilus influenzae y Streptococcus pneumoniae. Se sabe que causan trastornos respiratorios en condiciones particulares, es decir, si el sistema inmunitario humano está dañado. Se desconoce el mecanismo por el cual persisten en las vías aéreas inferiores en individuos sanos.
Los géneros de hongos que se encuentran comúnmente conforman el micobioma pulmonar, en la microbiota del pulmón, e incluyen Candida, Malassezia, Neosartorya, Saccharomyces y Aspergillus, entre otros.[4][5]
Papel de la barrera epitelial[editar]
El epitelio de la vía aérea junto con los macrófagos alveolares y las células dendríticas juegan un papel importante en el reconocimiento inicial de los productos bacterianos que ingresan a las vías aéreas inferiores con el aire. Dado que algunos de estos productos son potentes estímulos proinflamatorios, es extremadamente importante que el sistema inmune distinga entre patógenos y comensales no patógenos. Esto evita el desarrollo de inflamación constante y forma tolerancia contra la microbiota inofensiva.[6]
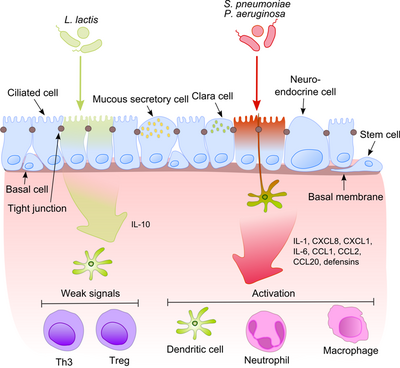
Este proceso se vuelve mucho más intrigante cuando se tiene en cuenta que los comensales a menudo comparten sus moléculas de superficie con los patógenos. Las células epiteliales están equipadas con herramientas de reconocimiento muy sensibles: receptores de tipo Toll (TLR), receptores similares al dominio de oligomerización de unión a nucleótidos (NOD) (NLR) y receptores similares al gen inducible por ácido retinoico (RIG) (RLR) que reconocen Una amplia variedad de componentes estructurales microbianos. Después del reconocimiento de las bacterias patógenas, se activan las vías proinflamatorias y los componentes celulares de la inmunidad adaptativa e innata se reclutan en el sitio de la infección.[7] Un regulador clave en este proceso es el NF-κB que se transloca desde el citoplasma al núcleo y activa genes proinflamatorios en las células epiteliales y los macrófagos. El complejo proteico de unión al ADN reconoce una secuencia de nucleótidos discreta (5'-GGG ACT TTC T-3 ') en la región aguas arriba de una variedad de genes de respuesta. La activación de NF-κB por una serie de estímulos: paredes celulares bacterianas o citocinas inflamatorias da como resultado su translocación al núcleo.
En contraste, las bacterias inofensivas no causan la translocación de NF-κB en el núcleo, evitando así la inflamación, aunque pueden expresar los mismos patrones moleculares asociados a microbios (MAMP). Neish sugirió un posible mecanismo que explica este efecto, mostrando que S. typhimurium PhoPc y S. pullorum no patógenos pueden prohibir la ubiquitinación del factor nuclear de la molécula inhibidora de NF-κB del potenciador del gen del polipéptido NF-κB en el inhibidor de células B alfa (IκB-κ).[8] Otra explicación de la tolerancia comensal del epitelio se refiere a la modificación postraduccional de una proteína mediante la unión covalente de uno o más monómeros de ubiquitina (Ub). La inhibición de la ubiquitinación conduce a la reducción de la inflamación, porque solo el poliubiquitinado (IκB-κ está dirigido a la degradación por el proteasoma 26 S, lo que permite la translocación de NF-κB al núcleo y la activación de la transcripción de genes efectores (por ejemplo, IL-8). Las bacterias probióticas, como los lactobacilos, pueden modular la actividad del sistema Ub-proteasoma mediante la inducción de la producción de especies reactivas de oxígeno (ROS) en las células epiteliales. En las células de mamíferos, se ha demostrado que las ROS sirven como segundos mensajeros críticos en múltiples vías de transducción de señales en respuesta a las citocinas proinflamatorias. Las ROS inducidas bacterianamente provocan la inactivación oxidativa del residuo de cisteína catalítica de Ub 12 que da como resultado una pérdida incompleta pero transitoria de nulilación de cullina-1 y los consiguientes efectos sobre la señalización de NF-κB y β-catenina. Otra especie comensal, B. thetaiotaomicron, atenúa la expresión de citocinas proinflamatorias al promover la exportación nuclear de la subunidad NF-κB RelA, a través de una vía dependiente del receptor γ (PPAR-γ) activado por el proliferador de peroxisomas. El PPAR-γ se dirige a Rel A transcripcionalmente activo e induce un aclaramiento nuclear temprano que limita la duración de la acción de NF-κB.
El equilibrio entre patógenos y comensales es extremadamente importante en el mantenimiento de la homeostasis en el tracto respiratorio.
Fisiología[editar]
Las vías respiratorias están continuamente expuestas a una multitud de microorganismos, algunos de los cuales pueden persistir e incluso colonizar las vías respiratorias. Esto es posible debido a la presencia de nutrientes, oxígeno y temperatura óptima de crecimiento. Existen varias fuentes de nutrientes derivadas del huésped para los residentes de microbios: secreciones de las células epiteliales de las vías respiratorias (especialmente las células caliciformes), secreciones de las glándulas submucosas y transudados del plasma. Además, el conjunto de nutrientes disponibles se ve incrementado por las actividades de algunos miembros de la microbiota. Los componentes macromoleculares de las secreciones respiratorias (proteínas, glicoproteínas, lípidos, ácidos nucleicos) se convierten en nutrientes (por ejemplo, carbohidratos, aminoácidos). Por lo tanto, la actividad metabólica de las bacterias presentes permite la colonización de nuevas especies. Las bacterias comensales no son patógenas y defienden nuestras vías respiratorias contra los patógenos. Hay varios mecanismos posibles. Los comensales son los competidores nativos de las bacterias patógenas, porque tienden a ocupar el mismo nicho ecológico dentro del cuerpo humano. En segundo lugar, pueden producir sustancias antibacterianas llamadas bacteriocinas que inhiben el crecimiento de patógenos.
Los géneros Bacillus, Lactobacillus, Lactococcus, Staphylococcus, Streptococcus y Streptomyces son los principales productores de bacteriocinas en las vías respiratorias. Además, se sabe que los comensales inducen la respuesta Th1 y la interleucina (IL) -10 antiinflamatoria, péptidos antimicrobianos, FOXP3, producción de inmunoglobulina A (sIgA).
Significación clínica[editar]
Los cambios en la composición de la comunidad microbiana parecen desempeñar un papel en la progresión de trastornos pulmonares como la enfermedad pulmonar obstructiva crónica (EPOC), el asma y la fibrosis quística.[9][10] En humanos, S. aureus es parte de la microbiota normal presente en el tracto respiratorio superior,[11] y en la piel y en la mucosa intestinal.[12] S. aureus, junto con especies similares que pueden colonizar y actuar simbióticamente pero pueden causar enfermedades si comienzan a tomar los tejidos que han colonizado o invaden otros tejidos, se les ha llamado "patobiontes". MRSA puede colonizar de manera similar a las personas sin enfermarlas.[13] La presencia de géneros tales como Mycoplasma, Pseudomonas y Staphylococcus se correlaciona con el estado estable de EPOC. Por otro lado, Prevotella, Mesorhizobium, Microbacterium, Micrococcus, Veillonella, Rhizobium, Stenotrophomonas y Lactococcus están presentes principalmente en una cohorte individual sana. La abundancia relativa de proteobacterias aumenta en niños asmáticos. Pseudomonas aeruginosa, Staphylococcus aureus y Burkholderia cepacia se encuentran con mayor frecuencia en pacientes con fibrosis quística.
La secuenciación de alto rendimiento y los enfoques de secuenciación del genoma completo proporcionarán más información sobre la complejidad y la implicación fisiológica de las bacterias comensales en el tracto respiratorio inferior.
Véase también[editar]
Referencias[editar]
- ↑ Erb-Downward, John R.; Thompson, Deborah L.; Han, Meilan K.; Freeman, Christine M.; McCloskey, Lisa; Schmidt, Lindsay A.; Young, Vincent B.; Toews, Galen B. et al. (2011). «Analysis of the Lung Microbiome in the "Healthy" Smoker and in COPD». En Bereswill, Stefan, ed. PLoS ONE 6 (2): e16384. PMC 3043049. PMID 21364979. doi:10.1371/journal.pone.0016384.
- ↑ Hilty, Markus; Burke, Conor; Pedro, Helder; Cardenas, Paul; Bush, Andy; Bossley, Cara; Davies, Jane; Ervine, Aaron et al. (2010). «Disordered Microbial Communities in Asthmatic Airways». En Neyrolles, Olivier, ed. PLoS ONE 5 (1): e8578. PMC 2798952. PMID 20052417. doi:10.1371/journal.pone.0008578.
- ↑ Beck, James M.; Young, Vincent B.; Huffnagle, Gary B. (1 de febrero de 2012). «The microbiome of the lung». Translational Research 160 (4): 258-66. PMC 3440512. PMID 22683412. doi:10.1016/j.trsl.2012.02.005.
- ↑ «The human mycobiome in health and disease». Genome Med 5 (7): 63. July 2013. PMC 3978422. PMID 23899327. doi:10.1186/gm467. «Figure 2: Distribution of fungal genera in different body sites».
- ↑ Richardson, M; Bowyer, P; Sabino, R (1 de abril de 2019). «The human lung and Aspergillus: You are what you breathe in?». Medical mycology 57 (Supplement_2): S145-S154. PMID 30816978. doi:10.1093/mmy/myy149.
- ↑ Kumar, Himanshu; Kawai, Taro; Akira, Shizuo (2011). «Pathogen Recognition by the Innate Immune System». International Reviews of Immunology 30 (1): 16-34. PMID 21235323. doi:10.3109/08830185.2010.529976.
- ↑ Sha, Q.; Truong-Tran, AQ; Plitt, JR; Beck, LA; Schleimer, RP (2004). «Activation of Airway Epithelial Cells by Toll-Like Receptor Agonists». American Journal of Respiratory Cell and Molecular Biology 31 (3): 358-64. PMID 15191912. doi:10.1165/rcmb.2003-0388OC.
- ↑ Kumar, Amrita; Wu, Huixia; Collier-Hyams, Lauren S; Hansen, Jason M; Li, Tengguo; Yamoah, Kosj; Pan, Zhen-Qiang; Jones, Dean P et al. (2007). «Commensal bacteria modulate cullin-dependent signaling via generation of reactive oxygen species». The EMBO Journal 26 (21): 4457-66. PMC 2063476. PMID 17914462. doi:10.1038/sj.emboj.7601867.
- ↑ Huang, Yvonne J.; Kim, Eugenia; Cox, Michael J.; Brodie, Eoin L.; Brown, Ron; Wiener-Kronish, Jeanine P.; Lynch, Susan V. (2010). «A Persistent and Diverse Airway Microbiota Present during Chronic Obstructive Pulmonary Disease Exacerbations». OMICS: A Journal of Integrative Biology 14 (1): 9-59. PMC 3116451. PMID 20141328. doi:10.1089/omi.2009.0100.
- ↑ Cox, Michael J.; Allgaier, Martin; Taylor, Byron; Baek, Marshall S.; Huang, Yvonne J.; Daly, Rebecca A.; Karaoz, Ulas; Andersen, Gary L. et al. (2010). «Airway Microbiota and Pathogen Abundance in Age-Stratified Cystic Fibrosis Patients». En Ratner, Adam J., ed. PLoS ONE 5 (6): e11044. PMC 2890402. PMID 20585638. doi:10.1371/journal.pone.0011044.
- ↑ Schenck, LP; Surette, MG; Bowdish, DM (November 2016). «Composition and immunological significance of the upper respiratory tract microbiota.». FEBS Letters 590 (21): 3705-3720. PMID 27730630. doi:10.1002/1873-3468.12455.
- ↑ Wollina, U (2017). «Microbiome in atopic dermatitis.». Clinical, Cosmetic and Investigational Dermatology 10: 51-56. PMC 5327846. PMID 28260936. doi:10.2147/ccid.s130013.
- ↑ Uhlemann, AC; Otto, M; Lowy, FD; DeLeo, FR (January 2014). «Evolution of community- and healthcare-associated methicillin-resistant Staphylococcus aureus.». Infection, Genetics and Evolution 21: 563-74. PMC 3884050. PMID 23648426. doi:10.1016/j.meegid.2013.04.030.
