Diferencia entre revisiones de «Evolución del lobo»
m →Canis mosbachensis: ; ortografía |
error, Vallonnet es con dos enes. |
||
| Línea 313: | Línea 313: | ||
<ref name=leonard2014>{{cite journal|author=Leonard, Jennifer|year=2014|title=Ecology drives evolution in grey wolves|journal=Evolution Ecology Research|volume=16|pages=461–473| url=http://www.evolutionary-ecology.com/open/v16n06/eear2913.pdf}}</ref> |
<ref name=leonard2014>{{cite journal|author=Leonard, Jennifer|year=2014|title=Ecology drives evolution in grey wolves|journal=Evolution Ecology Research|volume=16|pages=461–473| url=http://www.evolutionary-ecology.com/open/v16n06/eear2913.pdf}}</ref> |
||
<ref name=li1997>{{cite book|last=Li|first=Wen-Hsiung|year=1997|title=Molecular evolution|location=Sunderland, Massachusetts|publisher=Sinauer Associates|ISBN=978-0-87893-463-8}}{{page needed|date=July 2016}}</ref> |
<ref name=li1997>{{cite book|last=Li|first=Wen-Hsiung|year=1997|title=Molecular evolution|location=Sunderland, Massachusetts|publisher=Sinauer Associates|ISBN=978-0-87893-463-8}}{{page needed|date=July 2016}}</ref> |
||
<ref name=lumley1988>Lumley, H. de, Kahlke, H.D., Moigne, A.M., Moulle, P.E., 1988. Les faunes de grands mammifères de la grotte du |
<ref name=lumley1988>Lumley, H. de, Kahlke, H.D., Moigne, A.M., Moulle, P.E., 1988. Les faunes de grands mammifères de la grotte du Vallonnet Roquebrune-Cap-Martin, Alpes-Maritimes. L’Anthropologie 92, 465–496.</ref> |
||
<ref name=lindblad2005>{{Cite journal | last1 = Lindblad-Toh | first1 = K. | last2 = Wade | first2 = C. M. | last3 = Mikkelsen | first3 = T. S. | last4 = Karlsson | first4 = E. K. | last5 = Jaffe | first5 = D. B. | last6 = Kamal | first6 = M. | last7 = Clamp | first7 = M. | last8 = Chang | first8 = J. L. | last9 = Kulbokas | first9 = E. J. | last10 = Zody | doi = 10.1038/nature04338 | first10 = M. C. | last11 = Mauceli | first11 = E. | last12 = Xie | first12 = X. | last13 = Breen | first13 = M. | last14 = Wayne | first14 = R. K. | last15 = Ostrander | first15 = E. A. | last16 = Ponting | first16 = C. P. | last17 = Galibert | first17 = F. | last18 = Smith | first18 = D. R. | last19 = Dejong | first19 = P. J. | last20 = Kirkness | first20 = E. | last21 = Alvarez | first21 = P. | last22 = Biagi | first22 = T. | last23 = Brockman | first23 = W. | last24 = Butler | first24 = J. | last25 = Chin | first25 = C. W. | last26 = Cook | first26 = A. | last27 = Cuff | first27 = J. | last28 = Daly | first28 = M. J. | last29 = Decaprio | first29 = D. | last30 = Gnerre | first30 = S. | title = Genome sequence, comparative analysis and haplotype structure of the domestic dog | journal = Nature | volume = 438 | issue = 7069 | pages = 803–819 | year = 2005 | pmid = 16341006| pmc = | url =| bibcode = 2005Natur.438..803L | display-authors = 29 }}</ref> |
<ref name=lindblad2005>{{Cite journal | last1 = Lindblad-Toh | first1 = K. | last2 = Wade | first2 = C. M. | last3 = Mikkelsen | first3 = T. S. | last4 = Karlsson | first4 = E. K. | last5 = Jaffe | first5 = D. B. | last6 = Kamal | first6 = M. | last7 = Clamp | first7 = M. | last8 = Chang | first8 = J. L. | last9 = Kulbokas | first9 = E. J. | last10 = Zody | doi = 10.1038/nature04338 | first10 = M. C. | last11 = Mauceli | first11 = E. | last12 = Xie | first12 = X. | last13 = Breen | first13 = M. | last14 = Wayne | first14 = R. K. | last15 = Ostrander | first15 = E. A. | last16 = Ponting | first16 = C. P. | last17 = Galibert | first17 = F. | last18 = Smith | first18 = D. R. | last19 = Dejong | first19 = P. J. | last20 = Kirkness | first20 = E. | last21 = Alvarez | first21 = P. | last22 = Biagi | first22 = T. | last23 = Brockman | first23 = W. | last24 = Butler | first24 = J. | last25 = Chin | first25 = C. W. | last26 = Cook | first26 = A. | last27 = Cuff | first27 = J. | last28 = Daly | first28 = M. J. | last29 = Decaprio | first29 = D. | last30 = Gnerre | first30 = S. | title = Genome sequence, comparative analysis and haplotype structure of the domestic dog | journal = Nature | volume = 438 | issue = 7069 | pages = 803–819 | year = 2005 | pmid = 16341006| pmc = | url =| bibcode = 2005Natur.438..803L | display-authors = 29 }}</ref> |
||
Revisión del 09:25 19 mar 2017

La evolución del lobo tuvo lugar durante unos 800 000 años desde el Pleistoceno medio, cuando aparecen los primeros ejemplares morfológicamente similares al Canis lupus, hasta la aparición de las subespecies actuales Canis lupus dingo (dingo), Canis lupus lupus (lobo) y Canis lupus familiaris (perro). Los factores ecológicos, incluyendo el tipo de hábitat, el clima, la especialización en distintos tipos de presa y la competencia con otros depredadores has influenciado mucho la estructura genética de población de los lobos y su plasticidad craneo-dental. El número de lobos experimentó un brusco descenso —o «cuello de botella»— hace 20 000 años, lo que indica que muchas poblaciones de lobos se habían extinguido ya durante el último máximo glacial[cita requerida] y la expansión mundial de los humanos modernos con la tecnología para capturar caza mayor. El perro doméstico, el carnívoro más abundante en el mundo es descendiente de una población de lobos ya extinta[cita requerida].
Registro fósil
El registro fósil de vertebrados antiguos se compone de fragmentos dispersos de los que es a menudo imposible obtener material genético. Los investigadores, por tanto, recurren a los análisis morfológicos, pero es difícil estimar las variaciones entre los individuos de la misma y distintas especies y las relaciones entre especímenes en función del tiempo y de la ubicación. Existe debate sobre algunas observaciones, y las hipótesis apoyadas por algunos autores son rebatidas por otros.[2]
Hay un acuerdo general en los registros más antiguos, los cuales muestran que los feliformes y caniformes surgieron de la superfamilia Carnivoramorpha 43 millones de años antes del presente.[3] Entre los caniformes se incluye el género Leptocyon, de aspecto similar a los zorros, que existió hace 34 millones de años antes de ramificarse hace 11.9 millones de años en Vulpes (zorros) y Canis (coyotes, lobos y chacales). El eucyon, de tamaño parecido al de un chacal, existió en Norteamérica desde hace 10 millones de años, y hacia el Plioceno temprano, hace 5-6 millones de años, el Eucyon davisi,[4] similar al coyote, invadió Eurasia. Los primeros caninos aparecieron en América del Norte en el Mioceno —hace 6 millones de años—, concretamente en el suroeste de los Estados Unidos y México. Un millón de años más tarde, Canis lepophagus, de mayor tamaño, apareció en la misma región.[5]: p58
Los cánidos que habían migrado desde América del Norte a Eurasia -Eucyon, Vulpes y Nyctereutes—, eran depredadores de pequeño a mediano tamaño, que vivieron durante finales del Mioceno y principios del Plioceno, pero no eran superdepredadores. La posición de los cánidos en la cadena alimenticia cambió cuando los miembros de género Canis se volvieron los depredadores dominantes en el Holártico.
El C. chihilensis, con un tamaño similar al de un lobo moderno, apareció en el norte de China a mediados del Plioceno —hace 3-4 millones de años—. A continuación, la evolución del género se disparó por toda Eurasia a principios del Pleistoceno, hace 1.8 millones de años en lo que se conoce como «el evento del lobo», asociado con la formación del bioma llamado «estepa de mamuts», durante el último periodo glacial. Tres especies se expandieron por Europa: C. arnensis, C. eutruscus, y C. falconeri.[5]: p148
A pesar de las lagunas en el registro fósil, se considera probable que los lobos surgieran de un grupo de cánidos de pequeño tamaño.[6]: p241 La evidencia morfológica[6]: p239 [7] y genética [8] coinciden en que los lobos evolucionaron durante el Plioceno y a principios del Pleistoceno a partir del mismo linaje que también dio lugar a los coyotes.[6]: p239 Los especímenes fosilizados indican que el ancestro común de lobos y coyotes existió hace 1.5 millones de años,[6]: p240 [7] mientras que el ancestro de los chacales y de los otros miembros existentes del género ya se había separado del tronco común anteriormente.[6]: p240 A partir de la separación de los linajes de lobos y coyotes existen diferentes opiniones entre los paleontólogos sobre las relaciones entre las varias especies que conformaron la evolución del lobo.[6]: p240 Estas especies se describen a continuación.
Canis lepophagus
Canis lepophagus vivió a principios del Plioceno en Norteamérica[5]. Kurten propuso que evolucionó a partir de especies norteamericanas más pequeñas de Canis del Mioceno [9] que posteriormente se expandieron por Eurasia; se piensa que estaba cercanamente relacionado o era equivalente al C. arnensis de Europa.[6]: p241 [10][11]
Según Johnston, C. lepophagus tenía un cráneo y esqueleto más esbeltos que los del coyote moderno.[12]: 385 Nowak descubrió que las primeras poblaciones tenían cráneos pequeños, delicados y entrechos, similares a los de coyotes pequeños y que podrían ser los ancestros del C. latrans.[6]: p241 Johnson notó que algunos especímenes encontrados en el cañón Cita en Texas, tenían cráneos más grandes y amplios,[12] lo que llevó a Nowak a sugerir que estaban tranformándose en lobos.[6]: p241 [7]
Tedford discrepaba con estos autores por la ausencia de algunos rasgos en la morfología cráneo-dental de C. lepophagus compartidos por C. lupus y C. latrans, por lo que en su opinión no había una relación cercana con estas dos especies, aunque sugirió que era el ancestro tanto de lobos como de coyotes.[13]: p119
Canis priscolatrans
Canis priscolatrans habitó en Norteamérica a finales del Plioceno y principios del Pleistoceno.[7] El primer lobo como tal, apareció a finales del Blancano y principios del Irvintoniano,[6]: p240 [7][14] y era muy parecido o quizá idéntico al Canis edwardii.[6]: p241 [7]: 82 [15][16] Se parecía a C. rufus en el tamaño y proporciones del cráneo, pero con dentadura más compleja. [6]: p241 Sin embargo, los fósiles hallados de C. rufus no datan ese periodo, sino del finales del Rancholabreano.[6]: p242
Kurten no estaba seguro de si C. priscolatrans procedía de C. lepophagus y C. arnensis,[11] pero creía que era un ancestro de C. latrans. En su opinión, el C. arnensis de Europe mostraba similaridades notorias a C. priscolatrans, y ambas especies podrían representar lo que fue una población Holártica de coyotes. [10]: p27 Nowak se mostró en desacuerdo con esta interpretación y opinaba que que C. priscolatrans estaba relacionado con el C. etruscus. europeo[7] Posteriormente, Kurten propuso que tanto C. priscolatrans y C. etruscus eran parte de un grupo que dio origen a C. lupus pero no estaba seguro de si habían evolucionado de manera separada de C. lepophagus o de un descendiente de C. lepophagus.[11]
En el suoreste de los Estados Unidos se han hallado restos del Plioceno tardío de Canis edwardii, caracterizado por un tamaño similar al de coyotes grandes, junto con C. lepophagus, lo que indica una relación de descendencia.[5]: p60 Tedford reconoció la especie C. edwardii[17] y consideró que la morfología cráneo-dental de C. priscolatrans estaba dentro del rango de C. edwardii y quizá no serían especies distintas.[13]: p131
Canis ambrusteri
Durante el Pleistoceno medio, [7] los lobos de Norteamérica evolucionaron hacia un mayor tamaño y los restos de dientes hallados indican que C. priscolatrans dio lugar al gran lobo C. ambrusteri [6]: 242 [18] Martin, sin embargo, expresó la opinión de que C. ambrusteri[19] era C. lupus.[20] Nowak no estaba de acuerdo y sostuvo que C. ambrusteri no estaba relacionado con C. lupus sino con C. priscolatrans, que posteriormente originó a C. dirus. Tedford observó que el C. gezi sudamericano y C. nehringi compartían similaridades dentales y craneales, desarrolladas por el hipercarnivorismo, lo que indicaría que C. ambrusteri era el ancestro común de C. gezi, C. nehringi y C. dirus.[13]: 148
Canis dirus
Canis dirus vivió en América del Norte y del Sur desde el Pleistoceno tardío hasta el Holoceno.[21] C. dirus era la especie de mayor tamaño del género Canis.[5]: 52 Goulet sostuvo que C. dirus[22] era una variante de C. lupus,[23] al contrario que que Nowak, Kurten y Berta, que pensaban que C. dirus no se derivaba de C. lupus.[7][11][24]
Los paleontólogos Wang, Tedford y Nowak sugirieron que C. dirus había evolucionado de C. ambrusteri,[5]: p52 [13]: 181 Nowak sostenía que había especímenes de Cumberland Cave, (Maryland) que indicaban que C. ambrusteri se estaba convirtiendo en C. dirus.[6]: p243 [25] Los dos taxones compartían características propias (sinapomorfía), lo que situaría el origen de C. dirus en el Irvintoniano tardío en las llanuras centrales del continente, desde las que se expandió para desplazar a su ancestro C. ambrusteri.[13]: 181
Canis mosbachensis
Canis mosbachensis vivió a mediados del Pleistoceno en Eurasia.[26] El holotipo del C. mosbachensis (Soergel, 1925) fue hallado en Jockgrim (Alemania). Según un estudio de 2010, la diversidad del género Canis disminuyó entre el Pleistoceno temprano y medio, y los únicos representantes en Eurasia eran los lobos del grupo C. mosbachensis–C. variabilis, comparables en tamaño a la especie actual Canis lupus pallipes y Canis (Xenocyon) lycaonoides, de talla similar a los lobos grises modernos.[27] C. mosbachensis apareció entre los C. etruscus del Pleistoceno temprano y los C. lupus modernos.[6]: p242 C. mosbachensis era más pequeño que los lobos de Norteamérica y C. rufus,[6]: p242 [28][6]: p242 [26] pero a lo largo del timepo aumentó de tamaño.
Nowak propuso que C. mosbachensis era el ancestro de los lobos de Eurasia y América del Norte y que una población de C. mosbachensis invadió América del Norte donde quedó aislado por la glaciación y dio lugar a C. rufus. Otra población de C. mosbachensis permaneció en Eurasia donde se convirtió en C. lupus, que finalmente invadió Norteamérica. [6]: p242 Tedford comparó C. mosbachensis —que llegó a ocupar desde Europa del Este hasta Kazajistán— y C. variabilis —que habitaba desde Kazajistán a China—. La única diferencia entre ellos era la posición de los huesos nasales, que en C. variabilis «acababan en o antes de la posición más posterior de la sutura frontal-maxilar», por lo que ambos taxones podrían representar distintas variaciones de un único lobo del Pleistoceno medio con una amplia distribución geográfica.[13]: p181
Existe amplio acuerdo en que el lobo moderno, C. lupus desciende de C. etruscus vía C. mosbachensis.[2][26][27][29][30][31][32][33][34][35][36][37][38] Lumley consideraba a C. mosbachensis como subespecie del lobo gris, por lo que propuso el nombre de C. lupus mosbachensis.[39] Sin embargo, otros investigadores no aceptan la relación anatómica entre C. mosbachensis y C. etruscus y opinan que se parece más a C. arnensis, [40][41][42] con un tamaño y una dentadura más similares a los de un chacal omnívoro. [42]
Canis chihliensis
Wang y Tedford propusieron que el género Canis fue un descendiente del Eucyon davisi, y sus restos aparecieron primero en el Mioceno (hace 6 millones de años) en el sureste de EUA y México. En el Plioceno (5 millones de años), el Canis lepophagus de mayor tamaño, apareció en la misma región y para el Pleistoceno temprano (hace 1 millón de años) Canis latrans (el coyote) aún existía. Propusieron que la progresión de Eucyon davisi a C lepophagus a Coyote fue evolución linear.[5]: p58 Adicionalmente, C. edwardii, C. latrans y C. aureus forman un pequeño clado y porque C. edwardii apareció temprano abarcando medio Blancano (plioceno tardío) al Invintoniano (Pleistoceno tardío) es propuesto como el ancestro.[13]: p175, 180
Nowak y Tedford también creían que era posible que C. lupus había derivado del linaje de canidos del Mioceno o Pliocenoque precedían y que no estaban relacionados con C. lepophagus.[7][14] Wang y Tedford propusieron que al asumir los atributos geológicos del material, por lo que los restos más antiguos que se han podido identificar de C. lupus son de hace 800,000 millones de años[13]: 5 (mediados del Pleistoceno) con lobos similares a los de hoy en día [13]: p150 habitando en tanto la fauna Olioriana (inicios del Pleistoceno medio de Siberia) y en la fauna del sumidero Cripple Creek en Alaska, lo que apunta al origen de estos lobos en Beringia.[13]: p181
Basada en la morfología de China, el lobo C. chihliensis del Plioceno, pudo haber sido el ancestro de tanto C. armbrusteri y C. lupus antes de su migración a América del Norte.[5]: p148 [13]: p181 Al parecer C. chihliensis era más primitivo y pequeño que C. lupus, las medidass de su craneo y dientes eran similares a C. lupus, pero los elementos post-craneales son más pequeños. [43]
C. amrusteri apareció en América del Norte a mediados del Pleistoceno y su forma parecida a la de un lobo, es más grande que la de cualquier Canis de esta época. [7] A finales del retroceso glaciar más reciente, hace 30,000 años, el calentamiento derritió las barreras glaciales en todo el norte de Canadá permitiendo a los mamíferos árticos expanderse a latitudes medias en América del Norte, incluyendo alces, caribúes, bisontes y el lobo gris.[5]: p61 A mediados del Pleistoceno en Eurasia, de C. falconeri surgió el género hipercarnívoro Xenocyon, que después resultó en el género Cuon (el dhole) y el género Lycaon (el perro cazador africano).[5]: p105, 149 Justo antes de la aparición de C. dirus, Norteamérica fue invadida por el género Xenocyon que era un C. dirus más grande y más hipercarnivoro. [5]: p60 El lobo de gran tamaño C. antonii del Plioceno tardío al Pleistoceno temprano en China fue determinado como una variación del C. chihliensis,[13]: p197 y el de también gran tamaño C. falconeri surgió abruptamente en Europa en el Pleistoceno temprano, tal vez representando una extensión al este de C. antonii.[13]: p181
Canis variabilis
En 2012, un estudio sobre las especies Canis similares a los lobos de China antigua bajo la dirección de Xiaoming Wang, encontró que estos estaban relacionados con C. lupus tanto en dimensiones post-craneales como en dentales, excepto por C. variabilis, el cual fue comparado de manera "extraña" con otros Canis en China ya que tiene unas dimensiones craneo-dentales más pequeñas que las especies anteriores y posteriores.[43] El estudio concluyó que "Es bastante probable que estas especies sean el ancestro del perro doméstico Canis familiaris, una hipótesis que ya había sido propuesta antes por otros autores."[44][45][46][47][48]
Canis lupus familiaris (perro doméstico)
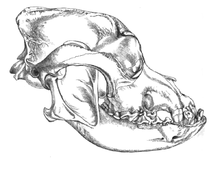

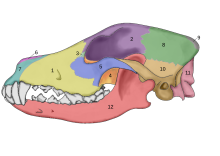
En el 2002, se realizó un estudio en los fósiles craneales de dos cánidos que se encontraron enterrados a metros de la entrada de lo que algún día fue una cabaña de huesos de mamuts en el en el sitio paleolítico superior Eliseevich-I en la región de Briansk en la Llanura Rusa, y usando una definición aceptada y basada morfologicamente de la domesticación, declaró que eran "perros de la era del hielo". La datación de carbón dio una antigüedad estimada de hace 16,945-13,905 .[49] En 2013, un estudió analizó un cráneo y su secuencia de ADN mitocondrial fue identificada como de Canis lupus familiaris.[50]
En 2015, un zooarqueólogo declaró que "En términos de fenotipos, perros y lobos son fundamentalmente animales diferentes."[51]
En 1986, un estudio de la morfología de un cráneo y encontró que el perro doméstico es morfologicamente diferente de todos los demás canidos excepto de aquellos similares a lobos. "La diferencia en tamaño y proporción entre algunas razas son tan grandes como aquellas entre géneros salvajes, pero todos los perros son claramente miembros de la misma especie." [52] En 2010, un estudio en la forma de los craneos de perros con carnívoros existentes que "La mayor diferencia en forma entre razas de perros claramente superan la máxima divergencia entre especies Carnivoras. Además, perros domésticos ocupan una gama de formas recientes fuera del dominio de carnívoros salvajes."[53]
El perro doméstico comparado con el lobo, muestra la variación más grande respecto a tamaño y forma del cráneo (Evans, 1979) el rango va desde 7 a 28 cm en longitud (McGreevy, 2004). Los lobos son dolicocéfalo (de cráneo largo) pero no tan extremo como algunas razas como los galgos y los loberos rusos (McGreevy 2004). La braquiocefalia de los caninos (cráneo corto) se encuentra solamente en los perros domésticos y se relaciona con el pedomorfismo (Goodwin 1997). Los cachorros nacen con hocicos cortos, con el cráneo más largo como perros dolicocéfalas surgiendo en el desarrollo posterior (Coppinger 1995). Otras diferencias en la forma de la cabeza entre los perros braquiocefalicos y dolicocéfalos son cambios entre el ángulo craniofacial (ángulo entre el eje basilar y el paladar duro) (Regodón 1993), la morfología de la articulación temporomandibular (Dickie 2001), y la anatomía radiográfica de la lámina cribosa (Schwarz 2000).[54]
Muchos autores han llegado a la conclusión de que en comparación con el lobo adulto existente, el perro doméstico adulto tiene un rostro relativamente reducido (parte frontal del cráneo), un hueso frontal elevada, un paladar más amplio, un cráneo amplio, y los dientes más pequeños (Hildebrand1954; Clutton- Brock, Corbet y Hills, 1976; Olsen 1985; Wayne 1986; Hemmer 1990; Morey 1990). Otros autores no concuerdan y han declarado que estos rasgos se pueden superponer y variar dentro de los dos (Crockford 1999; Harrison 1973). Los cachorros de los lobos tienen proporciones craneales relativamente similares como los perros adultos y esto fue propuesto como prueba de que el perro doméstico es un lobo neoténico.
La causa propuesta fue debido a la selección humana de apariencia juvenil o debido a un efecto pleiotrópico como resultado de la selección por el comportamiento juvenil (Clutton-Brock 1977; Belyaev 1979; Wayne 1986; Coppinger y Schneider 1995). Wayne (1986) llegó a la conclusión de que sus muestras de perros no tenían un acortamiento significativamente relativo del rostro en comparación con los lobos, poniendo en duda esta característica de identificación.[44]
En comparación con el lobo, la dentición del perro es relativamente menos robusta (Olsen 1985; Hemmer 1990), lo se propone que sea debido a la relajación de la selección natural cuando los lobos se convirtieron en comensales basureros, o por la selección artificial (Olsen 1985; Clutton-Brock 1995). Sin embargo, Kieser y Groeneveld (1992) compararon las mediciones mandibulo-dentales de chacales (C. adustus, C. mesomelas) y zorros Cabo (Vulpes chama) a perros equivalente de tamaño y se encontró que los caninos de estos otros cánidos tendían a ser ligeramente más pequeños y sus segundos molares más grandes en comparación con los perros, a parte de eso las proporciones eran esencialmente la misma en todas las especies. Concluyeron que "..los dientes de cánidos parecen haber evolucionado conjuntamente con los otros y con relativa independencia de las diferencias en el dimorfismo, demandas tamaño o funcionales". Esto pone en duda la suposición de que los dientes de perros son relativamente pequeños debido a la selección reciente, lo que sugiere que la dentición perro es plesiomorfica de un antepasado que era más pequeño que el lobo.[44]
La reducción del tamaño corporal de los primero perros en comparación con un lobo se cree debido a la selección de nicho (Olsen 1985; Morey 1992; Coppinger y Coppinger 2001). Morey (1992: 199) afirma que "Los resultados...son coherentes con la hipótesis de que los primeros perros domésticos son pedomorfos evolutivos, productos de fuerte selección para la reducción del tamaño de ontogenéticamente canalizado y alteraciones del calendario reproductivo asociado a la nueva forma doméstica de la vida."[44] Sin embargo, en un experimento domesticación, los zorros domesticados se mantuvieron el mismo tamaño que los zorros no seleccionados (Trutt 1999: 167).[51]
Wayne (1986) concluyó que el perro se encuentra más cercanamente relacionado en la morfología craneal de C. latrans, C. aureus, C. adustus, C. mesomelas, Cuon alpinus y Lycaon pictus que al lobo. Dahr (1942) llegó a la conclusión de que la forma de espacio para cerebro del perro es más cercano al de los coyotes que a la de los lobos. Manwell y Baker (1983) revisaron el trabajo de Dahr con la adición de los datos dentales para los cánidos y llegaron a la conclusión de que el ancestro del perro fue probablemente en el rango de 13.6-20.5 kg, que es menor que el rango de 27-54 kg para los lobos existentes (Mech 1970 ) y es comparable con el Dingo. [44]
La agitación auditiva del perro es relativamente más pequeña y más plana que la del lobo (Harrison 1973; Clutton-Brock, Corbet & Hill 1976; Nowak 1979; Olsen 1985; Wayne 1986), se propone que se debe a la selección relajada en estado doméstico como el perro ya no se requiere la audición aguda del lobo. Sin embargo, se ha demostrado que la forma agitada facilita el incremento de sensibilidad a las frecuencias específicas, pero la forma y el tamaño no se puede correlacionar con la agudeza (Ewer 1973). Por lo tanto, la diferencia observada podría ser que el perro con bulla ha conservado su forma ancestral.[44]
El borde ventral de la rama horizontal del perro de la mandíbula tiene una curva convexa que no existe en el lobo (Olsen 1985; Clutton-Brock, 1995), y no hay discusión de esta diferencia en la literatura. Sin embargo, Biknevicius y Van Valkenburgh (1997) notaron que la rama horizontal de los depredadores de procesamiento de hueso es más gruesa dorso-ventral en el punto de caudal al sitio de procesamiento de hueso. Este engrosamiento puede haber sido una función de nicho de adaptación por parte de los antepasados del perro.[44] Una descripción de la morfología del cerebro superficial de chacales (C. mesomelas, C. aureus), coyotes (C. latrans), lobos (C. lupus, C. rufus), y perros indicó que el cerebelo de perro se aproxima mucho a la de el coyote, está estrechamente alineados con los chacales y los lobos que muestran numerosos rasgos cerebrales distintas de las otras especies (Atkins y Dillon 1971). Los lobos también tienen serológica y caracteres bioquímicos distintos de los perros (Leona y Wiens 1956; Lauer, Kuyt & Baker 1969).[44]
Un autor ha propuesto que la explicación más probable para las diferentes características morfológicas del perro en comparación con el lobo, es que el antepasado del perro fue adaptado a un nicho diferente que el lobo.[44]
Récord genético
Secuencia de ADN
Las mitocondrias dentro de cada célula contienen muchas copias de un pequeño genoma de ADN circular y en los mamíferos es de una longitud de 16,000-18,000 pares de bases. Una célula contiene cientos o miles de mitocondrias y por lo tanto los genes contenidos dentro de esas mitocondrias son más abundantes que los genes que se producen en el núcleo de la célula. [55][56] La abundancia de ADN mitocondrial (mDNA) es útil para el análisis genético de restos antiguos en el que el ADN se ha degradado.[56][57]
Las secuencias de ADN mitocondrial tienen una tasa de mutación más alta que la tasa de mutación de los genes nucleares y para los mamíferos esta tasa es 5-10 veces más rápido..[56][58][59] Los genes que codifican proteínas mitocondriales evolucionan mucho más rápido y son marcadores de gran alcance para inferir la historia de la evolución a nivel de categoría, como las familias, géneros y especies. Sin embargo, han evolucionado a un ritmo más rápido que otros marcadores de ADN y hay una diferencia de tiempo en su reloj molecular que necesita ser validado frente a otras fuentes. La situación taxonómica de las especies de incertidumbre se resuelve a través de un mejor uso de ADN nuclear, que es más adecuado para el análisis de la historia reciente.[60] En la mayoría de los casos, mDNA se hereda del ancestro materno.[56][61] Por lo tanto, el análisis filogenético de las secuencias de mDNA dentro de las especies proporciona una historia de los linajes maternales que se pueden representar como un árbol filogenético.[56][62][63]
| De espécimen a árbol filogenético | |||||||||
| |||||||||
Las mutaciones que son diferentes en estos 4 secuencias se han numerado y resaltado. Estas mutaciones se pueden utilizar para construir un árbol filogenético para las cuatro cánidos. En este ejemplo, el perro y el lobo gris se diferencian por dos sustituciones (resaltadas en rojo), y cada uno de ellos difiere del coyote por cuatro sustituciones.[56]
1 2 3 4 5 67
Chacal dorado A-G-C-T-G-T-C-GA-T-TC-CA
Coyote A-G-C-T-A-T-C-GA-A-TC-GA
Lobo T-G-C-T-A-T-G-GA-T-TC-CT
Perro T-G-G-T-A-T-G-GA-T-TC-CA
Las secuencias de ADN mitocondrial del perro y el lobo se diferencian por sólo 0-12 sustituciones dentro de 261 pares de bases, mientras que los perros siempre difieren de los coyotes y chacales por lo menos 20 sustituciones.[56][65] Este hallazgo implica que el perro deriva del lobo y que ha habido mucho retrocruzamiento, [65] o que el perro pudo haber descendido de una especie ya extinta de cánido cuyo relativa vivo más cercano es el lobo moderno. [66]
Problemas de sincronización
Los estudios de ADN pueden dar resultados que no tienen solución, debido a los especímenes seleccionados, la tecnología utilizada, y las suposiciones hechas por los investigadores.[67] Cualquiera de un panel de marcadores genéticos puede ser elegir para su uso en un estudio, y las técnicas utilizadas para extraer, localizar y comparar las secuencias pueden ser aplicados utilizando los avances en la tecnología de observar longitudes más largas de pares de bases que dan mejor resolución filogenética. [68] Existen dos hipótesis principales que se hacen para explicar la divergencia de tiempo de las especies: el tiempo de generación y la tasa de mutación genética por generación. El tiempo entre generaciones de lobos se supone que es de tres años, basándose en el lobo gris existente, y dos años para el perro basado en el perro existente.[69] Un reciente e importante estudio supone un tiempo de generación de 2 años para el perro de hasta 10.000 años, y luego asumió un tiempo de generación de 3 años (el mismo que el lobo) antes de eso para calcular un tiempo de divergencia propuesta entre los dos.[70]
Se llevan a cabo estudios sobre el ADN, pero con "la tasa de mutación como la principal fuente de incertidumbre."[70] En 2005, Lindblad-Toh secuenció el primer borrador del genoma del perro existente, y se calcula una tasa de mutación propuesta de 1x10-8 mutaciones por generación. [69] En 2015, Skoglund fue capaz de secuenciar el primer borrador del genoma de un lobo del Pleistoceno y usó su fecha de radiocarbono para validar una tasa de mutación genética propuesta de 0.4x10-8 mutaciones por generación.[71] La diferencia es un factor de tiempo de 2,5, sin embargo, otro estudio afirmó que se debía a que sólo un ejemplar de lobo Pleistoceno hasta el momento había sido secuenciado, el resultado debe ser tratado con precaución, ese estudio proporcionó ambas estimaciones para calcular los tiempos de divergencia propuestas entre el lobo y el perro.[72]
Canidos similares a lobos
Los cánidos similares a lobos son un grupo de grandes carnívoros que están genéticamente relacionados por sus cromosomas número 78. El grupo incluye los géneros Canis, Cuon and Lycaon. los miembros son el perro (C. lupus familiaris), lobo gris (C. lupus), coyote (C. latrans), chacal dorado (C. aureus), lobo etiope (C. simensis), chacal de espalda negra (C. mesomelas), chacal del lado rayado(C. adustus), dhole (Cuon alpinus), y perro salvaja africano (Lycaon pictus).[73] Nuevos miembros propuestos incluyen el lobo rojo (Canis rufus), el lobo del este (Canis lycaon), y el lobo dorado africano (C. anthus). Ya que poseen 78 cromosomas, todos los miembros del género Canis (coyotes, lobos, chacales) son indistinguibles cariologicamente entre ellos, y desde el dhole y el perro de caza africano.[56]: p279 [74] Los miembros de Canis pueden potencialmente cruzarse [66], y hay pruebas de que el lobo etíope ha hibridado con los perros.[75] Según el zoólogo Reginald Pocock, un dhole se cruzóaron con un chacal dorado.[76] El perro de caza africano es grande, muy agil, conocido por estar disperso a través de grandes distancias y son raros en gran parte de su área de distribución geográfica, [77] que hace oportunidades para la hibridación difícil. Un estudio del ADN mitocondrial materno de los chacales de lomo negro no pudo encontrar evidencia de los genotipos de los compañeros más probables, el chacal lado rayado ni el chacal dorado - lo que indica que los chacales de lomo negro masculinos no habían criado con estos. [78] En la literatura científica no se pudieron encontrar pruebas de hibridación del raro chacal rayado lateral. Un alineamiento de secuencias de ADN de los cánidos similares a lobos dio el árbol filogenético más estrechamente relacionado de el lobo gris y el perro, seguido de una afiliación cercana con el coyote, chacal de oro y el lobo etíope, y el perro puede hibridar en la naturaleza con estas tres especies. Los otros más próximo a este grupo son el dhole y el perro salvaje africano ya que ambos tienen colmillos únicos, lo que sugiere que esta adaptación se perdió más tarde por los demás miembros.[69] Los dos chacales africanos se muestran como los miembros más basales de este clado, lo que significa que este árbol indica un origen africano para el clado.[69][79] El árbol ilustra la distinción genotipo-fenotipo, donde un genotipo está lleno de información hereditaria de un organismo y un fenotipo es la propiedad real observada de un organismo, como la morfología, desarrollo o comportamiento. Por fenotipo, el dhole (género Cuon) y el perro de caza africana (género Lycaon) no se clasifican como miembros del género Canis, pero por el genotipo están más cerca de los perros, lobos y coyotes que son los dos género chacales Canis - el chacal de lado rayado (C. adustus) y el chacal de espalda Negro (C. mesomelas). En 2014, un estudio de todo el genoma indicó que el linaje ancestral chacal dorado había divergido del lobo linaje / coyote ancestral hace 400.000 años, lo que es mucho más reciente que las estimaciones previas de 1,9 millones de años con base en el ADN mitocondrial, pero con "la tasa de mutación como la principal fuente de incertidumbre. "[70] Usando una tasa de mutación por generación alterna (Skoglund 2015),[71] el tiempo de divergencia se convierte en 955,000-1 millones.
Dos halogrupos de lobos
Un haplotipo (genotipo haploide) es un grupo de genes en un organismo que se heredan juntos de un solo padre.[80][81] Un haplogrupo es un grupo de haplotipos similares que comparten un ancestro común con una mutación de polimorfismo de un solo nucleótido.[82][83] El ADN mitocondrial pasa a lo largo de un linaje materno que se remontan a miles de años. [82] En 2010, un estudio comparó un par de secuencias de 230 bases de la región de control mitocondrial de 24 ejemplares de lobos antiguos de Europa occidental fechadas entre hace 44,000-1,200 con los de los lobos grises modernas. La mayor parte de las secuencias podrían ser representadas en un árbol filogenético. Sin embargo, los haplotipos del lobo del Himalaya y el lobo gris indio no podrían serlo porque eran 8 mutaciones aparte de los otros lobos, [84] indicando distintos linajes que previamente habían sido encontrados en otros estudios.[84][85][86] El estudio encontró que había 75 diferentes halotipos de mRNA del lobo gris que incluyen 23 en Europa, 30 en Asia, 18 en América del Norte, 3 en Europa y Asia y 1 en Europa y América del Norte.[84] Estos haplotipos formaron dos haplogrupos que se han separado entre ellos por 5 mutaciones. El haplogrupo 1 forma un clado monofilético (lo que indica que todos ellos tienen la misma mutación heredada de un único ancestro femenino). Todos los otros haplotipos fueron basales en el árbol, y éstos forman 2-3 clados más pequeños que fueron asignados al haplogrupo 2 que no era monofilético.[84][87] Los haplogrupos 1 y 2 se podían encontrar repartidas por toda Eurasia, pero sólo el haplogrupo 1 se encontró en América del Norte. Todas las antiguas muestras de lobos de Europa occidental pertenecían al haplogrupo 2, lo que indica que el haplogrupo 2 predominó en esta región durante más de 40.000 años antes y después del último máximo glacial. Una comparación de las frecuencias actuales y pasados indicó que en Europa el haplogrupo 2 fue superado en número por el haplogrupo 1 durante los últimos miles de años, pero en América del Norte el haplogrupo 2 se extinguió y fue reemplazado por el haplogrupo 1 después de que el último máximo glacial.[84][87] El acceso a América del Norte estaba disponible hace 20,000-11,000 años, después de que la glaciación de Wisconsin se había retirado, pero antes de que el puente de tierra de Bering se inundara. [88] Por lo tanto, el haplogrupo 1 era capaz de entrar en América del Norte durante este período. El análisis de isótopos estables realizado en una muestra de huesos permite a los investigadores para formar conclusiones acerca de la dieta, y por lo tanto la ecología, de las poblaciones de lobos extintos.
Este análisis sugiere que los lobos del Pleistoceno del haplogrupo 2 se encuentran en Beringia y Bélgica cazaban principalmente en la megafauna del Pleistoceno[84][89][90] , que escaceo al principio del Holoceno hace 12.000 años.[84][91] Se ha encontrado que los lobos euroasiáticos del Pleistoceno son morfológica y genéticamente comparables a los lobos del Pleistoceno ubicados en oriente-Beringia, [92] con algunos de los antiguos lobos europeos y Beringia que comparten un haplotipo común (a17),[84][89], que hace la probable ecológica similitud.[84] Dos lobos antiguos de Ucrania de fecha alrededor de 30.000 y 33.000 "perro de Altai" tenían la misma secuencia que seis lobos Beringia, y otro de la República Checa de hace 44.000 tenía la misma secuencia que dos lobos Beringia. [89] Se ha propuesto que los lobos del Pleistoceno de todo el norte de Eurasia y el norte de América del Norte representaban una población continua y casi panmíctica que era genéticamente y probablemente también ecológicamente distinta de los lobos que viven en esta área en la actualidad. [84][93] Los lobos especializados del Pleistoceno no contribuyen a la diversidad genética de los lobos modernos, y las poblaciones de lobos modernos de todo el Holartico es probable que sean los descendientes de los lobos de poblaciones que vinieron de refugios más al sur.[93] El haplogrupo 2 existente de lobos se encuentran en Italia, los Balcanes y los Cárpatos, pero es raro en otras partes de Europa. En Asia, sólo cuatro haplotipos se han identificado como pertenecientes a este haplogrupo, y dos de ellos habitan en el Medio Oriente.[94] El haplogrupo 2 no se extinguió en Europa, y si antes del glacial máximo pasado haplogrupo 2 se asocia exclusivamente con el lobo ecomorfo especializado en alimentarse de la megafauna, esto significaría que en Europa era capaz de adaptarse a los cambios de las presas.[84] En 2013, una secuenciación de ADN mitocondrial de los antiguos cánidos similares a lobos, reveló otro linaje separado de 3 haplotipos (que forman un haplogrupo) que se encuentran en 3 especímenes del Pleistoceno tardío de Bélgica; sin embargo, no se ha detectado en los lobos existentes.[50][94] Uno de ellos fue el "perro Goyet".[50]
En América y Japón

En 2016, un estudio construido en la obra de otro importante estudio [50] y se analizaron las secuencias de 12 genes que se encuentran en la cadena pesada del genoma mitocondrial de especies extintas y modernas C. lupus. El estudio excluyó a las secuencias del lobo del Himalaya divergente y el lobo gris indio. Los antiguos especímenes fueron datados por su radio de carbono y y estratagraficamente, y junto con las secuencias generaron un árbol filogenético basado en el tiempo. Por el árbol, el estudio fue capaz de inferir el ancestro común más reciente de todas las demás muestras de C. lupus - modernas y extintas - de hace 80.000 y esta fecha fue la misma que la del estudio anterior.[50][95] El estudio no pudo encontrar evidencia de una población "cuello de botella" de lobos hasta hace unos pocos miles de años.[95] El árbol filogenético mostró la polifilia de lobos americanos, el lobo mexicano fue divergente de otros lobos de América del Norte, y estos otros lobos norteamericanos formaron dos clados estrechamente relacionados. Un escenario consistente con los datos filogenéticos, la capa de hielo y el nivel del mar fue que durante la Edad de Hielo, cuando los niveles del mar estaban en su punto más bajo, había una sola onda de la colonización lobo en América del Norte a partir de la apertura del estrecho de Bering 70.000 y el cierre durante el máximo Glacial tardío del corredor Yukón que corría a través de la división entre la capa de hielo Laurentino y de la capa de hielo de la Cordillera hace 23,000. Los lobos mexicanos eran parte de la onda única y pudieron depararse de los otros lobos antes de entrar en América del Norte o una vez en América del Norte debido al cambio en su entorno.
Como lobos habían estado en el registro fósil de América del Norte, pero ascendencia de los lobos modernos sólo podría serrastreado hasta hace 80,000 años, los haplotipos de los lobos que ya estaban en América del Norte fueron reemplazados por estos invasores, ya sea mediante desplazamiento competitivo o mediante mezcla.[95] La sustitución en América del Norte de una población basal de lobos por una más reciente apoya las conclusiones de estudios anteriores.[84][87][89][95] Posiblemente existía una población de lobos panmíctica con el flujo de genes que abarca Eurasia y América del Norte hasta el cierre de las placas de hielo.[84][93][95]
Una vez que las puertas se cerraron, se aislaron los lobos del sur y en el norte solamente existían el lobo de Behring. El puente de tierra se inundó por el mar hace 10.000, las bardas se retiraron hace 12,000-6,000, el lobo de Behring se extinguió y los lobos del sur amplió para recolonizar el resto de América del Norte. Todos los lobos norteamericanos son descendientes de aquellos que fueron aislados al sur de las capas de hielo. Sin embargo, gran parte de su diversidad fue perdida durante el siglo XX.[95] Los estudios que utilizan ADN mitocondrial han indicado que los lobos de la costa sureste de Alaska son genéticamente distintos de los lobos grises del interior, lo que refleja un patrón observado también en otros taxones. Muestran una relación filogenética con los lobos extirpadas del sur (Oklahoma), lo que indica que estos lobos son los últimos restos de un grupo con gran amplitud que ha sido extirpado en gran parte durante el siglo pasado, y que los lobos del norte de América del Norte se habían expandido originalmente de refugios del sur por debajo de la glaciación de Wisconsin después de que el hielo se había derretido al final del último máximo glacial.[96][97][98] Un estudio de ADN de todo el genoma indicó que todos los lobos norteamericanos son monofilético y por lo tanto son los descendientes de un ancestro común.[99]
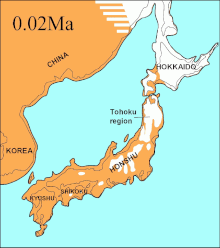
Durante el mismo período, el estrecho de Soya entre Hokkaido y la isla de Sakhalin estaba seco por 75.000 años y se propuso que el lobo de Ezo extinta (C. l. Hattai) llegó a Hokkaido desde Sajalín.[100][95][101] Sin embargo, las secuencias indicaron que llegó a Hokkaido hace menos de 10.000. El lobo de Ezo estaba estrechamente relacionado con uno de los clados de América del Norte,[100][95][102] pero diferente al lobo japonés más al sur (C. l. Hodophilax) que era basal para los lobos modernos.[100][95] El lobo japonés habitada Kyushu, Shikoku y en las islas Honshu [103][104], pero no la isla de Hokkaido.[104] Esto indica que su antepasado pudo haber migrado desde el continente asiático a través de la península de Corea en Japón.[100][104] Los últimos niveles del mar del estrecho de Corea, junto con la sincronización de las secuencias de lobos japoneses indicaron que llegó a las islas del sur hace menos de 20.000.[95]
Divergencia con los coyotes
En 2016, un estudio de ADN de todo el genoma propuso, suposiciones con fundamentos, que todos los lobos norteamericanos y coyotes divergieron de un ancestro común hace más de 6,000-117,000 años. El estudio también indica que todos los lobos América del Norte tienen una cantidad significativa de ascendencia de coyotes y todos los coyotes algún grado de ascendencia de lobos, y que el lobo y el lobo rojo oriental está altamente mezclado y tiene diferentes proporciones de lobo gris y de ascendencia coyote. Una de las pruebas indica un tiempo de divergencia de lobos/coyotes de 51.000 años antes del presente que coincidió con otros estudios que indican que el lobo existente comenzó a habitar al rededor de esa época. Otra prueba indicó que el lobo rojo se separaró de los coyotes entre 55,000-117,000 años antes de hoy y del lobo de los Grandes Lagos hace 32.000. Otras pruebas y modelos mostraron varias gamas de divergencia y la conclusión fue un intervalo de hace 6.000 y 117.000 años. [99][105] Este hallazgo no concuerda con el registro fósil que muestra un espécimen similar a un coyote de hace 1 millón de años. [5]
Perro doméstico
El perro doméstico (Canis lupus familiaris) es el carnívoro más grande y abundante. [50][72][106] Durante los últimos millones de años, existieron muchos espécimenes similares a lobos, pero su tasa ha aumentado, y los lobos modernos no son los antepasados directos de los perros.[50][72][70][107] Aunque investigaciones han sugerido que los perros y los lobos eran parientes genéticamente muy cercanos,[65][66][73] análisis filogenético posteriores apoyan firmemente la hipótesis de que los perros y los lobos son taxones recíprocamente monofílicos que forman dos clados hermanos,[65][70][108] lo que sugiere que ninguna población de lobo moderno está relacionada con los lobos que fueron domesticados por primera vez. [70][108] Análisis recientes de ADN mitocondrial de especímenes de lobos grises antiguos y modernos es compatible con un patrón de reducción de la población y el volumen de la tasa.[50][84][89]
En 2016, un estudio investigó por primera vez las subdivisiones de la población, la demografía y las relaciones de lobos grises en función de sus secuencias de todo el genoma. El estudio indicó que el perro era una subespecie divergente del lobo gris y se derivó de una población fantasma ya extinta de lobos Pleistoceno Tardío,[50][72][70] el perro y el dingo no son especies separadas.[72] El árbol filogenético de todo el genoma indica una divergencia genética entre los lobos del Nuevo Mundo y del Viejo Mundo, que luego fue seguido por una divergencia entre el perro y lobos del Viejo Mundo hace 27,000 [71][72] - 29.000. [72]
El perro tienen una taxonomía hermana con los lobos grises de Eurasia, pero no con los lobos de América del Norte. El perro tenía considerables ancestros después de su divergencia de los lobos del Viejo Mundo antes de que se separa en distintos linajes que son sumamentes distintos unos de otros, ya que son de los lobos. [72] El estudio sugiere que las dataciones anteriores basadas en la divergencia entre los lobos y coyotes de hace un millón de años usando fósiles de lo que parecían ser ejemplares similares a coyotes pueden no reflejar la ascendencia de las formas modernas.[109][71][72][70] El estudio indicó que el lobo mexicano era también una forma divergente del lobo gris, lo que sugiere que pudo haber sido parte de una invasión temprana a América del Norte.[72][107] Se encontró que el lobo tibetano era el más altamente divergente de los lobos del Viejo Mundo, había sufrido un cuello de botella poblacional histórico y sólo recientemente había vuelto a colonizar la meseta del Tíbet. La glaciación pudo haber causado la pérdida de hábitat y el aislamiento genético que a su vez llevó a la adaptación local. [72] El estudio indicó que ha habido una gran mezcla genética entre los perros domésticos y los lobos, con hasta un 25% del genoma de los lobos del Viejo Mundo que muestra signos de ascendencia perruna, posiblemente como resultado del flujo de genes de los perros en lobos que eran ancestrales a todos lobos modernos. Hubo muestras de significativ flujo de genes entre los lobos europeos con el lobo israelí con el basenji y el boxer, lo que sugiere una mezcla entre los linajes ancestrales de estas especies y las poblaciones de lobo.[72][70]
Para los lobos asiáticos de tierras bajas: los lobos de Rusia central y del este y todos los lobos chinas de tierras bajas tenido flujo de genes significativa con los perros indígenas chinos, el mastín tibetano y el dingo. Para los lobos asiáticos tierras altas: Los lobos tibetanos no mostraron mezcla significativa con los perros; sin embargo, los lobos Qinghai mostrarron flujo de genes con los dingo y uno de ellos tenía el flujo de genes con los perros chinos. Los lobos del Nuevo Mundo no mostraron ningún flujo de genes con el boxer, dingo o perros indígenas chinos pero no había indicacio de flujo de genes entre el lobo mexicano y el basenji africano.[72] Todas las especies dentro del género Canis, los cánidos similares a lobos, están estrechamente relacionados filogenéticamente con 78 cromosomas y, potencialmente, pueden cruzarse.[66] No había indicacio de flujo de genes al chacal dorado de la población ancestral a todos los lobos y los perros (11,3% -13,6%) y tasas mucho más bajas (hasta 2,8%) en las poblaciones de lobos existentes.[72][70]
Los datos indicaron que todos los lobos comparten trayectorias de población similares, seguido de disminución de la población que coincidió con la expansión de los humanos modernos en todo el mundo y su tecnología para aumentar la captura.[72][110] Los carnívoros del Pleistoceno tardío podrían haber vivido en grandes clanes y paquetes con el fin de aumentar la caza de animales disponible en ese momento, y estos grupos más grandes habrían sido blancos más visibles para los cazadores humanos. [110] Los perros grandes que acompañan a los seres humanos pueden haber acelerado el ritmo de descenso de los carnívoros que compitieron por la caza,[72][111] por lo tanto, los seres humanos se expandieron a través de Eurasia, se encontraron lobos, a algunos los domesticaron y posiblemente causaron el declive de otros.[72]
El estudio concluyó que la mezcla había confundido la capacidad de hacer inferencias sobre el lugar de la domesticación del perro. Estudios anteriores basados en SNPs, similitudes en todo el genoma con lobos chinos, y un menor desequilibrio de ligamiento podrían reflejar mezcla regional entre los perros con los lobos y el flujo de genes entre poblaciones de perros, con razas divergentes de perros, posiblemente manteniendo más ascendencia de lobo en su genoma. El estudio propone que el análisis de ADN antiguo podría ser un mejor enfoque.[72]
En el mismo año, un estudio encontró que sólo había 11 genes fijos que mostraron variación entre lobos y perros. Se cree que estos genes afectan a la docilidad y la capacidad de procesamiento emocional.[112] Otro estudio proporcionó una lista de todos los lobos y perros grises mDNA haplotipos combinados en el árbol filogenético.[113]
Lobo Taimyr
En mayo de 2015, se realizó un estudio en una costilla parcial de una muestra de lobo (nombrado Taimyr-1) encontrado cerca del río Bolshaya Balakhnaya en la Península de Taimyr del Ártico al norte de Asia, que data por el AMS radiocarbono hace 34.900. La muestra proveo el primer borrador del genoma nuclear de un carnívoro del Pleistoceno y la secuencia se depositó en el Archivo Europeo de nucleótidos y fue clasificada como Canis lupus.[71]
Usando la datación por radiocarbono de la muestra de Taimyr-1, su secuencia del genoma y la de un lobo moderno, se pudo hacer una estimación directa de la tasa de mutación de todo el genoma de los perros/lobos, para calcular el tiempo de divergencia. Los datos indicaron que el hasta ahora desconocido linaje de Taimyr-1 era una población de lobos diferente a los lobos modernos y perros e indicó que el genotipo de Taimyr-1, divergio de los lobos grises y perros de un ancestro común, ya extinta,[71][114][115] antes del auge del último máximo glacial hace 27,000-40,000 años. La separación del perro y lobo no tiene que coincidir con la cría selectiva por los seres humanos.[71][116] Una divergencia tan temprana es consistente con varios informes paleontológicos de cánidos similares a perros fecha hasta hace 36.000, así como la evidencia de que los perros domesticados más probablemente acompañaron a los primeros colonizadores en las Américas.[71]
La comparación con el linaje de lobo gris indicó que Taimyr-1 fue basal al gris lobos procedentes de Oriente Medio, China, Europa y América del Norte, pero compartió una cantidad sustancial de la historia con los lobos grises de hoy en día después de su divergencia de los coyotes. Esto implica que los ancestros de la mayoría de las poblaciones de lobos grises existentes,se derivan de una población ancestral que vivió hace menos de 35.000 años, pero antes de la inundación del puente de tierra de Bering con el posterior aislamiento de los lobos de Eurasia y América del Norte.[71] Una comparación de la ascendencia del linaje de Taimyr-1 y el del perro indica que algunas razas de perros modernos tienen una asociación más estrecha, ya sea con el lobo gris o de Taimyr-1 debido a la mezcla. El perro lobo de Saarloos mostró más asociación con el lobo gris, que concuerda con el cruzamiento histórico documentado con los lobos grises con esta raza. Taimyr-1 comparte más alelos (expresiones de genes) con aquellas razas que están asociadas con las altas latitudes, el Husky siberiano y perro de Groenlandia[71][115] que también están asociados con las poblaciones humanas del Ártico, y en menor medida al Shar Pei y de Pomerania finlandés.

An admixture graph of the Greenland dog indicates a best-fit of 3.5% shared material, although an ancestry proportion ranging between 1.4% and 27.3% is consistent with the data. This indicates admixture between the Taimyr-1 population and the ancestral dog population of these four high-latitude breeds. These results can be explained either by a very early presence of dogs in northern Eurasia or by the genetic legacy of Taimyr-1 being preserved in northern wolf populations until the arrival of dogs at high latitudes. This introgression could have provided early dogs living in high latitudes with phenotypic variation beneficial for adaption to a new and challenging environment. It also indicates that the ancestry of present-day dog breeds descends from more than one region.[71]
An attempt to explore admixture between Taimyr-1 and gray wolves produced unreliable results.[71]
Canis variabilis
En 2015, un estudio observó las secuencias de la región de control mitocondrial de los restos de 13 cánidos antiguos y un lobo moderno de cinco sitios del Ártico noreste de Siberia. Los cánidos revelaron nueve haplotipos mitocondriales, tres de los cuales estaban en el registro y los otros no habáin sido registrados. El árbol filogenético generado a partir de las secuencias mostró que cuatro de los cánidos siberianos dehace 28.000 y uno Canis c.f. variabilis de hace 360.000 eran muy divergentes. El haplotipo designado como S805 (hace 28.000) desde el río Yana era una mutación más de otro haplotipo S902 (8000 aap) que representa un clado del lobo moderno y linajes de perros domésticos. Estrechamente relacionados con este haplotipo están algunos de los que fue encontrado en el lobo japonés recientemente extinto. Varios haplotipos ancestrales estaban orientados hacia S805, incluyendo Canis c.f. variabilis (hace 360.000), en Bélgica (hace 36.000 - el "perro Goyet"), Bélgica (hace 30.000), y Konsteki, Rusia (hace 22.000). Dada la posición del haplotipo S805 en el árbol filogenético, puede representar un enlace potencialmente directo desde el progenitor (incluyendo Canis c.f. variabilis) con el perro doméstico y linajes lobo modernos. Se cree que el lobo gris que es ancestro del perro doméstico, sin embargo su relación con C. variabilis, y la contribución genética de C. variabilis al perro, es objeto de debate. [117]
Los haplotipos de los canidos de la isla Zhojov (hace 8.700) y Aachim (1700) pertenecen al clado del perro doméstico, llenos de S805, y también comparten sus haplotipos con - o son una mutación aparte de - el lobo tibetano (C l Chanco.) y el lobo japonés recientemente extinto (C. l. hodophilax). Esto puede indicar que estos cánidos retuvieron la firma genética de la mezcla con las poblaciones de lobos regionales. Otro haplotipo designado como S504 (hace 47.000) a partir de Duvanny Yar apareció en el árbol filogenético que no está conectado a los lobos (antiguos y modernos) todavía ancestrales a los perros, y puede representar una fuente genética para perros regionales. [117]
Ascenso a depredador dominante
En 2015, un estudio examinó la paleoecología de grandes carnívoros a través de la estepa mamut durante el Pleistoceno tardío mediante el uso de análisis de isótopos estables del colágeno de fósiles para reconstruir sus dietas. Basado en las pruebas en Bélgica, hace alrededor de 40.000 las hienas se aprovecharon de la cueva de los mamuts, rinocerontes lanudo, caballos y renos, con leones de las cavernas llevando renos y osos de las cavernas jóvenes. Los lobos parecen haber sido superados por números por las hienas cueva y su dieta fue restringida a renos, ciervos gigantes y ciervo. Sin embargo, después de que el último máximo glacial hace alrededor de 14.000, los lobos tenían acceso a todas clase de presas, el león de las cavernas fueron limitados a los renos, y la hiena de cueva se había extinguido.[118][119][120] Los datos sugieren que la extinción de la hiena de cueva permitió al lobo convertirse en el depredador dominante en lugar del león de las cavernas, justo antes de la extinción del león de las cavernas.[120]
Diferencias entre poblaciones de lobos
Los lobos grises tienen una amplia y natural distribución a través de la Holarctico que incluía muchos hábitats diferentes, que podían variar desde el Alto Ártico de densos bosques, estepas y desiertos. Las diferencias genéticas entre las diferentes poblaciones de lobos grises está estrechamente vinculada con el tipo de hábitat en el que viven.[121] Las diferencias en los marcadores genéticos entre la población de lobos escandinavos ha surgido en sólo poco más de una década debido a su pequeño tamaño de la población,[121][122] lo que indica que estas diferencias no dependen de un largo tiempo de permanencia en el aislamiento y que los patrones de población más grandes puede evolucionar en tan sólo unos pocos miles de años. [121] Estas diferencias también pueden incluir color de la piel, la densidad y el tamaño del cuerpo.[121][123][124] Las diferencias también pueden incluir comportamiento, como los lobos costeros se alimentan de peces [121][123] y los lobos de la tundra migran.[121][124] Se han observado estas diferencias entre dos poblaciones de lobos que viven cercanamente. Se ha demostrado que los lobos de la montaña no se cruzan con los lobos costeras cercanas, y los Alpes de Francia y Suiza se han repoblado con los lobos de las montañas de Italia [121][125] y de las lejanas montañas de Croacia[121][126] en vez de las tierras bajas cercanas, lo que indica que la distancia no es la fuerza motriz de las diferencias entre los dos ecomorfos.[121] Los factores ecológicos, incluyendo el tipo de hábitat, el clima, la especialización y la competencia depredadora tendrá gran influencia en la estructura genética de la población y la plasticidad cráneo-dental.[91][114][121][124][127][128][129][130][131] Dentro de la población de lobos grises del Pleistoceno, las variaciones entre los entornos locales habrían alentado una gama de ecotipos de lobos que eran genéticamente, morfológicamente y ecológicamente distintas una de otra[114].
Ecotipos
En 2016, dos estudios compararon las secuencias de 42.000 polimorfismos de nucleótido único en lobos grises de América del Norte y se encontró que se formaron 6 ecotipos - una población distinta genéticay ecológicamente separadas de otras poblaciones por su diferente tipo de hábitat. Los estudios encontraron que la precipitación y temperatura diurna media fueron las variables más influyentes en la variación de la secuencia..[132][133] Estos resultados concordaban con los hallazgos previos de que la precipitación influenciaba lA morfología[134] ,la vegetación [129] y el tipo de hábitat [123][135] influenciado diferencias lobo. El estudio ha identificado 1.040 genes que son potencialmente bajos de selección debido a la variación del hábitat. [133] La adaptación local de un lobo ecotipo, refleja probablemente la preferencia de un lobo de permanecer en el tipo de hábitat que nació. [132]
Lobos del Pleistoceno
Los restos más antiguos de Canis, encontrados en Europa eran de Francia y de hace a 3,1 millones,[136] seguido de Canis cf. etruscus (cf. en latín significa confieren, incierta) de Italia data de hace 2,2 millones.[137] C. lupus apareció por primera vez en Italia durante Etapa 9 de isotopos marinos [138] (hace 337.000). En Gran Bretaña, era la única especie de cánidos pesentes desde MIS 7 (hace 243.000), con el registro más antiguo de Pontnewydd en la cueva al norte de Gales.[139] Durante la era de hielo, Gran Bretaña estaba separada de Europa sólo por el Canal del Río.
Un estudio del C. lupus del Pleistoceno en Gran Bretaña, de diferentes períodos de tiempo encontró que sus habilidades para aplastar, rebanar carne y comer los huesos destacó su plasticidad cráneo-dental. Estas respuestas a los cambios en la dieta mostraron los cambios en un gran rango de especies, y no sólo ecomorfos locales, en respuesta a las variables climáticas y ecológicas. La supervivencia de C. lupus durante el Pleistoceno se puede atribuir en gran parte a su morfología plástica cráneo-dental. [131]



| Tiempo | Variables |
|---|---|
| 243,000 MIS 7 | El paleoambiente consistía en praderas abiertas con temperaturas de verano entre 16 °C y 23 °C y las temperaturas de invierno, entre -7 °C y -6 °C dominada por la estepa de mamuts y caballos. Los cazadores incluyen el león, el oso pardo, y rara vez la hiena manchada. Los lobos de MIS 7 fueron ligeramente más pequeños en el tamaño del cuerpo que los lobos de MIS 5 y los que se encuentran en Suecia hoy. Estos lobos estaban fuera de competencia por los competidores más grandes, lo que lleva a una dieta más omnívora con una mayor capacidad de trituración en un entorno abierto que admite más tipos de presas y más alimentos no cárnicos que el período de MIS 5. Tenían mandíbulas menos profundas y más estrechas que MIS 5 y los que se encuentran hoy en Suecia, lo que indicaba que era posible tomar sólo presas de tamaño pequeño a mediano. Ellos mostraron un menor porcentaje de roturas de dientes comparables con los lobos de MIS-3. Sin embargo, tuvieron el mayor porcentaje de moderado desgaste dental. [131] |
| 82,000 MIS 5A | El paleoambiente era frío, la tundra abierta tenía temperaturas de verano entre 7 °C y 11 °C y las temperaturas de invierno, entre -10 °C y -30 °C dominado por renos y bisontes. Una forma de oso pardo grande fue el máximo depredador, sin hienas en este momento. Los lobos de MIS 5 eran más grandes en el tamaño corporal que los encontrados en Suecia de hoy. Estos lobos sufrían de un clima severo, baja disponibilidad de presas en la dieta y el estrés conduce a una dieta más carnívora, con un aumento de la compactación de los cadáveres congelados y consumo de hueso. Ellos desarrollaron mandíbulas fuertes y la capacidad máxima de corte de carne en comparación con los otros lobos, con las mandíbulas menos profundas que el lobo moderno, pero más amplia y más profundas que las de MIS 7 y MIS 5. Mostraron el P4 superior más largo y angosto que sugiere la capacidad de corte mejorada y más larga M1 y M2 superior pero con anchura reducida, por lo tanto reduciendo la capacidad de trituración, lo que indica hipercarnivorismo. Exhiben un mayor porcentaje de rotura de dientes y los dientes muy desgastados en comparación con los otros lobos, y pueden haber estado utilizando su P4 superior y m1 inferiores para aplastar huesos en lugar de sus molares, que conduce a una mayor frecuencia de daños.[131] |
| 57,000 MIS 3 | Las praderas abiertas del paleoambiente, con la temperatura del verano de alrededor del 12 °C y en invierno la temperatura alrededor de -20 °C dominado por el mamut, rinoceronte lanudo, el caballo y ciervo gigante. Los cazadores incluyen el león, el oso pardo, y la hiena manchada como el carnívoro dominante. Los lobos de MIS 3 fueron más pequeños de cuerpo que MIS 5 lobos y los que se encuentran en Suecia hoy. Estos lobos estaban fuera de competencia por el león y la hiena, lo que lleva a una dieta más omnívora con una mayor capacidad de trituración en un entorno abierto que admite más tipos de presas y más alimentos no cárnicos que el período de MIS 5. Tenían mandíbulas menos profundas y más estrechas que los lobos deMIS 5 y los que se encuentran hoy en Suecia, lo que indicaba que era posible encontrar sólo presas de tamaño pequeño a mediano. Ellos mostraron un menor porcentaje de roturas de dientes comparables con el desgaste moderado dental de MIS-7.[131] |
| Hoy (Suecia) | Los lobos han sido extirpados de Gran Bretaña, pero no de Suecia, donde las temperaturas son similares a las de Gran Bretaña durante el período de MIS 7. Con medio ambiente como el del bosque boreal con temperaturas de verano entre 14 ° C y 18 °C y las temperaturas de invierno, entre 1 °C y -10 °C. Las especies cazadas son alces, renos, corzos, jabalíes, liebres, conejos y castores. Los cazadores son el oso pardo y el lince pero el lobo es el carnívoro superior. Los lobos que se encuentran en Suecia hoy son más pequeños de cuerpo que los lobos de MIS 5 pero más grandes que los de MIS 7 y MIS 3. La parte superior de M1 y M2 es más larga la de los lobos de MIS 7 y MIS 3, y las mandíbulas más amplias y profundas, lo que indica la capacidad de cazar y de someter a las presas grande. Sin embargo, los grandes molares tienen capacidad de trituración y para procesar los alimentos no cárnicos. Estos lobos viven en los bosques boreales, donde presas pequeñas y medianas son difícil de detectar y de trabajo intensivo para someter, lo que lleva a una adaptación para cazar más. Son hipercarnivoros similares a MIS 5 lobos, pero no con la misma capacidad de corte. |
Durante el último máximo glacial hace 20.000, la estepa del Pleistoceno se extendía a través de Eurasia septentrional y central y a través de Beringia hacia América del Norte. Los lobos del Pleistoceno de Beringia, y tal vez aquellos de la estepa, se adaptaron a este hábitat. La morfología de sus dientes y cráneos indica que se especializaron en aprovechar la ya extinta megafauna del Pleistoceno, y el desgaste de los dientes indica que su comportamiento era diferente a los lobos modernos.[89][121][140][141] Esto resalta el éxito de C. lupus como especie en la adaptación a diferentes condiciones ambientales.[131] Este lobo gris ecomorfo se extinguió al final de la glaciación, junto con el caballo y otras especies de la cual dependía, y fue reemplazado por los lobos del sur de América del Norte. Esto indica que los lobos ecomorfos especializados pueden extinguirse cuando su entorno cambia a pesar de que el hábitat todavía puede sustentar a otros lobos. [121] Los lobos pasaron por un cuello de botella poblacional hace 20.000 que coincide con el último máximo glacial[94][70][114][121] lo que indica que muchas poblaciones de lobos pueden haberse extinguido al mismo tiempo que los lobos Beringia.[121]
Se han encontrado pocos restos de Canis en la cueva Goyet, Bélgica (hace 36.500) [90], en la cueva Razboinichya, Rusia (hace 33.500)[142] Kostenki 8, Rusia (33,500-26,500)[143] Predmosti, República Checa (31.000) [144] y Eliseevichi 1, Rusia (17.000).[49] Basado en el estudio morfométrico craneal de las características que se cree están asociadas con el proceso de la domesticación, éstos se han propuesto como perros del Paleolítico temprano.[143] Estas características de rostro acortada, apiñamiento de los dientes, y la ausencia o la rotación de los premolares se han documentado en tanto lobos antiguos como modernos.[89][114][131][145][146][147] En lugar de representar a los perros tempranos, estas muestras pueden representar "una local morfológicamente diferente, ya extinta, población de lobos".[114][148]
Referencias
- ↑ Dawkins, William Boyd, Sir; Sanford, W. Ayshford; Reynolds, Sidney H. (1912). «British Pleistocene Hyænidæ, Ursidæ, Canidæ, and Mustelidæ». A Monograph of the British Pleistocene Mammalia 2. London: Palaeontographical Society.
- ↑ a b Sardella, Raffaele; Bertè, Davide; Iurino, Dawid Adam; Cherin, Marco; Tagliacozzo, Antonio (2014). «The wolf from Grotta Romanelli (Apulia, Italy) and its implications in the evolutionary history of Canis lupus in the Late Pleistocene of Southern Italy». Quaternary International. 328-329: 179-195. doi:10.1016/j.quaint.2013.11.016.
- ↑ Flynn, John J.; Wesley-Hunt, Gina D. (2005). «Phylogeny of the Carnivora: Basal Relationships Among the Carnivoramorphans, and Assessment of the Position of 'Miacoidea' Relative to Carnivora». Journal of Systematic Paleontology 3: 1-28.
- ↑ «Eucyon davisi». Fossilworks. Consultado el 11 de julio de 2016.
- ↑ a b c d e f g h i j k l Wang, Xiaoming; Tedford, Richard H.; Dogs: Their Fossil Relatives and Evolutionary History. New York: Columbia University Press, 2008.
- ↑ a b c d e f g h i j k l m n ñ o p q r R.M. Nowak (2003). «Chapter 9 - Wolf evolution and taxonomy». En Mech, L. David; Boitani, Luigi, eds. Wolves: Behaviour, Ecology and Conservation. University of Chicago Press. pp. 239-258. ISBN 978-0-226-51696-7.
- ↑ a b c d e f g h i j k Nowak, R. M. (1979). North American Quaternary Canis 6 (6). Monograph of the Museum of Natural History, University of Kansas. pp. 1-154. ISBN 978-0-89338-007-6.
- ↑ Wayne, R. K. (1995). «Red wolf: To conserve or not to conserve». En Macdonald, D.; Handoca, L., eds. Canid News 3. Newsletter of the IUCN/SSC Specialist Group. pp. 7-12.
- ↑ «Canis lepophagus». Fossilworks. Consultado el 11 de julio de 2016.
- ↑ a b Kurten, B. (1974). «A History of Coyote-Like Dogs (Canidae, Mamalia)». Acta zoologica Fennica (140): 1-38.
- ↑ a b c d Kurten, B.; Anderson, E. (1980). Pleistocene mammals of North America. New York: Columbia University Press. pp. 1-442. ISBN 978-0-231-03733-4.
- ↑ a b Johnston, C. S. (1938). «Preliminary report on the vertebrate type locality of Cita Canyon and the description of an ancestral coyote». American Journal of Science. 5 35 (209): 383-390. doi:10.2475/ajs.s5-35.209.383.
- ↑ a b c d e f g h i j k l m Tedford, Richard H.; Wang, Xiaoming; Taylor, Beryl E. (2009). «Phylogenetic Systematics of the North American Fossil Caninae (Carnivora: Canidae)». Bulletin of the American Museum of Natural History 325: 1-218. doi:10.1206/574.1.
- ↑ a b Tedford, R.H. & Qiu, Z.-X., 1996 - A new canid genus from the Pliocene of Yushe, Shanxi Province - Vertebrata PalAsiatica 34 (1): 27-40
- ↑ Anderson, E. (1996). «A preliminary report on the Carnivora of Porcupine Cave, Park County, Colorado». En Churcher, C. S. R.; Stewart, K. M.; Seymour, K. L., eds. Palaeoecology and palaeoenvironments of late Cenozoic mammals. Toronto: University of Toronto Press. pp. 259-282. ISBN 978-0-8020-0728-5.
- ↑ Albright, III, L. B. (2000). Biostratigraphy and Vertebrate Paleontology of the San Timoteo Badlands, Southern California. University of California Publications in Geological Sciences 144. University of California Press. pp. 1-121. ISBN 978-0-520-91598-5.
- ↑ «Canis edwardii». Fossilworks. Consultado el 11 de julio de 2016.
- ↑ Berta, A. (1995). «Fossil carnivores from the Leisey Shell Pits, Hillsborough County, Florida». En Hulbert, Jr., R. C.; Morgan, G. S.; Webb, S. D., eds. Paleontology and geology of the Leisey Shell Pits, early Pleistocene of Florida 37. Bulletin of the Florida Museum of Natural History. pp. 463-499.
- ↑ «Canis ambrusteri». Fossilworks. Consultado el 11 de julio de 2016.
- ↑ Martin, R. A.; Webb, S. D. (1974). «Late Pleistocene mammals from the Devil's Den fauna, Levy County». En Webb, S. D., ed. Pleistocene Mammals of Florida. Gainesville: University Presses of Florida. pp. 114-145. ISBN 978-0-8130-0361-0.
- ↑ Kurten, B. 1984: Geographic differentiation in the Rancholabrean dire wolf (Canis dirus Leidy) in North America. In Genoways, H. H. & Dawson, M. R. (eds.): Contributions in Quaternary Vertebrate Paleontology: A Volume in Memorial to John E. Guilday, 218–227. Carnegie Museum of Natural History Special Publication 8
- ↑ «Canis dirus». Fossilworks. Consultado el 11 de julio de 2016.
- ↑ Goulet, G. D. (1993). Comparison of temporal and geographical skull variation among Nearctic, modern, Holocene, and late Pleistocene gray wolves (Canis lupus) and selected Canis (Master's Degree). Winnipeg: University of Manitoba. pp. 1-116.
- ↑ Berta, A. (1988). Quaternary evolution and biogeography of the large South American Canidae (Mammalia: Carnivora) 132. University of California Publications in Geological Sciences. pp. 1-49. ISBN 978-0-520-09960-9.
- ↑ Nowak, R. M.; Federoff, N. E. (2002). «The systematic status of the Italian wolf Canis lupus». Acta theriol 47 (3): 333-338. doi:10.1007/BF03194151.
- ↑ a b c Kurtén, B. (1968). Pleistocene mammals of Europe. London: Weidenfeld and Nicolson. p. 317. ISBN 978-0-202-30953-8.
- ↑ a b Sotnikova, M (2010). «Dispersal of the Canini (Mammalia, Canidae: Caninae) across Eurasia during the Late Miocene to Early Pleistocene». Quaternary International 212 (2): 86-97. doi:10.1016/j.quaint.2009.06.008.
- ↑ Nowak, R. M. (1995). «Another look at wolf taxonomy». En Carbyn, L. H.; Fritts, S. H.; Seip, D. R., eds. Ecology and Conservation of Wolves in a Changing World. Edmonton, Canada: Canadian Circumpolar Institute. pp. 375-397. ISBN 978-0-919058-92-7.
- ↑ Torre, D. (1967). «I cani Villafranchiani della Toscana». Palaeontographia Italica (en italian) 63: 113-136.
- ↑ Torre, D. (1974). «Affinità dentali del cane della grotta di l’Escale». Rivista Italiana di Paleontologia e Stratigrafia (en italian) 80: 147-156.
- ↑ Torre, D. (1979). «The Ruscinian and Villafranchian dogs of Europe». Bollettino della Società Paleontologica Italiana (en italian) 18: 162-165.
- ↑ Martin, R. (1973). «Trois nouvelles espèces de Caninae (Canidae, Carnivora) des gisements Plio-Villafranchiens d’Europe. Documents des Laboratoires de Géologie de Lyon». Notes et Mémoires (en french) 57: 87-96.
- ↑ Sotnikova, M. (1989). «The carnivore mammals from the Pliocene to the early Pleistocene. Stratigraphic significance». Transactions of the Geological Institute of RAS 440: 1-122.
- ↑ Rook, L. (1993). I cani dell’Eurasia dal Miocene superiore al Pleistocene medio (Ph.D.). Universities of Modena, Bologna, Firenze, Roma “La Sapienza”, Italy.
- ↑ Sotnikova, M. (2001). «Remains of Canidae from the Lower Pleistocene site of Untermassfeld». En Kahlke, R. D., ed. Das Plestozan von Untermassfled bei Meiningen (Thuringen), part 2 40. Romisch-Germanisches Zentralmuseum. pp. 607-632. ISBN 978-3-7749-3080-3.
- ↑ Brugal, J. P.; Boudadi-Maligne, M. (2011). «Quaternary small to large canids in Europe: taxonomic status and biochronological contribution». Quaternary International 243: 171-182. doi:10.1016/j.quaint.2011.01.046.
- ↑ Cherin, M.; Bertè, D. F.; Sardella, R.; Rook, L. (2013). «Canis etruscus (Canidae, Mammalia) and its role in the faunal assemblage from Pantalla (Perugia, central Italy): comparison with the Late Villafranchian large carnivore guild of Italy». Bollettino della Società Paleontologica Italiana 52 (1): 11-18. doi:10.4435/BSPI.2013.05.
- ↑ Cherin, Marco; Bertè, Davide; Rook, Lorenzo; Sardella, Raffaele (2014). «Re-Defining Canis etruscus (Canidae, Mammalia): A New Look into the Evolutionary History of Early Pleistocene Dogs Resulting from the Outstanding Fossil Record from Pantalla (Italy)». Journal of Mammalian Evolution 21 (1): 95-110. doi:10.1007/s10914-013-9227-4.
- ↑ Lumley, H. de, Kahlke, H.D., Moigne, A.M., Moulle, P.E., 1988. Les faunes de grands mammifères de la grotte du Vallonnet Roquebrune-Cap-Martin, Alpes-Maritimes. L’Anthropologie 92, 465–496.
- ↑ Soergel, W., 1928. Ein Kleiner Wolf aus dem Kiesen von Süssenborn. Zeitschrift der Deutschen Geologische Gesellschaft 80, 227–255.
- ↑ Garrido, Guiomar; Arribas, Alfonso (2008). «Canis accitanus nov. sp., a new small dog (Canidae, Carnivora, Mammalia) from the Fonelas P-1 Plio-Pleistocene site (Guadix basin, Granada, Spain)». Geobios 41 (6): 751-761. doi:10.1016/j.geobios.2008.05.002.
- ↑ a b
- ↑ a b New remains of Canis chihliensis (Mammalia, Carnivora) from Shanshenmiaozui, a lower Pleistocene site in Yangyuan, Hebei Tong, H.-w., N. Hu, and X. Wang. 2012. Vertebrata PalAsiatica 50(4):335-360
- ↑ a b c d e f g h i Koler-Matznick, Janice (2002). «The origin of the dog revisited». Anthrozoos: A Multidisciplinary Journal of the Interactions of People & Animals 15 (2): 98. doi:10.2752/089279302786992595.
- ↑ Olsen, S. J. (1985). Origins of the domestic dog: the fossil record. Univ. of Arizona Press, Tucson, USA. pp. 88-89.
- ↑ Olsen S J, Olsen J W, Qi G Q, 1982. The position of Canis lupus variabilis, from Zhoukoudian, in the ancestral lineage of the domestic dog, Canis familiaris. Vert PalAsia, 20(3): 264-267(in Chinese with last page abstract in English)
- ↑ Error en la cita: Etiqueta
<ref>no válida; no se ha definido el contenido de las referencias llamadaslawrence1967b - ↑ Pei, W.C. (1934). The carnivora from locality 1 of Choukoutien. Palaeontologia Sinica, Series C, vol. 8, Fascicle 1. Geological Survey of China, Beijing. pp. 1-45.
- ↑ a b Sablin, Mikhail V.; Khlopachev, Gennady A. (2002). The Earliest Ice Age Dogs:Evidence from Eliseevichi I. Wenner-Gren Foundation for Anthropological Research. Consultado el 10 de enero de 2015.
- ↑ a b c d e f g h i Thalmann, O.; Shapiro, B.; Cui, P.; Schuenemann, V. J.; Sawyer, S. K.; Greenfield, D. L.; Germonpre, M. B.; Sablin, M. V.; Lopez-Giraldez, F.; Domingo-Roura, X.; Napierala, H.; Uerpmann, H.-P.; Loponte, D. M.; Acosta, A. A.; Giemsch, L.; Schmitz, R. W.; Worthington, B.; Buikstra, J. E.; Druzhkova, A.; Graphodatsky, A. S.; Ovodov, N. D.; Wahlberg, N.; Freedman, A. H.; Schweizer, R. M.; Koepfli, K.- P.; Leonard, J. A.; Meyer, M.; Krause, J.; Paabo, S. et al. (2013). «Complete Mitochondrial Genomes of Ancient Canids Suggest a European Origin of Domestic Dogs». Science 342 (6160): 871-4. PMID 24233726. doi:10.1126/science.1243650.
- ↑ a b Morey, Darcy F.; Jeger, Rujana (2015). «Paleolithic dogs: Why sustained domestication then?». Journal of Archaeological Science: Reports 3: 420. doi:10.1016/j.jasrep.2015.06.031.
- ↑ Wayne, Robert K. (1986). «Cranial Morphology of Domestic and Wild Canids: The Influence of Development on Morphological Change». Evolution 40 (2): 243. JSTOR 2408805. doi:10.2307/2408805.
- ↑ Drake, Abby Grace; Klingenberg, Christian Peter (2010). «Large‐Scale Diversification of Skull Shape in Domestic Dogs: Disparity and Modularity». The American Naturalist 175 (3): 289. PMID 20095825. doi:10.1086/650372.
- ↑ Roberts, Taryn; McGreevy, Paul; Valenzuela, Michael (2010). «Human Induced Rotation and Reorganization of the Brain of Domestic Dogs». PLoS ONE 5 (7): e11946. PMID 20668685. doi:10.1371/journal.pone.0011946.
- ↑ Rand, David M. (2001). «The Units of Selection on Mitochondrial DNA». Annual Review of Ecology and Systematics 32: 415-448. doi:10.1146/annurev.ecolsys.32.081501.114109.
- ↑ a b c d e f g h Robert K. Wayne, Jennifer A. Leonard, Carles Vila (2006). «Chapter 19:Genetic Analysis of Dog Domestication». En Melinda A. Zeder, ed. Documenting Domestication:New Genetic and Archaeological Paradigms. University of California Press. pp. 279-295. ISBN 978-0-520-24638-6.
- ↑ Hofreiter, Michael; Serre, David; Poinar, Hendrik N.; Kuch, Melanie; Pääbo, Svante (2001). «Ancient DNA». Nature Reviews Genetics 2 (5): 353-9. PMID 11331901. doi:10.1038/35072071.
- ↑ Li, Wen-Hsiung (1997). Molecular evolution. Sunderland, Massachusetts: Sinauer Associates. ISBN 978-0-87893-463-8.[página requerida]
- ↑ Pesole, G; Gissi, C; De Chirico, A; Saccone, C (1999). «Nucleotide substitution rate of mammalian mitochondrial genomes». Journal of Molecular Evolution 48 (4): 427-34. PMID 10079281. doi:10.1007/pl00006487.
- ↑ Wan, Qiu-Hong; Wu, Hua; Fujihara, Tsutomu; Fang, Sheng-Guo (2003). «Which genetic marker for which conservation genetics issue?». Electrophoresis 25 (14): 2165. PMID 15274000. doi:10.1002/elps.200305922.
- ↑ Birky, C. William (2001). «The Inheritance of Genes in Mitochondria and Chloroplasts: Laws, Mechanisms, and Models». Annual Review of Genetics 35: 125-48. PMID 11700280. doi:10.1146/annurev.genet.35.102401.090231.
- ↑ Avise, J. C. (1994). Molecular Markers, Natural History, and Evolution. New York: Chapman & Hall. ISBN 978-0-412-03781-8.
- ↑ Avise, J. C. (2000). Phylogeography: The History and Formation of Species. Cambridge, Massachusetts: Harvard University Press. ISBN 978-0-674-66638-2.
- ↑ Tedford, Richard H.; Wang, Xiaoming; Taylor, Beryl E. (2009). «Phylogenetic Systematics of the North American Fossil Caninae (Carnivora: Canidae)». Bulletin of the American Museum of Natural History 325: 1-218. doi:10.1206/574.1.
- ↑ a b c d Vila, C. (1997). «Multiple and ancient origins of the domestic dog». Science 276 (5319): 1687-9. PMID 9180076. doi:10.1126/science.276.5319.1687.
- ↑ a b c d Wayne, R. & Ostrander, Elaine A. (1999). «Origin, genetic diversity, and genome structure of the domestic dog». BioEssays 21 (3): 247-57. PMID 10333734. doi:10.1002/(SICI)1521-1878(199903)21:3<247::AID-BIES9>3.0.CO;2-Z.
- ↑ Boyko, A. (2009). «Complex population structure in African village dogs and its implications for inferring dog domestication history». Proceedings of the National Academy of Sciences 106 (33): 13903-13908. PMC 2728993. PMID 19666600. doi:10.1073/pnas.0902129106.
- ↑ Pang, J. (2009). «mtDNA data indicate a single origin for dogs south of Yangtze River, less than 16,300 years ago, from numerous wolves». Molecular Biology and Evolution 26 (12): 2849-64. PMC 2775109. PMID 19723671. doi:10.1093/molbev/msp195.
- ↑ a b c d Lindblad-Toh, K.; Wade, C. M.; Mikkelsen, T. S.; Karlsson, E. K.; Jaffe, D. B.; Kamal, M.; Clamp, M.; Chang, J. L.; Kulbokas, E. J.; Zody, M. C.; Mauceli, E.; Xie, X.; Breen, M.; Wayne, R. K.; Ostrander, E. A.; Ponting, C. P.; Galibert, F.; Smith, D. R.; Dejong, P. J.; Kirkness, E.; Alvarez, P.; Biagi, T.; Brockman, W.; Butler, J.; Chin, C. W.; Cook, A.; Cuff, J.; Daly, M. J.; Decaprio, D. et al. (2005). «Genome sequence, comparative analysis and haplotype structure of the domestic dog». Nature 438 (7069): 803-819. Bibcode:2005Natur.438..803L. PMID 16341006. doi:10.1038/nature04338.
- ↑ a b c d e f g h i j k Freedman, Adam H.; Gronau, Ilan; Schweizer, Rena M.; Ortega-Del Vecchyo, Diego; Han, Eunjung; Silva, Pedro M.; Galaverni, Marco; Fan, Zhenxin; Marx, Peter; Lorente-Galdos, Belen; Beale, Holly; Ramirez, Oscar; Hormozdiari, Farhad; Alkan, Can; Vilà, Carles; Squire, Kevin; Geffen, Eli; Kusak, Josip; Boyko, Adam R.; Parker, Heidi G.; Lee, Clarence; Tadigotla, Vasisht; Siepel, Adam; Bustamante, Carlos D.; Harkins, Timothy T.; Nelson, Stanley F.; Ostrander, Elaine A.; Marques-Bonet, Tomas; Wayne, Robert K. et al. (2014). «Genome Sequencing Highlights the Dynamic Early History of Dogs». PLoS Genetics 10 (1): e1004016. PMC 3894170. PMID 24453982. doi:10.1371/journal.pgen.1004016.
- ↑ a b c d e f g h i j k l Skoglund, P. (2015). «Ancient wolf genome reveals an early divergence of domestic dog ancestors and admixture into high-latitude breeds». Current Biology 25 (11): 1515-9. PMID 26004765. doi:10.1016/j.cub.2015.04.019.
- ↑ a b c d e f g h i j k l m n ñ o p q Fan, Zhenxin; Silva, Pedro; Gronau, Ilan; Wang, Shuoguo; Armero, Aitor Serres; Schweizer, Rena M.; Ramirez, Oscar; Pollinger, John; Galaverni, Marco; Ortega Del-Vecchyo, Diego; Du, Lianming; Zhang, Wenping; Zhang, Zhihe; Xing, Jinchuan; Vilà, Carles; Marques-Bonet, Tomas; Godinho, Raquel; Yue, Bisong; Wayne, Robert K. (2016). «Worldwide patterns of genomic variation and admixture in gray wolves». Genome Research 26 (2): 163-73. PMID 26680994. doi:10.1101/gr.197517.115.
- ↑ a b Wayne, R. (1993). «Molecular evolution of the dog family». Trends in Genetics 9 (6): 218-24. PMID 8337763. doi:10.1016/0168-9525(93)90122-X.
- ↑ Wurster-Hill, D. H.; Centerwall, W. R. (1982). «The interrelationships of chromosome banding patterns in canids, mustelids, hyena, and felids». Cytogenetics and Cell Genetics 34: 178-192. PMID 7151489. doi:10.1159/000131806.
- ↑ Gottelli, D.; Sillero-Zubiri, C.; Applebaum, G. D.; Roy, M. S.; Girman, D. J.; Garcia-Moreno, J.; Ostrander, E. A.; Wayne, R. K. (1994). «Molecular genetics of the most endangered canid: The Ethiopian wolf Canis simensis». Molecular Ecology 3 (4): 301-12. PMID 7921357. doi:10.1111/j.1365-294X.1994.tb00070.x.
- ↑ Pocock, R. I. (1941). Fauna of British India: Mammals 2. Taylor & Francis. pp. 146-163.
- ↑ Girman, D. J.; Vilà, C.; Geffen, E.; Creel, S.; Mills, M. G. L.; McNutt, J. W.; Ginsberg, J.; Kat, P. W.; Mamiya, K. H.; Wayne, R. K. (2001). «Patterns of population subdivision, gene flow and genetic variability in the African wild dog (Lycaon pictus)». Molecular Ecology 10 (7): 1703-23. PMID 11472538. doi:10.1046/j.0962-1083.2001.01302.x.
- ↑ Wayne, R.K.; Meyer, A.; Lehman, N.; van Valkeburgh, B.; Kat, P.W.; Fuller, T.K.; Girman, D.; O'Brien, S.J. (1990). «Large sequence divergence among mitochondrial DNA genotypes within populations of eastern African black-backed jackals». Proceedings of the National Academy of Sciences of the United States of America 87: 1772-1776. doi:10.1073/pnas.87.5.1772. Consultado el 21 de diciembre de 2011.
- ↑ Juliane Kaminski and Sarah Marshall-Pescini (2014). «Chapter 1 - The Social Dog:History and Evolution». The Social Dog:Behavior and Cognition. Elsevier. p. 4. ISBN 978-0-12407-931-1.
- ↑ Cox, C. B.; Moore, Peter D.; Ladle, Richard (2016). Biogeography: An Ecological and Evolutionary Approach. Wiley-Blackwell. p. 106. ISBN 978-1-118-96858-1.
- ↑ Editorial Board (2012). Concise Dictionary of Science. New Delhi: V&S Publishers. p. 137. ISBN 978-93-81588-64-2.
- ↑ a b Arora, Devender; Singh, Ajeet; Sharma, Vikrant; Bhaduria, Harvendra Singh; Patel, Ram Bahadur (2015). «Hgs Db: Haplogroups Database to understand migration and molecular risk assessment». Bioinformation 11 (6): 272-5. PMC 4512000. PMID 26229286. doi:10.6026/97320630011272.
- ↑ «Genetics Glossary». International Society of Genetic Genealogy. 2015. Consultado el 22 de julio de 2016.
- ↑ a b c d e f g h i j k l m n Pilot, M. (2010). «Phylogeographic history of grey wolves in Europe». BMC Evolutionary Biology 10: 104. PMC 2873414. PMID 20409299. doi:10.1186/1471-2148-10-104.
- ↑ Aggarwal, R. K.; Kivisild, T.; Ramadevi, J.; Singh, L. (2007). «Mitochondrial DNA coding region sequences support the phylogenetic distinction of two Indian wolf species». Journal of Zoological Systematics and Evolutionary Research 45 (2): 163-172. doi:10.1111/j.1439-0469.2006.00400.x.
- ↑ Sharma, D. K.; Maldonado, J. E.; Jhala, Y. V.; Fleischer, R. C. (2004). «Ancient wolf lineages in India». Proceedings of the Royal Society B: Biological Sciences 271 (Suppl 3): S1-S4. PMC 1809981. PMID 15101402. doi:10.1098/rsbl.2003.0071.
- ↑ a b c Randi, Ettore (2011). «Genetics and conservation of wolves Canis lupus in Europe». Mammal Review 41 (2): 99-111. doi:10.1111/j.1365-2907.2010.00176.x.
- ↑ Tamm, E.; Kivisild, T.; Reidla, M.; Metspalu, M.; Smith, D. G.; Mulligan, C. J.; Bravi, C. M.; Rickards, O.; Martinez-Labarga, C.; Khusnutdinova, E. K.; Fedorova, S. A.; Golubenko, M. V.; Stepanov, V. A.; Gubina, M. A.; Zhadanov, S. I.; Ossipova, L. P.; Damba, L.; Voevoda, M. I.; Dipierri, J. E.; Villems, R.; Malhi, R. S. (2007). «Beringian Standstill and Spread of Native American Founders». En Carter, Dee, ed. PLoS ONE 2 (9): e829. PMC 1952074. PMID 17786201. doi:10.1371/journal.pone.0000829.
- ↑ a b c d e f g Leonard, J. A.; Vilà, C; Fox-Dobbs, K; Koch, P. L.; Wayne, R. K.; Van Valkenburgh, B (2007). «Megafaunal extinctions and the disappearance of a specialized wolf ecomorph». Current Biology 17 (13): 1146-50. PMID 17583509. doi:10.1016/j.cub.2007.05.072.
- ↑ a b Germonpré, Mietje; Sablin, Mikhail V.; Stevens, Rhiannon E.; Hedges, Robert E.M.; Hofreiter, Michael; Stiller, Mathias; Despre´s, Viviane R. (2009). «Fossil dogs and wolves from Palaeolithic sites in Belgium, the Ukraine and Russia: osteometry, ancient DNA and stable isotopes». Journal of Archaeological Science 36 (2): 473-490. doi:10.1016/j.jas.2008.09.033.
- ↑ a b Hofreiter, Michael; Barnes, Ian (2010). «Diversity lost: Are all Holarctic large mammal species just relict populations?». BMC Biology 8: 46. PMC 2858106. PMID 20409351. doi:10.1186/1741-7007-8-46.
- ↑ Germonpré, Mietje; Sablin, Mikhail V.; Després, Viviane; Hofreiter, Michael; Lázničková-Galetová, Martina; Stevens, Rhiannon E.; Stiller, Mathias (2013). «Palaeolithic dogs and the early domestication of the wolf: A reply to the comments of Crockford and Kuzmin (2012)». Journal of Archaeological Science 40: 786-792. doi:10.1016/j.jas.2012.06.016.
- ↑ a b c Hofreiter, Michael (2007). «Pleistocene Extinctions: Haunting the Survivors». Current Biology 17 (15): R609-11. PMID 17686436. doi:10.1016/j.cub.2007.06.031.
- ↑ a b c Pilot, M; Greco, C; Vonholdt, B M; Jędrzejewska, B; Randi, E; Jędrzejewski, W; Sidorovich, V E; Ostrander, E A et al. (2013). «Genome-wide signatures of population bottlenecks and diversifying selection in European wolves». Heredity 112 (4): 428-42. PMID 24346500. doi:10.1038/hdy.2013.122.
- ↑ a b c d e f g h i j Koblmüller, Stephan; Vilà, Carles; Lorente-Galdos, Belen; Dabad, Marc; Ramirez, Oscar; Marques-Bonet, Tomas; Wayne, Robert K.; Leonard, Jennifer A. (2016). «Whole mitochondrial genomes illuminate ancient intercontinental dispersals of grey wolves (Canis lupus)». Journal of Biogeography. doi:10.1111/jbi.12765.
- ↑ Weckworth, Byron V.; Talbot, Sandra; Sage, George K.; Person, David K.; Cook, Joseph (2005). «A Signal for Independent Coastal and Continental histories among North American wolves». Molecular Ecology 14 (4): 917. PMID 15773925. doi:10.1111/j.1365-294X.2005.02461.x.
- ↑ Weckworth, Byron V.; Talbot, Sandra L.; Cook, Joseph A. (2010). «Phylogeography of wolves (Canis lupus) in the Pacific Northwest». Journal of Mammalogy 91 (2): 363. doi:10.1644/09-MAMM-A-036.1.
- ↑ Weckworth, Byron V.; Dawson, Natalie G.; Talbot, Sandra L.; Flamme, Melanie J.; Cook, Joseph A. (2011). «Going Coastal: Shared Evolutionary History between Coastal British Columbia and Southeast Alaska Wolves (Canis lupus)». PLoS ONE 6 (5): e19582. PMID 21573241. doi:10.1371/journal.pone.0019582.
- ↑ a b Vonholdt, B. M.; Cahill, J. A.; Fan, Z.; Gronau, I.; Robinson, J.; Pollinger, J. P.; Shapiro, B.; Wall, J. et al. (2016). «Whole-genome sequence analysis shows that two endemic species of North American wolf are admixtures of the coyote and gray wolf». Science Advances 2 (7): e1501714. doi:10.1126/sciadv.1501714.
- ↑ a b c d Matsumura, Shuichi; Inoshima, Yasuo; Ishiguro, Naotaka (2014). «Reconstructing the colonization history of lost wolf lineages by the analysis of the mitochondrial genome». Molecular Phylogenetics and Evolution 80: 105-12. PMID 25132126. doi:10.1016/j.ympev.2014.08.004.
- ↑ Abe, H. (1999). «Diversity and conservation of mammals of Japan». En Yokohata, Y.; Nakamura, S., eds. Recent Advances in the Biology of Japanese Insectivore. Shōbara: Hiba Society of Natural History. pp. 89-104.
- ↑ Ishiguro, Naotaka; Inoshima, Yasuo; Shigehara, Nobuo; Ichikawa, Hideo; Kato, Masaru (2010). «Osteological and Genetic Analysis of the Extinct Ezo Wolf (Canis Lupus Hattai) from Hokkaido Island, Japan». Zoological Science 27 (4): 320-4. PMID 20377350. doi:10.2108/zsj.27.320.
- ↑ Walker, Brett (2008). The Lost Wolves of Japan. University of Washington Press. p. 42.
- ↑ a b c Ishiguro, Naotaka; Inoshima, Yasuo; Shigehara, Nobuo (2009). «Mitochondrial DNA Analysis of the Japanese Wolf (Canis lupus hodophilax Temminck, 1839) and Comparison with Representative Wolf and Domestic Dog Haplotypes». Zoological Science 26 (11): 765-70. PMID 19877836. doi:10.2108/zsj.26.765.
- ↑ Morell, Virginia (2016). «How do you save a wolf that's not really a wolf?». Science 353 (6300). doi:10.1126/science.aag0699.
- ↑ Vila, C.; Amorim, I. R.; Leonard, J. A.; Posada, D.; Castroviejo, J.; Petrucci-Fonseca, F.; Crandall, K. A.; Ellegren, H. et al. (1999). «Mitochondrial DNA phylogeography and population history of the grey wolf Canis lupus». Molecular Ecology 8 (12): 2089-103. PMID 10632860. doi:10.1046/j.1365-294x.1999.00825.x.
- ↑ a b Leonard, Jennifer A.; Vilà, Carles; Wayne, Robert K. (2004). «Fast Track: Legacy lost: Genetic variability and population size of extirpated US grey wolves (Canis lupus)». Molecular Ecology 14 (1): 9-17. PMID 15643947. doi:10.1111/j.1365-294X.2004.02389.x.
- ↑ a b Larson G, Bradley DG (2014). «How Much Is That in Dog Years? The Advent of Canine Population Genomics». PLOS Genetics 10 (1): e1004093. doi:10.1371/journal.pgen.1004093.
- ↑ Koepfli, K.-P.; Pollinger, J.; Godinho, R.; Robinson, J.; Lea, A.; Hendricks, S.; Schweizer, R. M.; Thalmann, O.; Silva, P.; Fan, Z.; Yurchenko, A. A.; Dobrynin, P.; Makunin, A.; Cahill, J. A.; Shapiro, B.; Álvares, F.; Brito, J. C.; Geffen, E.; Leonard, J. A.; Helgen, K. M.; Johnson, W. E.; O’Brien, S. J.; Van Valkenburgh, B.; Wayne, R. K. (17 de agosto de 2015). «Genome-wide Evidence Reveals that African and Eurasian Golden Jackals Are Distinct Species». Current Biology 25 (16): 2158-65. PMID 26234211. doi:10.1016/j.cub.2015.06.060.
- ↑ a b Van Valkenburgh, Blaire; Hayward, Matthew W.; Ripple, William J.; Meloro, Carlo; Roth, V. Louise (2016). «The impact of large terrestrial carnivores on Pleistocene ecosystems». Proceedings of the National Academy of Sciences 113 (4): 862-867. doi:10.1073/pnas.1502554112.
- ↑ Shipman, P. (2015). The Invaders:How humans and their dogs drove Neanderthals to extinction. Harvard University Press. ISBN 978-0-674-73676-4.
- ↑ Cagan, Alex; Blass, Torsten (2016). «Identification of genomic variants putatively targeted by selection during dog domestication». BMC Evolutionary Biology 16. doi:10.1186/s12862-015-0579-7.
- ↑ Song, Jiao-Jiao; Wang, Wen-Zhi; Otecko, Newton O.; Peng, Min-Sheng; Zhang, Ya-Ping (2016). «Reconciling the conflicts between mitochondrial DNA haplogroup trees of Canis lupus». Forensic Science International: Genetics 23: 83-85. doi:10.1016/j.fsigen.2016.03.008.
- ↑ a b c d e f Perri, Angela (2016). «A wolf in dog's clothing: Initial dog domestication and Pleistocene wolf variation». Journal of Archaeological Science 68: 1-4. doi:10.1016/j.jas.2016.02.003.
- ↑ a b Gross, Michael (2015). «Are dogs just like us?». Current Biology 25 (17): R733-6. PMID 26561653. doi:10.1016/j.cub.2015.08.018.
- ↑ «Ancient wolf genome pushes back dawn of the dog». Nature. 2015.
- ↑ a b Lee, E. (2015). «Ancient DNA analysis of the oldest canid species from the Siberian Arctic and genetic contribution to the domestic dog». PLoS ONE 10 (5): e0125759. PMC 4446326. PMID 26018528. doi:10.1371/journal.pone.0125759.
- ↑ Bocherens, Hervé; Drucker, Dorothée G.; Bonjean, Dominique; Bridault, Anne; Conard, Nicholas J.; Cupillard, Christophe; Germonpré, Mietje; Höneisen, Markus; Münzel, Susanne C.; Napierala, Hannes; Patou-Mathis, Marylène; Stephan, Elisabeth; Uerpmann, Hans-Peter; Ziegler, Reinhard (2011). «Isotopic evidence for dietary ecology of cave lion (Panthera spelaea) in North-Western Europe: Prey choice, competition and implications for extinction». Quaternary International 245 (2): 249-261. doi:10.1016/j.quaint.2011.02.023.
- ↑ Yeakel, J. D.; Guimaraes, P. R.; Bocherens, H.; Koch, P. L. (2013). «The impact of climate change on the structure of Pleistocene food webs across the mammoth steppe». Proceedings of the Royal Society B: Biological Sciences 280 (1762): 20130239. doi:10.1098/rspb.2013.0239.
- ↑ a b Bocherens, Hervé (2015). «Isotopic tracking of large carnivore palaeoecology in the mammoth steppe». Quaternary Science Reviews 117: 42-71. doi:10.1016/j.quascirev.2015.03.018.
- ↑ a b c d e f g h i j k l m n Leonard, Jennifer (2014). «Ecology drives evolution in grey wolves». Evolution Ecology Research 16: 461-473.
- ↑ Vila, C.; Sundqvist, A.-K.; Flagstad, O.; Seddon, J.; Rnerfeldt, S. B.; Kojola, I.; Casulli, A.; Sand, H.; Wabakken, P.; Ellegren, H. (2003). «Rescue of a severely bottlenecked wolf (Canis lupus) population by a single immigrant». Proceedings of the Royal Society B: Biological Sciences 270 (1510): 91-97. doi:10.1098/rspb.2002.2184.
- ↑ a b c Muñoz-Fuentes, Violeta; Darimont, Chris T.; Wayne, Robert K.; Paquet, Paul C.; Leonard, Jennifer A. (2009). «Ecological factors drive differentiation in wolves from British Columbia». Journal of Biogeography 36 (8): 1516-1531. doi:10.1111/j.1365-2699.2008.02067.x.
- ↑ a b c Musiani, Marco; Leonard, Jennifer A.; Cluff, H. Dean; Gates, C. Cormack; Mariani, Stefano; Paquet, Paul C.; Vilà, Carles; Wayne, Robert K. (2007). «Differentiation of tundra/taiga and boreal coniferous forest wolves: Genetics, coat colour and association with migratory caribou». Molecular Ecology 16 (19): 4149-70. PMID 17725575. doi:10.1111/j.1365-294X.2007.03458.x.
- ↑ Valière, Nathaniel; Fumagalli, Luca; Gielly, Ludovic; Miquel, Christian; Lequette, Benoît; Poulle, Marie-Lazarine; Weber, Jean-Marc; Arlettaz, Raphaël et al. (2003). «Long-distance wolf recolonization of France and Switzerland inferred from non-invasive genetic sampling over a period of 10 years». Animal Conservation 6: 83-92. doi:10.1017/S1367943003003111.
- ↑ Fabbri, E.; Caniglia, R.; Kusak, J.; Galov, A.; Gomerčić, T.; Arbanasić, H.; Huber, D.; Randi, E. (2014). «Genetic structure of expanding wolf (Canis lupus) populations in Italy and Croatia, and the early steps of the recolonization of the Eastern Alps». Mammalian Biology - Zeitschrift für Säugetierkunde 79 (2): 138-148. doi:10.1016/j.mambio.2013.10.002.
- ↑ Carmichael, L. E.; Nagy, J. A.; Larter, N. C.; Strobeck, C. (2001). «Prey specialization may influence patterns of gene flow in wolves of the Canadian Northwest». Molecular Ecology 10 (12): 2787-98. PMID 11903892. doi:10.1046/j.0962-1083.2001.01408.x.
- ↑ Carmichael, L. E. (2006). Ecological Genetics of Northern Wolves and Arctic Foxes. Ph.D. Dissertation. University of Alberta.
- ↑ a b Geffen, ELI; Anderson, Marti J.; Wayne, Robert K. (2004). «Climate and habitat barriers to dispersal in the highly mobile grey wolf». Molecular Ecology 13 (8): 2481-90. PMID 15245420. doi:10.1111/j.1365-294X.2004.02244.x.
- ↑ Pilot, Malgorzata; Jedrzejewski, Wlodzimierz; Branicki, Wojciech; Sidorovich, Vadim E.; Jedrzejewska, Bogumila; Stachura, Krystyna; Funk, Stephan M. (2006). «Ecological factors influence population genetic structure of European grey wolves». Molecular Ecology 15 (14): 4533-53. PMID 17107481. doi:10.1111/j.1365-294X.2006.03110.x.
- ↑ a b c d e f g h Flower, Lucy O.H.; Schreve, Danielle C. (2014). «An investigation of palaeodietary variability in European Pleistocene canids». Quaternary Science Reviews 96: 188-203. doi:10.1016/j.quascirev.2014.04.015.
- ↑ a b Schweizer, Rena M.; Vonholdt, Bridgett M.; Harrigan, Ryan; Knowles, James C.; Musiani, Marco; Coltman, David; Novembre, John; Wayne, Robert K. (2016). «Genetic subdivision and candidate genes under selection in North American grey wolves». Molecular Ecology 25 (1): 380-402. PMID 26333947. doi:10.1111/mec.13364.
- ↑ a b Schweizer, Rena M.; Robinson, Jacqueline; Harrigan, Ryan; Silva, Pedro; Galverni, Marco; Musiani, Marco; Green, Richard E.; Novembre, John et al. (2016). «Targeted capture and resequencing of 1040 genes reveal environmentally driven functional variation in grey wolves». Molecular Ecology 25 (1): 357-79. PMID 26562361. doi:10.1111/mec.13467.
- ↑ O'Keefe, F. Robin; Meachen, Julie; Fet, Elizabeth V.; Brannick, Alexandria (2013). «Ecological determinants of clinal morphological variation in the cranium of the North American gray wolf». Journal of Mammalogy 94 (6): 1223-1236. doi:10.1644/13-MAMM-A-069.
- ↑ Carmichael, L. E.; Krizan, J.; Nagy, J. A.; Fuglei, E.; Dumond, M.; Johnson, D.; Veitch, A.; Berteaux, D. et al. (2007). «Historical and ecological determinants of genetic structure in arctic canids». Molecular Ecology 16 (16): 3466-83. PMID 17688546. doi:10.1111/j.1365-294X.2007.03381.x.
- ↑
- ↑ Rook, L.; Torre, D. (1996). «The wolf-event in western Europe and the beginning of the Late Villafranchian». Neues Jahrbuch fur Geologie und Palaontologie Monatshefte 8: 495-501.
- ↑ Gliozzi, E.; Abbazzi, L.; Argenti, A.; Azzaroli, A.; Caloi, L.; Capasso Barbato, L.; di Stefano, G.; Esu, D.; Ficcarelli, G.; Girotti, O.; Kotsakis, T.; Masini, F.; Mazza, P.; Mezzabotta, C.; Palombo, M. R.; Petronio, C.; Rook, L.; Sala, B.; Sardella, R.; Zanalda, E.; Torre, D. (1997). «Biochronology of selected mammals, Molluscs and ostracodes from the Middle Pliocene to the Late Pleistocene in Italy. The state of the art». Rivista Italiana di Paleontologia e Stratigrafia 103 (3): 369-388. doi:10.13130/2039-4942/5299.
- ↑ Currant, A. P. (1984). The mammalian remains. En Green, H. S., ed. «Pontnewydd Cave: A Lower Palaeolithic Hominid Site in Wales: the First Report». Journal of Archaeological Science 12 (4) (Amgueddfa Genedlaethol Cymru). pp. 177-181. ISBN 978-0-7200-0282-9. doi:10.1016/0305-4403(85)90038-X.
- ↑ Fox-Dobbs, Kena; Leonard, Jennifer A.; Koch, Paul L. (2008). «Pleistocene megafauna from eastern Beringia: Paleoecological and paleoenvironmental interpretations of stable carbon and nitrogen isotope and radiocarbon records». Palaeogeography, Palaeoclimatology, Palaeoecology 261: 30-46. doi:10.1016/j.palaeo.2007.12.011.
- ↑ Baryshnikov, Gennady F.; Mol, Dick; Tikhonov, Alexei N (2009). «Finding of the Late Pleistocene carnivores in Taimyr Peninsula (Russia, Siberia) with paleoecological context». Russian Journal of Theriology (Russian Journal of Theriology) 8 (2): 107-113. Consultado el 23 de diciembre de 2014.
- ↑ Ovodov, N. (2011). «A 33,000-year-old incipient dog from the Altai Mountains of Siberia: Evidence of the earliest domestication disrupted by the Last Glacial Maximum». PLoS ONE 6 (7): e22821. PMC 3145761. PMID 21829526. doi:10.1371/journal.pone.0022821.
- ↑ a b Germonpré, Mietje; Lázničková-Galetová, Martina; Losey, Robert J.; Räikkönen, Jannikke; Sablin, Mikhail V. (2015). «Large canids at the Gravettian Předmostí site, the Czech Republic: The mandible». Quaternary International. 359-360: 261-279. doi:10.1016/j.quaint.2014.07.012.
- ↑ Germonpré, Mietje; Laznickova-Galetova, Martina; Sablin, Mikhail V. (2012). «Palaeolithic dog skulls at the Gravettian Predmosti site, the Czech Republic». Journal of Archaeological Science 39: 184-202. doi:10.1016/j.jas.2011.09.022.
- ↑ Boudadi-Maligne, M. (2010). «Les Canis pleistocenes du sud de la France: approche biosystematique, evolutive et biochronologique. Ph.D. dissertation». Arch´eologie et Pr´ehistoire (Universitie Bordeaux) (4126).
- ↑ Dimitrijević, V.; Vuković, S. (2015). «Was the Dog Locally Domesticated in the Danube Gorges? Morphometric Study of Dog Cranial Remains from Four Mesolithic-Early Neolithic Archaeological Sites by Comparison with Contemporary Wolves». International Journal of Osteoarchaeology 25: 1-30. doi:10.1002/oa.2260.
- ↑ Germonpréa, Mietje; Sablinb, Mikhail V.; Stevensc, Rhiannon E.; Hedgesd, Robert E. M.; Hofreitere, Michael; Stillere, Mathias; Desprése, Viviane R. (2008). «Fossil dogs and wolves from Palaeolithic sites in Belgium, the Ukraine and Russia: osteometry, ancient DNA and stable isotopes». Journal of Archaeological Science 36 (2): 473-490. doi:10.1016/j.jas.2008.09.033.
- ↑ Larson G (2012). «Rethinking dog domestication by integrating genetics, archeology, and biogeography». PNAS 109 (23): 8878-8883. PMC 3384140. PMID 22615366. doi:10.1073/pnas.1203005109.
<ref> definida en las <references> con nombre «soergel1925» no se utiliza en el texto anterior.





