KFERQ
KFERQ representa el código pentapéptido de la secuencia de aminoácidos Lys-Phe-Glu-Arg-Gln. Esta secuencia se encuentra en proteínas citosólicas específicas para la autofagia mediada por chaperonas (acompañantes), por lo que constituye la señal que llevan estas proteínas para que se degraden en el lisosoma. El KFERQ es reconocido por un complejo de proteínas chaperonas. La secuencia KFERQ se encuentran principalmente en proteínas citosólicas y se estima que alrededor del 30% de estas proteínas contienen esta secuencia.
Descubrimiento
[editar]Todos los sustratos de la autofagia mediada por chaperonas (CMA), contienen una secuencia de pentapéptidos para que sean degradados en el sistema lisosomal. Esta secuencia se denomina KFERQ y fue identificada por primera vez en una ribonucleasa microinyectada A (RNasa).[1] Fue descubierta gracias al siguiente experimento: a partir de una microinyección de glóbulos rojos, se introdujeron proteínas marcadas en un cultivo de fibroblasto humano, con el fin de seguir su catabolismo: La “bovine pancreatic ribonuclease A” se encontrava inicialmente en el citosol de la célula inyectada. Ésta fue transportada y degradada en el lisosoma, a causa del rechazo del suero. A continuación, se observaron dos fragmentos: RNase S peptide (residuo 1-20) y RNase S proteine(residuo 21-124). Este último se degradó de manera independiente, aplicándole un suero. En cambio el RNase peptide mostró un doble incremento de degradación sin presencia de suero. Es más, el enlace covalente del RNase S péptide con proteínas de la microinyección, causó la degradación del conjugado para convertirse en suero sensible. Estos resultados mostraron que el reconocimiento de la RNase con otras proteínas, sin aplicación de suero, está basado en algunas características de la secuencia terminal de los 20 aminoácidos contenidos en el RNase S péptide. A partir de un estudió, se detectó la secuencia esencial contenida en el RNase peptide (Lys-Phe-Glu-Arg-Gln KFERQ) responsable de que las proteínas fueran degradadas hacia el lisosoma. Para poder confirmar esta evidencia, se incrementó la cantidad de anticuerpos que reaccionavan sobre ese penptapéptido (KFERQ), y se observó que gran parte de las proteínas inmunoprecipitaron, sin presencia de suero.[2] Por lo tanto, se afirmó que la secuencia de aminoácidos KFERQ indica la degradación de la proteína citosólicas hacia los lisosomas.
Estructura
[editar]Todos los sustratos del CMA contienen una secuencia de 5 aminoácidos (Lys-PHe-Glu-Arg-Gln), que permite la degradación de las proteínas a través de los lisosomas. Esta secuencia se denomina KFERQ y fue identificada por primera vez en una ribonucleasa microinyectada. Esta secuencia consiste en un conjunto de Q seguido por cuatro aminoácidos seguidos que consisten en un ácido (+), un básico (-), una cadena voluminosa hidrófuga (φ) y un aminoácido hidrofóbico básico o voluminoso. Representación: (+φ-+Q).[3] Además, un pequeño número de estos péptidos permiten la substitución de Q por una N relacionada.
| Aminoácido | Símbolo | Abreviatura |
|---|---|---|
| Lisina | K | Lys |
| Fenilalanina | F | Phe |
| Ácido glutámico | E | Glu |
| Arginina | R | Arg |
| Glutamina | Q | Gln |
Propiedades
[editar]La secuencia KFERQ de las proteínas tiene la propiedad de ser reconocida por las chaperonas Hcs70,[4] las cuales dirigen a las proteínas marcadas por ella hacia el sistema lisosomal. El hcs70 se une a un péptido de 20 aminoácidos que contiene KFERQ unida a Sepharose, y ésta a su vez se une a la membrana citosólica del lisosoma. De esta forma las proteínas son capaces de llegar a este orgánulo de la célula para ser degradadas.[5][6]
| Secuencia | KFERQ |
|---|---|
| Longitud | 5 |
| Masa | 706.3751 |
| Punto Isoeléctrico | 9.84 |
| Carga Neta | +1 |
| Coeficiente de Extinción | 0 M-1 * cm-1 |
Función
[editar]El pentapéptido KFERQ son secuencias peptídicas que marcan proteínas citosólicas para su proteólisis lisosómica, siendo esta secuencia una de las señales para que las proteínas entren en los lisosomas. Es decir, la señal KFERQ marca a determinadas proteínas que encontramos en el citoplasma para su degradación en el lisosoma. Participa en la autofagia mediada por chaperonas o Autofagia mediada acompañante o AMA (en inglés: Chaperone mediated autofagia o en la sigla también inglesa: CMA).
La secuencia KFERQ en la autofagia mediada por chaperonas (CMA)
[editar]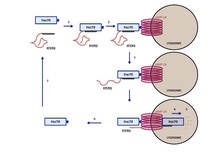
En la autofagia mediada por chaperonas se degradan determinadas proteínas citoplásmáticas que son introducidas a los lisosomas molécula por molécula. Las proteínas se identifican por contener la secuencia KFERQ, que es reconocida por un complejo citoplásmático formado por la chaperona hsc70. En la membrana del lisosoma encontramos un receptor llamado LAMP-2A que interactúa con este complejo. Este receptor está compuesto por varias subunidades que forman un poro, a través del cual pasa la proteína para su degradación. En el lumen de los lisosomas se encuentra una variante de la hcp70 (ly-hsc70) que facilita la entrada de la proteína a la matriz del lisosoma, donde es finalmente degradada por enzimas hidrolíticas. El complejo de las chaperonas hsp70 se desprende de la membrana del lisosoma para unirse a otra proteína citoplásmatica que contenga la secuencia KFERQ.
Estudios Relacionados
[editar]Proteínas con KFERQ selectivamente agotadas
[editar]La secuencia KFERQ es necesaria para el proceso de la autofagia mediada por chaperonas. El CMA puede ser activado por factores de estrés fisiológico como la privación prolongada de alimentos en varios tejidos como el hígado, riñón o corazón.[7] Se ha demostrado que las proteínas que contienen secuencias proteicas relacionadas con el KFERQ son selectívamente agotadas en el hígado y corazón, pero no en el músculo esquelético de ratas en ayunas. Esto demuestra que la falta de proteínas en diferentes tejidos resulta de una activación selectiva de rutas proteolíticas diferentes. El incremento de proteólisis en el corazón e hígado de animales en ayunas incluye la activación de la vía selectiva del lisosoma del KFERQ, mientras que la proteólisis del músculo esquelético no.
Degradación de anexinas por CMA
[editar]La secuencia KFERQ es de vital importancia en sustratos de la autofagia mediada por chaperonas como se evidencia en los resultados de un estudio de anexinas.[8] Los análisis de las secuencias de diferentes miembros de la familia de anexinas han revelado la presencia de KFERQ en algunas anexinas pero no en otras. Utilizando lisosomas aislados, solo las anexinas que contienen KFERQ fueron degradados por la CMA. La asociación de anexinas sin la secuencia KFERQ a la membrana del lisosoma está regulada por mecanismos diferentes que para la KFERQ y por tanto esta asociación no resultó en el transporte de la proteína a la matriz del lisosoma.
Patologías Relacionadas
[editar]En la degradación selectiva de los lisosomas, estos orgánulos captan y degradan las proteínas que contienen el pentapéptido Lys-Phe-Glu-Arg-Gln (KFERQ). Esta secuencia de aminoácidos es reconocida por chaperonas citosólicas (hsc70) y posteriormente el complejo KFERQ y hsc70 son identificados en la membrana lisosomal debido a la proteína de reconocimiento (LAMP-2A). Procesos patológicos como la diabetes mellitus o la enfermedad de Huntington resultan estar relacionados frecuentemente con un funcionamiento anómalo de los mecanismos de degradación de la célula (actividad lisosomal).
El KFERQ ha sido identificado como un agente involucrado en esta clase de patologías. Con motivo ha sido detectado en concentraciones anómalas en células de enfermos por diabetes u otras patologías como la enfermedad del Huntington.
Enfermedad de Huntington
[editar]La enfermedad del Huntington es una patología genética neurodegenerativa causada por una mutación en el gen que codifica la proteína huntingtina. Esta proteína estructural se encuentra en significativas cantidades en terminaciones nerviosas (como las neuronas), y los últimos estudios demuestran que el extremo aminoterminal de la proteína, con poliglutamina de la proteína mutada, se reconoce como proteína anormal y el extremo es suprimido por caspasas (enzimas que median la ruptura de proteínas) específicas.[9]
Por tanto, el KFERQ queda separado de las proteínas y estas no podrán degradarse en los lisosomas (debido a la ausencia de la señal identificador), y en consecuencia se inicia un proceso de acumulación progresiva de residuos proteicos. Este fenómeno resultaría tóxico para la célula, alterando la regulación de degradación de estas proteínas y causando una degeneración progresiva de las células que llevaría a largo plazo a la apoptosis celular del tejido neuronal (causando la enfermedad).
Diabetes Mellitus
[editar]La diabetes Mellitus es una patología definida por un conjunto de trastornos metabólicos caracterizados por la hiperglicemia, es decir la presencia (crónica o constante) de concentraciones elevadas de glucosa en la sangre.
En relación con el KFERQ, según estudios recientes, la disminución de la proteolisi celular (degradación de proteínas) característica de la diabetes mellitus, está asociada a una reducción significativa en el número de proteínas que regulan la degradación lisosomal (a través del mecanismo de autofagia mediada por chaperonas). Además, la diabetes está relacionada al mismo tiempo con un aumento en el número de proteínas destinadas a esta degradación mediada por chaperonas, proteínas las cuales contienen la señal KFERQ.
Consecuentemente se establece un vínculo entre estos factores como contribución a la acumulación de proteínas en hipertrofias renales en diabéticos.[10]
Atrofia muscular
[editar]En caso de ayuno prolongado, las células no poseen los nutrientes necesarios, y en consecuencia se activa una vía, la autofagia mediada por chaperonas, CMA, que importa y degrada las proteínas citosólicas que contienen el pentapéptido KFERQ. Estas se pierden de manera selectiva en los tejidos, los cuales se atrofian, como sucede en el tejido muscular.
Referencias
[editar]- ↑ Majeski, A. E. & Dice, J. F. (2004). Mechanisms of chaperone-mediated autophagy. The International Journal of Biochemistry & Cell Biology, 36, 2435–44.
- ↑ "Microinjected Ribonuclease A as a Probe for Lysosomal Pathway of Intro Celullar Protein degradation" Journal of Protein Chemistry, Vol 7, Nº2, 1998, J.Fred Dice
- ↑ Majeski, A. E. & Dice, J. F. (2004). Mechanisms of chaperone-mediated autophagy. The International Journal of Biochemistry & Cell Biology, 36, 2435–44.
- ↑ Bioquimica, 3 edición, editorial medica panamerica, Voet
- ↑ Dice, J., Terlecky, S., (1993) Polypeptide Import and Degradation by Isolated Lysosomes. The Journal of Biological Chemistry Vol. 268 No. 31 pp. 23490-23495
- ↑ Salvador, N., Aguado, C., Horst, M., Knecht, E., (2000) Import of a Cytosolic Protein into Lysosomes by Chaperone-mediated Autophagy Depends on its Folding State. The Journal of Biological Chemistry 275:27447-27456
- ↑ Wing, S., Chiang, H.-L-, Goldberg, A., & Dice, J. (1991). Proteins containing peptide sequences related to KFERQ are selectively depleted in liver and heart, but not skeletal muscle, of fasted rets. Biochemical Journal, 275, 165-169.
- ↑ Cuervo, A., Gomes, A., Barnes, J., & Dice, J. (2000). Selective degradation of annexins by chaperone-mediated autophagy. Journal of Biological Chemistry, 275, 33329-33335.
- ↑ Lin Qi y Xing-Ding Zhang (2013). Role of chaperone-mediated autophagy in degrading Huntington's disease-associated huntingtin protein. Oxford Journal (Acta Biochim Biophys Sin), 46 (2): 83-91.
- ↑ Sooparb, S., Russ Price, S., Shaoguang, J., Franch, A., (2004) Kidney International, Vol. 65 pp. 2135–2144
Bibliografía
[editar]- «Fundamentos De Bioquímica/ Fundamental of Biochemistry». Consultado el 20 de octubre de 2014.
- Rodríguez López, José Neptuno. «Proteólisis Intracelular Intracelular: Recambio Recambio Proteico». Archivado desde el original el 12 de marzo de 2016. Consultado el 20 de octubre de 2014.
- Cuervo, Ana Maria; Aldrin V. Gomes, Junor A. Barnes, Fred Dice (27 de junio). «Selective Degradation of Annexines by Chaperone-mediated Autophagy» (en inglés). Archivado desde el original el 25 de octubre de 2014. Consultado el 22 de octubre de 2014.
- «Autophagy genes and ageing» (en inglés). 12 de septiembre de 2008. Consultado el 23 de octubre de 2014.
- Badadani, Mallikarjun (30 de julio de 2012). «Autophagy Mechanism, Regulation, Functions, and Disorders». International Scholarly Research Notice (en inglés). Consultado el 23 de octubre de 2014.
