Operón de triptófano
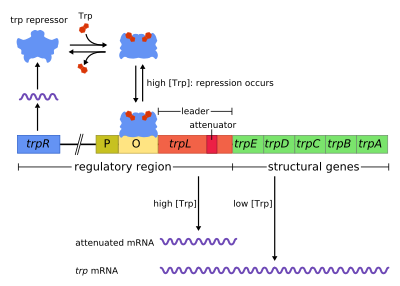
El trp operon es un operón — un grupo de genes que es empleado o transcrito en conjunto — que codifica para los componentes necesarios para producción de triptófano. El operón trp se encuentra en muchas bacterias, pero fue caracterizado primero en Escherichia coli. El operón se encuentra regulado de modo que cuando el triptófano está presente en el medio, los genes para la síntesis del triptófano no son expresados. El operón fue un importante sistema experimental para aprender acerca de la regulación de los genes, y es generalmente empleado para enseñar regulación génica.
Descubierto en 1953 por Jacques Monod y colegas, el operón de trp en E. coli era el primer operón inhibible en ser descubierto. Mientras el operón lac puede ser activado por un disacárido (alolactosa) el operón trp es inhibido por el mismo aminoácido triptófano. Este operón contiene cinco genes estructurales: trp E, trp D, trp C, trp B, y trp A, los cuales codifican para la proteína triptófano sintetasa. El operón también contiene un gen regulador represivo llamado trp R. Trp R Tiene un promotor donde la ARN polimerasa se une y sintetiza RNAm para la síntesis de una proteína reguladora. La proteína que es sintetizada por trp R se une a la región operadora y en presencia de Trp la bloquea, por lo que no hay transcripción de los genes codificantes para la Trp sintetasa. En el operón de lactosa, la alolactosa ata a la proteína represora, permitiendo la transcripción de gen, mientras en el operón de trp, el triptófano ata a la proteína represora a la zona operadora bloqueando la transcripción de los genes. En ambas situaciones, la represión impide la transcripción de los genes del operón por parte de la ARN polimerasa. Además, a diferencia del operón lac, el operón trp contiene una secuencia péptido dirigente y una secuencia atenuadora que permite una regulación graduada.[1]
Es un ejemplo de regulación negativa por represión de expresión de un gen. Dentro de la secuencia reguladora del operón, el operador es bloqueado por la proteína represora en presencia de triptófano (impidiendo la transcripción) y es liberado en ausencia de triptófano (permitiendo la transcripción). El proceso de inhibición (explicado abajo) complementa esta acción reguladora.
Represión[editar]
El operón opera por un mecanismo inhibidor de retroalimentación negativo. El represor para el operón trp es codificado por gen trpR en la región previa a la zona operadora del operón, el cual es constitutivamente expresado en un nivel bajo. Los monómeros de TrpR sintetizados se asocian formando tetrámeros. Estos tetrámeros, disueltos en el citoplasma, se encuentran inactivos. Cuándo triptófano está presente, estos tetrámeros de represión de triptófano se unen al aminoácido causando un cambio en la conformación del represor, permitiendo que el represor se una a la región operadora. Esto impide que ARN polimerasa se una a la región operadora y transcriba los genes del operón , entonces el triptófano no es producido de su precursor. Cuando el triptófano no está presente, el represor se encuentra en su conformación inactiva y no puede unirse a la región operadora, así que la transcripción no es inhibida por el represor.
Atenuación[editar]

La atenuación es un segundo mecanismo de retroalimentación negativa en el operón trp . El sistema de represión actúa bajo el efecto de la concentración intracelular del trp mientras que la atenuación responde a la concentración de tRNAtrp cargado.[2] Así, el represor TrpR disminuye la expresión genética por alteración de la transcripción en su inicio, mientras que la atenuación altera el proceso de transcripción una vez que se ha iniciado este proceso.[2] Mientras el represor TrpR disminuye la transcripción en un factor de 70, la atenuación puede disminuirla más en un factor de 10, permitiendo una represión acumulada de 700 veces.[3] La atenuación es posible por el hecho de que en procariotas (los cuales carecen núcleo), los ribosomas empiezan traducir el mRNA mientras la polimerasa de ARN todavía está transcribiendo la secuencia de ADN. Esto permite que el proceso de traducción afecte directamente la transcripción del operón.
Véase también[editar]
Enlaces externos[editar]
Referencias[editar]
- ↑ William Klug, Cummings, and Spencer.
- ↑ a b Yanofsky, C. (1981) Attenuation in the control of expression of bacterial operons.
- ↑ Lehninger, Albert L.; Nelson, David L.; Cox, Michael M. (2008). Principles of Biochemistry (5th edición). New York, NY: W.H. Freeman and Company. p. 1128. ISBN 978-0-7167-7108-1.
- Morse, DE; Mosteller RD; Yanofsky C (1969). «Dynamics of synthesis, translation, and degradation of trp operon messenger RNA in E. coli.». Cold Spring Harb Symp Quant Biol. 34: 725-40. PMID 4909527. doi:10.1101/sqb.1969.034.01.082. Morse, DE; Mosteller RD; Yanofsky C (1969). «Dynamics of synthesis, translation, and degradation of trp operon messenger RNA in E. coli.». Cold Spring Harb Symp Quant Biol. 34: 725-40. PMID 4909527. doi:10.1101/sqb.1969.034.01.082.
- Yanofsky, Charles (1981). «Attenuation in the control of expression of bacterial operons». Nature 289 (5800): 751-58. PMID 7007895. doi:10.1038/289751a0. Yanofsky, Charles (1981). «Attenuation in the control of expression of bacterial operons». Nature 289 (5800): 751-58. PMID 7007895. doi:10.1038/289751a0.
