Ku (proteína)
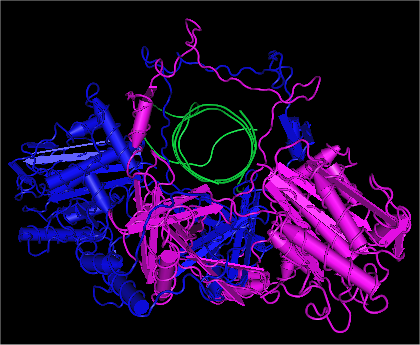
Ku es una proteína que se une a terminaciones quebradas de ADN de doble hebra y es necesaria para la ruta de reparación del ADN por recombinación no homóloga (NHEJ). Ku se encuentra evolutivamente conservada desde las bacterias hasta los humanos. La proteína Ku ancestral conservada en bacterias es una homodímero (dos copias de la misma proteína unidas entre sí).[1] Las Ku eucarióticas son un heterodímero de dos polipéptidos, Ku70 y Ku80, llamada así porque el peso molecular de las proteínas Ku humanas es de alrededor de 70 kDa y 80 kDa. Las dos subunidades Ku forman una estructura similar a una canasta que se une al extremo del ADN.[2] Una vez unidos, Ku pueden deslizarse hacia abajo en la hebra de ADN, lo que permite a otras moléculas de Ku unirse al extremo libre. En eucariotas superiores, Ku forma un complejo con la subunidad catalítica de las proteínas quinasa dependiente de ADN (ADN-PKcs) para formar la quinasa dependiente de ADN (ADN-PK).[3] Se cree que Ku funciona como un andamio molecular al cual otras proteínas que participan en NHEJ pueden unirse. Ambas subunidades del Ku han sido experimentadas en ratones knock out. Estos ratones muestran inestabilidad cromosómica, lo que indica que NHEJ es importante para el mantenimiento del genoma.[4][5] En muchos organismos, Ku tiene funciones adicionales en los telómeros, además de su papel en la reparación del ADN.[6]
Referencias[editar]
- ↑ Doherty AJ, Jackson SP, Weller GR. Identification of bacterial homologues of the Ku DNA repair proteins. FEBS Lett. 2001 Jul 6;500(3):186-8. PMID 11445083.
- ↑ Walker JR, Corpina RA, Goldberg J. Structure of the Ku heterodimer bound to DNA and its implications for double-strand break repair. Nature. 2001 Aug 9;412(6847):607-14. PMID 11493912
- ↑ Carter T, Vancurova I, Sun I, Lou W, DeLeon S. A DNA-activated protein kinase from HeLa cell nuclei. Mol Cell Biol. 1990 Dec;10(12):6460-71. PMID 2247066
- ↑ Difilippantonio MJ, Zhu J, Chen HT, Meffre E, Nussenzweig MC, Max EE, Ried T, Nussenzweig A. DNA repair protein Ku80 suppresses chromosomal aberrations and malignant transformation. Nature. 2000 Mar 30;404(6777):510-4. PMID 1076192
- ↑ Ferguson DO, Sekiguchi JM, Chang S, Frank KM, Gao Y, DePinho RA, Alt FW. The nonhomologous end-joining pathway of DNA repair is required for genomic stability and the suppression of translocations. Proc Natl Acad Sci U S A. 2000 Jun 6;97(12):6630-3. PMID 10823907
- ↑ Boulton SJ, Jackson SP. 1998. Components of the Ku-dependent non-homologous endjoining pathway are involved in telomeric length maintenance and telomeric silencing. EMBO J. 17:1819–28 PMID 9501103
