ADN polimerasa
| ADN polimerasa ADN-dirigida | ||||
|---|---|---|---|---|
 | ||||
| Estructuras disponibles | ||||
| PDB | ||||
| Identificadores | ||||
| Identificadores externos |
Bases de datos de enzimas
| |||
| Número EC | 2.7.7.7 | |||
| Número CAS | 9012-90-2 | |||
| Ortólogos | ||||
| Especies |
| |||
| PubMed (Búsqueda) |
| |||
| PMC (Búsqueda) |
| |||
Las ADN polimerasas son enzimas (celulares o virales) que intervienen en el proceso de replicación del ADN. Llevan a cabo la síntesis de la nueva cadena de ADN emparejando los desoxirribonucleótidos trifosfato (dNTP) con los desoxirribonucleótidos complementarios correspondientes del ADN molde. Los dNTP que se usan en la replicación del ADN contienen tres fosfatos unidos al grupo hidroxilo 5' de la desoxirribosa y dependiendo de la base nitrogenada serán dATP, dTTP, dCTP o dGTP. La reacción fundamental es una transferencia de un grupo fosfato en la que el grupo 3'-OH actúa como nucleófilo en el extremo 3' de la cadena que está en crecimiento. El ataque nucleofílico se produce sobre el fosfato α (el más próximo a la desoxirribosa) del desoxirribonucleósido 5' trifosfato que entra, liberándose pirofosfato inorgánico y alargándose el ADN (al formarse un nuevo enlace fosfodiéster). A diferencia de la mayoría de procesos biológicos que ocurren en la célula en los que solo se separa un grupo fosfato (Pi), durante la replicación se separan los dos últimos grupos fosfato, en forma de grupo pirofosfato (PPi)
Este proceso se puede resumir en una ecuación química:
(DNA)n + dNTP ↔ (DNA)n+1 + PPi
A pesar de que la ADN polimerasa solo tiene un sitio activo para emparejar los cuatro dNTPs diferentes, la unión correcta de los pares de bases A:T, C:G es posible basándose en la geometría de estos: si la unión es incorrecta se produce un desplazamiento del fosfato α haciendo más difícil su unión al extremo 3'-OH y ralentizando así el ritmo de catalísis, lo que da lugar a que la ADN polimerasa añada preferentemente las bases correctas.
Las ADN polimerasas pueden añadir hasta 1000 nucleótidos por segundo. Esto es debido a su naturaleza, es decir, el número de nucleótidos que son capaces de añadir cada vez que se asocian al molde de ADN que van a copiar. Dado que la adición de los nucleótidos es un proceso que dura unos milisegundos, la velocidad de catálisis va a depender del tiempo que la ADN polimerasa permanece unida al ADN, esto es, de su procesividad.

El crecimiento de la cadena se produce en dirección 5' → 3', ya que se requiere de un grupo 3'-OH libre para el inicio de la síntesis puesto que este es el que realiza el ataque nucleofílico sobre el fosfato α del dNTP, de forma que las ADN polimerasas requieren de un iniciador 3'-OH (que puede ser de ADN o ARN) llamado cebador que es sintetizado por la ARN primasa. El extremo 3' del cebador se denomina extremo cebador.
Las ADN polimerasas también realizan otras funciones durante el proceso de replicación. Además de participar en la elongación, desempeñan una función correctora y reparadora gracias a su actividad exonucleasa 3', que les confiere la capacidad de degradar el ADN partiendo de un extremo de este. Es importante que existan estos mecanismos de corrección ya que de lo contrario los errores producidos durante la copia del ADN darían lugar a mutaciones.
Funciones
Las ADN polimerasas pueden agregar nucleótidos libres solo al extremo 3 ' de la cadena de ADN en formación. Esto provoca que el alargamiento de la cadena en formación se realice en la dirección 5'-3 '. Ninguna ADN polimerasa conocida puede iniciar una hebra de ADN nuevamente, es decir, no puede colocar el primer nucleótido en la hebra, luego de unir el segundo, luego el tercero, y así sucesivamente. Todo lo que pueden hacer es alargar una cadena existente por su extremo 3'-OH, por lo que siempre debe haber un fragmento inicial ya formado. Esta es la razón por la que la ADN polimerasa necesita un cebador pre-formado al que agregar nucleótidos. Los cebadores pueden estar formados por ARN o ADN. En la replicación del ADN, las dos primeras bases son siempre ARN y son sintetizadas por otra enzima llamada primasa. También se requiere una enzima llamada helicasa para desenredar el ADN y deshacer en esa región su estructura de doble hebra y formar la estructura de dos hebras simples bifurcadas (esta región es la horquilla de replicación o horquilla de replicación), lo que facilita la replicación de cada una de las hebras siguiendo el modelo semiconservador de replicación del ADN.
Otra propiedad que tienen algunas ADN polimerasas, pero no todas, es la corrección de errores. Este proceso corrige los errores producidos durante la formación del ADN neosintetizado. Cuando se reconoce un par de bases incorrectamente colocado, la ADN polimerasa invierte la dirección de su movimiento al retrasar un par de bases. Esta actividad exonucleasa 3'-5' de la enzima permite eliminar el par de bases incorrecto para luego ser reemplazado por el correcto, la propia polimerasa que continuará la replicación. Esta actividad se llama corrección de pruebas. Las ADN polimerasas se utilizan ampliamente en experimentos de biología molecular.
Las ADN polimerasas tienen una estructura muy conservada, lo que significa que sus subunidades catalíticas varían muy poco de una especie a otra. Las estructuras conservadas generalmente realizan funciones importantes e insustituibles en la célula, cuyo mantenimiento produce una serie de ventajas.
Familias de ADN polimerasas
Según la homología de su estructura y la secuencia de aminoácidos las ADN polimerasas se pueden clasificar en 6 familias. Las ADN polimerasas de los virus de ADN son difíciles de clasificar en los siguientes grupos debido a la alta divergencia de las secuencias virales.
- Familia A: Incluye las ADN polimerasas eucariotas γ, θ, ν, la procariota I y la viral T7.
- Familia B: Incluye las ADN polimerasas eucariotas ζ, α, δ, ε y las procariotas II y B. También se ha propuesto la inclusión de algunas ADN polimerasas virales.
- Familia D: Incluye la ADN polimerasa procariota D y también se ha propuesto la inclusión de algunas ADN polimerasas virales.
- Familia X: Incluye las ADN polimerasas eucariotas β, σ, λ, μ, TDT y la procariota III.
- Familia Y: Incluye las ADN polimerasas eucariotas ι, κ, η y las procariotas IV y V.
- Familia RT: Incluye la telomerasa exclusiva de los eucariotas y las transcriptasas inversas virales, eucariotas y procariotas.
ADN polimerasas procariotas

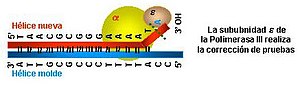
Las ADN polimerasas de los procariotas (arqueas y bacterias) son: las Pol I, II, III, IV, V, B, D y la RT cada una de ellas está especializada en una o más de éstas funciones según cuál sea su papel en la replicación. Por ejemplo en Escherichia coli la procesividad se relaciona con cuánto tiempo permanece unida la ADNpol al ADN cuando está agregando nucleótidos. Así, la ADN Pol I que es poco procesiva (añade entre 20 y 100 nucleótidos por acontecimiento de unión), es la encargada de la eliminación de los cebadores y el "relleno" del espacio que dejan con ADN (actividad exonucleasa 5' → 3' y actividad polimerasa 5' → 3'). Este proceso se conoce como traslación de la mella. La ADN Pol II está encargada de la reparación de los quiebres producidos en el ADN (actividades polimerasa 5' → 3' y exonucleasa 3' → 5'), la ADN Pol III que es muy procesiva es la principal encargada de la elongación del ADN (actividad polimerasa 5' → 3') durante la cual también realiza tareas de corrección (actividad exonucleasa 3' → 5'). Es preciso notar que actividad correctora y de reparación no son lo mismo. Solo la ADN pol I y beta tienen actividad de reparación, en cambio todas las polimerasas tienen actividad correctora (poseen 2 sitios activos: un sitio de polimerización y otro de corrección). ¿Por qué ocurre? ¿Cómo se da cuenta la ADNpol de que hubo un error en la replicación del ADN? Ocurre por el fenómeno de tautomerización. Los nucleótidos cambian a su forma química alternativa, la ADNpol las confunde con otro nucleótido y lo elonga. Cuando este vuelve a su conformación más estable provoca un cambio en el ancho de la hebra (20A) y termina abriéndose. Esto es detectado por la polimerasa, quien cambia conformacionalmente y deja la hebra posicionada en el sitio de corrección. Las ADN Pol IV y V, identificadas en 1999, están involucradas en una forma poco común de reparación del ADN.
A diferencia de lo que ocurre con la ADN Pol I, que solo debe añadir unos 5-10 nucleótidos una vez eliminado el cebador, es importante que la ADN Pol III sí sea muy procesiva, y por esa razón suele formar parte de un complejo denominado holoenzima ADN Pol III que le da una mayor procesividad. Este complejo consta de diversas subunidades polipeptídicas encargadas cada una de una función, y que constituyen en su conjunto un dímero asimétrico: una mitad se encarga de la síntesis de la hebra adelantada y la otra mitad de la hebra rezagada. El corazón catalítico lo componen las subunidades α que corresponden a dos copias de la ADN Pol III, la subunidad ε con actividad correctora exonucleasa 3' → 5' y la subunidad θ cuya función podría ser la de ensamblar las otras dos; las subunidades τ mantienen la estructura dimérica; el complejo γ lo conforman las subunidades γ y δ cuya su función es la de aumentar la procesividad de la ADN Pol III; la subunidad β sujeta la ADN Pol III al ADN.
| ADN Pol I | ADN Pol II | ADN Pol III | ||
|---|---|---|---|---|
| Estructura | Masa molecular (dalton) | 109.000 | 90.000 | 900.000 |
| Constitución | Monómero | Monómero | Dímero asimétrico | |
| Número de polimerasas / célula | 400 | 100 | 10 - 20 | |
| Actividad / Función | Polimerasa 5' → 3' / Elongación | SI | SI | SI |
| Exonucleasa 3' → 5' / Corrección | SI | SI | SI | |
| Exonucleasa 5' → 3' / Reparación | SI | NO | NO | |
ADN polimerasas eucariotas
Hay varios tipos de ADN polimerasa en los eucariotas: son las Pol ζ, α, δ, γ, θ, ν, ε, β, σ, λ, μ, ι, κ, η, TDT y la RT, aunque las más importantes son cuatro.
- ADN polimerasa α (alfa): se encuentra en el núcleo celular, se inhibe por afidicolina y en humanos presenta 4 subunidades, aunque las más importantes son dos:
- Subunidad mayor (130 kDa): tiene actividad polimerasa.
- Subunidad menor (48 kDa): tiene un centro activo de primasa para sintetizar el cebador necesario para la replicación. Como todas las polimerasas poseen un dominio común, representado como una mano, en la cual se incluye un dominio exonucleasa. Por lo tanto sí posee una facultad correctora.
- ADN polimerasa σ (sigma): se localiza en el núcleo celular y se inhibe mediante alidilcolina. Tiene una o dos subunidades dependiendo del organismo. Tiene una alta capacidad de polimerización y actividad correctora de errores (exo 3'→5'). Carece de actividad primasa.
La primasa (que es parte de la molécula de ADN polimerasa α) sintetiza cebadores de ARN y además comienza la elongación con ADN de las dos cadenas. Después se produce un cambio de polimerasa y entra la ADN polimerasa σ, que continúa la síntesis.
ADN polimerasas virales
Los virus de ADN sintetizan una gran variedad de ADN polimerasas, que en su mayoría no están emparentadas con las ADN polimerasas celulares o con las de otros virus de ADN. Los virus retrotranscritos como el VIH se caracterizan por sintetizar transcriptasas inversas, las cuales si están emparentadas entre sí y también con las transcriptasas inversas celulares en especial con las eucariotas.
El fago Φ29 sintetiza una ADN polimerasa propia. Esta enzima es muy utilizada en biología molecular para múltiples procedimientos de desplazamiento de amplificación de ADN, y tiene una serie de características que la hacen particularmente adecuada para esta aplicación.
Reacción en cadena de la polimerasa
La propiedad de las ADN polimerasas para replicar hebras de ADN se utiliza para la reacción en cadena de la polimerasa, conocida como PCR por sus siglas en inglés, para obtener un gran número de copias de un fragmento de ADN particular, amplificándolo para propósitos de investigación. En los procesos de PCR se requiere la utilización de polimerasas termoestables, como la polimerasa Taq, debido a que es necesario aplicar altas temperaturas para desnaturalizar la molécula de ADN.
