Diferencia entre revisiones de «GENCODE»
Creado al traducir la página «GENCODE» |
(Sin diferencias)
|
Revisión del 12:33 20 dic 2020
GENCODE es un proyecto científico de investigación de genomas que forma parte de la plataforma ENCODE (del inglés ENCyclopedia Of DNA Elements).
El consorcio GENCODE se creó inicialmente como una parte de la fase piloto del proyecto ENCODE, para identificar y mapear todos los genes codificantes de proteínas de las regiones ENCODE (aproximadamente un 1% del genoma humano). [1] Tras el éxito obtenido, GENCODE ahora pretende crear una “Enciclopedia de genes y variantes genéticas” identificando todas los las características genéticas del genoma humano y de ratón, mediante una combinación de análisis computacional, anotación manual y validación experimental. También tiene como objetivo anotar todas las características de genes basadas en la evidencia en el genoma humano completo con la máxima precisión posible. [2]
El resultado será una recopilación de anotaciones, que incluirá todos los loci que codifican para proteínas con variantes transcritas alternativamente [2] , loci no codificantes [3] con tránscritos encontrados y pseudogenes. [4]
En la actualidad
Actualmente GENCODE sigue avanzando hacia sus objetivos en la fase 2 del proyecto, que son:
- Continuar aumentando el coverage y la precisión con la que se presentan los genes humanos en GENCODE, mediante la mejora y la extensión de la anotación de todas las características de genes basadas en la evidencia. Esto incluye loci que codifican para proteínas con variantes con splicing alternativo, loci no codificantes y pseudogenes. [5]
- Crear un set de genes de ratón en GENCODE que incluya regiones que codifican para proteína asociados a variantes con splicing alternativo, loci no codificantes con evidencia de tránscritos y pseudogenes.
La última versión de anotaciones de genes humanos es la Gencode 36, a diciembre de 2020. Este último lanzamiento utiliza el genoma de referencia humano GRCh38 y las anotaciones genéticas de ratón de Gencode M25, también a diciembre de 2020.
Desde septiembre de 2009, el proyecto Ensembl utiliza la base de datos GENCODE, y por tanto cada nuevo lanzamiento de GENCODE se corresponde con una actualización de Ensembl.
Historia

Septiembre 2003El NHGRI (del inglés National Human Genome Research Institute) inició un consorcio de investigación pública llamado ENCODE, la “Enciclopedia de los elementos de DNA”, con el objetivo de identificar todos los elementos funcionales en la secuencia del genoma humano. Este proyecto se diseñó con 3 fases: piloto, desarrollo tecnológico y fase de producción.[6] La fase piloto se centró en investigar en profundidad, computacional y experimentalmente, 44 regiones de un total de 30Mb de secuencia (aproximadamente un 1% del genoma humano). Además, es en este momento cuando se crea el consorcio GENCODE para identificar y mapear todos los genes que codifican para proteínas presentes en las regiones ENCODE.[1] Los resultados de las primeras dos fases fueron pensados para poder determinar el mejor camino de cara al análisis del 99% restante del genoma humano de forma eficiente y comprensiva en la fase de producción.
Abril 2005La primera anotación de las 44 regiones ENCODE fue publicada el 29 de abril de 2005 y se usó en el primer taller del ENCODE Genome Annotation Assessment Project (E-GASP).[1] El primer lanzamiento de GENCODE contenía 416 loci conocidos, 26 nuevas secuencias codificantes de DNA (CDS, inglés coding DNA sequence), 82 nuevos loci de tránscritos, 78 loci putativos, 104 pseudogenes procesados y 66 pseudogenes no procesados.
Octubre 2005Esta anotación fue actualizada (lanzamiento 02) el 14 de octubre de 2005, y contenía nuevos descubrimientos de validaciones experimentales por medio de las técnicas RACE (del inglés Rapid amplification cDNA ends) y RT-PCR.[1] En total se añadieron 411 loci conocidos, 30 nuevos loci CDS, 81 nuevo loci con tráscrito, 83 loci putativos, 104 pseudogenes procesados y 66 pseudogenes no procesados.
Junio 2007Se publicaron las conclusiones del proyecto piloto.[7] Estos descubrimientos resaltaron el éxito de dicho proyecto en crear una plataforma y nuevas tecnologías para caracterizar elementos funcionales en el genoma humano, abriendo un camino para la investigación en estudios del genoma completo.
Octubre 2007El NHGRI concedió una subvención al Wellcome Trust Sanger Institute por sus buenos resultados en la fase piloto con el 1% del genoma. Esta subvención permitiría realizar una ampliación del proyecto GENCODE para integrar la anotación de las características de genes. [8]
Esta nueva inversión fue parte del esfuerzo del NHGRI por avanzar el Proyecto ENCODE a la fase de producción en el genoma entero.
Diciembre 2011Se lanzó la versión GENCODE 7, que combinaba la anotación genética manual del Análisis y Anotación de Humanos y Vertebrados (HAVANA, por sus siglas en inglés) y la última anotación automática de Ensembl (Ensembl 62). En el momento de su publicación, GENCODE 7 era la anotación más exhaustiva de loci de RNA largos no codificantes (lncRNA) disponible. El consorcio GENCODE publicó un artículo discutiendo los resultados de esta versión en septiembre de 2012. [2]
2013 - 2017Tras el éxito de la nueva anotación definitiva de elementos funcionales del genoma humano, el grupo de GENCODE recibió una segunda subvención en 2013 para continuar con su trabajo de anotación y amplió sus esfuerzos al genoma del ratón.[9] Se espera que los datos de anotación del genoma del ratón permitan estudios comparativos entre este y el humano, para mejorar la calidad de anotación de ambos.
2018
Una de las últimas adiciones es el seguimiento de CRISPR/Cas9 en construcciones genómicas de humanos y de organismos modelo. La técnica CRISPR de edición de genomas depende de la presencia de secuencias de RNA guía que se unen a la región a editar con elevada especificidad. La nueva herramienta de GENCODE está diseñada para ayudar a encontrar la secuencia de RNA guía apropiada, mediante una lista de potenciales sitios de unión para el complejo CRISPR/Cas9 que se encuentran cerca (entre 0 y 200 bp) de regiones codificantes. Para cada sitio, se dan las posibles secuencias guía junto con una predicción de su eficiencia y puntuaciones de especificidad. También se da información sobre potenciales sitios de unión inespecífica para cada secuencia guía, según el número de mismatches que haya entre el sitio inespecífico y la secuencia guía.
2020 - Actualidad
Se introdujeron mejoras en la infraestructura del sistema de anotaciones, en las herramientas bioinformáticas y en el análisis, tanto para genoma humano como de ratón. Algunas de estas fueron: la compleción del primer pase manual de anotación del genoma de referencia de ratón, el inicio de proyectos colaborativos para conseguir la convergencia entre diferentes bases de datos de anotación para genes codificantes de proteínas en humanos y ratones, y la compleción de la primera anotación de lncRNAs supervisada manualmente. Además, se ha trabajado en la mejora de la anotación de genes asociados con la infección por SARS-CoV-2, el virus causante de la pandemia de COVID-19.
Participantes principales
Los participantes principales de GENCODE se han mantenido relativamente consistentes a lo largo de sus fases, siendo el Wellcome Trust Sanger Institute el que ahora lidera los esfuerzos del proyecto.
A continuación se incluye un resumen de las principales instituciones que contribuyeron en cada fase:
| GENCODE Fase 2 (Actual)[10] | GENCODE Fase de ampliación[8] | GENCODE
Fase piloto[11] | |
|---|---|---|---|
| Wellcome Trust Sanger Institute, Cambridge, Reino Unido | Wellcome Trust Sanger Institute, Cambridge, Reino Unido | Wellcome Trust Sanger Institute, Cambridge, Reino Unido
| |
| Centre de Regulació Genòmica (CRG), Barcelona, Cataluña, España | Centre de Regulació Genòmica (CRG), Barcelona, Cataluña, España | Institut Municipal d'Investigació Mèdica (IMIM), Barcelona, Cataluña, España | |
| Universidad de Lausanne, Suiza | Universidad de Lausanne, Suiza | Universidad de Ginebra, Suiza | |
| Universidad de California, Santa Cruz (UCSC), California, EE.UU. | Universidad de California Santa Cruz(UCSC), EE.UU. | Universidad de Washington (WashU), St Louis, EE.UU. | |
| Instituto de Massachusetts de Tecnología (MIT), Boston, EE.UU. | Instituto de Massachusetts de Tecnología (MIT), Boston, EE.UU. | Universidad de California, Berkeley, EE.UU. | |
| Universidad de Yale(Yale), New Haven, EE.UU. | Universidad de Yale (Yale), New Haven EE.UU. | Instituto Europeo de Bioinformática, Hinxton, Reino Unido | |
| Centro Nacional de Investigaciones Oncológicas (CNIO), Madrid, España | Centro Nacional de Investigaciones Oncológicas (CNIO), Madrid, España | ||
| Universidad de Washington (WashU), St Louis, EE.UU. |
Estadísticas
Desde su creación, GENCODE ha publicado 20 versiones de anotaciones del genoma humano, con pequeñas actualizaciones adicionales.
Debajo se muestran las estadísticas principales de la anotación del genoma humano en GENCODE en la versión 20 (2014), que fue la primera en utilizar la última actualización del Human Genome Assembly (GRCh38):[12]
| Categorías | Total | Categorías | Total |
|---|---|---|---|
| Total No de Genes | 58,688 | Total No de Transcripts | 194,334 |
| Proteína-genes de codificación | 19,942 | Proteína-codificación transcripts | 79,460 |
| Mucho tiempo no-genes de ARN de la codificación | 14,470 | - Proteína de longitud llena-codificación: | 54,447 |
| Pequeño no-genes de ARN de la codificación | 9,519 | - Proteína de longitud parcial-codificación: | 25,013 |
| Pseudogenes | 14,363 | Barbaridad decadencia mediada transcripts | 13,229 |
| - Procesado pseudogenes: | 10,736 | Mucho tiempo no-ARN de codificación loci transcripts | 24,489 |
| - unprocessed pseudogenes: | 3,202 | ||
| - Unitario pseudogenes: | 171 | ||
| - Polimórfico pseudogenes: | 26 | ||
| - pseudogenes: | 2 | ||
| Inmunoglobulina/T-segmentos de gen de receptor de célula | 618 | Total No de traducciones distintas | 59,575 |
| - Segmentos de codificación de la proteína: | 392 | Genes que tiene más de uno traducciones distintas | 13,579 |
| - pseudogenes: | 226 |
Para más detalles en la clasificación del set de genes mostrado arriba, se puede consultar las páginas de GENCODE Statistics README y GENCODE biotypes.
Gracias a los avances en técnicas de secuenciación, como RT-PCR-seq, al aumento de la cobertura de las anotaciones manuales (HAVANA), y las mejoras en algoritmos de anotación automática con Ensembl, la precisión de las anotaciones en GENCODE se ha ido refinando continuamente a lo largo de los distintos lanzamientos.
Una comparación de estadística clave de 3 importante GENCODE las liberaciones está mostrada abajo.[12] Es evidente que a pesar de que la cobertura, en plazos del número total de genes descubrió, es firme
- Gracias a los avances en técnicas de secuenciación, como RT-PCR-seq, al aumento de la cobertura de las anotaciones manuales (HAVANA), y las mejoras en algoritmos de anotación automática con Ensembl, la precisión de las anotaciones en GENCODE se ha ido refinando continuamente a lo largo de los distintos lanzamientos.
-
Comparación de las distintas versiones de GENCODE de humanos (tránscritos)
-
Comparación de versiones de GENCODE humanas (genes)
-
Comparación de versiones de GENCODE humanas (traducciones)
Metodología
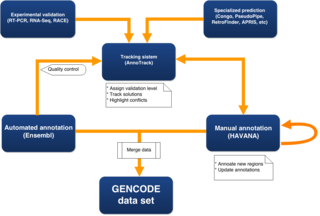
El proceso general para crear una anotación de GENCODE incluye trabajo manual, análisis computacionales y enfoques experimentales específicos. Los loci putativos se pueden verificar en un laboratorio experimental y las predicciones computacionales se analizan manualmente.[5]Actualmente, para asegurar que la anotación cubre el genoma completo y no solo regiones que han sido anotadas manualmente, se fusionan los datos de anotaciones manuales de HAVANA junto con las anotaciones automáticas obtenidas de Ensembl correspondientes. Este proceso también añade predicciones únicas de CDS de tamaño completo para proporcionar la anotación más precisa y actualizada posible del genoma.[13]
Anotación automática (Ensembl)
Los tránscritos Ensembl son producto de su sistema anotación automática. Todos los tránscritos Ensembl están basados en evidencia experimental, y por lo tanto la información automatizada se basa en los mRNAs y las secuencias de proteínas depositadas en bases de datos públicas.[14]
Además, se incluyen:
- Los niveles 1 y 2 de Proteínas de UniProt
- Las regiones no traducidas (UTR, del inglés Untranslated Regions)
- RNA intergenómico no codificante (lincRNA por sus siglas en inglés: long intergenomic non-coding RNA)
- Genes, que se anotan usando una combinación de secuencias de cDNA e información regulatoria del proyecto Ensembl
- RNAs cortos no codificantes, anotados usando los datos para RNA no codificante de Ensembl
[2]
Anotación manual (grupo HAVANA)
La principal aproximación a la anotación de genes manual es anotar los tránscritos alineados con el genoma y tomar las secuencias genómicas como referencia, en lugar de los cDNAs. La secuencia genómica final se analiza usando datos de Ensembl modificados. Los resultados de BLAST de los cDNAs/ESTs (del inglés Expressed Sequence Tags) y las proteínas, junto con varias predicciones ab initio, se pueden analizar manualmente en la herramienta de buscador de anotaciones Otterlace. Por lo tanto, se pueden predecir más variantes de splicing alternativo en comparación con la anotación cDNA. Además, la anotación genómica produce un análisis más exhaustivo de los pseudogenes. [2]
Hay diversos grupos de análisis en el consorcio GENCODE que trabajan en herramientas de mejora de la asistencia para anotaciones manuales en el momento de producir modelos en regiones sin anotar, así como para identificar potenciales regiones que no se hayan anotado o que se hayan anotado incorrectamente, incluyendo loci, isoformas, sitios de splicing y biotipos incorrectos. Esta información se devuelve a los anotadores manuales usando el sistema de seguimiento AnnoTrack.[15]
Algunas de estas herramientas usan información de otros subgrupos de ENCODE, incluyendo información de RNASeq y modificación de histonas, entre otros. Los datos de RNASeq son importantes como nueva fuente de evidencia, pero la generación de modelos genéticos completos a partir de ellos es complicado. Como parte de GENCODE, se realizó un concurso para determinar la calidad de las predicciones producidas por diversas herramientas de predicción que usaban RNAseq (ver RGASP más abajo). Para confirmar algunos modelos inciertos, GENCODE también tiene una herramienta de validación experimental usando secuenciación de RNA y RACE.[13]
Fusión Ensembl/HAVANA
Durante el proceso de fusión, todos los modelos de tránscritos de HAVANA y Ensembl se comparan, primero solapando los exones codificantes en una misma hebra, y después mediante comparaciones por pares de cada exón en un conjunto de tránscritos. El módulo usado para fusionar el conjunto de información genética es HavanaAdder. Si alguna anotación descrita en los sets de información externa no se encuentra en el manual, entonces se almacena en el sistema AnnoTrack para que sea revisada. [2]
Valoración de la calidad
Para GENCODE 7, a los modelos de tránscritos se les asigna un nivel elevado o bajo de soporte basado en un nuevo método desarrollado para puntuar la calidad de los tránscritos. Este método se basa en alineamientos de mRNA y de EST proporcionados por la UCSC y Ensembl. Los alineamientos de mRNA y EST se comparan con los tránscritos de GENCODE, y los tráscritos se puntúan de acuerdo con el alineamiento en su longitud completa. En la figura de la derecha se muestra un resumen de los niveles de soporte para cada cromosoma en GENCODE 7. Las anotaciones se dividen en las producidas por el proceso automatizado, por el método manual y las anotaciones combinadas, donde ambos procesos resultan en la misma anotación. [2]
Métodos generales utilizados en GENCODE 7
GENCODE 7 fue la versión lanzada en diciembre de 2010. Fue la primera en la que se hizo una reconstrucción completa desde su creación, usando Ensembl y fusionando los datos con los de HAVANA, tal como se hace en cada actualización. En el momento de su publicación, GENCODE 7 era la anotación más exhaustiva de loci de RNA largos no codificantes (lncRNA) disponible.
Amplificación, secuenciación, mapeo y validación de la unión exón–exón
Se generó cDNA de doble cadena de ocho tejidos humanos (cerebro, corazón, riñón, testículo, hígado, bazo, pulmón y músculo esquelético) con una amplificación de cDNA. El DNA purificado se utilizó directamente para generar una biblioteca de secuenciación con el '' Kit de preparación de muestras de DNA genómico '' (Illumina).
A continuación, esta biblioteca fue secuenciada en una plataforma Genome Analyzer 2 de Illumina. Se obtuvieron lecturas de 35 o 75 nucleótidos y entonces se mapearon sobre el genoma humano de referencia (hg19) y sobre los amplicones con splicing predichos usando el software Bowtie.
Para validar un sitio de splicing en un tránscrito, se consideraron sólo las lecturas mapeadas una sola vez y sin ningún mismatch. Para lecturas de 35 y 75 nt, las uniones de splicing se validaron cuando había un mínimo de 10 lecturas con al menos 4 y 8 nucleótidos, respectivamente, en cada lado del punto de rotura. [2]
Comparación entre RefSeq, UCSC, AceView y tránscritos GENCODE
Los tránscritos pertenecientes a los cuatro grupos (GENCODE, RefSeq, UCSC y AceView) fueron comparados para determinar hasta qué punto se superpone la información que contienen. Concretamente, las versiones que se compararon fueron: GENCODE 7, RefSeq y UCSC de julio de 2011, y la versión de AceView de 2010. Las superposiciones entre las diferentes combinaciones de bases de datos fueron representadas gráficamente como diagramas de Venn de tres conjuntos usando Vennerable R, y editadas manualmente. [2]
Análisis PhyloCSF
Se usó PhyloCSF para identificar potenciales nuevos genes codificantes en tránscritos modelo de RNA-seq, basándose en marcadores evolutivos. Para cada tránscrito modelo generado por la información obtenida de Illumina HBM usando Exonerate o Scripture, se generó un alineamiento de mamífero extrayendo el alineamiento de cada exón de los alineamientos de vertebrados de la UCSC, que incluye a 33 mamíferos placentarios. [2]
APPRIS (CNIO)
APPRIS es un sistema que presenta un rango de métodos computacionales para proporcionar valor a las anotaciones del genoma humano. También selecciona una de las CDS para cada gen como isoforma principal. Además, define la principal variante mediante la combinación de proteínas estructurales e información funcional con datos de la conservación de las especies relacionadas. El servidor APPRIS se usa en el contexto de la escalada del proyecto ENCODE para anotar el genoma humano, pero también se usa en otras especies como ratón, rata y pez cebra. [16] La herramienta se compone de módulos separados que combinan la estructura y la función de una proteína con evidencia evolutiva. Cada módulo está implementado como un servicio web separado.
Uso/Acceso
La versión actual del GENCODE humano (GENCODE versión 36) incluye archivos de anotación (en formato GTF y GFF3), archivos FASTA y archivos METADATA asociados con la anotación de GENCODE en todas las regiones genómicas (cromosomas de referencia/ patches/ scaffolds/ haplotipos). La información de la anotación se traslada a cromosomas de referencia y se almacena en archivos separados que incluyen: anotación genética, características PolyA anotadas por HAVANA, pseudogenes (retrotransposones o no) predichos por las herramientas de Yale y UCSC pero no por HAVANA, lncRNAs, y estructuras tRNA predichas por tRNA-Scan. Algunos ejemplos de líneas en formato GTF están a continuación:
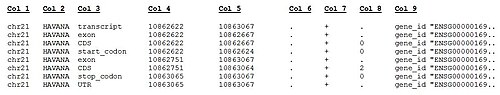
Las columnas en el formato GTF se describen debajo.
Descripción de formato de GENCODE GTF archivo. Columnas: TABULADOR-separado estándar GTF
| Número de columna | Contenido | Formato/de valores |
|---|---|---|
| 1 | Nombre de cromosoma | chr{1,2,3,4,5,6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,X,Y,M} |
| 2 | Fuente de anotación | {ENSEMBL,HAVANA} |
| 3 | Característica-tipo | {Gen,transcript,exon,CDS,UTR,inicio_codon,parón_codon,Selenocysteine} |
| 4 | Ubicación de inicio genómico | Entero-valor (1-basado) |
| 5 | Ubicación de fin genómico | Entero-valor |
| 6 | Puntuación (no utilizado) | . |
| 7 | Hebra genómica | {+,-} |
| 8 | Fase genómica (para CDS características) | {0,1,2,.} |
| 9 | Información adicional como pares de valor clave | Ve explicación en someter abajo. |
Descripción de pares de valor clave en 9.ª columna del GENCODE GTF archivo (formato: valor "clave")
| Nombre clave | Formato de valor |
|---|---|
| Gen_id | ENSGXXXXXXXXXXX |
| transcript_id | ENSTXXXXXXXXXXX |
| Tipo_de gen | Lista de biotipos |
| Estado_de gen | {SABIDO,NOVELA,PUTATIVE} |
| Nombre_de gen | Cuerda |
| transcript_Tipo | Lista de biotipos |
| transcript_Estado | {SABIDO,NOVELA,PUTATIVE} |
| transcript_Nombre | Cuerda |
| exon_Número | Indica la posición biológica del exon en el transcript |
| exon_id | ENSEXXXXXXXXXXX |
| Nivel |
|
Definición del nivel
Cada gen en la base de datos de GENCODE se clasifica en tres niveles según el tipo de anotación
- Nivel 1 (loci verificados): incluye tránscritos que han sido anotados manualmente y validados experimentalmente por RT-PCR-seq, y pseudogenes que han sido validados por distintas metodologías.[2]
- Nivel 2 (loci anotados manualmente): contiene tránscritos que han sido anotados manualmente sólo por HAVANA, y tránscritos que han sido fusionados con modelos producidos por la herramienta automática Ensembl.[2]
- Nivel 3 (loci anotados automáticamente): indica predicciones de tránscritos y pseudogenes generados por la herramienta automática de anotación de Ensembl.[2]
Definición de estados de genes y tránscritos
A los genes y tránscritos se les asigna el estatus “conocido”, “nuevo” o “putativo” dependiendo de su presencia en otras bases de datos principales y de la evidencia usada para construir sus componentes:
- Conocidos: representados en la base de datos del HUGO Gene Noenclature Committee (HGNC) y en RefSeq. [2]
- Nuevos: actualmente no representados en las bases de datos de HGNC ni RefSeq, pero bien respaldados por evidencia de especificidad de locus, o por evidencia de tránscrito más escasa.[2]
- Putativos: actualmente no representados en las bases de datos de HGNC ni RefSeq, pero apoyados por evidencia de tránscrito más escasa. [2]
La web de GENCODE también contiene un buscador de genomas para humano y ratón donde se puede llegar a cualquier región genómica sabiendo el número del cromosoma y la posición de inicio y final (ejemplo: 22:30,700,000..,30,900,000), así como con la identificación del tránscrito o del gen en ENS (con o sin versión), o con el nombre del gen. Este navegador lo proporciona Biodalliance.
Retos
Definición de un "gen"
La definición de “gen” nunca ha sido una cuestión trivial, pues ha contado con numerosas definiciones y nociones propuestas a lo largo de los años desde que se descubrió el genoma humano. Inicialmente, a principios del siglo XX, los genes se concebían como unidades discretas de herencia; más adelante se pensó en ellos como el “mapa” de la síntesis de proteínas, y recientemente, se definieron como el código genético que se transcribe a RNA. A pesar de que la definición de gen ha evolucionado mucho a lo largo del último siglo, ha permanecido como motivo de controversia entre muchos investigadores. Con la llegada del proyecto ENCODE/GENCODE, se han ido desvelando aspectos incluso más problemáticos, como el splicing alternativo y transcripciones intergénicas, patrones complejos de regulación dispersa, junto con la conservación no génica y la abundancia de RNAs no codificantes. GENCODE se dedica a construir una enciclopedia de genes y variantes genéticas, por lo que encontrar una noción actualizada del concepto de “gen” se presenta como un reto para el proyecto.[17]
Pseudogenes
Los pseudogenes tienen secuencias de DNA que son similares a las de genes codificantes funcionales, pero sus tránscritos normalmente se identifican con un cambio en la pauta de lectura o una deleción, y son generalmente anotados como producto de genes codificantes en la mayoría de bases de datos genéticos. De todos modos, análisis recientes de pseudogenes retrotranspuestos han encontrado que algunos de ellos se expresan y son funcionales, y de hecho tienen un impacto funcional o regulatorio en la biología humana. Para lidiar con las incógnitas y la complejidad que rodean a los pseudogenes, GENCODE ha creado una ontología de pseudogenes usando una combinación de métodos automáticos, manuales y experimentales para asociar una variedad de propiedades biológicas (secuencia, características, evolución y potencial función) a los pseudogenes. [1]
Proyectos Relacionados
ENCODE
La ENCyclopedia Of DNA Elements (ENCODE) es un consorcio de investigación pública lanzado por el National Human Genome Research Institute (NHGRI) en septiembre de 2003 (en fase piloto). La meta de ENCODE es construir una lista exhaustiva de elementos funcionales en el genoma humano, incluyendo elementos que actúan a nivel de las proteínas y el RNA, y de elementos regulatorios que controlan las células y las circunstancias en las que un gen determinado se encuentra activo. [18] El análisis de la información durante la fase piloto (2003-2007) fue coordinado por el grupo Ensembl, un proyecto conjunto entre el EBI y el Wellcome Trust Sanger Institute. Durante las fases piloto y de desarrollo inicial de tecnologías, 44 regiones (aproximadamente el 1% del genoma humano) fueron seleccionadas para ser analizadas usando una variedad de métodos experimentales y computacionales.[19]Toda la información producida por investigadores de ENCODE y los resultados del análisis del proyecto ENCODE entre 2003 y 2012 se encuentran en el navegador de genomas y la base de datos de la UCSC. Los resultados a partir de 2013 se encuentran libremente disponibles para ser descargados y analizados en el portal del proyecto ENCODE. (CITA ENCODE) Para anotar toda la evidencia basada en rasgos genéticos (genes, tránscritos, secuencias codificantes, etc.) en la totalidad del genoma humano con gran precisión, el consorcio ENCODE creó el subproyecto GENCODE.
Proyecto Genoma Humano
El Proyecto Genoma Humano fue un esfuerzo internacional de investigación para determinar la secuencia del genoma humano e identificar los genes que contiene. El proyecto fue coordinado por los National Institutes of Health y el departamento de energía de los Estados Unidos. También hubo contribuciones adicionales de varias universidades a lo largo de los Estados Unidos, y algunos participantes internacionales en Reino Unido, Francia, Alemania, Japón y China. El Proyecto Genoma Humano empezó formalmente en 1990 y fue completado en 2003, 2 años antes de lo previsto.[20]Tras la publicación de la secuencia completa del genoma humano en abril del 2003, la comunidad científica aumentó sus esfuerzos para explorar toda la información disponible, buscando pistas para entender cómo funciona el cuerpo humano en un estado fisiológico y patológico. Un requerimiento básico para la comprensión de la biología humana es la habilidad para identificar y caracterizar los elementos funcionales basados en la secuencia a través de la experimentación y el análisis computacional. En septiembre de 2003, el NHGRI introdujo el proyecto ENCODE para facilitar la identificación y el análisis del set completo de elementos funcionales en la secuencia del genoma humano.[19]
Sub Proyectos
Ensembl
Ensembl es parte del proyecto GENCODE, ya que ha jugado un papel esencial para la obtención de anotación automática en el constructo del genoma humano de referencia, y para unir su anotación con la anotación manual del equipo HAVANA. Para la anotación del genoma humano, Ensembl utiliza los datos de GENCODE.[21]
Diseño de microarrays para la expresión de lncRNA
Una investigación clave en el área del proyecto GENCODE fue la significancia biológica de los lncRNA. Para entender mejor su expresión en humanos, GENCODE creó un subproyecto para desarrollar plataformas de microarray capaces de cuantificar los tránscritos en la anotación de lncRNAs en GENCODE.[22] Algunos de los diseños se generaron mediante el sistema eArray de Agilent Technologies, y se encuentran disponibles en el formato estándar Agilent[23]
RGASP
RGASP es el RNA-seq Genome Annotation Assesment Project, un proyecto diseñado para demostrar la efectividad de varios métodos computacionales en obtener un RNA-seq de calidad. Los objetivos principales de RGASP son: la evaluación no sesgada del alineamiento de RNA-seq, la caracterización de software de tránscritos (descubrimiento, reconstrucción y cuantificación), y la determinación de la factibilidad de las anotaciones automatizadas de genoma basadas en la secuenciación del transcriptoma.[24]
RGASP se organiza en un marco de trabajo de consorcio modelado después del taller EGASP (ENCODE Genome Annotation Assessment Project) de predicción de genomas, y se han llevado a cabo dos rondas de talleres para abordar distintos aspectos del RNA-seq, así como cambiar tecnologías de secuenciación y formatos. Uno de los descubrimientos principales de estas dos rondas del proyecto fue la importancia del alineamiento de los fragmentos para la calidad de las predicciones producidas. Además, una tercera ronda de talleres RGASP se llevó a cabo en 2014 para centrarse en el mapeado del genoma.[25]
Ver también
- Anotación de genomas
- Anotación de Genomas y Vertebrados
Referencias
- ↑ a b c d «GENCODE: producing a reference annotation for ENCODE.». Genome Biol. 7 Suppl 1: S4.1-9. 2006. PMC 1810553. PMID 16925838. doi:10.1186/gb-2006-7-s1-s4.
- ↑ Frankish, A; Mudge, JM; Thomas, M; Harrow, J (2012). «The importance of identifying alternative splicing in vertebrate genome annotation.». Database 2012: bas014. PMC 3308168. PMID 22434846. doi:10.1093/database/bas014.
- ↑ Derrien, T; Johnson, R; Bussotti, G; Tanzer, A; Djebali, S; Tilgner, H; Guernec, G; Martin, D; Merkel, A; Knowles, DG; Lagarde, J; Veeravalli, L; Ruan, X; Ruan, Y; Lassmann, T; Carninci, P; Brown, JB; Lipovich, L; Gonzalez, JM; Thomas, M; Davis, CA; Shiekhattar, R; Gingeras, TR; Hubbard, TJ; Notredame, C; Harrow, J; Guigó, R (Sep 2012). «The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression.». Genome Research 22 (9): 1775-89. PMC 3431493. PMID 22955988. doi:10.1101/gr.132159.111.
- ↑ Pei, B; Sisu, C; Frankish, A; Howald, C; Habegger, L; Mu, XJ; Harte, R; Balasubramanian, S; Tanzer, A; Diekhans, M; Reymond, A; Hubbard, TJ; Harrow, J; Gerstein, MB (5 de septiembre de 2012). «The GENCODE pseudogene resource.». Genome Biology 13 (9): R51. PMC 3491395. PMID 22951037. doi:10.1186/gb-2012-13-9-r51.
- ↑ a b «GENCODE – Goals». GENCODE. Wellcome Trust Sanger Institute. c. 2013. Consultado el 5 September 2014.
- ↑ The ENCODE Project Consortium (22 October 2004). «The ENCODE (ENCyclopedia of DNA Elements) Project». Science 306 (5696): 636-640. Bibcode:2004Sci...306..636E. PMID 15499007. doi:10.1126/science.1105136.
- ↑ The ENCODE Project Consortium (14 June 2007). «Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project.». Nature 447 (7146): 799-816. Bibcode:2007Natur.447..799B. PMC 2212820. PMID 17571346. doi:10.1038/nature05874.
- ↑ a b «Researchers Expand Efforts to Explore Functional Landscape of the Human Genome». Wellcome Trust Sanger Institute. Wellcome Trust Sanger Institute. 9 October 2007. Consultado el 8 September 2014.
- ↑ «GENCODE – Home page». GENCODE. Wellcome Trust Sanger Institute. c. 2013. Consultado el 8 September 2014.
- ↑ «Participants, all funded personnel». GENCODE. Wellcome Trust Sanger Institute. c. 2014. Consultado el 8 September 2014.
- ↑ «GENCODE Project Participants». Genome BioInformatics Research Lab. Genome BioInformatics Research Lab. c. 2005. Consultado el 8 September 2014.
- ↑ a b «GENCODE – Statistics». GENCODE. Wellcome Trust Sanger Institute. c. 2014. Consultado el 8 September 2014.
- ↑ a b Searle, S; Frankish, A; Bignell, A; Aken, B; Derrien, T; Diekhans, M (2010). «The GENCODE human gene set.». Genome Biology 11 (Suppl 1): 36. PMC 3026266. doi:10.1186/gb-2010-11-S1-P36.
- ↑ «Ensembl Gene Set». Ensembl. August 2014. Consultado el 6 September 2014.
- ↑ Kokocinski, F; Harrow, J; Hubbard, T (2010). «AnnoTrack - a tracking system for genome annotation.». BMC Genomics 11: 538. PMC 3091687. PMID 20923551. doi:10.1186/1471-2164-11-538.
- ↑ «A system for annotating alternative splice isoforms». APPRIS. July 2014. Consultado el 6 September 2014.
- ↑ «What is a gene, post-ENCODE? History and updated definition.». Genome Res 17 (6): 669-81. 2007. PMID 17567988. doi:10.1101/gr.6339607.
- ↑ «ENCODE: Encyclopedia of DNA Elements». ENCODE. c. 2014. Consultado el 7 September 2014.
- ↑ a b «ENCODE: Pilot Project at UCSC». ENCODE. c. 2007. Consultado el 7 September 2014.
- ↑ «The Human Genome Project». Genetics Home Reference. U.S. National Library of Medicine (NLM). 1 September 2014. Consultado el 7 September 2014.
- ↑ «ENCODE data in Ensembl». Ensembl. August 2014. Consultado el 7 September 2014.
- ↑ «The GENCODE v7 catalog of human long noncoding RNAs: analysis of their gene structure, evolution, and expression.». Genome Res 22 (9): 1775-89. 2012. PMC 3431493. PMID 22955988. doi:10.1101/gr.132159.111.
- ↑ «GENCODE - lncRNA microarray». GENCODE. c. 2013. Consultado el 10 September 2014.
- ↑ «GENCODE - RGASP 1/2 Guidelines». GENCODE. c. 2013. Consultado el 10 September 2014.
- ↑ «GENCODE - RGASP 1/2 Guidelines». GENCODE. c. 2013. Consultado el 10 September 2014.



