Diferencia entre revisiones de «Robustez (evolución)»
Creado al traducir la página «Robustness (evolution)» |
(Sin diferencias)
|
Revisión del 00:13 11 jul 2019

La robustez de un sistema biológico (también llamada robustez biológica o genética[1]) es la persistencia de cierta característica o rasgo en un sistema bajo perturbaciones o condiciones de incertidumbre.[2][3] La robustez en el desarrollo se conoce como canalización.[4][5] Según el tipo de perturbación involucrada, la robustez puede clasificarse como robustez mutacional, ambiental, de recombinación o de comportamiento, etc.[6][7][8] La robustez se logra a través de la combinación de muchos mecanismos genéticos y moleculares y puede evolucionar por selección directa o indirecta. Se han desarrollado varios sistemas modelo para estudiar experimentalmente la robustez y sus consecuencias evolutivas.
Clasificación
Robustez mutacional
La robustez mutacional (también llamada tolerancia a la mutación) describe hasta qué punto el fenotipo de un organismo permanece constante a pesar de la mutación.[9] La robustez puede medirse empíricamente para varios genomas[10][11] y genes individuales[12] al inducir mutaciones y medir qué proporción de mutantes retienen el mismo fenotipo, función o aptitud. Más generalmente, esto se conoce como la distribución de los efectos de la condición física de la mutación (es decir, las frecuencias de diferentes formas físicas de mutantes). Las proteínas investigadas hasta ahora han demostrado una tolerancia a mutaciones de aproximadamente el 66% (es decir, dos tercios de las mutaciones son neutrales).[13]
Por el contrario, la robustez mutacional medida de los organismos varía ampliamente. Por ejemplo,> 95% de las mutaciones puntuales en C. elegans no tienen un efecto detectable[14] e incluso el 90% de las inactivaciones genéticas únicas en E. coli no son letales.[15] Sin embargo, los virus solo toleran el 20-40% de las mutaciones y, por lo tanto, son mucho más sensibles a las mutaciones.[10]
Robustez ambiental
En diferentes entornos, la adaptación perfecta a una condición puede venir a expensas de la adaptación a otra. En consecuencia, la presión de selección total en un organismo es la selección promedio en todos los entornos ponderada por el porcentaje de tiempo empleado en ese entorno. Por lo tanto, el entorno variable puede seleccionar la robustez ambiental en la que los organismos pueden funcionar en una amplia gama de condiciones con pocos cambios en el fenotipo o la aptitud (biología). Algunos organismos muestran adaptaciones para tolerar grandes cambios de temperatura, disponibilidad de agua, salinidad o disponibilidad de alimentos. Las plantas, en particular, no pueden moverse cuando el entorno cambia y, por lo tanto, muestran una gama de mecanismos para lograr la solidez ambiental. De manera similar, esto se puede ver en las proteínas como tolerancia a una amplia gama de solventes, concentraciones de iones o temperaturas.
Causas genéticas y moleculares

Los genomas mutan por el daño ambiental y la replicación imperfecta, sin embargo, muestran una tolerancia notable. Esto proviene de la robustez tanto a nivel del genoma como a nivel de proteínas.
Robustez mutacional del organismo
Hay muchos mecanismos que proporcionan robustez al genoma. Por ejemplo, la redundancia genética reduce el efecto de las mutaciones en cualquier copia de un gen de copias múltiples.[16] Además, el flujo a través de una vía metabólica suele estar limitado por solo algunos de los pasos, lo que significa que los cambios en la función de muchas de las enzimas tienen poco efecto sobre la aptitud física.[17][18] De manera similar, las redes metabólicas tienen múltiples vías alternativas para producir muchos metabolitos clave.[19]
Robustez mutacional de la proteína
La tolerancia a la mutación de proteínas es el producto de dos características principales: la estructura del código genético y la robustez estructural de las proteínas.[20][21] Las proteínas son resistentes a las mutaciones porque muchas secuencias se pueden plegar en pliegues estructurales muy similares.[22] Una proteína adopta un conjunto limitado de conformaciones nativas porque esos conformadores tienen menos energía que los estados desplegados y mal plegados (ΔΔG de plegado).[23][24] Esto se logra mediante una red interna distribuida de interacciones cooperativas (hidrófobas, polares y covalentes).[25] La robustez estructural de la proteína resulta de que pocas mutaciones individuales son lo suficientemente disruptivas para comprometer la función. Las proteínas también han evolucionado para evitar la agregación[26] ya que las proteínas parcialmente plegadas pueden combinarse para formar grandes fibrillas y masas de proteínas insolubles y repetitivas.[27] Hay evidencia de que las proteínas muestran características de diseño negativas para reducir la exposición de los motivos de hoja beta propensos a la agregación en sus estructuras. [28] Además, existe cierta evidencia de que el código genético en sí mismo puede optimizarse de tal manera que la mayoría de las mutaciones puntuales conducen a aminoácidos similares (conservadores).[29][30] Juntos, estos factores crean una distribución de los efectos de aptitud física de las mutaciones que contiene una alta proporción de mutaciones neutrales y casi neutrales. [12]
Consecuencias evolutivas
Dado que los organismos están constantemente expuestos a perturbaciones genéticas y no genéticas, la robustez es importante para garantizar la estabilidad de los fenotipos . Además, bajo el equilibrio mutación-selección, la solidez mutacional puede permitir que se acumule una variación genética críptica en una población. Aunque fenotípicamente neutrales en un entorno estable, estas diferencias genéticas pueden revelarse como diferencias de rasgos en una forma dependiente del entorno (capacitancia evolutiva ), lo que permite la expresión de un mayor número de fenotipos hereditarios en poblaciones expuestas a un entorno variable.[31]
Ser robusto puede incluso ser favorecido a expensas de la condición física total como una estrategia evolutivamente estable (también llamada supervivencia del más plano). [32] Un pico alto pero estrecho de un paisaje de aptitud física confiere una buena aptitud física pero una robustez baja, ya que la mayoría de las mutaciones llevan a una pérdida masiva de la aptitud física. Las altas tasas de mutación pueden favorecer a la población de picos de condición física más bajos pero más amplios. Los sistemas biológicos más críticos también pueden tener una mayor selección de robustez, ya que las reducciones en la función son más perjudiciales para la aptitud física.[33] Se cree que la robustez mutacional es un factor determinante de la formación de especies teóricas virales.
Robustez mutacional emergente
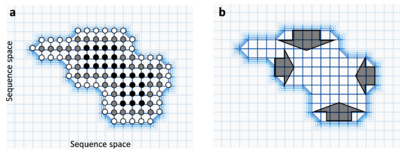
La selección natural puede seleccionar directa o indirectamente por robustez. Cuando las tasas de mutación son altas y el tamaño de la población es grande, se prevé que las poblaciones se trasladen a regiones más densamente conectadas de la red neutra, ya que las variantes menos robustas tienen menos descendientes mutantes supervivientes.[34] Las condiciones bajo las cuales la selección podría actuar para aumentar directamente la robustez mutacional de esta manera son restrictivas y, por lo tanto, se cree que dicha selección se limita a solo unos pocos virus [35] y microbios [36] tienen un gran tamaño de población y una alta mutación tarifas Dicha robustez emergente se ha observado en la evolución experimental del citocromo P450s [37] y la B-lactamasa.[38] Por el contrario, la robustez mutacional puede evolucionar como un subproducto de la selección natural para la robustez de las perturbaciones ambientales.[39][40][41][42] [43]
Robustez y evolucionabilidad
Se ha pensado que la robustez mutacional tiene un impacto negativo en la capacidad de evolución porque reduce la accesibilidad mutacional de fenotipos hereditarios distintos para un genotipo único y reduce las diferencias selectivas dentro de una población genéticamente diversa. Sin embargo, contrariamente a la intuición, se ha planteado la hipótesis de que la solidez fenotípica hacia las mutaciones en realidad puede aumentar el ritmo de la adaptación fenotípica hereditaria cuando se observa durante largos períodos de tiempo. [44] [45] [46] [47]
Una hipótesis sobre cómo la robustez promueve la capacidad de evolución en poblaciones asexuales es que las redes conectadas de genotipos neutrales para la aptitud física dan como resultado una robustez mutacional que reduce la accesibilidad de nuevos fenotipos hereditarios en escalas de tiempo cortas. Durante periodos de tiempo más largos, la mutación neutral y la deriva genética hacen que la población se extienda sobre una red neutral más grande en el espacio de genotipos. Esta diversidad genética le da a la población acceso mutacional a un mayor número de fenotipos hereditarios distintos que se pueden alcanzar desde diferentes puntos de la red neutral.[44][45][47][48][49][50][51] Sin embargo, este mecanismo puede estar limitado a fenotipos dependientes de un solo locus genético; para los rasgos poligénicos, la diversidad genética en las poblaciones asexuales no aumenta significativamente la capacidad de evolución.[52]
En el caso de las proteínas, la robustez promueve la evolvibilidad en forma de un exceso de energía libre de plegamiento.[53] Dado que la mayoría de las mutaciones reducen la estabilidad, un exceso de energía libre de plegado permite la tolerancia de mutaciones que son beneficiosas para la actividad pero que de otro modo desestabilizarían la proteína.
En las poblaciones sexuales, la robustez conduce a la acumulación de variaciones genéticas crípticas con alto potencial evolutivo.[54][55]
La capacidad de evolución puede ser alta cuando la robustez es reversible, con una capacitancia evolutiva que permite un cambio entre la alta robustez en la mayoría de las circunstancias y la baja robustez en momentos de estrés.[56]
Métodos y sistemas modelo
Hay muchos sistemas que se han utilizado para estudiar la robustez. Los modelos in silico se han utilizado para modelar la estructura secundaria del ARN, los modelos de red de proteínas o las redes de genes. Los sistemas experimentales para genes individuales incluyen la actividad enzimática del citocromo P450,[37] B-lactamasa, [38] ARN polimerasa,[13] y LacI[13] se han utilizado. Se ha investigado la robustez de todo el organismo en la aptitud del virus de ARN,[10] quimiotaxis bacteriana, aptitud de Drosophila,[15] red de polaridad de segmento, red neurogénica y gradiente de proteína morfogenética ósea, aptitud de C. elegans[14] y desarrollo de la vulva y reloj circadiano de los mamíferos.[9]
Véase también
- Distribución de los efectos de fitness.
- Evolucionabilidad
- Canalización
- Red neutra (evolución)
- Epistasis
- Capacitancia evolutiva
- Paisaje de fitness
- Biología evolutiva evolutiva.
Referencias
- ↑ Kitano, Hiroaki (2004). «Biological robustness». Nature Reviews Genetics 5 (11): 826-37. PMID 15520792. doi:10.1038/nrg1471.
- ↑ Stelling, Jörg; Sauer, Uwe; Szallasi, Zoltan; Doyle, Francis J.; Doyle, John (2004). «Robustness of Cellular Functions». Cell 118 (6): 675-85. PMID 15369668. doi:10.1016/j.cell.2004.09.008.
- ↑ Félix, M-A; Wagner, A (2006). «Robustness and evolution: Concepts, insights and challenges from a developmental model system». Heredity 100 (2): 132-40. PMID 17167519. doi:10.1038/sj.hdy.6800915.
- ↑ Waddington, C. H. (1942). «Canalization of Development and the Inheritance of Acquired Characters». Nature 150 (3811): 563-5. Bibcode:1942Natur.150..563W. doi:10.1038/150563a0.
- ↑ De Visser, JA; Hermisson, J; Wagner, GP; Ancel Meyers, L; Bagheri-Chaichian, H; Blanchard, JL; Chao, L; Cheverud, JM et al. (2003). «Perspective: Evolution and detection of genetic robustness». Evolution; international journal of organic evolution 57 (9): 1959-72. PMID 14575319.
- ↑ Fernandez-Leon, Jose A. (2011). «Evolving cognitive-behavioural dependencies in situated agents for behavioural robustness». Biosystems 106 (2–3): 94-110. PMID 21840371. doi:10.1016/j.biosystems.2011.07.003.
- ↑ Fernandez-Leon, Jose A. (2011). «Behavioural robustness: A link between distributed mechanisms and coupled transient dynamics». Biosystems 105 (1): 49-61. PMID 21466836. doi:10.1016/j.biosystems.2011.03.006.
- ↑ Fernandez-Leon, Jose A. (2011). «Evolving experience-dependent robust behaviour in embodied agents». Biosystems 103 (1): 45-56. PMID 20932875. doi:10.1016/j.biosystems.2010.09.010.
- ↑ a b Wagner A (2005). Robustness and evolvability in living systems. Princeton University Press. ISBN 0-691-12240-7.[página requerida]
- ↑ a b c Sanjuán, R (27 de junio de 2010). «Mutational fitness effects in RNA and single-stranded DNA viruses: common patterns revealed by site-directed mutagenesis studies.». Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences 365 (1548): 1975-82. PMC 2880115. PMID 20478892. doi:10.1098/rstb.2010.0063.
- ↑ Eyre-Walker, A; Keightley, PD (Aug 2007). «The distribution of fitness effects of new mutations.». Nature Reviews Genetics 8 (8): 610-8. PMID 17637733. doi:10.1038/nrg2146.
- ↑ a b Hietpas, RT; Jensen, JD; Bolon, DN (10 de mayo de 2011). «Experimental illumination of a fitness landscape.». Proceedings of the National Academy of Sciences of the United States of America 108 (19): 7896-901. Bibcode:2011PNAS..108.7896H. PMC 3093508. PMID 21464309. doi:10.1073/pnas.1016024108.
- ↑ a b c Guo, HH; Choe, J; Loeb, LA (22 de junio de 2004). «Protein tolerance to random amino acid change.». Proceedings of the National Academy of Sciences of the United States of America 101 (25): 9205-10. Bibcode:2004PNAS..101.9205G. PMC 438954. PMID 15197260. doi:10.1073/pnas.0403255101.
- ↑ a b Davies, E. K.; Peters, A. D.; Keightley, P. D. (10 September 1999). «High Frequency of Cryptic Deleterious Mutations in Caenorhabditis elegans». Science 285 (5434): 1748-1751. PMID 10481013. doi:10.1126/science.285.5434.1748.
- ↑ a b Baba, T; Ara, T; Hasegawa, M; Takai, Y; Okumura, Y; Baba, M; Datsenko, KA; Tomita, M et al. (2006). «Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection.». Molecular Systems Biology 2 (1): 2006.0008. PMC 1681482. PMID 16738554. doi:10.1038/msb4100050.
- ↑ Gu, Z; Steinmetz, LM; Gu, X; Scharfe, C; Davis, RW; Li, WH (Jan 2, 2003). «Role of duplicate genes in genetic robustness against null mutations.». Nature 421 (6918): 63-6. Bibcode:2003Natur.421...63G. PMID 12511954. doi:10.1038/nature01198.
- ↑ Kauffman, Kenneth J; Prakash, Purusharth; Edwards, Jeremy S (October 2003). «Advances in flux balance analysis». Current Opinion in Biotechnology 14 (5): 491-496. PMID 14580578. doi:10.1016/j.copbio.2003.08.001.
- ↑ Nam, H; Lewis, NE; Lerman, JA; Lee, DH; Chang, RL; Kim, D; Palsson, BO (Aug 31, 2012). «Network context and selection in the evolution to enzyme specificity.». Science 337 (6098): 1101-4. Bibcode:2012Sci...337.1101N. PMC 3536066. PMID 22936779. doi:10.1126/science.1216861.
- ↑ Krakauer, DC; Plotkin, JB (5 de febrero de 2002). «Redundancy, antiredundancy, and the robustness of genomes.». Proceedings of the National Academy of Sciences of the United States of America 99 (3): 1405-9. Bibcode:2002PNAS...99.1405K. PMC 122203. PMID 11818563. doi:10.1073/pnas.032668599.
- ↑ Taverna, DM; Goldstein, RA (Jan 18, 2002). «Why are proteins so robust to site mutations?». Journal of Molecular Biology 315 (3): 479-84. PMID 11786027. doi:10.1006/jmbi.2001.5226.
- ↑ Tokuriki, N; Tawfik, DS (Oct 2009). «Stability effects of mutations and protein evolvability.». Current Opinion in Structural Biology 19 (5): 596-604. PMID 19765975. doi:10.1016/j.sbi.2009.08.003.
- ↑ Meyerguz, L; Kleinberg, J; Elber, R (10 de julio de 2007). «The network of sequence flow between protein structures.». Proceedings of the National Academy of Sciences of the United States of America 104 (28): 11627-32. Bibcode:2007PNAS..10411627M. PMC 1913895. PMID 17596339. doi:10.1073/pnas.0701393104.
- ↑ Karplus, M (17 de junio de 2011). «Behind the folding funnel diagram.». Nature Chemical Biology 7 (7): 401-4. PMID 21685880. doi:10.1038/nchembio.565.
- ↑ Tokuriki, N; Stricher, F; Schymkowitz, J; Serrano, L; Tawfik, DS (22 de junio de 2007). «The stability effects of protein mutations appear to be universally distributed.». Journal of Molecular Biology 369 (5): 1318-32. PMID 17482644. doi:10.1016/j.jmb.2007.03.069.
- ↑ Shakhnovich, BE; Deeds, E; Delisi, C; Shakhnovich, E (Mar 2005). «Protein structure and evolutionary history determine sequence space topology.». Genome Research 15 (3): 385-92. PMC 551565. PMID 15741509. doi:10.1101/gr.3133605.
- ↑ Monsellier, E; Chiti, F (Aug 2007). «Prevention of amyloid-like aggregation as a driving force of protein evolution.». EMBO Reports 8 (8): 737-42. PMC 1978086. PMID 17668004. doi:10.1038/sj.embor.7401034.
- ↑ Fink, AL (1998). «Protein aggregation: folding aggregates, inclusion bodies and amyloid.». Folding & Design 3 (1): R9-23. PMID 9502314. doi:10.1016/s1359-0278(98)00002-9.
- ↑ Richardson, JS; Richardson, DC (5 de marzo de 2002). «Natural beta-sheet proteins use negative design to avoid edge-to-edge aggregation.». Proceedings of the National Academy of Sciences of the United States of America 99 (5): 2754-9. Bibcode:2002PNAS...99.2754R. PMC 122420. PMID 11880627. doi:10.1073/pnas.052706099.
- ↑ «Directed evolution of a model primordial enzyme provides insights into the development of the genetic code.». PLOS Genetics 9 (1): e1003187. 2013. PMC 3536711. PMID 23300488. doi:10.1371/journal.pgen.1003187. Plantilla:Open access
- ↑ Firnberg, E; Ostermeier, M (Aug 2013). «The genetic code constrains yet facilitates Darwinian evolution.». Nucleic Acids Research 41 (15): 7420-8. PMC 3753648. PMID 23754851. doi:10.1093/nar/gkt536.
- ↑ Masel J Siegal ML (2009). «Robustness: mechanisms and consequences». Trends in Genetics 25 (9): 395-403. PMC 2770586. PMID 19717203. doi:10.1016/j.tig.2009.07.005.
- ↑ Wilke, CO; Wang, JL; Ofria, C; Lenski, RE; Adami, C (19 de julio de 2001). «Evolution of digital organisms at high mutation rates leads to survival of the flattest.». Nature 412 (6844): 331-3. PMID 11460163. doi:10.1038/35085569.
- ↑ Van Dijk; Van Mourik, Simon; Van Ham, Roeland C. H. J. (2012). «Mutational Robustness of Gene Regulatory Networks». PLoS ONE 7 (1): e30591. Bibcode:2012PLoSO...730591V. PMC 3266278. PMID 22295094. doi:10.1371/journal.pone.0030591. Consultado el 10 de junio de 2013. Plantilla:Open access
- ↑ «Neutral evolution of mutational robustness». PNAS 96 (17): 9716-9720. 1999. Bibcode:1999PNAS...96.9716V. PMC 22276. PMID 10449760. doi:10.1073/pnas.96.17.9716.
- ↑ «Evolution of mutational robustness in an RNA virus». PLoS Biology 3 (11): 1939-1945. 2005. doi:10.1371/journal.pbio.0030381. Plantilla:Open access
- ↑ Masel J, Maughan H; Maughan (2007). «Mutations Leading to Loss of Sporulation Ability in Bacillus subtilis Are Sufficiently Frequent to Favor Genetic Canalization». Genetics 175 (1): 453-457. PMC 1775008. PMID 17110488. doi:10.1534/genetics.106.065201.
- ↑ a b Bloom, JD; Lu, Z; Chen, D; Raval, A; Venturelli, OS; Arnold, FH (17 de julio de 2007). «Evolution favors protein mutational robustness in sufficiently large populations.». BMC Biology 5: 29. PMC 1995189. PMID 17640347. doi:10.1186/1741-7007-5-29. Plantilla:Open access
- ↑ a b Bershtein, Shimon; Goldin, Korina; Tawfik, Dan S. (June 2008). «Intense Neutral Drifts Yield Robust and Evolvable Consensus Proteins». Journal of Molecular Biology 379 (5): 1029-1044. PMID 18495157. doi:10.1016/j.jmb.2008.04.024.
- ↑ «A single mode of canalization». Trends in Ecology & Evolution 17: e9035. 2002. doi:10.1016/s0169-5347(02)02596-x.
- ↑ «Plasticity, evolvability, and modularity in RNA». Journal of Experimental Zoology 288 (3): 242-283. 2000. PMID 11069142. doi:10.1002/1097-010X(20001015)288:3<242::AID-JEZ5>3.0.CO;2-O.
- ↑ «Congruent Evolution of Genetic and Environmental Robustness in Micro-RNA». Molecular Biology & Evolution 26 (4): 867-874. 2009. PMID 19168567. doi:10.1093/molbev/msp008.
- ↑ «A population genetic theory of canalization». Evolution 51 (2): 329-347. 1997. doi:10.2307/2411105.
- ↑ Lehner B (2010). «Genes Confer Similar Robustness to Environmental, Stochastic, and Genetic Perturbations in Yeast». PLoS ONE 5 (2): 468-473. Bibcode:2010PLoSO...5.9035L. PMC 2815791. PMID 20140261. doi:10.1371/journal.pone.0009035. Plantilla:Open access
- ↑ a b Draghi, Jeremy A.; Parsons, Todd L.; Wagner, Günter P.; Plotkin, Joshua B. (2010). «Mutational robustness can facilitate adaptation». Nature 463 (7279): 353-5. Bibcode:2010Natur.463..353D. PMC 3071712. PMID 20090752. doi:10.1038/nature08694.
- ↑ a b Wagner, A. (2008). «Robustness and evolvability: A paradox resolved». Proceedings of the Royal Society B: Biological Sciences 275 (1630): 91-100. PMC 2562401. PMID 17971325. doi:10.1098/rspb.2007.1137.
- ↑ «Robustness and evolvability». Trends in Genetics 26 (9): 406-414. 2010. PMC 3198833. PMID 20598394. doi:10.1016/j.tig.2010.06.002.
- ↑ a b Aldana; Balleza, E; Kauffman, S; Resendiz, O (2007). «Robustness and evolvability in genetic regulatory networks». Journal of Theoretical Biology 245 (3): 433-448. PMID 17188715. doi:10.1016/j.jtbi.2006.10.027.
- ↑ Babajide; Hofacker, I. L.; Sippl, M. J.; Stadler, P. F. (1997). «Neutral networks in protein space: A computational study based on knowledge-based potentials of mean force». Folding & Design 2 (5): 261-269. PMID 9261065. doi:10.1016/s1359-0278(97)00037-0.
- ↑ van Nimwegen and Crutchfield (2000). «Metastable evolutionary dynamics: Crossing fitness barriers or escaping via neutral paths?». Bulletin of Mathematical Biology 62 (5): 799-848. PMID 11016086. doi:10.1006/bulm.2000.0180.
- ↑ Ciliberti (2007). «Innovation and robustness in complex regulatory gene networks». Proceedings of the National Academy of Sciences, USA 104 (34): 13591-13596. Bibcode:2007PNAS..10413591C. doi:10.1073/pnas.0705396104.
- ↑ Andreas Wagner (2008). «Neutralism and selectionism: a network-based reconciliation». Nature Reviews Genetics 9 (12): 965-974. PMID 18957969. doi:10.1038/nrg2473.
- ↑ Rajon, E.; Masel, J. (18 January 2013). «Compensatory Evolution and the Origins of Innovations». Genetics 193 (4): 1209-1220. PMC 3606098. PMID 23335336. doi:10.1534/genetics.112.148627.
- ↑ Bloom (2006). «Protein stability promotes evolvability». Proceedings of the National Academy of Sciences 103 (15): 5869-74. Bibcode:2006PNAS..103.5869B. PMC 1458665. PMID 16581913. doi:10.1073/pnas.0510098103.
- ↑ Waddington CH (1957). The strategy of the genes. George Allen & Unwin.
- ↑ Masel, J. (30 December 2005). «Cryptic Genetic Variation Is Enriched for Potential Adaptations». Genetics 172 (3): 1985-1991. PMC 1456269. PMID 16387877. doi:10.1534/genetics.105.051649.
- ↑ Masel, J (30 de septiembre de 2013). «Q&A: Evolutionary capacitance.». BMC Biology 11: 103. PMC 3849687. PMID 24228631. doi:10.1186/1741-7007-11-103. Plantilla:Open access
