Dominios asociados topológicamente

Un dominio asociado topológicamente o TAD (por sus siglas en inglés: Topologically Associated Domain) es una región genómica que interactúa consigo misma, lo que significa que las secuencias de ADN dentro de un TAD interactúan físicamente entre sí con más frecuencia que con secuencias fuera del TAD.[1] El tamaño medio de un TAD en células de ratón es de 880 kb, y tienen tamaños similares en especies no mamíferas.[2] Los límites a ambos lados de estos dominios se conservan entre diferentes tipos de células de mamíferos e incluso entre especies[2] y están altamente enriquecidos con el factor de unión a CCCTC (CTCF) y sitios de unión a cohesina.[1] Además, algunos tipos de genes (como los genes de transferencia de ARN y los genes de mantenimiento) aparecen cerca de los límites de TAD con más frecuencia de lo que cabría esperar al azar.[3][4]
Las funciones de los TAD no se comprenden completamente y todavía es un tema de debate. La mayoría de los estudios indican que los TAD regulan la expresión génica al limitar la interacción potenciador-promotor a cada TAD,[5] sin embargo, un estudio reciente desacopla la organización del TAD y la expresión génica.[6] La alteración de los límites del TAD se asocia con una amplia gama de enfermedades como el cáncer,[7][8][9] una variedad de malformaciones de las extremidades como la sinpolidactilia, el síndrome de Cooks y el síndrome F,[10] y el número de trastornos como el cuerpo calloso hipoplásico y la leucodistrofia desmielinizante de inicio en la edad adulta.[10]
Los mecanismos subyacentes a la formación de TAD también son complejos y aún no se han aclarado por completo, aunque varios complejos de proteínas y elementos de ADN están asociados con los límites de TAD. Sin embargo, el modelo de esposas y el modelo de extrusión de bucle se describen para describir la formación de TAD con la ayuda de CTCF y proteínas de cohesina.[11] Además, se ha propuesto que la rigidez de los límites del TAD en sí misma podría causar el aislamiento del dominio y la formación del TAD.
Descubrimiento y diversidad[editar]
Los TAD se definen como regiones cuyas secuencias de ADN se contactan preferentemente entre sí. Fueron descubiertos en 2012 utilizando métodos de captura de la conformación de los cromosomas, incluida Hi-C.[3][4][12] Se ha demostrado que están presentes en múltiples especies,[13] incluyendo moscas de la fruta (Drosophila),[14] ratón,[3] plantas, hongos y genomas humanos.[4] En las bacterias, se les conoce como dominios de interacción cromosómica (CID).[13]
Herramientas analíticas y bases de datos[editar]
Las ubicaciones de TAD se definen aplicando un algoritmo a los datos Hi-C. Por ejemplo, los TAD a menudo se denominan de acuerdo con el llamado "índice de direccionalidad".[4] El índice de direccionalidad se calcula para contenedores individuales de 40 kb, recopilando las lecturas que caen en el contenedor y observando si sus lecturas emparejadas se asignan aguas arriba o aguas abajo del contenedor (los pares de lectura deben abarcar no más de 2 Mb). Un valor positivo indica que hay más pares de lectura en sentido descendente que en sentido ascendente, y un valor negativo indica lo contrario. Matemáticamente, el índice de direccionalidad es una estadística de chi-cuadrado con signo.
El desarrollo de navegadores y bases de datos del genoma 3D como The 3D Genome Browser,[15] 3DIV,[16] 3D-GNOME,[17] y TADKB[18] nos han permitido visualizar la organización TAD de las regiones de interés en diferentes tipos celulares.
Mecanismos de formación[editar]
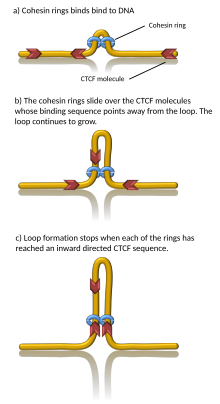
Se sabe que varias proteínas están asociadas con la formación de TAD, incluida la proteína CTCF y la cohesina del complejo proteico.[1] También se desconoce qué componentes se requieren en los límites de TAD; sin embargo, en células de mamíferos, se ha demostrado que estas regiones límite tienen niveles comparativamente altos de unión a CTCF. Además, algunos tipos de genes (como los genes codificantes de ARN de transferencia y los genes constitutivos) aparecen cerca de los límites de TAD con más frecuencia de lo que cabría esperar por casualidad.[3][4]
Las simulaciones por computadora han demostrado que la extrusión de bucles de cromatina impulsada por el superenrollamiento generado por transcripción asegura que la cohesina se reubique rápidamente y los bucles crezcan con una velocidad razonable y en una buena dirección. Además, el mecanismo de extrusión de bucle impulsado por superenrollamiento es consistente con explicaciones anteriores que proponen por qué los TAD flanqueados por sitios de unión CTCF convergentes forman bucles de cromatina más estables que los TAD flanqueados por sitios de unión CTCF divergentes. En este modelo, el superenrollamiento también estimula los contactos del promotor potenciador y se propone que la transcripción del eRNA envía la primera ola de superenrollamiento que puede activar la transcripción del mRNA en un TAD determinado.[19][20] Los modelos computacionales también mostraron que los anillos de cohesina actúan como un peine molecular muy eficiente, empujando nudos y enredos como en los catenanos hacia el borde de los TAD, donde estos son eliminados por la acción de las topoisomerasas. De manera constante, la eliminación de enredos durante la extrusión del asa también aumenta el grado de segregación entre los cromosomas.[21] Sin embargo, la prueba de la extrusión del bucle de ADN se limita hasta ahora a la condensina (complejo de proteína hermana de la cohesina).[22]
Propiedades[editar]
Conservación[editar]
Se ha informado que los TAD son relativamente constantes entre diferentes tipos de células (en células madre y células sanguíneas, por ejemplo), e incluso entre especies en casos específicos.[23][24]
Relación con la interacción Promotor-Potenciador[editar]
La mayoría de interacciones observadas entre promotores y potenciadores no cruzan los límites de TAD. La eliminación de un límite de TAD (por ejemplo, utilizando CRISPR para eliminar la región relevante del genoma) puede permitir que se formen nuevos contactos promotor-potenciador. Esto puede afectar la expresión génica cercana; se ha demostrado que tal mala regulación causa malformaciones en las extremidades (por ejemplo, polidactilia) en humanos y ratones.[23]
Las simulaciones por computadora han demostrado que el superenrollamiento inducido por la transcripción de las fibras de cromatina puede explicar cómo se forman los TAD y cómo pueden asegurar interacciones muy eficientes entre los potenciadores y sus promotores afines ubicados en el mismo TAD.[20]
Relación con otras características estructurales del genoma[editar]
Se ha demostrado que los dominios de tiempo de replicación están asociados con los TAD, ya que su límite está colocalizado con los límites de los TAD que se encuentran a ambos lados de los compartimentos.[25] Se propone que los vecindarios aislados, bucles de ADN formados por regiones unidas a CTCF / cohesina, subyacen funcionalmente a los TAD.[26]
Rol en enfermedades[editar]
La alteración de los límites de TAD puede afectar la expresión de genes cercanos y esto puede causar enfermedades.[27]
Por ejemplo, se ha informado que las variantes genómicas estructurales que alteran los límites del TAD causan trastornos del desarrollo como malformaciones de las extremidades humanas.[28][29][30] Además, varios estudios han proporcionado evidencia de que la alteración o reordenación de los límites de TAD puede proporcionar ventajas de crecimiento para ciertos cánceres, como la leucemia linfoblástica aguda de células T (LLA-T),[31] gliomas,[32] y cáncer de pulmón.[33]
Dominios asociados a la lámina nuclear[editar]
Articulo principal: Lámina nuclear

Los dominios asociados a la lámina (LAD) son partes de la cromatina que interactúan en gran medida con la lámina, una estructura en forma de red en la membrana interna del núcleo.[34] Los LAD consisten principalmente en cromatina transcripcionalmente silenciosa, enriquecida con Lys27 trimetilada en la histona H3 (también denominada H3K27me3), que es una modificación de histona postraduccional común de la heterocromatina.[35] Los LAD tienen sitios de unión a CTCF en su periferia
Referencias[editar]
- ↑ a b c Pombo, Ana; Dillon, Niall (Abril de 2015). «Three-dimensional genome architecture: players and mechanisms». Nature Reviews Molecular Cell Biology (en inglés) 16 (4): 245-257. ISSN 1471-0080. PMID 25757416. doi:10.1038/nrm3965. Consultado el 25 de febrero de 2023.
- ↑ a b Yu, Miao; Ren, Bing (6 de octubre de 2017). «The Three-Dimensional Organization of Mammalian Genomes». Annual Review of Cell and Developmental Biology 33: 265-289. ISSN 1530-8995. PMC 5837811. PMID 28783961. doi:10.1146/annurev-cellbio-100616-060531. Consultado el 25 de febrero de 2023.
- ↑ a b c d Nora, Elphège P.; Lajoie, Bryan R.; Schulz, Edda G.; Giorgetti, Luca; Okamoto, Ikuhiro; Servant, Nicolas; Piolot, Tristan; van Berkum, Nynke L. et al. (11 de abril de 2012). «Spatial partitioning of the regulatory landscape of the X-inactivation centre». Nature 485 (7398): 381-385. ISSN 1476-4687. PMC 3555144. PMID 22495304. doi:10.1038/nature11049. Consultado el 25 de febrero de 2023.
- ↑ a b c d e Dixon, Jesse R.; Selvaraj, Siddarth; Yue, Feng; Kim, Audrey; Li, Yan; Shen, Yin; Hu, Ming; Liu, Jun S. et al. (11 de abril de 2012). «Topological domains in mammalian genomes identified by analysis of chromatin interactions». Nature 485 (7398): 376-380. ISSN 1476-4687. PMC 3356448. PMID 22495300. doi:10.1038/nature11082. Consultado el 25 de febrero de 2023.
- ↑ Krijger, Peter Hugo Lodewijk; de Laat, Wouter (Diciembre de 2016). «Regulation of disease-associated gene expression in the 3D genome». Nature Reviews. Molecular Cell Biology 17 (12): 771-782. ISSN 1471-0080. PMID 27826147. doi:10.1038/nrm.2016.138. Consultado el 25 de febrero de 2023.
- ↑ Ghavi-Helm, Yad; Jankowski, Aleksander; Meiers, Sascha; Viales, Rebecca R.; Korbel, Jan O.; Furlong, Eileen E. M. (Agosto de 2019). «Highly rearranged chromosomes reveal uncoupling between genome topology and gene expression». Nature Genetics (en inglés) 51 (8): 1272-1282. ISSN 1546-1718. PMC 7116017. PMID 31308546. doi:10.1038/s41588-019-0462-3. Consultado el 25 de febrero de 2023.
- ↑ Corces, M. Ryan; Corces, Victor G. (Febrero de 2016). «The three-dimensional cancer genome». Current Opinion in Genetics & Development 36: 1-7. ISSN 1879-0380. PMC 4880523. PMID 26855137. doi:10.1016/j.gde.2016.01.002. Consultado el 25 de febrero de 2023.
- ↑ Valton, Anne-Laure; Dekker, Job (Febrero de 2016). «TAD disruption as oncogenic driver». Current Opinion in Genetics & Development 36: 34-40. ISSN 1879-0380. PMC 4880504. PMID 27111891. doi:10.1016/j.gde.2016.03.008. Consultado el 25 de febrero de 2023.
- ↑ Achinger-Kawecka, Joanna; Clark, Susan J. (Enero de 2017). «Disruption of the 3D cancer genome blueprint». Epigenomics 9 (1): 47-55. ISSN 1750-192X. PMID 27936932. doi:10.2217/epi-2016-0111. Consultado el 25 de febrero de 2023.
- ↑ a b Spielmann, Malte; Lupiáñez, Darío G.; Mundlos, Stefan (Julio de 2018). «Structural variation in the 3D genome». Nature Reviews. Genetics 19 (7): 453-467. ISSN 1471-0064. PMID 29692413. doi:10.1038/s41576-018-0007-0. Consultado el 25 de febrero de 2023.
- ↑ Dixon, Jesse R.; Gorkin, David U.; Ren, Bing (2 de junio de 2016). «Chromatin Domains: The Unit of Chromosome Organization». Molecular Cell 62 (5): 668-680. ISSN 1097-4164. PMC 5371509. PMID 27259200. doi:10.1016/j.molcel.2016.05.018. Consultado el 25 de febrero de 2023.
- ↑ de Laat, Wouter; Duboule, Denis (24 de octubre de 2013). «Topology of mammalian developmental enhancers and their regulatory landscapes». Nature 502 (7472): 499-506. ISSN 1476-4687. PMID 24153303. doi:10.1038/nature12753. Consultado el 25 de febrero de 2023.
- ↑ a b Szabo, Quentin; Bantignies, Frédéric; Cavalli, Giacomo (2019-04). «Principles of genome folding into topologically associating domains». Science Advances 5 (4): eaaw1668. ISSN 2375-2548. PMC 6457944. PMID 30989119. doi:10.1126/sciadv.aaw1668. Consultado el 25 de febrero de 2023.
- ↑ Sexton, Tom; Yaffe, Eitan; Kenigsberg, Ephraim; Bantignies, Frédéric; Leblanc, Benjamin; Hoichman, Michael; Parrinello, Hugues; Tanay, Amos et al. (3 de febrero de 2012). «Three-dimensional folding and functional organization principles of the Drosophila genome». Cell 148 (3): 458-472. ISSN 1097-4172. PMID 22265598. doi:10.1016/j.cell.2012.01.010. Consultado el 25 de febrero de 2023.
- ↑ Wang, Yanli; Song, Fan; Zhang, Bo; Zhang, Lijun; Xu, Jie; Kuang, Da; Li, Daofeng; Choudhary, Mayank N. K. et al. (4 de octubre de 2018). «The 3D Genome Browser: a web-based browser for visualizing 3D genome organization and long-range chromatin interactions». Genome Biology 19 (1): 151. ISSN 1474-760X. PMC 6172833. PMID 30286773. doi:10.1186/s13059-018-1519-9. Consultado el 25 de febrero de 2023.
- ↑ Dang, Dongchan; Jang, Insu; Choi, Jinhyuk; Kim, Min-Seo; Lee, Andrew J.; Kim, Hyunwoong; Eom, Junghyun; Kim, Dongsup et al. (2 de noviembre de 2017). «3DIV: A 3D-genome Interaction Viewer and database». Nucleic Acids Research 46 (D1): D52-D57. doi:10.1093/nar/gkx1017.
- ↑ Szalaj, Przemyslaw; Michalski, Paul J.; Wróblewski, Przemysław; Tang, Zhonghui; Kadlof, Michal; Mazzocco, Giovanni; Ruan, Yijun; Plewczynski, Dariusz (8 de julio de 2016). «3D-GNOME: an integrated web service for structural modeling of the 3D genome». Nucleic Acids Research 44 (W1): W288-293. ISSN 1362-4962. PMC 4987952. PMID 27185892. doi:10.1093/nar/gkw437. Consultado el 25 de febrero de 2023.
- ↑ Liu, Tong; Porter, Jacob; Zhao, Chenguang; Zhu, Hao; Wang, Nan; Sun, Zheng; Mo, Yin-Yuan; Wang, Zheng (14 de marzo de 2019). «TADKB: Family classification and a knowledge base of topologically associating domains». BMC Genomics 20 (1): 217. ISSN 1471-2164. PMC 6419456. PMID 30871473. doi:10.1186/s12864-019-5551-2. Consultado el 25 de febrero de 2023.
- ↑ Racko, Dusan; Benedetti, Fabrizio; Dorier, Julien; Stasiak, Andrzej (28 de febrero de 2018). «Transcription-induced supercoiling as the driving force of chromatin loop extrusion during formation of TADs in interphase chromosomes». Nucleic Acids Research 46 (4): 1648-1660. ISSN 1362-4962. PMC 5829651. PMID 29140466. doi:10.1093/nar/gkx1123. Consultado el 25 de febrero de 2023.
- ↑ a b Racko, Dusan; Benedetti, Fabrizio; Dorier, Julien; Stasiak, Andrzej (5 de noviembre de 2018). «Are TADs supercoiled?». Nucleic Acids Research 47 (2): 521-532. doi:10.1093/nar/gky1091.
- ↑ Racko, Dusan; Benedetti, Fabrizio; Goundaroulis, Dimos; Stasiak, Andrzej (11 de octubre de 2018). «Chromatin Loop Extrusion and Chromatin Unknotting». Polymers 10 (10): 1126. ISSN 2073-4360. PMC 6403842. PMID 30961051. doi:10.3390/polym10101126. Consultado el 25 de febrero de 2023.
- ↑ Ganji, Mahipal; Shaltiel, Indra A.; Bisht, Shveta; Kim, Eugene; Kalichava, Ana; Haering, Christian H.; Dekker, Cees (6 de abril de 2018). «Real-time imaging of DNA loop extrusion by condensin». Science (New York, N.Y.) 360 (6384): 102-105. ISSN 1095-9203. PMC 6329450. PMID 29472443. doi:10.1126/science.aar7831. Consultado el 25 de febrero de 2023.
- ↑ a b Jost, Daniel; Vaillant, Cédric; Meister, Peter (2017-02). «Coupling 1D modifications and 3D nuclear organization: data, models and function». Current Opinion in Cell Biology 44: 20-27. ISSN 1879-0410. PMID 28040646. doi:10.1016/j.ceb.2016.12.001. Consultado el 25 de febrero de 2023.
- ↑ Yang, Yang; Zhang, Yang; Ren, Bing; Dixon, Jesse R.; Ma, Jian (26 de junio de 2019). «Comparing 3D Genome Organization in Multiple Species Using Phylo-HMRF». Cell Systems 8 (6): 494-505.e14. ISSN 2405-4720. PMC 6706282. PMID 31229558. doi:10.1016/j.cels.2019.05.011. Consultado el 25 de febrero de 2023.
- ↑ Marchal, Claire; Sima, Jiao; Gilbert, David M. (Diciembre de 2019). «Control of DNA replication timing in the 3D genome». Nature Reviews. Molecular Cell Biology 20 (12): 721-737. ISSN 1471-0080. PMID 31477886. doi:10.1038/s41580-019-0162-y. Consultado el 25 de febrero de 2023.
- ↑ Ji, Xiong; Dadon, Daniel B.; Powell, Benjamin E.; Fan, Zi Peng; Borges-Rivera, Diego; Shachar, Sigal; Weintraub, Abraham S.; Hnisz, Denes et al. (4 de febrero de 2016). «3D Chromosome Regulatory Landscape of Human Pluripotent Cells». Cell Stem Cell 18 (2): 262-275. ISSN 1875-9777. PMC 4848748. PMID 26686465. doi:10.1016/j.stem.2015.11.007. Consultado el 25 de febrero de 2023.
- ↑ Lupiáñez, Darío G.; Spielmann, Malte; Mundlos, Stefan (Abril de 2016). «Breaking TADs: How Alterations of Chromatin Domains Result in Disease». Trends in genetics: TIG 32 (4): 225-237. ISSN 0168-9525. PMID 26862051. doi:10.1016/j.tig.2016.01.003. Consultado el 25 de febrero de 2023.
- ↑ Lupiáñez, Darío G.; Kraft, Katerina; Heinrich, Verena; Krawitz, Peter; Brancati, Francesco; Klopocki, Eva; Horn, Denise; Kayserili, Hülya et al. (21 de mayo de 2015). «Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions». Cell 161 (5): 1012-1025. ISSN 1097-4172. PMC 4791538. PMID 25959774. doi:10.1016/j.cell.2015.04.004. Consultado el 25 de febrero de 2023.
- ↑ Angier, Natalie (9 de enero de 2017). «A Family’s Shared Defect Sheds Light on the Human Genome». The New York Times (en inglés estadounidense). ISSN 0362-4331. Consultado el 25 de febrero de 2023.
- ↑ Franke, Martin; Ibrahim, Daniel M.; Andrey, Guillaume; Schwarzer, Wibke; Heinrich, Verena; Schöpflin, Robert; Kraft, Katerina; Kempfer, Rieke et al. (13 de octubre de 2016). «Formation of new chromatin domains determines pathogenicity of genomic duplications». Nature 538 (7624): 265-269. ISSN 1476-4687. PMID 27706140. doi:10.1038/nature19800. Consultado el 25 de febrero de 2023.
- ↑ Hnisz, Denes; Weintraub, Abraham S.; Day, Daniel S.; Valton, Anne-Laure; Bak, Rasmus O.; Li, Charles H.; Goldmann, Johanna; Lajoie, Bryan R. et al. (25 de marzo de 2016). «Activation of proto-oncogenes by disruption of chromosome neighborhoods». Science (New York, N.Y.) 351 (6280): 1454-1458. ISSN 1095-9203. PMC 4884612. PMID 26940867. doi:10.1126/science.aad9024. Consultado el 25 de febrero de 2023.
- ↑ Flavahan, William A.; Drier, Yotam; Liau, Brian B.; Gillespie, Shawn M.; Venteicher, Andrew S.; Stemmer-Rachamimov, Anat O.; Suvà, Mario L.; Bernstein, Bradley E. (7 de enero de 2016). «Insulator dysfunction and oncogene activation in IDH mutant gliomas». Nature 529 (7584): 110-114. ISSN 1476-4687. PMC 4831574. PMID 26700815. doi:10.1038/nature16490. Consultado el 25 de febrero de 2023.
- ↑ Weischenfeldt, Joachim; Dubash, Taronish; Drainas, Alexandros P.; Mardin, Balca R.; Chen, Yuanyuan; Stütz, Adrian M.; Waszak, Sebastian M.; Bosco, Graziella et al. (Enero de 2017). «Pan-cancer analysis of somatic copy-number alterations implicates IRS4 and IGF2 in enhancer hijacking». Nature Genetics 49 (1): 65-74. ISSN 1546-1718. PMC 5791882. PMID 27869826. doi:10.1038/ng.3722. Consultado el 25 de febrero de 2023.
- ↑ Gonzalez-Sandoval, Adriana; Gasser, Susan M. (2016-08). «On TADs and LADs: Spatial Control Over Gene Expression». Trends in genetics: TIG 32 (8): 485-495. ISSN 0168-9525. PMID 27312344. doi:10.1016/j.tig.2016.05.004. Consultado el 25 de febrero de 2023.
- ↑ Li, Mo; Liu, Guang-Hui; Izpisua Belmonte, Juan Carlos (23 de julio de 2012). «Navigating the epigenetic landscape of pluripotent stem cells». Nature Reviews. Molecular Cell Biology 13 (8): 524-535. ISSN 1471-0080. PMID 22820889. doi:10.1038/nrm3393. Consultado el 25 de febrero de 2023.
