Grafo ponderado
Una red ponderada, pesada, o con costos es una red donde los vínculos entre nodos tienen asignado un valor de peso. Una red es un sistema cuyos elementos están conectados de alguna manera (Wasserman y Faust, 1994).[1] Los elementos del sistema se representan como nodos (también conocidos como vértices) y las conexiones entre los elementos que interactúan son llamadas vínculos, aristas, arcos o enlaces. Los nodos pueden representar entidades como neuronas, individuos, grupos, organizaciones, aeropuertos o países. Asimismo, los vínculos pueden tomar la forma de amistad, comunicación, colaboración, alianza, flujo o comercio, por nombrar algunas relaciones entre distintas componentes.
En redes del mundo real, no siempre todos los vínculos tienen la misma importancia o capacidad. En muchos casos, se les asocia a los vínculos un valor que los diferencia en términos de fuerza, intensidad o capacidad (Barrat et al., 2004)[2] y Horvath (2011).[3] Acerca de las redes sociales, Mark Granovetter (1973)[4] argumentó que la importancia de las relaciones es función de su duración, intensidad emocional, intimidad e intercambio de servicios. Para las redes no sociales, con frecuencia los pesos refieren a la función que cumplen los vínculos. Ejemplos de esto son diferentes valores de flujos de carbono (mg / m² / día) entre especies en las redes alimentarias (Luczkowich et al., 2003), [5] la cantidad de sinapsis y las uniones de brechas en redes neuronales (Watts y Strogatz, 1998),[6] o la cantidad de tráfico vehicular a lo largo de las conexiones en redes de transporte (Opsahl et al., 2008).[7]
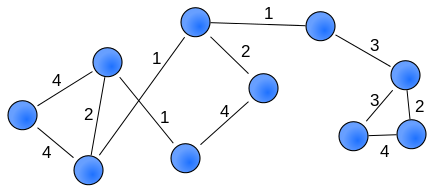
Medir o registrar la importancia de los diferentes enlaces,[8] permite crear una red ponderada que captura más información que una red sin pesos. A continuación se muestra un ejemplo de una red de este tipo (los pesos también se pueden visualizar con vínculos de diferente espesor):
Las redes ponderadas también se usan ampliamente en aplicaciones genómicas y de sistemas biológicos. (Horvath, 2011).[3] Por ejemplo, el análisis de redes ponderadas de coexpresión de genes (WGCNA, por sus siglas en inglés) se usa a menudo para construir una red entre diferentes genes o productos genéticos, basados en datos de expresión de genes como micromatrices (Zhang y Horvath 2005).[9] De manera más general, pueden definirse otras redes ponderadas de correlación a partir de determinar un umbral entre pares entre variables, como activación de distintas partes del cerebro o expresión de diferentes genes).[10]
Métricas aplicables a redes ponderadas
Aunque las redes ponderadas son más difíciles de analizar que aquellas donde los vínculos simplemente fueran booleanos (1 o 0, presentes o ausentes), se han propuesto varias medidas posibles para ellas:
- Fuerza del nodo: la suma de los pesos unidos a las ataduras que pertenecen a un nodo (Barrat et al., 2004)[2]
- Cercanía: se redefine utilizando el algoritmo de distancia de Dijkstra (Newman, 2001)[11]
- Betweenness: redefinida utilizando el algoritmo de distancia de Dijkstra (Brandes, 2001)[12] (detalles)
- El coeficiente de agrupamiento o clustering (global): se redefine utilizando una terna de valores (Opsahl and Panzarasa, 2009)[13]
- El coeficiente de agrupamiento o clustering (local): se redefine utilizando una terna de valores (Barrat et al., 2004)[2] o una fórmula algebraica (Zhang and Horvath 2005) [9]
Una ventaja teórica de las redes ponderadas es que permiten a uno derivar relaciones entre diferentes métricas de la red, también llamados conceptos de red, estadísticas o índices.[3] Por ejemplo, Dong and Horvath (2007)[14] muestran que simples relaciones entre métricas de la red pueden derivarse a partir de grupos de nodos (comunidades) en las redes ponderadas. Para las redes ponderadas de correlación, puede usarse la interpretación angular de las correlaciones para proporcionar una interpretación geométrica de los conceptos teóricos de la red, y derivar relaciones inesperadas entre ellas (Horvath y Dong, 2008).[15]
Software para analizar redes ponderadas
Existe un gran número de paquetes de software que pueden usarse para analizar redes ponderadas. Entre estos se encuentran el software propietario UCINET y el paquete de código abierto tnet. El artículo (no existente en español, sí su versión en inglés) de Software para el análisis de redes sociales lista algunos de ellos.
El paquete de R WGCNA implementa funciones para construir y analizar redes ponderadas, particularmente redes de correlación.[10]
Vea también
Referencias
- ↑ Wasserman, S., Faust, K., 1994. Social Network Analysis: Methods and Applications. Cambridge University Press, New York, NY.
- ↑ a b c A. Barrat and M. Barthelemy and R. Pastor-Satorras and A. Vespignani (2004). «The architecture of complex weighted networks». Proceedings of the National Academy of Sciences 101 (11): 3747-3752. Bibcode:2004PNAS..101.3747B. PMC 374315. PMID 15007165. arXiv:cond-mat/0311416. doi:10.1073/pnas.0400087101.
- ↑ a b c Horvath, S., 2011. Weighted Network Analysis. Applications in Genomics and Systems Biology. Springer Book. ISBN 978-1-4419-8818-8.
- ↑ Granovetter, M (1973). «The strength of weak ties». American Journal of Sociology 78 (6): 1360-1380. doi:10.1086/225469.
- ↑ Luczkowich, J.J.; Borgatti, S.P.; Johnson, J.C.; Everett, M.G. (2003). «Defining and measuring trophic role similarity in food webs using regular equivalence». Journal of Theoretical Biology 220 (3): 303-321. PMID 12468282. doi:10.1006/jtbi.2003.3147. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ D. J. Watts and Steven Strogatz (June 1998). «Collective dynamics of 'small-world' networks». Nature 393 (6684): 440-442. Bibcode:1998Natur.393..440W. PMID 9623998. doi:10.1038/30918. Archivado desde el original el 21 de febrero de 2007.
- ↑ Tore Opsahl and Vittoria Colizza and Pietro Panzarasa and Jose J. Ramasco (2008). «Prominence and control: The weighted rich-club effect». Physical Review Letters 101 (16): 168702. Bibcode:2008PhRvL.101p8702O. PMID 18999722. arXiv:0804.0417. doi:10.1103/PhysRevLett.101.168702.
- ↑ «Operationalisation of tie strength in social networks». 6 de febrero de 2009.
- ↑ a b Zhang, Bin; Horvath, Steve (2005). «A general framework for weighted gene co-expression network analysis». Statistical Applications in Genetics and Molecular Biology 4: Article17. PMID 16646834. doi:10.2202/1544-6115.1128.
- ↑ a b Langfelder, Peter; Horvath, Steve (2008). «WGCNA: an R package for weighted correlation network analysis». BMC Bioinformatics 9: 559. PMC 2631488. PMID 19114008. doi:10.1186/1471-2105-9-559.
- ↑ Mark E J Newman (2001). «Scientific collaboration networks: II. Shortest paths, weighted networks, and centrality». Physical Review E 64 (1): 016132. Bibcode:2001PhRvE..64a6132N. PMID 11461356. arXiv:cond-mat/0011144. doi:10.1103/PhysRevE.64.016132.
- ↑ U Brandes (2008). «On variants of shortest-path betweenness centrality and their generic computation». Social Networks 30 (2): 136-145. doi:10.1016/j.socnet.2007.11.001. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Tore Opsahl and Pietro Panzarasa (2009). «Clustering in Weighted Networks». Social Networks 31 (2): 155-163. doi:10.1016/j.socnet.2009.02.002. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Dong J, Horvath S (2007) Understanding Network Concepts in Modules. BMC Systems Biology 2007, June 1:24
- ↑ Dong, Jun; Horvath, Steve (2008). «Geometric interpretation of gene coexpression network analysis». En Miyano, Satoru, ed. PLoS Computational Biology 4 (8): e1000117. Bibcode:2008PLSCB...4E0117H. PMC 2446438. PMID 18704157. doi:10.1371/journal.pcbi.1000117.