Diferencia entre revisiones de «Aprendizaje automático en bioinformática»
Saving progress... |
→Bases de Datos: Translated databases from en:wiki |
||
| Línea 68: | Línea 68: | ||
El aumento de las publicaciones científicas dificulta la búsqueda y compilación de toda la información relevante disponible sobre un tema determinado. Esto, a su vez, dificulta la recopilación de datos biológicos necesaria para todas las aplicaciones anteriormente mencionadas.<ref name=":5">{{Cite journal|url=http://www.sciencedirect.com/science/article/pii/S1359644605033763|title=Text-mining approaches in molecular biology and biomedicine|last2=Erhardt|first2=Ramon Alonso-Allende|date=15 de marzo de 2005|journal=Drug Discovery Today|volume=10|issue=6|pages=439–445|doi=10.1016/S1359-6446(05)03376-3|last3=Valencia|first3=Alfonso|last=Krallinger|first=Martin}}</ref> El [[procesamiento del lenguaje natural]] permite extraer la información útil dentro de los informes generados por humanos en una base de datos dada. Además, como las anotaciones de proteínas en las bases de datos de proteínas a menudo no reflejan el conjunto de conocimiento conocido, se puede extraer información adicional de la literatura biomédica, permitiendo funciones como la anotación automática, la determinación de la [[Hipótesis de la señal|localización subcelular de una proteína]], el análisis de interacción de proteínas a gran escala o la detección y visualización de regiones que comparten un alto grado de semejanza.<ref name="sing">{{Cita publicación|url=https://link.springer.com/chapter/10.1007/978-3-319-58838-4_26|título=Visualization of Distinct DNA Regions of the Modern Human Relatively to a Neanderthal Genome|apellidos2=Hosseini, M|fecha=June 20–23, 2017|publicación=Iberian Conference on Pattern Recognition and Image Analysis. Springer|páginas=235–242|apellidos3=Silva, R|apellidos4=Pinho, A|apellidos5=Ferreira, P}}</ref> |
El aumento de las publicaciones científicas dificulta la búsqueda y compilación de toda la información relevante disponible sobre un tema determinado. Esto, a su vez, dificulta la recopilación de datos biológicos necesaria para todas las aplicaciones anteriormente mencionadas.<ref name=":5">{{Cite journal|url=http://www.sciencedirect.com/science/article/pii/S1359644605033763|title=Text-mining approaches in molecular biology and biomedicine|last2=Erhardt|first2=Ramon Alonso-Allende|date=15 de marzo de 2005|journal=Drug Discovery Today|volume=10|issue=6|pages=439–445|doi=10.1016/S1359-6446(05)03376-3|last3=Valencia|first3=Alfonso|last=Krallinger|first=Martin}}</ref> El [[procesamiento del lenguaje natural]] permite extraer la información útil dentro de los informes generados por humanos en una base de datos dada. Además, como las anotaciones de proteínas en las bases de datos de proteínas a menudo no reflejan el conjunto de conocimiento conocido, se puede extraer información adicional de la literatura biomédica, permitiendo funciones como la anotación automática, la determinación de la [[Hipótesis de la señal|localización subcelular de una proteína]], el análisis de interacción de proteínas a gran escala o la detección y visualización de regiones que comparten un alto grado de semejanza.<ref name="sing">{{Cita publicación|url=https://link.springer.com/chapter/10.1007/978-3-319-58838-4_26|título=Visualization of Distinct DNA Regions of the Modern Human Relatively to a Neanderthal Genome|apellidos2=Hosseini, M|fecha=June 20–23, 2017|publicación=Iberian Conference on Pattern Recognition and Image Analysis. Springer|páginas=235–242|apellidos3=Silva, R|apellidos4=Pinho, A|apellidos5=Ferreira, P}}</ref> |
||
== Bases de |
== Bases de datos == |
||
Una parte importante de la bioinformática es la gestión de grandes conjuntos de datos, conocidos como bases de datos de referencia. Existen bases para cada tipo de datos biológicos, por ejemplo |
Una parte importante de la bioinformática es la gestión de grandes conjuntos de datos, conocidos como bases de datos de referencia. Existen bases de datos para cada tipo de datos biológicos, por ejemplo para los grupos de genes biosintéticos y los metagenomas. |
||
* Centro Nacional de Información Biotecnológica: El NCBI ofrece un amplio conjunto de recursos en línea de información y datos biológicos, como la base de datos de secuencias de ácidos nucleicos [[GenBank]] y la base de datos de citas y resúmenes de revistas de ciencias de la vida [[PubMed]].<ref>{{Cita web|url=https://www.ncbi.nlm.nih.gov/|título=National Center for Biotechnology Information|fechaacceso=2021-11-20|apellido=Information|nombre=National Center for Biotechnology|sitioweb=www.ncbi.nlm.nih.gov|idioma=en}}</ref> Muchas de las aplicaciones web se complementan con implementaciones personalizadas del programa BLAST, optimizadas para la búsqueda de conjuntos de datos especializados. Los recursos incluyen la gestión de datos de PubMed, los elementos funcionales de RefSeq, la descarga de datos del genoma, la API de servicios de variación, Magic-BLAST, QuickBLASTp y los grupos de proteínas idénticas. Se puede acceder a todos estos recursos a través del NCBI. <ref><nowiki>{cite journal | vauthors = Agarwala R, Barrett T, Beck J, Benson DA, Bollin C, Bolton E, etal | collaboration = NCBI Resource Coordinators | title = Database resources of the National Center for Biotechnology Information | journal = Nucleic Acids Research | volume = 46 | issue = D1 | pages = D8-D13 | date = January 2018 | pmid = 29140470 | pmc = 5753372 | doi = 10.1093/nar/gkx1095 }}</nowiki></ref> |
|||
| ⚫ | |||
{{Listaref}} |
|||
* antiSMASH: Permite la rápida identificación, anotación y análisis de grupos de genes de biosíntesis de metabolitos secundarios en genomas bacterianos y fúngicos. Se integra y cruza con un gran número de herramientas de análisis in silico de [[metabolitos secundarios]].<ref>{{cite web|url=https://antismash-db.secondarymetabolites.org/|title=antiSMASH database|website=antismash-db.secondarymetabolites.org}}</ref> |
|||
{{Control de autoridades}} |
|||
* gutSMASH: Evalúa sistemáticamente el potencial metabólico bacteriano mediante la predicción de clústeres de genes metabólicos (MGC), tanto conocidos como novedosos, del [[microbioma]] intestinal. |
|||
* MIBiG: Son las siglas de Minimal Identifier for Biosintetic Gene clusters,<ref>{Cite web|title=MIBiG: Información mínima sobre un clúster de genes biosintéticos|url=https://mibig.secondarymetabolites.org/%7Caccess-date=2021-07-30%7Cwebsite=mibig.secondarymetabolites.org%7Clanguage=en}}</ref> y proporciona un estándar para las anotaciones y [[metadatos]] sobre los clústeres de genes biosintéticos y sus productos moleculares.<ref>{{cite journal|title=MIBiG 2. 0: a repository for biosynthetic gene clusters of known function|date=January 2020|journal=Nucleic Acids Research|volume=48|issue=D1|pages=D454-D458|doi=10.1093/nar/gkz882|pmc=7145714|pmid=31612915|display-authors=6|vauthors=Kautsar SA, Blin K, Shaw S, Navarro-Muñoz JC, Terlouw BR, van der Hooft JJ, van Santen JA, Tracanna V, Suarez Duran HG, Pascal Andreu V, Selem-Mojica N, Alanjary M, Robinson SL, Lund G, Epstein SC, Sisto AC, Charkoudian LK, Collemare J, Linington RG, Weber T, Medema MH}}</ref> Se trata de un proyecto del [[Consorcio de Estándares Genómicos]] que se basa en el marco de información mínima sobre cualquier secuencia (MIxS).<ref>[https://mibig.secondarymetabolites.org/ MiBiG ]</ref> |
|||
* SILVA: es un proyecto interdisciplinario entre biólogos e informáticos que reúne una base de datos completa de secuencias de genes de ARN ribosómico (ARNr), tanto de las subunidades pequeñas ([[ARN ribosomal 16S|16S]],18S, SSU) como de las grandes ([[23S]], 28S, LSU),<ref>{Cite web|title=Silva|url=https://www.arb-silva.de/%7Caccess-date=2021-07-30%7Cwebsite=www.arb-silva<nowiki>. de}}</nowiki></ref> que pertenecen a los dominios de las bacterias, las arqueas y las eucariotas. Estos datos están disponibles gratuitamente para uso académico y comercial. <ref><nowiki>{cite journal | vauthors = Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Glöckner FO | display-authors = 6 | title = The SILVA ribosomal RNA gene database project: procesamiento de datos mejorado y herramientas basadas en la web | journal = Nucleic Acids Research | volume = 41 | issue = Database issue | pages = D590-6 | date = January 2013 | pmid = 23193283 | pmc = 3531112 | doi = 10. 1093/nar/gks1219 }}</nowiki></ref> |
|||
* Greengenes: es una base de datos de genes de [[ARN ribosomal 16S]] de longitud completa que proporciona cribado de quimeras,<ref>{{Cita web|url=https://greengenes.secondgenome.com/|título=greengenes.secondgenome.com|fechaacceso=2021-11-20|sitioweb=greengenes.secondgenome.com}}</ref> alineación estándar y una taxonomía curada basada en la inferencia de árboles de novo. <ref><nowiki>{cite journal | vauthors = DeSantis TZ, Hugenholtz P, Larsen N, Rojas M, Brodie EL, Keller K, Huber T, Dalevi D, Hu P, Andersen GL | display-authors = 6 | title = Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB | journal = Applied and Environmental Microbiology | volume = 72 | issue = 7 | pages = 5069-72 | date = July 2006 | pmid = 16820507 | pmc = 1489311 | doi = 10. 1128/AEM.03006-05 | Código de barras = 2006ApEnM..72. 5069D | doi-access = free }}</nowiki></ref><ref><nowiki>{cite journal | vauthors = McDonald D, Price MN, Goodrich J, Nawrocki EP, DeSantis TZ, Probst A, Andersen GL, Knight R, Hugenholtz P | display-authors = 6 | title = Una taxonomía Greengenes mejorada con rangos explícitos para los análisis ecológicos y evolutivos de bacterias y arqueas | journal = The ISME Journal | volume = 6 | issue = 3 | pages = 610-8 | date = March 2012 | pmid = 22134646 | pmc = 3280142 | doi = 10. 1038/ismej.2011 .139 }}</nowiki></ref> |
|||
* Open Tree of life Taxonomy: Tiene como objetivo construir un [[Árbol de la vida (biología)|Árbol de la Vida]] completo, dinámico y disponible digitalmente, sintetizando los árboles filogenéticos publicados junto con los datos taxonómicos. Aunque menos utilizada, tiene un mayor número de secuencias clasificadas taxonómicamente hasta el nivel de género en comparación con SILVA y Greengenes.<ref>{{cite journal|title=Síntesis de la filogenia y la taxonomía en un árbol completo de la vida|date=October 2015|journal=Proceedings of the National Academy of Sciences of the United States of America|volume=112|issue=41|pages=12764-9|bibcode=2015PNAS..11212764H|doi=10. 1073/pnas.1423041112|pmc=4611642|pmid=26385966|display-authors=6|vauthors=Hinchliff CE, Smith SA, Allman JF, Burleigh JG, Chaudhary R, Coghill LM, Crandall KA, Deng J, Drew BT, Gazis R, Gude K, Hibbett DS, Katz LA, Laughinghouse HD, McTavish EJ, Midford PE, Owen CL, Ree RH, Rees JA, Soltis DE, Williams T, Cranston KA|doi-access=free}}</ref> |
|||
* Ribosomal Database Project: Es una base de datos que proporciona secuencias de ARN ribosómico (ARNr) de subunidades pequeñas de dominio bacteriano y arqueológico (16S); y secuencias de ARNr fúngico de subunidades grandes (28S). <ref>{{cite journal|title=Ribosomal Database Project: Datos y herramientas para el análisis de ARNr de alto rendimiento|fecha=enero de 2014|volumen=42|número=Base de datos|páginas=D633-42|doi=10. 1093/nar/gkt1244|pmc=3965039|pmid=24288368|display-authors=6|vauthors=Cole JR, Wang Q, Fish JA, Chai B, McGarrell DM, Sun Y, Brown CT, Porras-Alfaro A, Kuske CR, Tiedje JM|revista=Nucleic Acids Research}}</ref> |
|||
| ⚫ | |||
{{Listaref}}{{Traducido ref|en|Machine learning in bioinformatics|oldid=1050209319}}{{Control de autoridades}} |
|||
[[Categoría:Bioinformática]] |
[[Categoría:Bioinformática]] |
||
[[Categoría:Aprendizaje automático]] |
[[Categoría:Aprendizaje automático]] |
||
Revisión del 19:19 20 nov 2021

El aprendizaje automático en bioinformática consiste en la aplicación de algoritmos de aprendizaje automático, en entornos de bioinformática, como, por ejemplo, la genómica, la proteómica, los microarrays, la biología de sistemas, la biología evolutiva y la minería de textos.[2] Esto permite automatizar la búsqueda de patrones complejos en series de datos, facilitando la comprensión de procesos biológicos tan complejos como la estructura de las proteínas,[3] lo que diferencia a esta disciplina de los enfoques tradicionales de bioinformática, que requieren supervisión y que dificultan la aparición de patrones inesperados u ocultos. [4]
Se trata de un campo emergente debido al gran crecimiento de la cantidad de conjuntos de datos biológicos disponibles, necesarios para entrenar algoritmos con un nivel de detalle alto.[5]
Métodos de clasificación y predicción
- La clasificación y la predicción son las tareas más utilizadas en el análisis de datos.
- Los métodos para lograr esta tarea son variados y abarcan muchas disciplinas, siendo las más conocidas el aprendizaje automático y la estadística.
- La clasificación predice una clase valorada categóricamente.
- La predicción predice una característica con valor numérico.
- Las tareas de clasificación y predicción tienen como objetivo la construcción de modelos que describen y distinguen clases o conceptos para predicciones futuras.
- Lo que diferencia entre los métodos es el tipo de algoritmo o proceso utilizado para construir los modelos predictivos a partir de datos, ya sea que se basen en analogías, reglas, redes neuronales, probabilidades o estadísticas.
Aplicaciones
Genómica
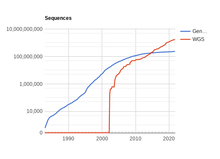
Aunque los datos de genómica han sido históricamente escasos debido a la dificultad técnica de secuenciar un fragmento de ADN, en los últimos años el número de secuencias indexadas en bases de datos está creciendo exponencialmente.[6] Sin embargo, la interpretación de estos datos está ocurriendo a un ritmo mucho más lento,[7] por lo que existe una necesidad creciente de desarollar sistemas que puedan determinar automáticamente qué partes de una secuencia de ADN dada codifican proteínas y cuáles no: se trata de la predicción de gen.
Esta predicción se puede realizar mediante búsqueda extrínseca e intrínseca:
- En la extrínseca, una secuencia de ADN se pasa por una base de datos de secuencias anotadas, buscando homólogos a genes ya existentes
- En la intrínseca, se buscan regiones codificantes en la cadena "por sí misma", sin ayuda de agentes externos.
El aprendizaje automático también puede usarse para la alineación de secuencias, que junta regiones de semejanza para detectar una historia evolutiva compartida, así como regiones recombinantes.[8]
Proteómica
Las proteínas son cadenas de aminoácidos cuya funcionalidad depende en gran medida de su estructura tridimensional. Esta estructura se va "plegando" progresivamente, pasando desde la estructura primaria (la cadena lineal de aminoácidos) a la estructura secundaria que predice la terciaria, y si se asocia con otras cadenas peptídicas, la cuaternaria. Así, la predicción de la estructura secundaria directamente desde la primaria es un campo con grandes aplicaciones en el área de la farmacodinámica, aunque se trata de un proceso increíblemente costoso y que requiere mucho tiempo, y que, hasta la llegada del aprendizaje automático, debía realizarse manualmente.[9]
Hoy en día, mediante el uso del aprendizaje automático se puede alcanzar una precisión del 82-84%, usando redes neuronales artificiales para clasificar regiones del ADN en función de si codifican para una hélice alfa, lámina beta o espiral).[10] El aprendizaje automático también se puede usar para la predicción de cadenas laterales, el modelado de bucles de proteínas, y la generación de mapas de contacto.
Microarrays
Los microarrays son un tipo de chip de laboratorio utilizado para recopilar datos de forma automática sobre grandes cantidades de material biológico. Esta tecnología permite controlar la expresión diferencial de genes, ayudando a diagnosticar enfermedades como el cáncer.[11] Sin embargo, debido a la enorme cantidad de datos recopilados, se hace necesario discernir cuales son de interés y cuales no para el diagnóstico, para lo que se pueden usar métodos de aprendizaje automático tales como el aprendizaje profundo o los árboles de decisión.
Biología de sistemas
La biología de sistemas se centra en el estudio de los comportamientos emergentes a partir de interacciones complejas de componentes biológicos simples en un sistema. Dichos componentes pueden incluir moléculas como ADN, ARN, proteínas y metabolitos.[12]
El aprendizaje automático se ha utilizado para ayudar a modelar estas interacciones complejas en sistemas biológicos en dominios tales como redes genéticas, redes de transducción de señales y vías metabólicas. Los modelos gráficos probabilistas, una técnica de aprendizaje automático para determinar la estructura entre diferentes variables, son uno de los métodos más utilizados para modelar redes genéticas. Además, el aprendizaje automático se ha aplicado a problemas de biología de sistemas tales como la identificación de sitios de unión del factor de transcripción utilizando una técnica conocida como optimización de cadena de Markov. Los algoritmos genéticos, técnicas de aprendizaje automático que se basan en el proceso natural de la evolución, se han utilizado para modelar redes genéticas y estructuras reguladoras.
Otras aplicaciones de biología de sistemas de aprendizaje automático incluyen la tarea de predicción de función enzimática, análisis de datos de micromatriz de alto rendimiento, análisis de estudios de asociación de genoma completo para comprender mejor los marcadores de esclerosis múltiple, predicción de función de proteína e identificación de sensibilidad de genes NCR en levadura.[13]
Decodificación de estructuras químicas de RiPPs
El aumento acelerado de péptidos ribosomales sintetizados y modificados post-traduccionalmente (RiPPs) que se han logrado caracterizar experimentalmente, en conjunto con la disponibilidad de información de la secuencia y estructura química de un gran número de ellos, seleccionados de bases de datos como BAGEL, BACTIBASE, MIBIG y THIOBASE brinda la oportunidad de desarrollar herramientas de aprendizaje automático para descifrar la estructura química de los RiPPs y lograr una clasificación entre ellos.
En 2017, Priyesh Agrawal, Shradha Khater, Money Gupta, Neetu Sain y Debasisa Mohanty del Instituto Nacional de Inmunología "Aruna Asaf Ali Marg" de Nueva Deli, India, desarrollan el software RiPPMiner,[14] un recurso bioinformático para descifrar estructuras químicas de RiPPs mediante minería del genoma. El servidor web RiPPMiner consta de dos componentes principales, su interfaz de consulta y la base de datos RiPPDB. RiPPMiner logra la clasificación en 12 subclases de RiPPs prediciendo el sitio de división del péptido líder y el enlace de cruzamiento final de la estructura química del RiPP. Las herramientas de análisis de RiPPs como antiSMASH y RiPP-PRISM usan Modelos de Markov Ocultos de enzimas modificadoras presentes en los clústeres de genes biosintéticos en el RiPP para predecir la subclase RiPP. A diferencia de estas herramientas, RiPPMiner utiliza un modelo de aprendizaje automático entrenado con 513 RiPPs que usa la secuencia de aminoácidos del gen RiPP únicamente para identificar RiPPs y posteriormente predecir su subclase RiPP. RiPPMiner primero distingue RiPPs de otras proteínas y péptidos usando un modelo de máquina de soporte vectorial que está entrenado usando 293 RiPPs caracterizados experimentalmente como conjunto de datos positivo y 8140 polipéptidos no RiPPs codificados en el genoma como conjunto de datos negativos. El conjunto de datos negativo incluyó entradas de SWISSProt con una longitud similar a RiPPs, por ejemplo, proteínas ribosómicas de 30s, proteínas de matriz y proteínas de citocromo b, etc. Los vectores soporte del modelo de máquina de soporte vectorial consisten en la composición de aminoácidos y frecuencias dipéptidas.
La evaluación comparativa de estos métodos de identificación de RiPPs en un conjunto de datos no incluido en el conjunto de entrenamiento utilizando un enfoque de validación cruzada doble indicó los siguientes valores:
| Sensibilidad | 0.93 |
| Especificidad | 0.90 |
| Precisión | 0.90 |
| Coeficiente de correlación de Matthews | 0.85 |
Estos valores indican un buen poder predictivo del modelo de máquina de soporte vectorial de RiPPMiner para distinguir entre los RiPPs y los no RiPPs. Para la predicción de la subclase RiPP, una máquina de soporte vectorial multiclase usó la composición de aminoácidos y frecuencias dipéptidas como vectores soporte del conjunto de entrenamiento. Las secuencias precursoras RiPP disponibles pertenecientes a una clase determinada, por ejemplo, el péptido de lazo, se usan como conjunto de datos positivo, mientras que RiPPs pertenecientes a las subclases restantes se usan como conjunto de datos negativo.
RiPPMiner permite la visualización de la estructura química de un RiPP seleccionado de su base de datos, además de otras características del péptido líder, péptido núcleo residuos modificados, enlaces cruzados y genes vecinos en el clúster de genes del RiPP.
Minería de textos
El aumento de las publicaciones científicas dificulta la búsqueda y compilación de toda la información relevante disponible sobre un tema determinado. Esto, a su vez, dificulta la recopilación de datos biológicos necesaria para todas las aplicaciones anteriormente mencionadas.[15] El procesamiento del lenguaje natural permite extraer la información útil dentro de los informes generados por humanos en una base de datos dada. Además, como las anotaciones de proteínas en las bases de datos de proteínas a menudo no reflejan el conjunto de conocimiento conocido, se puede extraer información adicional de la literatura biomédica, permitiendo funciones como la anotación automática, la determinación de la localización subcelular de una proteína, el análisis de interacción de proteínas a gran escala o la detección y visualización de regiones que comparten un alto grado de semejanza.[16]
Bases de datos
Una parte importante de la bioinformática es la gestión de grandes conjuntos de datos, conocidos como bases de datos de referencia. Existen bases de datos para cada tipo de datos biológicos, por ejemplo para los grupos de genes biosintéticos y los metagenomas.
- Centro Nacional de Información Biotecnológica: El NCBI ofrece un amplio conjunto de recursos en línea de información y datos biológicos, como la base de datos de secuencias de ácidos nucleicos GenBank y la base de datos de citas y resúmenes de revistas de ciencias de la vida PubMed.[17] Muchas de las aplicaciones web se complementan con implementaciones personalizadas del programa BLAST, optimizadas para la búsqueda de conjuntos de datos especializados. Los recursos incluyen la gestión de datos de PubMed, los elementos funcionales de RefSeq, la descarga de datos del genoma, la API de servicios de variación, Magic-BLAST, QuickBLASTp y los grupos de proteínas idénticas. Se puede acceder a todos estos recursos a través del NCBI. [18]
- antiSMASH: Permite la rápida identificación, anotación y análisis de grupos de genes de biosíntesis de metabolitos secundarios en genomas bacterianos y fúngicos. Se integra y cruza con un gran número de herramientas de análisis in silico de metabolitos secundarios.[19]
- gutSMASH: Evalúa sistemáticamente el potencial metabólico bacteriano mediante la predicción de clústeres de genes metabólicos (MGC), tanto conocidos como novedosos, del microbioma intestinal.
- MIBiG: Son las siglas de Minimal Identifier for Biosintetic Gene clusters,[20] y proporciona un estándar para las anotaciones y metadatos sobre los clústeres de genes biosintéticos y sus productos moleculares.[21] Se trata de un proyecto del Consorcio de Estándares Genómicos que se basa en el marco de información mínima sobre cualquier secuencia (MIxS).[22]
- SILVA: es un proyecto interdisciplinario entre biólogos e informáticos que reúne una base de datos completa de secuencias de genes de ARN ribosómico (ARNr), tanto de las subunidades pequeñas (16S,18S, SSU) como de las grandes (23S, 28S, LSU),[23] que pertenecen a los dominios de las bacterias, las arqueas y las eucariotas. Estos datos están disponibles gratuitamente para uso académico y comercial. [24]
- Greengenes: es una base de datos de genes de ARN ribosomal 16S de longitud completa que proporciona cribado de quimeras,[25] alineación estándar y una taxonomía curada basada en la inferencia de árboles de novo. [26][27]
- Open Tree of life Taxonomy: Tiene como objetivo construir un Árbol de la Vida completo, dinámico y disponible digitalmente, sintetizando los árboles filogenéticos publicados junto con los datos taxonómicos. Aunque menos utilizada, tiene un mayor número de secuencias clasificadas taxonómicamente hasta el nivel de género en comparación con SILVA y Greengenes.[28]
- Ribosomal Database Project: Es una base de datos que proporciona secuencias de ARN ribosómico (ARNr) de subunidades pequeñas de dominio bacteriano y arqueológico (16S); y secuencias de ARNr fúngico de subunidades grandes (28S). [29]
Referencias
- ↑ Farrell, Daniel P.; Anishchenko, Ivan; Shakeel, Shabih; Lauko, Anna; Passmore, Lori A.; Baker, David; DiMaio, Frank (3 de mayo de 2020). Deep learning enables the atomic structure determination of the Fanconi Anemia core complex from cryoEM (en inglés). pp. 2020.05.01.072751. doi:10.1101/2020.05.01.072751v1. Consultado el 11 de noviembre de 2021.
- ↑ «Machine learning in bioinformatics». academic.oup.com. Consultado el 11 de noviembre de 2021.
- ↑ Yang, Yuedong; Gao, Jianzhao; Wang, Jihua; Heffernan, Rhys; Hanson, Jack; Paliwal, Kuldip; Zhou, Yaoqi (31 de diciembre de 2016). «Sixty-five years of the long march in protein secondary structure prediction: the final stretch?». Briefings in Bioinformatics: bbw129. doi:10.1093/bib/bbw129.
- ↑ Chicco, Davide (2017). «Ten quick tips for machine learning in computational biology». BioData Mining 10 (1): 35. doi:10.1186/s13040-017-0155-3.
- ↑ Larrañaga, Pedro; Calvo, Borja; Santana, Roberto; Bielza, Concha; Galdiano, Josu; Inza, Iñaki; Lozano, José A.; Armañanzas, Rubén; Santafé, Guzmán; Pérez, Aritz; Robles, Victor (1 de marzo de 2006). «Machine learning in bioinformatics». Briefings in Bioinformatics 7 (1): 86-112. doi:10.1093/bib/bbk007.
- ↑ «GenBank and WGS Statistics». www.ncbi.nlm.nih.gov. Consultado el 19 de noviembre de 2021.
- ↑ Mathé, Catherine; Sagot, Marie-France; Schiex, Thomas; Rouzé, Pierre (1 de octubre de 2002). «SURVEY AND SUMMARY: Current methods of gene prediction, their strengths and weaknesses». Nucleic Acids Research 30 (19): 4103-4117. ISSN 0305-1048. PMID 12364589. Consultado el 19 de noviembre de 2021.
- ↑ Pratas, D; Silva, R; Pinho, A; Ferreira, P (18 de mayo de 2015). «An alignment-free method to find and visualise rearrangements between pairs of DNA sequences.». Scientific Reports (Group Nature) 5 (10203): 10203. PMC 4434998. PMID 25984837. doi:10.1038/srep10203.
- ↑ Pauling, L.; Corey, R. B.; Branson, H. R. (1 de abril de 1951). «The structure of proteins; two hydrogen-bonded helical configurations of the polypeptide chain». Proceedings of the National Academy of Sciences of the United States of America 37 (4): 205-211. ISSN 0027-8424. PMC 1063337. PMID 14816373. doi:10.1073/pnas.37.4.205.
- ↑ Wang, Sheng; Peng, Jian; Ma, Jianzhu; Xu, Jinbo (1 de diciembre de 2015). «Protein secondary structure prediction using deep convolutional neural fields». .
- ↑ Pirooznia, Mehdi; Yang, Jack Y.; Yang, Mary Qu; Deng, Youping (2008). «A comparative study of different machine learning methods on microarray gene expression data». BMC Genomics 9 (1): S13. ISSN 1471-2164. PMC 2386055. PMID 18366602. doi:10.1186/1471-2164-9-S1-S13.
- ↑ «Machine Learning in Molecular Systems Biology». Frontiers (en inglés). Consultado el 9 de junio de 2017.
- ↑ «Machine Learning in Systems Biology». BMC Proceedings 2 (4): S1. 2008. ISSN 1753-6561. doi:10.1186/1753-6561-2-S4-S1.
- ↑ Agrawal, Priyesh; Khater, Shradha; Gupta, Money; Sain, Neetu; Mohanty,, Debasisa (12 de mayo de 2017). «RiPPMiner: a bioinformatics resource for deciphering chemical structures of RiPPs based on prediction of clevage and cross-links». Nucleic Acids Research 45 (W1): W80-W88. PMID 28499008. doi:10.1093/nar/gkx408. Consultado el 11 de mayo de 2021.
- ↑ Krallinger, Martin; Erhardt, Ramon Alonso-Allende; Valencia, Alfonso (15 de marzo de 2005). «Text-mining approaches in molecular biology and biomedicine». Drug Discovery Today 10 (6): 439-445. doi:10.1016/S1359-6446(05)03376-3.
- ↑ «Visualization of Distinct DNA Regions of the Modern Human Relatively to a Neanderthal Genome». Iberian Conference on Pattern Recognition and Image Analysis. Springer: 235-242. June 20–23, 2017.
- ↑ Information, National Center for Biotechnology. «National Center for Biotechnology Information». www.ncbi.nlm.nih.gov (en inglés). Consultado el 20 de noviembre de 2021.
- ↑ {cite journal | vauthors = Agarwala R, Barrett T, Beck J, Benson DA, Bollin C, Bolton E, etal | collaboration = NCBI Resource Coordinators | title = Database resources of the National Center for Biotechnology Information | journal = Nucleic Acids Research | volume = 46 | issue = D1 | pages = D8-D13 | date = January 2018 | pmid = 29140470 | pmc = 5753372 | doi = 10.1093/nar/gkx1095 }}
- ↑ «antiSMASH database». antismash-db.secondarymetabolites.org.
- ↑ {Cite web|title=MIBiG: Información mínima sobre un clúster de genes biosintéticos|url=https://mibig.secondarymetabolites.org/%7Caccess-date=2021-07-30%7Cwebsite=mibig.secondarymetabolites.org%7Clanguage=en}}
- ↑ «MIBiG 2. 0: a repository for biosynthetic gene clusters of known function». Nucleic Acids Research 48 (D1): D454-D458. January 2020. PMC 7145714. PMID 31612915. doi:10.1093/nar/gkz882. Parámetro desconocido
|vauthors=ignorado (ayuda) - ↑ MiBiG
- ↑ {Cite web|title=Silva|url=https://www.arb-silva.de/%7Caccess-date=2021-07-30%7Cwebsite=www.arb-silva. de}}
- ↑ {cite journal | vauthors = Quast C, Pruesse E, Yilmaz P, Gerken J, Schweer T, Yarza P, Peplies J, Glöckner FO | display-authors = 6 | title = The SILVA ribosomal RNA gene database project: procesamiento de datos mejorado y herramientas basadas en la web | journal = Nucleic Acids Research | volume = 41 | issue = Database issue | pages = D590-6 | date = January 2013 | pmid = 23193283 | pmc = 3531112 | doi = 10. 1093/nar/gks1219 }}
- ↑ «greengenes.secondgenome.com». greengenes.secondgenome.com. Consultado el 20 de noviembre de 2021.
- ↑ {cite journal | vauthors = DeSantis TZ, Hugenholtz P, Larsen N, Rojas M, Brodie EL, Keller K, Huber T, Dalevi D, Hu P, Andersen GL | display-authors = 6 | title = Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB | journal = Applied and Environmental Microbiology | volume = 72 | issue = 7 | pages = 5069-72 | date = July 2006 | pmid = 16820507 | pmc = 1489311 | doi = 10. 1128/AEM.03006-05 | Código de barras = 2006ApEnM..72. 5069D | doi-access = free }}
- ↑ {cite journal | vauthors = McDonald D, Price MN, Goodrich J, Nawrocki EP, DeSantis TZ, Probst A, Andersen GL, Knight R, Hugenholtz P | display-authors = 6 | title = Una taxonomía Greengenes mejorada con rangos explícitos para los análisis ecológicos y evolutivos de bacterias y arqueas | journal = The ISME Journal | volume = 6 | issue = 3 | pages = 610-8 | date = March 2012 | pmid = 22134646 | pmc = 3280142 | doi = 10. 1038/ismej.2011 .139 }}
- ↑ «Síntesis de la filogenia y la taxonomía en un árbol completo de la vida». Proceedings of the National Academy of Sciences of the United States of America 112 (41): 12764-9. October 2015. Bibcode:2015PNAS..11212764H. PMC 4611642. PMID 26385966. doi:10. 1073/pnas.1423041112
|doi=incorrecto (ayuda). Parámetro desconocido|vauthors=ignorado (ayuda); Parámetro desconocido|doi-access=ignorado (ayuda) - ↑ «Ribosomal Database Project: Datos y herramientas para el análisis de ARNr de alto rendimiento». Nucleic Acids Research 42 (Base de datos): D633-42. enero de 2014. PMC 3965039. PMID 24288368. doi:10. 1093/nar/gkt1244
|doi=incorrecto (ayuda). Parámetro desconocido|vauthors=ignorado (ayuda)
- Esta obra contiene una traducción derivada de «Machine learning in bioinformatics» de Wikipedia en inglés, concretamente de esta versión, publicada por sus editores bajo la Licencia de documentación libre de GNU y la Licencia Creative Commons Atribución-CompartirIgual 4.0 Internacional.