Diferencia entre revisiones de «PCR en tiempo real»
Retiro {{enobras}} |
m →Rectas de calibrado: +imagen |
||
| Línea 58: | Línea 58: | ||
;Gráfica de la derecha (B) |
;Gráfica de la derecha (B) |
||
[[Image:Log qPCR Mason2002.jpg|thumb|Lectura del logaritmo de la fluroscencia respecto del número de ciclo (arriba) y recta de calibrado (abajo).<ref name=Mason2002>{{citation | last1 = Mason | first1 = G. | last2 = Provero | first2 = P. | last3 = Vaira | first3 = A.M. | last4 = Accotto | first4 = G.P. | year = 2002 | journal = BMC Biotechnology | volume = 2 | issue = 20 | pages = 203–212 | doi = 10.1186/1472-6750-2-20 | url = http://www.biomedcentral.com/1472-6750/2/20/}}</ref>]] |
|||
Los valores CT medios en función de la cantidad de ADN inicial de todas las réplicas pueden representarse en un gráfico semilogarítmico. De este modo, la recta de calibrado para estos valores medios puede definirse gracias a una [[regresión lineal]] con un determinado [[coeficiente de correlación]] (r<sup>2</sup>) que, para ser considerado de calidd, debe poseer un valor de 0,9999. No obstante, han de representarse los errores, generalmente definidos según el [[rango (estadística)|rango]] de valores obtenidos para un punto. |
Los valores CT medios en función de la cantidad de ADN inicial de todas las réplicas pueden representarse en un gráfico semilogarítmico. De este modo, la recta de calibrado para estos valores medios puede definirse gracias a una [[regresión lineal]] con un determinado [[coeficiente de correlación]] (r<sup>2</sup>) que, para ser considerado de calidd, debe poseer un valor de 0,9999. No obstante, han de representarse los errores, generalmente definidos según el [[rango (estadística)|rango]] de valores obtenidos para un punto. |
||
Revisión del 12:06 20 dic 2008
PCR cuantitativa, PCR en tiempo real o qPCR (del inglés quatitative polimerase chain reaction) es una variante de la reacción en cadena de la polimerasa (PCR) en la cual se cuantifica de forma absoluta o relativa el producto de la amplificación de ácido desoxirribonucleico (ADN). Para ello emplea, del mismo modo que la PCR convencional, un molde de ADN, al menos un par de cebadores específicos, dNTPs, un tampón de reacción adecuado, y una ADN polimerasa termoestable; a dicha mezcla se le adiciona una sustancia marcada con un fluorocromo que, en un termociclador que albergue sensores para medir fluorescencia tras excitar el fluorocromo a la longitud de onda apropiada, permita medir la tasa de generación de uno o más productos específicos.[1] En muchos casos el molde que se emplea para la PCR cuantitativa no es desde el principio ADN, sino que puede ser ADN complementario (ADNc), de hebra simple, obtenido por retrotranscripción de ácido ribonucleico (ARN); en este caso, la técnica es una RT-PCR cuantitativa.
La PCR cuantitativa es la metodología más moderna para el estudio de la expresión génica, junto a los chip de ADN, si bien otros métodos tradicionales, como el Northern blot, también permiten su abordaje.[2] La variabilidad que se introduce en la cuantificación y que conlleva a un error en la estima deriva de la integridad de ADN, eficiencia enzimática y otros muchos factores, por lo que se han desarrollado numerosos sistemas de estandarización. Los hay para cuantificar de forma absoluta la expresión génica, pero, de forma más común, se orientan a la cuantificación relativa del gen de estudio respecto de otro, denominado «normalizador», que se selecciona debido a su expresión casi constante; estos genes suelen denominarse, en inglés, house-keeping genes debido a que suelen estar involucrados en funciones básicas en la supervivencia celular, lo cual suele implicar una expresión constitutiva.[3][4] De este modo, efectuando en cada experimento la medición de los genes de interés y dividiéndolos por la expresión del gen normalizador seleccionado es posible comparar los primeros aun sin conocer en términos absolutos su nivel de expresión. Los genes normalizadores más empleados son: tubulinas, gliceraldehído-3-fosfato deshidrogenasa, albúmina, ciclofilina, RNA ribosomales, etc.[2]
Clasificación
Podemos clasificar las técnicas de PCR cuantitativa según el empleo de fluorocromos no específicos o bien de sondas moleculares dependientes de la secuencia.
- En las técnicas basadas en fluorocromos inespecíficos se detecta la generación exponencial de ADN de doble cadena empleando un fluorocromo que se une inespecíficamente a aquél. Un ejemplo de colorante que permite esta detección es el SYBR Green), que, excitado mediante luz azul (λmax = 488 nm) emite luz verde (λmax = 522 nm).[5] Posee la ventaja de requerir sólo un par de cebadores para efectuar la amplificación, lo que abarata su coste; sin embargo, sólo es posible amplificar un producto en cada reacción.
- Las técnicas basadas en sondas específicas utilizan al menos un oligonucleótido marcado fluorescentemente. Típicamente esta sonda está unida a dos fluorocromos e hibrida en la zona intermedia entre el cebador directo (forward) y el inverso (reverse); esto es, en el amplicón. De este modo, cuando la sonda está intacta, presentan una transferencia energética de fluorescencia por resonancia (FRET). Dicha FRET no se produce cuando los dos fluorocromos están distantes debido a la degradación de la sonda mediante la actividad 5'-3' exonucleasa de la ADN polimerasa, o bien debido a la separación física de los fluorocromos por un cambio en la conformación de la sonda. Esto permite monitorizar el cambio del patrón de fluorescencia y deducir el nivel de amplificación del gen.
Fundamento
La PCR cuantitativa se realiza en un termociclador con capacidad de hacer incidir sobre cada muestra un haz de luz de una longitud de onda determinada y de detectar la fluorescencia emitida por el fluorocromo excitado. Este termociclador es un aparato con capacidad para calentar y enfriar rápidamente las muestras, de modo que se aprovechen las cualidades fisicoquímicas de los ácidos nucleicos y las enzimáticas de la ADN polimerasa.
El proceso de PCR por lo general consiste en una serie de 25 a 40 cambios repetidos de temperatura llamados ciclos, donde cada ciclo posee un mínimo de tres etapas a distinta temperatura: el primero, en torno a los 95 °C, permite la separación de los ácidos nucleicos de doble cadena; el segundo, a una temperatura en torno a los 50-60 °C, permite el alineamiento de los cebadores al ADN molde;[6] el tercero, a 68 ó 72 °C, facilita la polimerización por parte de la ADN polimerasa. Debido al pequeño tamaño de los fragmentos amplificados usualmente en este tipo de PCR puede omitirse el último paso, pues la enzima es capaz de amplificar durante la rampa entre la temperatura de alineamiento y la de desnaturalización. Además, algunos termocicladores añaden a cada ciclo unos segundos a otra temperatura, por ejemplo 80 °C, a fin de reducir el ruido por la presencia de dímeros de cebadores cuando se emplea un colorante inespecífico. Las temperaturas usadas y el tiempo aplicado en cada ciclo dependen de gran variedad de parámetros, como: la enzima usada para la síntesis de ADN, la concentración de iones divalentes y desoxirribonucleótidos (dNTPs) en la reacción y la temperatura de unión de los cebadores.[7]
Análisis de temperatura de fusión

La qPCR permite, empleando un fluorocromo de unión inespecífica a la doble hebra de ADN, generalmente SYBR Green,identificar fragmentos amplificados de DNA concretos a partir de la temperatura de fusión (también denominado valor Tm, del inglés melting temperature), que es especifica para el fragmento amplificado que se esta buscando; y cuyos resultados son obtenidos a partir de la observación de la curva de disociación de las muestras de DNA analizadas. [9]
Ello permite, a diferencia de PCR convencional, prescindir del posterior empleo de técnicas de electroforesis para la visualización de los resultados de todas las muestras. Porque, pese a que la PCR cuantitativa es una técnica cinética, suele ser evaluada a punto final. Así esta técnica lleva a la obtención de resultados más rápidos, y/o en el menos gasto de reactivos empleados en las técnicas de electroforesis; si según el criterio del investigador, posteriormente es necesario correr en geles solo las muestras cuyo resultados previos en el PCR en tiempo real se pueden considerar dudosos y/o para ratificar resultados en muestras positivas.
Cuantificación de la expresión génica
La cuantificación puede realizarse en términos absolutos o relativos. En el primer caso, la estrategia es relacionar la señal de amplificación obtenida con el contenido en ADN empleando una curva de calibrado; para este enfoque es vital que la PCR de la muestra y de los elementos de la recta de calibrado posean una misma eficiencia de amplificación. En el segundo caso, se expresa el cambio en los niveles de expresión de ARN mensajero (ARNm) interpretado como ADN complementario (ADNc, generado por retrotranscripción del ARNm); esta cuantificación relativa es más fácil de realizar, puesto que no requiere curva de calibrado, y se sustenta en la comparación entre el nivel de expresión del gen a estudiar versus un gen control (también llamado de referencia, interno o normalizador o, en inglés, housekeeping gene.
Por tanto, en la cuantificación relativa es irrelevante en qué unidades se expresa la cuantificación, y sus resultados son comparables entre múltiples experimentos de RT-qPCR. De hecho, el propósito de emplear uno o más genes de normalización es corregir la variación no específica, como las diferencias en la cantidad y calidad del ARN empleado, que pueden afectar a las eficiencias de retrotranscripción y de PCR. No obstante, el aspecto crucial es que la estabilidad del gen de referencia sea una realidad.[10]
La selección de los genes internos se ha realizado clásicamente en Biología Molecular analizando la estabilidad de la expresión en estudios cualitativos o de baja sensiblidad, como el examen visual de geles de ARN, densitometría de Northern blots o qPCR semicuantitativa. En plena era de la genómica, es posible realizar una aproximación a gran escala empleando los chips de ADN para muchos organismos. [11] No obstante, se ha descrito que la mayoría de los genes empleados como normalizadores en la cuantificación de la expresión de ARN mensajero varían según las condiciones experimentales. [12][13][14] Por ello, es preciso realizar un estudio metodológico previo a emplear a fin de seleccionar, con ayuda de herramientas estadísticas, los más apropiados.
Se han desarrollado varios algoritmos estadísticos que detectan qué gen o genes son los más apropiados para la normalización de un conjunto de tejidos bajo unas condiciones dadas: algunos, como geNORM o BestKeeper, realizan, sobre una matriz de expresión de genes de referencia para diversos tejidos, comparaciones por pares y medias geométricas.[15][16]
Modelización
A diferencia de la PCR de punto final (PCR convencional), la PCR en tiempo real permite cuantificar el nivel de producto obtenido en cualquier momento de la amplificación mediante la señal de fluorescencia (en realidad, mediante su nivel sobre un umbral). Los valores de fluorescencia, expresados como logaritmos a fin de estudiar fácilmente la fase exponencial de amplificación, que aparece como una línea recta al representar gráficamente el logaritmo de la fluorescencia frente al número de ciclo; este segmento, denominado segmento cuantificable, permite valorar la cantidad de ADN inicial.
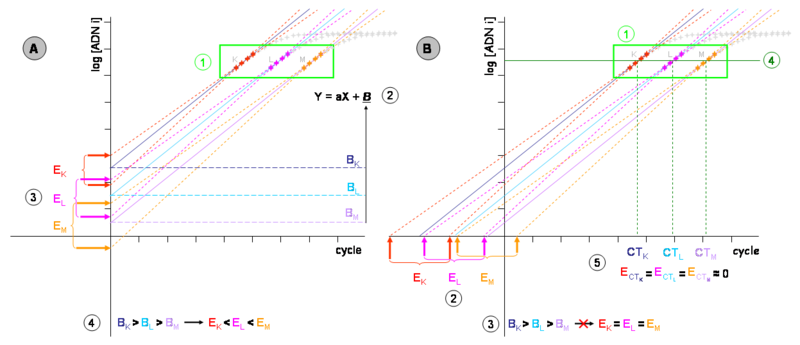
- Gráfica e la izquierda (A)
Las cinéticas de tres PCR de tres muestras distintas (K, L y M) de concentraciones de ADN inicial decrecientes (concretamente, se reducen a la décima parte cada vez) se representan en un gráfico del logaritmo de la fluorescencia (en ordenadas) frente al ciclo de PCR (en abscisas). No se representan las fases previas a la exponencial.
- Zona de segmentos cuantificables de cada muestra. En gris la zona de saturación, fuera ya de la zona lineal.
- Cada uno de los segmentos cuantificables permite definir una ecuación de la recta de tipo U=ax+b, que permite modelizar la eficiencia de la amplificación (la pendiente a) y, mediante la ordenada en el origen b, la cantidad de ADN en el ciclo 0, al menos teóricamente.
- Si bien, puesto que las medidas experimentales conllevan un error, dicha cuantificación arrastra un error estocástico. Si las muestras de concentraciones K, L y M fueran amplificadas durante más ciclos, se obtendrían cinéticas diferentes, si bien bastante próximas (ser representan sus segmentos cuantificables en rojo, rosa y naranja). Cada una de estas medidas permite establecer una nueva ecuación, representada en puntos del mismo color. Las ordenadas en el origen (b) diferentes son también medidas. Así, podemos evaluar el error o incertidumbre de la técnica evaluando las diferencias entre las pendientes b (EK, EL, EM).
- No obstante, dicha imprecisión en la medida varía para cada muestra (EK es inferior a EL, que es, a su vez, más pequeña que EM). Una proyección de la ecuación de la derecha sobre el eje de ordenadas o una de sus paralelas dará lugar a un «error dependiente de la concentración de ADN inicial».
- Gráfica de la derecha (B)
- Las ecuaciones obtenidas según los segmentos cuantificables pueden extrapolarse hacia el eje de abscisas, aunque se trate de un valor carente de significado bioquímico sino simplemente matemático.
- Los ángulos que modelizan el error experimental (líneas de puntos rojos, naranjas o rosas) permiten definir las nuevas incertidumbres EK, EL y EM. Estas incertidumbres son mayores que en la gráfica de la izquierda (gráfica A), si bien se mantiene su proporcionalidad. Por tanto, sobre la medida ahora generamos un «error independiente de la concentración de ADN inicial».
- Es posible proyectar rectas paralelas a la recta definida sobre una paralela al eje de abscisas de modo que se corten los segmentos cuantificables por la mitad. Este segmento es denominado «umbral de detección».
- Los valores en X (número de ciclos) de estas intersecciones son denominados «CT» (o «Ct», del inglés cycle threshold, cuya traducción es ciclo umbral), aunque también pueden llamarse «CP» (del inglés crossing point, o punto de cruce). Se trata, pues, de los valores matemáticos definidos sobre el espacio de reales positivos y no de los enteros positivos (aunque una fracción del ciclo no posea realidad experimental). Estos valores, inversamente proporcionales a la cantidad de ADN inicial, suelen poseer una incertidumbre sobre la medida mínima, en general inferior al 5%.
Rectas de calibrado
Como se indicaba en el apartado de cuantificación, emplear el CT, como valor matemático, permite obtener resultados fiables, pero este hecho puede no ser explotado directamente. A fin de conocer la cantidad de ADN inicial, es precioso pues relaizar nuevas transformaciones matemáticas que requieren conocer la eficiencia de PCR, que suele determinarse gracias a una recta de calibrado.

- Gráfica de la derecha (A)
Las nuevas muestras F, G, H, I, J, K, L, M y N, de concentración de ADN inicial decreciente (un orden de magnitud cada vez) son amplificadas mediante PCR en un mismo experimento. Cada cinética permite determinar un CT para cada uno (es decir, un número en referencia a un ciclo en concreto). Se representan las concentraciones de ADN como número de moléculas por tubo (en este caso, F posee 70 millones y N, 0,7).
Los rendimientos representados corresponden a «n» reacciones, cuya fiabilidad se debe a la existencia de réplicas independientes en la reacción, personal y reactivos. La fluorescencia se muestra en unidades arbitrarias y el ruido de fondo ha sido limpiado. Cabe destacar que la muestra Nn no ha amplificado en absoluto.
- Gráfica de la derecha (B)

Los valores CT medios en función de la cantidad de ADN inicial de todas las réplicas pueden representarse en un gráfico semilogarítmico. De este modo, la recta de calibrado para estos valores medios puede definirse gracias a una regresión lineal con un determinado coeficiente de correlación (r2) que, para ser considerado de calidd, debe poseer un valor de 0,9999. No obstante, han de representarse los errores, generalmente definidos según el rango de valores obtenidos para un punto.
Con estos datos, es posible estudiar:
- La fase «cuantitativa y detectable de la PCR». En la cual todas las muestras son detectables y se alinean en todos sus puntos con su recta particular. Generalmente, esta fase comprende las ocncentraciones de ADN iniciales en torno a 102 o 108 copias del ácido nucleico. Por debajo de estas cifras los fenómenos estocásticos alteran perceptiblemente los resultados, si bien es posible compensarlos con un alto número de réplicas en las medidas. Por encima de estos valores, el «ruido de fondo» es suficientemente bajo como para no poder ser determinado; en este caso, es posible compensar esta deficiencia mediante un protocolo de PCR con una eficiencia muy alta o, de forma más simple, diluyendo la muestra inicial.
- La fase «a veces detectable pero no cuantitativa de la PCR». Que comprende las concentraciones de ADN inicial entre una copia y la decena de copias. El porcentaje de muestras medidas, en el ejemplo de la gráfica, se indica mediante las concentraciones M y N, o sea 83% para una concentración media de siete copias y 28% cuando tres de los cuatro tubos contienen una copia (0,7 de concentración, o -0,15 en logaritmo). La dispersión de las medidas, y por ello el margen de error, se incrementan perceptiblemente. Nótese que un buen número de medidas M y N no se alinean con su recta particular, aunque no lo hagan por motivos de azar. Por tanto, sólo replicando más veces las medidas es posible determinar con precisión esta fase.
- La recta de calibrado, por tanto, representada en un gráfico semilogarítmico da lugar a una recta definida por la fórmula Y = aX + B donde:
- Y es el CT medido por el termociclador.
- La pendiente a deriva de la eficiencia de la PCR, y puede ser calculada mediante la ecuación
- Nótese que en el esquema se representa el caso más frecuente en el que la concentración está expresada en logaritmos decimales. Esta pendiente está a menudo considerada como una constante de amplificación para cada gen, par de cebadores y condición de PCR particular. Por ello esta pendiente o eficiencia de amplificación se emplea para cuantificar en la PCR cuantitativa.
- X es la concentración de ADN inicial expresada en logaritmo de copias/tubo, ng/µL, unidades arbitrarias, etc.
- B es un puunto matemático sin realidad experimental (log 0 = 1/∞) que, sin embargo, puede ser empleado para calibrar cada experimento de PCR (los «runs», en inglés) con sus semejantes. De este modo, si las rectas de calibrado lo fueron por su intersección al origen B, la dispersión será menor, salvo para los puntos M y N.
- La recta de regresión no pasa por el punto dentral de la dispersión en cada medida para las concentraciones M y N, pero sí según la pendiente y un factor de «amortiguación». Esto es: si consideramos CT medios para L y N serán modelizados de mejor manera mediante un polinomio de segundo grado. El software de algunos termocicladores permite tener en cuenta esta «amortiguación», si bien hay que tener en cuenta que:
- Dicha amortiguación es extremadamente variable de una recta de calibrado a otra, y la corrección que se modeliza no corresponde probablemente a aquélla que pasa por la muestra cuantificada. El software comercial lo modeliza para una sola muestr
- La imprecisión en la cuantificación a estas concentraciones es tan importante que la modelización de la «amortiguación» puede ser necesaria.
- Esta «amortiguación» corresponde, cuando se dibujan las rectas, a una reducción de la pendiente luego de un aumento de la eficacia de PCR, así que el incremento de los efecos estocásticos puede reducirse. Nótese además que esta «amortiguación» se define principalmente según la concentración más baja (N, 0,7 copias en el ejemplo). O lo que es lo mismo, no podrá realizarse amplificación alguna si no se tiene al menos una molécula completa del ADN a amplificar. El sesgo producido en la distribución gaussiana del error puede provocar esta aparición de datos extraños o «amortiguaciones medias».
Luego la recta de calibrado permite una cuantificación para un protocolo experimental dado pero hay que tener en cuenta que existen multitud de fuentes de error potenciales, como diferencias de composición química del tampón de reacción, de las muestras (presencia de proteínas, ARN, etc) e incluso del diluyente (el agua, generalmente).
Sondas
La PCR en tiempo real puede realizarse marcando fluorescentemente oligonucleótidos que detectan específicamente la aparición del producto deseado. El fundamento de esta técnica se basa en el empleo del FRET o transmisión de energía de resonancia, que es un mecanismo de transferencia de energía entre cromóforos. EL FRET se fundamenta en que la excitación de un cromóforo puede transferirse a otro cercano, generalmente cuando ambos se sitúan en la misma molécula, mediante un mecanismo acoplador dipolo-dipolo.[18]. En el caso de que los cromóforos sean fluorescentes (esto es, fluorocromos), el mecanismo subyacente continúa siendo el mismo: la energía se transfiere, lo que desemboca en la aparición de fluorescencia (cabe destacar que no es la fluorescencia la transferida)[19][20] Las sondas empleadas en PCR a tiempo real existentes son:

- Las sondas TaqMan permiten medir la producción de productos de PCR mediante un sistema de sondas marcadas mediante dos fluorocromos. Su utilidad radica en que poseen un fluoróforo en su extremo 3' y una molécula en el 5' que bloquea su emisión de fluorescencia (denominada en inglés «quencher»); esta sonda marcada hibrida específicamente en la parte central del producto de PCR a obtener. De este modo, cuando se efectúa la PCR (con la sonda más el par de cebadores específicos), la sonda hibrida en el amplicón, pero, debido a la cercanía del fluoróforo al quencher, no se emite fluorescencia; cuando la polimerasa se topa con la sonda la hidroliza mediante su actividad exonucleasa 5'-3', lo cual provoca la separación del quencher del fluorocromo y, por tanto, la emisión de fluorescencia. Fluorescencia que está relacionada con la cantidad de amplicón producido.[21]

- Las sondas tipo Molecular Beacons son también olignucleótidos de cadena simple que, por su estructura, poseen una zona de apareamiento de bases interna y, que, por tanto, forman una horquilla. En presencia del amplicón la sonda se abre y se une preferentemente a aquél, lo que produce la emisión de fluorescencia. Estructuralmente, poseen la zona complementaria al amplicón en el giro de la horquilla, la de complementariedad interna en el cuello y los fluoróforos en sus extremos: el donador en el 5' y el aceptor en el 3'. Cuando la sonda está cerrada en horquilla, el fluoróforo del 3' impide la emisión de fluorescencia por parte del propio del 5', cosa que no sucede al unirse al amplicón.[22]

- Sondas Scorpion. Se trata de moléculas mixtas conteniendo un cebador específico para el amplicón unido covalentemente a una horquilla similar a las sondas del tipo molecular beacon que, en su zona de giro, poseen un elemento complementario al amplicón. Los fluoróforos donador y aceptor se encuentran en la estructura de horquilla; de este modo, cuando ésta se encuentra cerrada no se produce la emisión de fluorescencia, cosa que sí sucede cuando ambos se separan debido a la presencia del amplicón y la apertura de la zona en horquilla.[23]
Referencias
- Elyse Poitras et Alain Houde (2002). La PCR en temps réel: principes et applications. Reviews in Biology and Biotechnology. 2(2):2-11.
- Bustin SA (2000). Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J Mol Endocrinol. 25(2):169-93.
- Higuchi, R., Dollinger, G., Walsh, P.S., Griffith, R. (1992). Simultaneous amplification and detection of specific DNA-sequences. Bio-Technology 10 (4), 413–417.
- Holland, P.M., Abramson, R.D., Watson, R., Gelfand, D.H. (1991). Detection of specific polymerase chain reaction product by utilizing the 50 !30 exonuclease activity of Thermus aquaticus DNA polymerase. Proc. Natl. Acad. Sci. USA 88 (16), 7276–7280.
- Kubista M, Andrade JM, Bengtsson M, Forootan A, Jonak J, Lind K, Sindelka R, Sjoback R, Sjogreen B, Strombom L, Stahlberg A, Zoric N (2006). The real-time polymerase chain reaction. Mol Aspects Med. 27(2-3):95-125.
Citas en línea
- ↑ Watson, J, D.; Baker, T. A.; Bell, S. P.; Gann, A.; Levine, M. et Losick, R (2004). «Molecular Biology of the Gene». Benjamin Cummings (Fifth edition edición) (San Francisco). ISBN 0-321-22368-3.
- ↑ a b Michael W. Pfaff, Ales Tichopad, Christian Prgomet and Tanja P. Neuvians (2005). Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper – Excel-based tool using pair-wise correlations Biotechonology Letters 26:509-515
- ↑ Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, Speleman F (2002) Accurate normalisation of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Gen. Biol. 3: 1–12.
- ↑ Pfaffl MW, Horgan GW, Dempfle L (2002) Relative Expression Software Tool (REST©) for group wise comparison and statistical analysis of relative expression results in real-time PCR. Nucl. Acids Res. 30: e36.
- ↑ Zipper, H. et al. (2004). Investigations on DNA intercalation and surface binding by SYBR Green I, its structure determination and methodological implications. Nucleic Acids Res. 32, e103. PMID 15249599
- ↑ Rychlik W, Spencer WJ, Rhoads RE (1990). «Optimization of the annealing temperature for DNA amplification in vitro». Nucl Acids Res 18: 6409-6412. PMID 2243783. doi:10.1093/nar/18.21.6409.
- ↑ Joseph Sambrook and David W. Russel (2001). Molecular Cloning: A Laboratory Manual (3rd ed. edición). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 0-87969-576-5.
- ↑ Ponchel, F.; Toomes, C.; Bransfield, K.; Leong, F.T.; Douglas, S.H.; Field, S.L.; Bell, S.M.; Combaret, V.; Puisieux, A.; Mighell, A.J.; Others (2003), [http://www.biomedcentral.com/1472-6750/3/18/ «� … for a relative quantification of gene rearrangements, gene amplifications and micro gene deletions»] (w), BMC Biotechnol 3 (1): 18, doi:10.1186/1472-6750-3-18.
- ↑ Ririe, K.M.; Rasmussen, R.P.; Wittwer, C.T. (1997), «Product Differentiation by Analysis of DNA Melting Curves during the Polymerase Chain Reaction», Analytical Biochemistry 245 (2): 154-160, doi:10.1006/abio.1996.9916.
- ↑ Brunner AM, Yakovlev IA, Strauss SH (2004) Validating internal controls for quantitative plant gene expression studies. BMC Plant Biol 4: 14
- ↑ Czechowski T, Stitt M, Altmann T, Udvardi MK, Scheible WR (2005) Genome-wide identification and testing of superior reference genes for transcript normalization in Arabidopsis. Plant Physiol 139: 5–17
- ↑ Thellin O, Zorzi W, Lakaye B, De Borman B, Coumans B, Henne G, Grisar T, Igout A, Heinen E (1999) Housekeeping genes as internal standards: use and limits. J Biotechnol 75: 197–200
- ↑ Radonic A, Thulke S, Mackay IM, Landt O, Siegert W, Nitsche A (2004) Guideline for reference gene selection for quantitative real-time PCR. Biochem Biophys Res Commun 313: 856–862
- ↑ Dheda K, Huggett JF, Bustin SA, Johnson MA, Rook G, Zumla A (2004) Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques 37: 112–119
- ↑ Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, Speleman F (2002) Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes. Genome Biol 37: RESEARCH0034
- ↑ Pfaffl MW, Tichopad A, Prgomet C, Neuvians TP (2004) Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper—Excel-based tool using pair-wise correlations. Biotechnol Lett 26: 509–515
- ↑ Mason, G.; Provero, P.; Vaira, A.M.; Accotto, G.P. (2002), BMC Biotechnology 2 (20): 203-212, doi:10.1186/1472-6750-2-20 http://www.biomedcentral.com/1472-6750/2/20/
|url=sin título (ayuda). - ↑ Förster T. Zwischenmolekulare Energiewanderung und Fluoreszenz, Ann. Physik 1948, 437, 55. doi 10.1002/andp.19484370105
- ↑ Joseph R. Lakowicz, "Principles of Fluorescence Spectroscopy", Plenum Publishing Corporation, 2nd edition (July 1, 1999)
- ↑ FRET microscopy tutorial from Olympus
- ↑ Heid, C.A.; Stevens, J.; Livak, K.J.; Williams, P.M. (1996), «Real time quantitative PCR», Genome Research 6 (10): 986-994, doi:10.1101/gr.6.10.986.
- ↑ Tyagi, S.; Kramer, F.R.; Others (1996), «Molecular Beacons: Probes that Fluoresce upon Hybridization», Nature Biotechnology 14 (3): 303-308, doi:10.1038/nbt0396-303.
- ↑ Whitcombe, D.; Theaker, J.; Guy, S.P.; Brown, T.; Little, S. (1999), Nature Biotechnology 17: 804-807, doi:10.1038/11751 http://www.gene-quantification.de/whitcombe-1999.pdf
|url=sin título (ayuda).

![{\displaystyle E=(indice\;del\;logaritmo)^{\frac {-1}{pendiente}}={\sqrt[{-pendiente}]{indice\;del\;logaritmo}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a62e8b7798886375544d6e9f3b6238fc2567ad75)