Microcompartimiento bacteriano
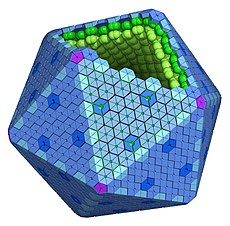

Los microcompartimientos bacterianos (MCB o BMC por sus siglas en inglés) son orgánulos bacterianos que consisten en una cubierta proteica que encierra ciertas enzimas y otras proteínas. Estos microcompartimientos tienen generalmente entre 40 y 200 nanómetros de diámetro y están compuestos íntegramente por proteínas. La función de las cubiertas es similar a la de una membrana, ya que son selectivamente permeables.[1][2][3][4] Otros compartimientos a base de proteínas que se encuentran en bacterias y arqueas son los nanocompartimientos de encapsulina y las vesículas de gas. Los eucariotas también tienen orgánulos proteicos como el complejo de bóveda.
Estructuralmente son similares a las cápsides de los virus con los que no guardan ni una relación, sin embargo los estudios sugieren que los virus surgieron de microcompartimientos como este en la era precelular (antes que el último antepasado común universal).[5]
Origen de los BMC y su relación con las cápsides virales[editar]
Si bien la cubierta de los microcompartimientos bacterianos es arquitectónicamente similar a las cápsides virales, no se ha encontrado que las proteínas de la cubierta tengan homología estructural o de secuencia con proteínas de la cápside de ciertos virus. En cambio, las comparaciones estructurales y de secuencia sugieren que los microcompartimientos bacterianos, muy probablemente, han evolucionado a partir de proteínas celulares genuinas, como, proteínas de señalización PII y proteínas que contiene el dominio de pliegues OB, respectivamente. Las geometrías de la membrana de los microcompartimientos bacterianos son poliedros que se explican al considerar capas de varios componentes.[6][7]
Descubrimiento[editar]
Los primeros BMC se observaron en la década de 1950 en micrografías electrónicas de cianobacterias, y luego se denominaron carboxisomas después de que se estableció su papel en la fijación de carbono. Hasta la década de 1990, se pensaba que los carboxisomas eran una rareza confinada a ciertas bacterias autótrofas. Pero entonces los genes que codifican para proteínas homólogas a las de la cáscara carboxisoma se identificaron en la PDU (utilización propanodiol) y eut (utilización de etanolamina). Posteriormente, micrografías electrónicas de transmisión de células de Salmonella cultivadas en propanodiol, oetanolamina mostró la presencia de cuerpos poliédricos similares a los carboxisomas. El término metabolosoma se usa para referirse a tales BMC catabólicas (en contraste con el carboxisoma autotrófico).
Aunque el carboxisoma, el uso de propanodiol (PDU) y el uso de etanolamina (EUT) encapsulan diferentes enzimas y, por lo tanto, tienen diferentes funciones, los genes que codifican las proteínas de la cubierta son muy similares. La mayoría de los genes (que codifican las proteínas del caparazón y las enzimas encapsuladas) de las CMM caracterizadas experimentalmente se encuentran cerca unos de otros en distintos loci u operones genéticos. Actualmente hay más de 20.000 genomas bacterianos secuenciados, y se pueden utilizar métodos bioinformáticos para encontrar todos los genes de caparazón de BMC y observar qué otros genes hay en las proximidades, lo que produce una lista de posibles BMC. En 2014, una encuesta exhaustiva identificó 23 loci diferentes que codifican hasta 10 BMC funcionalmente distintos en 23 filos bacterianos.
Cubierta y capas[editar]
La cubierta de los BMC es icosaédrica o cuasi-icosaédrica, y está formada por subunidades proteicas (pseudo) hexámeras y pentaméricas. Los componentes principales de la cubierta de BMC son proteínas que contienen dominio (s) Pfam00936. Estas proteínas forman oligómeros de forma hexagonal y se cree que forman las facetas de la cáscara.
Proteínas de dominio único (BMC-H)[editar]
Las proteínas BMC-H, que contienen una sola copia del dominio Pfam00936, son el componente más abundante de las facetas de la cáscara. Se han determinado las estructuras cristalinas de varias de estas proteínas, lo que demuestra que se ensamblan en hexámeros cíclicos, normalmente con un pequeño poro en el centro. Se propone que esta apertura interviene en el transporte selectivo de los pequeños metabolitos a través de la cáscara.
Proteínas de dominio en tándem (BMC-T)[editar]
Un subconjunto de proteínas de capa está compuesto por copias en tándem (fusionadas) del dominio Pfam00936 (proteínas BMC-T). Las proteínas BMC-T estructuralmente caracterizadas forman trímeros que son de forma pseudohexámera. Algunas estructuras cristalinas de BMC-T muestran que los trímeros pueden apilarse cara a cara. En tales estructuras, un poro de un trímero está en una conformación "abierta", mientras que el otro está cerrado, lo que sugiere que puede haber un mecanismo similar a una esclusa de aire que modula la permeabilidad de algunas conchas de BMC. Otro subconjunto de proteínas BMC-T contiene un grupo [4Fe-4S] y puede estar involucrado en el transporte de electrones a través de la capa de BMC.
La familia EutN / CcmL (BMC-P)[editar]
Se necesitan doce unidades pentagonales para cubrir los vértices de una capa icosaédrica. Se han resuelto las estructuras cristalinas de proteínas de la familia EutN / CcmL (Pfam03319) y normalmente forman pentámeros (BMC-P). La importancia de las proteínas BMC-P en la formación de la capa parece variar entre las diferentes BMC. Se demostró que son necesarios para la formación de la capa de la PDU BMC ya que los mutantes en los que el gen de la proteína BMC-P se eliminó no puede formar capas, pero no para el alfa-carboxisoma: sin BMC-P proteínas, los carboxisomas todavía se ensamblarán y muchos se alargarán; estos carboxisomas mutantes parecen tener "fugas".
Permeabilidad de la cubierta[editar]
Está bien establecido que las enzimas están empaquetadas dentro de la cubierta de BMC y que debe producirse algún grado de secuestro de metabolitos y cofactores. Sin embargo, también se debe permitir que otros metabolitos y cofactores crucen la capa para que las BMC funcionen. Por ejemplo, en los carboxisomas, la ribulosa-1,5-bisfosfato, el bicarbonato y el fosfoglicerato deben cruzar la capa, mientras que la difusión de dióxido de carbono y oxígeno es aparentemente limitada. De manera similar, para el PDU BMC, la cáscara debe ser permeable al propanodiol, propanol, propionilfosfato y potencialmente también vitamina B12, pero está claro que el propionaldehído se secuestra de alguna manera para prevenir daño celular. Existe alguna evidencia de que el ATP también debe atravesar algunos proyectiles de BMC.
Se ha propuesto que el poro central formado en las placas de proteína hexagonales de la cubierta son los conductos a través de los cuales los metabolitos se difunden en la cubierta. Por ejemplo, los poros de la capa carboxisoma tienen una carga positiva general, que se ha propuesto para atraer sustratos cargados negativamente como el bicarbonato. En el microcompartimento de la PDU, los experimentos de mutagénesis han demostrado que el poro de la proteína de la cubierta de PduA es la ruta de entrada del sustrato de propanodiol. Para metabolitos más grandes, es evidente un mecanismo de activación en algunas proteínas BMC-T. En el microcompartimento de EUT, la apertura del poro grande en la proteína de la cubierta de EutL está regulada por la presencia del principal sustrato metabólico, la etanolamina.
La presencia de grupos de hierro-azufre en algunas proteínas de la capa, presumiblemente en el poro central, ha llevado a la sugerencia de que pueden servir como un conducto a través del cual los electrones pueden transportarse a través de la capa.
Tipos[editar]
Un estudio exhaustivo reciente de los datos de la secuencia del genoma bacteriano indicó hasta diez funciones metabólicas diferentes encapsuladas por conchas de BMC. La mayoría está involucrada en la fijación de carbono (carboxisomas) o en la oxidación de aldehídos (metabolosomas).
Carboxisomas: fijación de carbono[editar]
Los carboxisomas encapsulan ribulosa-1,5-bisfosfato carboxilasa / oxigenasa (RuBisCO) y anhidrasa carbónica en bacterias fijadoras de carbono como parte de un mecanismo de concentración de carbono. El bicarbonato se bombea al citosol y se difunde en el carboxisoma, donde la anhidrasa carbónica lo convierte en dióxido de carbono, el sustrato de RuBisCO. Se cree que el caparazón del carboxisoma es poco permeable al dióxido de carbono, lo que da como resultado un aumento efectivo de la concentración de dióxido de carbono alrededor de RuBisCO, mejorando así la fijación de carbono. Los mutantes que carecen de genes que codifican la cáscara del carboxisoma muestran un fenotipo de alto requerimiento de carbono debido a la pérdida de la concentración de dióxido de carbono, lo que resulta en una mayor fijación de oxígeno por RuBisCO. Las conchas también se han propuesto para restringir la difusión de oxígeno, evitando así la reacción de oxigenasa, reduciendo la fotorrespiración derrochadora.
Metabolosomas: oxidación de aldehídos[editar]
Además de los carboxisomas anabólicos, se han caracterizado varias BMC catabólicas que participan en el metabolismo heterotrófico a través de aldehídos de cadena corta; se denominan colectivamente metabolosomas.
Estos BMC comparten una química encapsulada común impulsada por tres enzimas centrales: aldehído deshidrogenasa, alcohol deshidrogenasa y fosfotransacilasa. Debido a que los aldehídos pueden ser tóxicos para las células y / o volátiles, se cree que están secuestrados dentro del metabolosoma. El aldehído se fija inicialmente a la coenzima A por una aldehído deshidrogenasa dependiente de NAD +, pero estos dos cofactores deben reciclarse, ya que aparentemente no pueden cruzar la capa. Estas reacciones de reciclado son catalizadas por una alcohol deshidrogenasa (NAD +), y una fosfotransacetilasa (coenzima A), dando como resultado un compuesto de acilo fosforilado que puede ser fácilmente una fuente de fosforilación a nivel de sustrato o entrar en el metabolismo central, dependiendo de si el organismo está creciendo aeróbicamente o anaeróbicamente. Parece que la mayoría, si no todos, los metabolosomas utilizan estas enzimas centrales. Los metabolosomas también encapsulan otra enzima que es específica del sustrato inicial del BMC, que genera el aldehído; esto se considera la enzima distintiva del BMC.
BMC de PDU[editar]
Algunas bacterias pueden usar 1,2-propanodiol como fuente de carbono. Usan un BMC para encapsular varias enzimas utilizadas en esta vía. El PDU BMC generalmente está codificado por un locus de 21 genes. Estos genes son suficientes para el ensamblaje de las BMC, ya que pueden trasplantarse de un tipo de bacteria a otro, lo que da como resultado un metabolosoma funcional en el receptor. Este es un ejemplo de bioingeniería que también proporciona evidencia en apoyo de la hipótesis del operón egoísta. El 1,2-propanodiol se deshidrata a propionaldehído por la propanodiol deshidratasa, que requiere vitamina B12 como cofactor. El propionaldehído causa mutaciones en el ADN y, como resultado, es tóxico para las células, lo que posiblemente explica por qué este compuesto está secuestrado dentro de un BMC, los productos finales del PDU BMC son propanol y propionil-fosfato, que luego se desfosforila a propionato, generando un ATP. El propanol y el propionato se pueden utilizar como sustratos para el crecimiento.
BMC de PDU / EUT bifuncionales[editar]
Algunas bacterias, codifican un único locus en el que están presentes genes para las BMC de PDU y EUT. Aún no está claro si se trata realmente de un BMC quimérico con una mezcla de ambos conjuntos de proteínas, o si se forman dos BMC separados.
BMC que contienen enzimas de radicales glicilo (GRM)[editar]
Se han identificado varios loci de BMC diferentes que contienen enzimas radicales glicilo, que obtienen el radical catalítico de la escisión de s-adenosilcobalamina. Se ha demostrado que un locus GRM está involucrado en la fermentación de fucosa y ramnosa, que inicialmente se degradan a 1,2-propanodiol en condiciones anaeróbicas. Se propone que la enzima de radical glicilo deshidrate el propanodiol a propionaldehído, que luego se procesa de una manera idéntica a la PDU canónica BMC.
BMC de función desconocida (BUF)[editar]
Un tipo de locus de BMC no contiene RuBisCO ni ninguna de las enzimas del metabolosoma central, y se ha propuesto para facilitar una tercera categoría de transformaciones bioquímicas (es decir, no la fijación de carbono ni la oxidación de aldehídos). La presencia de genes que se predice que codificarán amidohidrolasas y desaminasas podría indicar que este BMC está involucrado en el metabolismo de compuestos nitrogenados.
Ensamblaje[editar]
Carboxisomas[editar]
Se ha identificado la ruta de ensamblaje de los beta-carboxisomas y comienza con la proteína CcmM que nuclea RuBisCO. CcmM tiene dos dominios: un dominio de anhidrasa gamma-carbónica N-terminal seguido de un dominio que consta de tres a cinco repeticiones de secuencias similares a subunidades pequeñas de RuBisCO. El dominio C-terminal agrega RuBisCO, probablemente sustituyendo las pequeñas subunidades reales de RuBisCO en la holoenzima L8-S8, reticulando efectivamente RuBisCO en la célula en un agregado grande, denominado procarboxisoma. El dominio N-terminal de CcmM interactúa físicamente con el dominio N-terminal de la proteína CcmN, que, a su vez, recluta las subunidades de la proteína de la cubierta hexagonal a través de un péptido de encapsulación en su C-terminal. Luego, los carboxisomas se alinean espacialmente en la célula cianobacteriana mediante la interacción con el citoesqueleto bacteriano, lo que garantiza su distribución equitativa en las células hijas.
El ensamblaje de alfa-carboxisomas puede ser diferente al de los beta-carboxisomas, ya que no tienen proteínas homólogas a CcmN o CcmM ni péptidos de encapsulación. Se han observado carboxisomas vacíos en micrografías electrónicas. Algunas micrografías indican que su ensamblaje ocurre como una coalescencia simultánea de enzimas y proteínas de la cáscara en oposición a la forma aparentemente escalonada observada para los beta-carboxisomas. Se ha demostrado que la formación de alfa-carboxisomas simples en sistemas heterólogos requiere solo subunidades grandes y pequeñas de Rubisco, la proteína de anclaje interna CsoS2 y la proteína de capa principal CsoS1A.
Metabolosomas[editar]
El ensamblaje del metabolosoma es probablemente similar al del beta-carboxisoma, a través de una agregación inicial de las proteínas a encapsular. Las proteínas centrales de muchos metabolosomas se agregan cuando se expresan solas. Además, muchas proteínas encapsuladas contienen extensiones terminales que son sorprendentemente similares al péptido C-terminal de CcmN que recluta proteínas de caparazón. Estos péptidos de encapsulación son cortos (alrededor de 18 residuos) y se prevé que formen hélices alfa anfipáticas. Se ha demostrado que algunas de estas hélices median en la encapsulación de enzimas nativas en BMC, así como en proteínas heterólogas (como GFP).
Regulación (genética)[editar]
Con la excepción de los carboxisomas, en todos los casos probados, las BMC están codificadas en operones que se expresan solo en presencia de su sustrato.
Los BMC de PDU son inducidos por la presencia de propanodiol o glicerol en condiciones anaeróbicas y solo propanodiol en condiciones aeróbicas. Esta inducción está mediada por las proteínas reguladoras globales Crp y ArcA (que detectan AMP cíclico y condiciones anaeróbicas, respectivamente), y la proteína reguladora PocR, que es el activador transcripcional para los loci pdu y cob (el operón necesaria para la síntesis de vitamina B12, un cofactor necesario para la propanodiol deshidratasa).
Las CMO de EUT se inducen a través de la proteína reguladora EutR por la presencia simultánea de etanolamina y vitamina B12, lo que puede ocurrir en condiciones aeróbicas o anaeróbicas. Solo puede producir vitamina B12 endógena en condiciones anaeróbicas, aunque puede importar cianobalamina y convertirla en vitamina B12 en condiciones aeróbicas o anaeróbicas.
Los BMC de PVM son inducidos por la presencia de fucosa o ramnosa en condiciones aeróbicas, pero no por glucosa. Se obtuvieron resultados similares para el GRM BMC, el cual ambos azúcares inducen los genes que codifican el BMC, así como los que codifican las enzimas disimilatorias de fucosa y ramnosa.
Además de los sistemas reguladores caracterizados, las encuestas bioinformáticas han indicado que existen potencialmente muchos otros mecanismos reguladores, incluso dentro de un tipo funcional de BMC (por ejemplo, PDU), incluidos los sistemas reguladores de dos componentes.
Aplicaciones biotecnológicas[editar]
Varias características de las BMC las hacen atractivas para aplicaciones biotecnológicas. Debido a que los carboxisomas aumentan la eficiencia de la fijación de carbono, se han realizado muchos esfuerzos de investigación para introducir carboxisomas y transportadores de bicarbonato necesarios en los cloroplastos de las plantas para diseñar un mecanismo de concentración de CO2 cloroplástico con cierto éxito.
De manera más general, debido a que las proteínas de caparazón de BMC se autoensamblan, se pueden formar capas vacías, lo que impulsa esfuerzos para diseñarlas para contener carga personalizada. El descubrimiento del péptido de encapsulación en los extremos de algunas proteínas asociadas a BMC proporciona un medio para comenzar a diseñar BMC personalizadas fusionando proteínas extrañas a este péptido y co-expresándolo con proteínas de la cubierta. Por ejemplo, al agregar este péptido a la piruvato descarboxilasa y la alcohol deshidrogenasa, los investigadores han diseñado un biorreactor de etanol. Finalmente, los poros presentes en las proteínas de la cáscara controlan la permeabilidad de la cáscara: estos pueden ser un objetivo para la bioingeniería, ya que pueden modificarse para permitir el cruce de sustratos y productos seleccionados.
Referencias[editar]
- ↑ Kerfeld, Cheryl A.; Heinhorst, Sabine; Cannon, Gordon C. (2010). «Bacterial Microcompartments». Annual Review of Microbiology (Submitted manuscript) 64 (1): 391-408. ISSN 0066-4227. PMID 20825353. doi:10.1146/annurev.micro.112408.134211.
- ↑ Parsons, J. B.; Dinesh, S. D.; Deery, E.; Leech, H. K.; Brindley, A. A.; Heldt, D.; Frank, S.; Smales, C. M.; Lunsdorf, H.; Rambach, A.; Gass, M. H.; Bleloch, A.; McClean, K. J.; Munro, A. W.; Rigby, S. E. J.; Warren, M. J.; Prentice, M. B. (2008). «Biochemical and Structural Insights into Bacterial Organelle Form and Biogenesis». Journal of Biological Chemistry 283 (21): 14366-14375. ISSN 0021-9258. PMID 18332146. doi:10.1074/jbc.M709214200.
- ↑ Kerfeld CA, Sawaya MR, Tanaka S, Nguyen CV, Phillips M, Beeby M, Yeates TO (August 2005). «Protein structures forming the shell of primitive bacterial organelles». Science 309 (5736): 936-938. PMID 16081736. doi:10.1126/science.1113397. Parámetro desconocido
|citeseerx=ignorado (ayuda) - ↑ Lassila, Jonathan K.; Bernstein, Susan L.; Kinney, James N.; Axen, Seth D.; Kerfeld, Cheryl A. (2014). «Assembly of Robust Bacterial Microcompartment Shells Using Building Blocks from an Organelle of Unknown Function». Journal of Molecular Biology 426 (11): 2217-2228. ISSN 0022-2836. PMID 24631000. doi:10.1016/j.jmb.2014.02.025.
- ↑ Krupovic, M; Dolja, VV; Koonin, EV (14 de julio de 2020). «The LUCA and its complex virome.». Nat Rev Microbiol. PMID 32665595. doi:10.1038/s41579-020-0408-x. Consultado el 16 de agosto de 2020.
- ↑ Kinney, James N.; Axen, Seth D.; Kerfeld, Cheryl A. (2011). «Comparative analysis of carboxysome shell proteins». Photosynthesis Research 109 (1–3): 21-32. ISSN 0166-8595. PMC 3173617. PMID 21279737. doi:10.1007/s11120-011-9624-6.
- ↑ Fan, C.; Cheng, S.; Liu, Y.; Escobar, C. M.; Crowley, C. S.; Jefferson, R. E.; Yeates, T. O.; Bobik, T. A. (2010). «Short N-terminal sequences package proteins into bacterial microcompartments». Proceedings of the National Academy of Sciences 107 (16): 7509-7514. ISSN 0027-8424. PMC 2867708. PMID 20308536. doi:10.1073/pnas.0913199107.
