HB36.6
La proteína HB36.6 es una proteína diseñada de manera computacional por investigadores y cuya función básica es la protección ante el virus de la gripe A. Se trata de una versión madurada y perfeccionada de la previa proteína HB36.5 de la que difiere en 9 aminoácidos.[1]
A diferencia de los fármacos antigripales convencionales, tales como el Oseltamivir, esta proteína no desencadena una respuesta inmunitaria en el enfermo, sino que se une a la región de Hemaglutinina (HA) del virus influenza (región que no experimenta cambios generalmente en las distintas cepas del virus, lo que le aporta un gran interés) para que éste no pueda unirse a las células sanas del individuo (Figura 1). Es por eso que se prevé que desarrollará un papel importante en la prevención de la enfermedad, así como en la curación de ésta en pacientes inmunodeprimidos como los ancianos.[2]
Asimismo, la HB36.6 representa una muestra clara de que proteínas diseñadas de manera computacional tienen la suficiente estabilidad y potencia para aportar protección in vivo.[3]
Estructura[editar]
Estructura del virus de la gripe A[editar]
El virus de la gripe A tiene una estructura de forma globular. En el núcleo encontramos ocho cadenas de RNA así como moléculas de polimerasa asociados a cuatro proteínas distintas. Éste está envuelto de una membrana lipídica en donde podemos encontrar asociadas diversas proteínas de membrana, como son HA y otro tipo llamada neuraminidasa, cuya función es deshacer la unión entre HA y el ácido siálico.[1]
Estructura de la proteína de membrana[editar]
HB36.6 es una proteína sintetizada artificialmente que se une a la hemaglutinina o HA (Figura 2), que es una proteína de membrana del virus de la gripe A. Su función es el reconocimiento específico con la célula, y también contiene los mecanismos de fusión de membranas.
Estructuralmente, HA es una proteína homotrimérica, es decir, compuesta por tres monómeros idénticos, compuestos a su vez cada uno por dos subunidades unidas entre sí por puentes disulfuro. Estas subunidades provienen de la escisión proteótica de un precursor la cual tiene lugar en la síntesis de esta proteína. Se denominan HA1 y HA2. La cadena HA1 forma la cabeza globular de la proteína, así como parte del tronco. HA2, en cambio, forma el resto del tronco.[4]
Funcionalmente, en los procesos de unión a la membrana celular, la cabeza se encarga de la unión específica con un receptor compuesto por ácido siálico, el hecho de que su receptor específico sea éste explica su mayor proliferación en el tracto respiratorio, rico en este compuesto. Para ello, tiene una región aminoácidos conservada con glicosilaciones.[5] Esta es inaccesible por los anticuerpos ya que está envuelta por su entorno, el cual es altamente mutable. Al ser esta la región más expuesta es la que ataca el sistema humoral. Esto explica la excesiva especificidad para cada cepa del sistema immunitario. En cuanto al tronco, contiene los mecanismos de fusión entre membranas. El extremo N-terminal de HA2 es el péptido que se encarga de esta función, y se encuentra rodeado por las alfa hélices del tallo.[6] Al entrar en contacto con el medio ácido de cerca de la célula existente por el ácido siálico, este péptido padece unos cambios conformacionales que empujan el receptor específico escondido en el globo para que enlace con el ácido siálico así como la membrana vírica para permitir esta fusión. En este proceso se desestructuran las alfa hélices y la cabeza globular que contenían estos aminoácidos clave. Esta parte, bastante conservada, sí es accesible por algunos anticuerpos, por lo cual es el centro de investigación en la búsqueda de artificiales.[7]
La mayor parte de los anticuerpos están dirigidos a la región de la cabeza de esta molécula. El problema en el momento de buscar un antiviral es que las regiones de los códigos genéticos que deberán codificar estos epítopos (determinantes antigénicos) son muy plásticos y susceptibles a mutaciones. En contraste, la parte del tronco es mucho más estable en las diferentes cepas del virus. En consecuencia, un anticuerpo (la mayoría de los producidos por el cuerpo atacan la región de la cabeza) dirigido a esta región podría afectar a la mayoría de cepas del virus de la gripe A, e incluso de la A y la B.[4]
Estructura de la proteína artificial[editar]
La proteína HB36.6 es una proteína que consta de 97 aminoácidos y, como tal, es considerada una proteína pequeña. Asimismo, presenta una estructura de hélice alfa, lo que le proporciona polaridad.
HB36.6 surgió de la mejora de la proteína previa HB36.5 de la que difiere en 9 aminoácidos. Estos cambios en la secuencia hacen que HB36.6 presente una afinidad mucho más elevada con el segmento HA del virus de la gripe que su precursora. En los virus del grupo 1, por ejemplo, la afinidad puede aumentar hasta 40 veces.[8]
Los cambios de aminoácidos que se produjeron fueron los siguientes:
| HB36.5 | HB36.6 | Posición del aminoácido | |
|---|---|---|---|
| W17N | Triptófano (Trp) | Asparagina (Asn) | 17 |
| D32T | Ácido aspártico (Asp) | Treonina (Thr) | 32 |
| E34Q | Ácido glutámico (Glu) | Glutamina (Gln) | 34 |
| E46N | Ácido glutámico (Glu) | Asparagina (Asn) | 46 |
| S47R | Serina (Ser) | Arginina (Arg) | 47 |
| Q66R | Glutamina (Gln) | Arginina (Arg) | 66 |
| H71R | Histidina (His) | Arginina (Arg) | 71 |
| A72T | Alanina (Ala) | Treonina (Thr) | 72 |
| L75F | Leucina (Leu) | Fenilalanina (Phe) | 75 |
Analizando estos datos, se observa que en la proteína HB36.6 aumentó la proporción de aminoácidos polares (de 5 iniciales en su precursora a 8). Este grupo de aminoácidos se caracteriza por presentar cadenas lateral hidrófilas. Entre los 8 aminoácidos polares hay 3 (Arg) que, además,tienen carga positiva, es decir,en sus cadenas laterales hidrófilas tienen un grupo básico. Estas propiedades favorecen la unión de la proteína con la Hemaglutinina y, en consecuencia, permiten que pueda desempeñar de manera óptima su función.[9]
Sin embargo, estos cambios no proporcionan mayor eficacia ante todos los diferentes grupos de la gripe, pero sí que contra la mayoría de ellos. Es por eso que se la considera una proteína más madura y optimizada.
Asimismo, cabe destacar el papel de la Arginina de la posición 54 de la proteína. Este aminoácido se une mediante una red de enlaces de hidrógeno al Ácido aspártico y la Arginina de las HAs 1-4 (Figura 3). En las Has 5-7 sucede lo mismo, pero en lugar de Ácido aspártico encontramos Ácido glutámico.[5]
Función[editar]
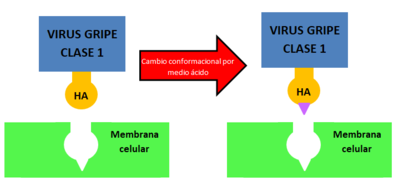
Figura 4: Secuencia normal de los cambios que experimenta la región HA del virus para la posterior infección de la célula. - La función biólogica de la proteína es la neutralización del virus de la gripe A de clase 1 (Figura 4). Esto lo consigue mediante la unión al tallo de la hemaglutinina, responsable de la fusión de la membrana vírica con la membrana celular.

- La función biólogica de la proteína es la neutralización del virus de la gripe A de clase 1. Esto lo consigue mediante la unión al tallo de la hemaglutinina, responsable de la fusión de la membrana vírica con la membrana celular.[10]
- La proteína al enlazar con el tallo impide que se produzcan los cambios conformacionales en el extremo N-terminal de HA2 al entrar en contacto con un medio más ácido (Figura 5). De esta manera, impide la fusión de las membranas vírica y celular impidiendo así la proliferación del virus.[11]
- El motivo por el que únicamente funciona con virus con hemaglutininas de clase 1 es que las diferencias entre la clase 1 y la 2 se deben a la fusión por puente disulfuro de HA1 y HA2, lo cual crea diferencias conformacionales que hace que el diseño de HB36.6 solo actúe con la clase 1. Sin embargo, los científicos no descartan la posibilidad de sintetizar otra proteína capaz de afectar a la hemaglutinina de clase 2.[12] Asimismo, cabe añadir que mientras que la proteína artificial es capaz de evitar la mortalidad por completo y la pérdida de peso prácticamente por completo en los ratones cuando se ha usado como prevención, pierde el 50% de su eficacia cuando se administra una vez han aparecido los primeros síntomas. Esto se puede explicar teniendo en cuenta que el virus ha tenido y tiene la capacidad de replicarse mientras no ha sido afectado por la proteína, mientras el cuerpo no tiene la capacidad de sintetizar más proteína. En consecuencia, si hay más virus que proteínas, el virus libre podrá replicarse y proliferar sin más dosis de este fármaco
- Paralelamente, induce una respuesta de citocina transitoria no relacionada con la protección, en concreto de IL-6, IL-10, IL-12 (p70), TNF-α y INF-γ, que desaparece a las 48 horas.[13]
Características[editar]
- Está constituida por 97 aminoácidos, generalmente polares.[14]
- Tiene una masa molecular de 10670 Da.
- La proteína HB36.6 se desnaturaliza a temperaturas superiores a los 95 °C.[14]
- Actúa de manera independiente al sistema immune, lo cual puede ser beneficioso en pacientes immunodeprimidos, ancianos o niños.
- Es más eficaz que la Ribavirina y el anticuerpo FI6v3 en cuanto a la reducción de replicación del virus de la gripe, puesto que se requieren concentraciones menores de ésta para lograr los mismos resultados. Por ejemplo, para que la cepa de la gripe H1N1 reduzca su replicación a la mitad, se necesitarían las siguientes concentraciones:
- HB36.6: 0,18 µg/mL
- Ribavirina: 15 µg/mL
- FI6v3: 0,27 µg/mL[15]
Síntesis artificial de la proteína[editar]
Para conseguir la síntesis artificial de una proteína con unas características concretas deseadas, hoy en día se pueden sintetizar artificialmente oligonucleótidos. Una vez se ha creado la cadena, se multiplica mediante la técnica PCR. Una vez se obtienen las copias, se insertan en células de levadura, ya que, dado su naturaleza eucariota, tiene sistemas de procesamiento proteíco similares a los humanos (lo único en lo que difiere el procesamiento es en la glicosilación, aunque dado que HB36.6 no es una glicoproteína, puede sintetizarse con este método). Para encontrar la secuencia deseada, se hizo una mutagénesis saturada de la HB36.5, procedimiento consistente en cambiar aminoácidos en 12 posiciones clave a todos los posibles. A continuación, se observó la Kd de cada una al unirse con HA de diversas cepas del virus, sacando al final una variante con nueve cambios, la HB36.6. Esta proteína puede acabar con la clase 1 del virus de la gripe A, aunque su eficacia disminuye al enfrentarse a virus gripe A de clase 2, y aún más con la gripe B.[16]
Conclusión[editar]
La proteína HB36.6 es un ejemplo que demuestra el progreso de la llamada evolución dirigida, a través de la cual se pretende sintetizar biomoléculas, principalmente proteínas, capaces de desarrollar eficazmente una función biológica deseada, ya que la molécula ha sido obtenida de la optimización de HB36.5. En este caso se ha logrado superar con una eficacia que supera la de los antivirales desarrollados en la paralización de la replicación del virus de la gripe A de clase 1, mostrando una capacidad de actuación contra diversas cepas de este virus.
Referencias externas[editar]
- ↑ a b «Un ordenador ayuda a los investigadores hacer frente a múltiples cepas de la gripe a la vez | Diagnostrum». Archivado desde el original el 24 de octubre de 2019. Consultado el 24 de octubre de 2019.
- ↑ Stu Borman. «Una proteína creada "a la carta" es un prometedor agente antigripal | February 8, 2016 Issue - Vol. 94 Issue 6 | Chemical & Engineering News». cen.acs.org. Consultado el 24 de octubre de 2019.
- ↑ «Fighting Flu with Imitation Antibodies». The Medicine Maker (en inglés). Consultado el 24 de octubre de 2019.
- ↑ a b Whitehead, Timothy A.; Chevalier, Aaron; Song, Yifan; Dreyfus, Cyrille; Fleishman, Sarel J.; De Mattos, Cecilia; Myers, Chris A.; Kamisetty, Hetunandan et al. (2012-06). «Optimization of affinity, specificity and function of designed influenza inhibitors using deep sequencing». Nature Biotechnology (en inglés) 30 (6): 543-548. ISSN 1546-1696. doi:10.1038/nbt.2214. Consultado el 26 de octubre de 2019.
- ↑ a b Baranovich, Tatiana; Burnham, Andrew J.; Marathe, Bindumadhav M.; Armstrong, Jianling; Guan, Yi; Shu, Yuelong; Peiris, Joseph Malik Sriyal; Webby, Richard J. et al. (1 de mayo de 2014). «The neuraminidase inhibitor oseltamivir is effective against A/Anhui/1/2013 (H7N9) influenza virus in a mouse model of acute respiratory distress syndrome». The Journal of Infectious Diseases 209 (9): 1343-1353. ISSN 1537-6613. PMC 3982843. PMID 24133191. doi:10.1093/infdis/jit554. Consultado el 26 de octubre de 2019.
- ↑ «Hélice alfa». Wikipedia, la enciclopedia libre.
- ↑ Ekiert, Damian C; Wilson, Ian A (1 de abril de 2012). «Broadly neutralizing antibodies against influenza virus and prospects for universal therapies». Current Opinion in Virology. Virus structure and function / Emerging viruses 2 (2): 134-141. ISSN 1879-6257. doi:10.1016/j.coviro.2012.02.005. Consultado el 26 de octubre de 2019.
- ↑ [No title found], doi:10.1371/journal.ppat.1005409.g001 .
- ↑ «Hemaglutinina». bioinformatica.uab.es. Consultado el 24 de octubre de 2019.
- ↑ Leyva-Grado, Victor H.; Tan, Gene S.; Leon, Paul E.; Yondola, Mark; Palese, Peter (1 de julio de 2015). «Direct Administration in the Respiratory Tract Improves Efficacy of Broadly Neutralizing Anti-Influenza Virus Monoclonal Antibodies». Antimicrobial Agents and Chemotherapy (en inglés) 59 (7): 4162-4172. ISSN 0066-4804. PMID 25941218. doi:10.1128/AAC.00290-15. Consultado el 26 de octubre de 2019.
- ↑ Shrestha, Sourya; Foxman, Betsy; Weinberger, Daniel M.; Steiner, Claudia; Viboud, Cécile; Rohani, Pejman (26 de junio de 2013). «Identifying the Interaction Between Influenza and Pneumococcal Pneumonia Using Incidence Data». Science Translational Medicine (en inglés) 5 (191): 191ra84-191ra84. ISSN 1946-6234. PMID 23803706. doi:10.1126/scitranslmed.3005982. Consultado el 26 de octubre de 2019.
- ↑ Chen, Jianjun; Yan, Bin; Chen, Quanjiao; Yao, Yanfeng; Wang, Huadong; Liu, Qian; Zhang, Shaoqiong; Wang, Hanzhong et al. (2014-3). «Evaluation of neutralizing efficacy of monoclonal antibodies specific for 2009 pandemic H1N1 influenza A virus in vitro and in vivo». Archives of Virology 159 (3): 471-483. ISSN 1432-8798. PMID 24057757. doi:10.1007/s00705-013-1852-y. Consultado el 26 de octubre de 2019.
- ↑ Walsh, Kevin B.; Teijaro, John R.; Wilker, Peter R.; Jatzek, Anna; Fremgen, Daniel M.; Das, Subash C.; Watanabe, Tokiko; Hatta, Masato et al. (19 de julio de 2011). «Suppression of cytokine storm with a sphingosine analog provides protection against pathogenic influenza virus». Proceedings of the National Academy of Sciences of the United States of America 108 (29): 12018-12023. ISSN 1091-6490. PMC 3142000. PMID 21715659. doi:10.1073/pnas.1107024108. Consultado el 26 de octubre de 2019.
- ↑ a b Koday, Merika Treants; Nelson, Jorgen; Chevalier, Aaron; Koday, Michael; Kalinoski, Hannah; Stewart, Lance; Carter, Lauren; Nieusma, Travis et al. (4 de febrero de 2016). «A Computationally Designed Hemagglutinin Stem-Binding Protein Provides In Vivo Protection from Influenza Independent of a Host Immune Response». PLOS Pathogens (en inglés) 12 (2): e1005409. ISSN 1553-7374. doi:10.1371/journal.ppat.1005409. Consultado el 24 de octubre de 2019.
- ↑ «A Computationally Designed Hemagglutinin Stem-Binding Protein Provides In Vivo Protection from Influenza Independent of a Host Immune Response». PLOS Pathogens (en inglés) 12 (2): e1005409. 4 de febrero de 2016. ISSN 1553-7374. doi:10.1371/journal.ppat.1005409. Consultado el 24 de octubre de 2019.
- ↑ Ichinohe, Takeshi; Lee, Heung Kyu; Ogura, Yasunori; Flavell, Richard; Iwasaki, Akiko (16 de enero de 2009). «Inflammasome recognition of influenza virus is essential for adaptive immune responses». The Journal of Experimental Medicine 206 (1): 79-87. ISSN 1540-9538. PMC 2626661. PMID 19139171. doi:10.1084/jem.20081667. Consultado el 26 de octubre de 2019.
Enlaces externos[editar]
- Esta obra contiene una traducción derivada de «[[:{{{1}}}:{{{2}}}|{{{2}}}]]» de Wikipedia en Idioma no definido en la plantillaError: parámetro de idioma no válido.
{{obtener idioma}}., publicada por [{{FULLURL:{{{1}}}:{{{2}}}|action=history}} sus editores] bajo la Licencia de documentación libre de GNU y la Licencia Creative Commons Atribución-CompartirIgual 4.0 Internacional.

