Flora de la piel
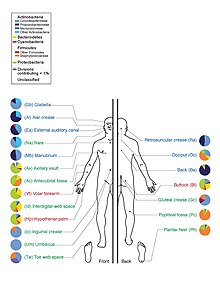
El término microbiota de la piel también conocido comúnmente como flora de la piel, se refiere a los microorganismos que residen en la piel, en este caso la piel humana. La microbiota cutánea está formada por bacterias, hongos y parásitos que constituyen un complejo ecosistema en permanente interacción con la piel del huésped.[1] De las bacterias hay alrededor de 1000 especies en la piel humana de diecinueve filos.[2][3] La mayoría se encuentran en las capas superficiales de la epidermis y en las partes superiores de los folículos capilares.
La flora de la piel generalmente no es patógena y es comensal (no es dañina para su huésped) o mutualista (ofrece un beneficio). Los beneficios que las bacterias pueden ofrecer incluyen evitar que los organismos patógenos transitorios colonicen la superficie de la piel, ya sea compitiendo por nutrientes, secretando químicos contra ellos o estimulando el sistema inmunológico de la piel.[4] Sin embargo, los microbios residentes pueden causar enfermedades de la piel e ingresar al sistema sanguíneo, creando enfermedades potencialmente mortales, particularmente en personas inmunodeprimidas.
Variedad de especies[editar]
En la superficie cutánea cohabitan bacterias, hongos y parásitos, que constituyen un complejo ecosistema que participa activamente en una función protectora de la piel, como barrera física y como barrera inmunológica.[1]
Bacterias[editar]

El número estimado de especies presentes entre las bacterias de la piel ha cambiado radicalmente mediante el uso de ARN ribosómico 16S para identificar las especies bacterianas presentes en las muestras de piel directamente de su material genético. Anteriormente, dicha identificación dependía del cultivo microbiológico en el que muchas variedades de bacterias no crecían y, por lo tanto, estaban ocultas a la ciencia.[2]
Staphylococcus epidermidis y Staphylococcus aureus se consideraron dominantes a partir de investigaciones de cultivos. Sin embargo, la investigación del ARN ribosómico 16S revela que, si bien son comunes, estas especies representan solo el 5% de las bacterias de la piel.[5] Sin embargo, la variedad de piel proporciona un hábitat rico y diverso para las bacterias. La mayoría proviene de cuatro filos: actinobacterias (51.8%), firmicutes (24.4%), proteobacterias (16.5%) y bacteroidetes (6.3%).

Hay tres áreas ecológicas principales: sebáceas, húmedas y secas. Las especies de propionibacterias y estafilococos fueron las principales especies en áreas sebáceas. En lugares húmedos del cuerpo dominan las cornebacterias junto con los estafilococos. En las zonas secas, hay una mezcla de especies, pero Betaproteobacteria y Flavobacteriales son dominantes. Ecológicamente, las áreas sebáceas tenían mayor riqueza de especies que las húmedas y secas. Las áreas con menor similitud de especies entre personas fueron los espacios entre los dedos, los espacios entre los dedos de los pies, las axilas y el muñón del cordón umbilical. La mayoría eran de manera similar al lado de los orificios nasales, cavidad nasal (dentro de la fosa nasal), y en la espalda.[2]
Frecuencia de los microbios cutáneos mejor estudiados[4][1] Organismo Observaciones Patogenicidad Staphylococcus epidermidis Común ocasionalmente patógeno Staphylococcus aureus Infrecuente usualmente patógeno Staphylococcus warneri Infrecuente ocasionalmente patógeno Streptococcus pyogenes Infrecuente usualmente patógeno Streptococcus mitis Frecuente ocasionalmente patógeno Cutibacterium acnes Frecuente ocasionalmente patógeno Corynebacterium spp. Frecuente ocasionalmente patógeno Acinetobacter johnsonii Frecuente ocasionalmente patógeno Pseudomonas aeruginosa Infrecuente ocasionalmente patógeno
Hongos[editar]
Un estudio del área entre los dedos de los pies en 100 adultos jóvenes encontró 14 géneros diferentes de hongos. Estos incluyen levaduras como Candida albicans, Rhodotorula rubra, Torulopsis y Trichosporon cutaneum, dermatofitos (hongos vivos de la piel) como Microsporum gypseum y Trichophyton rubrum y hongos no dermatofitos (hongos oportunistas que pueden vivir en la piel) como Rhizopus stolonifer, Trichosporon cutaneum, Trichosporon cutaneum Fusarium, Scopulariopsis brevicaulis, Curvularia, Alternaria alternata, Paecilomyces, Aspergillus flavus y Penicillium.[6]
Un estudio realizado por el Instituto Nacional de Investigación del Genoma Humano en Bethesda, Maryland, investigó el ADN de hongos de la piel humana en 14 lugares diferentes del cuerpo. Estos fueron el canal auditivo, entre las cejas, la parte posterior de la cabeza, detrás de la oreja, el talón, las uñas de los pies, entre los dedos de los pies, el antebrazo, la espalda, la ingle, las fosas nasales, el pecho, la palma y la curva del codo. El estudio mostró una gran diversidad de hongos en todo el cuerpo, siendo el hábitat más rico el talón, que alberga alrededor de 80 especies de hongos. Por el contrario, hay unas 60 especies en recortes de uñas y 40 entre los dedos. Otras áreas ricas son la palma, el antebrazo y el interior del codo, con 18 a 32 especies. La cabeza y el tronco albergan entre 2 y 10 cada uno.[7]
Una de las principales especies de la piel no humana es Batrachochytrium dendrobatidis, un hongo zoospórico quítrido y no hifal que causa la quitridiomicosis, una enfermedad infecciosa que se considera responsable de la disminución de las poblaciones de anfibios.[8]
Microbioma umbilical[editar]
El ombligo es un área del cuerpo que rara vez está expuesta a la luz ultravioleta, jabones o secreciones corporales[9] (el ombligo no produce secreciones ni aceites)[10] y porque es una comunidad casi intacta de bacterias[11] es una excelente parte del microbioma de la piel para estudiar.[12] El ombligo es un microbioma húmedo del cuerpo[13] (con alta humedad y temperaturas),[14] que contiene una gran cantidad de bacterias,[15] especialmente bacterias que favorecen condiciones húmedas como Corynebacterium[16] y Estafilococo.
El Proyecto de Biodiversidad Belly Button comenzó en la Universidad Estatal de Carolina del Norte a principios de 2011 con dos grupos iniciales de 35 y 25 voluntarios.[11] Los voluntarios recibieron hisopos de algodón estériles y se les pidió que insertaran los hisopos de algodón en sus ombligos, que giraran el hisopo de algodón tres veces y luego devolvieran el hisopo de algodón a los investigadores en un vial[17] que contenía 0,5 ml de solución buffer salina de fosfato al 10%. Investigadores de la Universidad Estatal de Carolina del Norte, liderados por Jiri Hulcr,[18] luego cultivaron las muestras hasta que las colonias bacterianas fueron lo suficientemente grandes como para ser fotografiadas y luego estas imágenes fueron publicadas en el sitio web del Proyecto de Biodiversidad de Belly Button (se dieron muestras a los voluntarios números para que puedan ver sus propias muestras en línea). Estas muestras se analizaron luego utilizando bibliotecas de ADNr 16S para poder identificar las cepas que no crecían bien en los cultivos.
Los investigadores de la Universidad Estatal de Carolina del Norte descubrieron que, si bien era difícil predecir cada cepa de bacterias en el microbioma del ombligo, podían predecir qué cepas serían frecuentes y qué cepas de bacterias serían bastante raras en el microbioma.[11] Se descubrió que los microbiomas del ombligo solo contenían unos pocos tipos prevalentes de bacterias (Staphylococcus, Corynebacterium, Actinobacteria, Clostridiales y Bacilli) y muchos tipos diferentes de bacterias raras. Se descubrieron otros tipos de organismos raros dentro de los ombligos de los voluntarios, incluidos tres tipos de Archaea (un organismo que generalmente vive en ambientes extremos) y dos de los tres tipos de Archaea se encontraron en un voluntario que afirmó no haberse bañado o duchado durante muchos años.
Staphylococcus y Corynebacterium se encuentran entre los tipos más comunes de bacterias que se encuentran en los ombligos de los voluntarios de este proyecto y estos tipos de bacterias son los tipos más comunes de bacterias que se encuentran en la piel humana en estudios más amplios del microbioma de la piel[19] (del cual forma parte el Proyecto de Biodiversidad de Belly Button).[11] (En estos estudios más amplios se ha encontrado que las mujeres generalmente tienen más Staphylococcus viviendo en sus microbiomas de la piel (generalmente Staphylococcus epidermidis)[17] y que los hombres tienen más Corynebacterium viviendo en sus microbiomas de la piel.)
Según el Proyecto de Biodiversidad del ombligo[11] en la Universidad Estatal de Carolina del Norte, se encuentran dos tipos de microorganismos en el ombligo y las áreas circundantes. Las bacterias transitorias (bacterias que no se reproducen)[13] forman la mayoría de los organismos que se encuentran en el ombligo, y se estima que se encontraron 1400 cepas diferentes en el 95% de los participantes del estudio.[20]
El Proyecto de Biodiversidad Belly Button está en curso y ahora ha tomado muestras de más de 500 personas.[11] El proyecto fue diseñado con el objetivo de contrarrestar esa idea errónea de que las bacterias siempre son dañinas para los humanos[21] y que los humanos están en guerra con las bacterias.[22] En realidad, la mayoría de las cepas de bacterias son inofensivas[14] si no son beneficiosas para el cuerpo humano.[23] Otro de los objetivos del proyecto es fomentar el interés público en la microbiología.[18] Trabajando en concierto con el Proyecto del Microbioma Humano, el Proyecto de Biodiversidad de la Ombligo también estudia las conexiones entre los microbiomas humanos y los factores de edad, sexo, origen étnico, ubicación y salud en general.[24]
Relación con el anfitrión[editar]
La microflora de la piel puede ser comensal, mutualista o patógena. A menudo pueden ser los tres, dependiendo de la fortaleza del sistema inmunitario de la persona.[4] La investigación sobre el sistema inmune en el intestino y los pulmones ha demostrado que la microflora ayuda al desarrollo de la inmunidad: sin embargo, dicha investigación solo ha comenzado sobre si este es el caso con la piel. Pseudomonas aeruginosa es un ejemplo de una bacteria mutualista que puede convertirse en un patógeno y causar una enfermedad: si ingresa al sistema circulatorio puede provocar infecciones en los sistemas óseo, articular, gastrointestinal y respiratorio. También puede causar dermatitis. Sin embargo, Pseudomonas aeruginosa produce sustancias antimicrobianas como el ácido pseudomónico (que se explotan comercialmente como la mupirocina). Esto funciona contra las infecciones por estafilococos y estreptococos. Pseudomonas aeruginosa también produce sustancias que inhiben el crecimiento de especies de hongos como Candida krusei, Candida albicans, Torulopsis glabrata, Saccharomyces cerevisiae y Aspergillus fumigatus.[25] También puede inhibir el crecimiento de Helicobacter pylori.[26] Sus acciones antimicrobianas son tan importantes que se ha observado que "la eliminación de P. aeruginosa de la piel, mediante el uso de antibióticos orales o tópicos, puede permitir inversamente la colonización e infección por levaduras aberrantes".
Otro aspecto de las bacterias es la generación de olor corporal. El sudor es inodoro, sin embargo, varias bacterias pueden consumirlo y crear subproductos que los humanos pueden considerar pútridos (en contraste con las moscas, por ejemplo, que pueden encontrarlas atractivas / atractivas). Varios ejemplos son:
- Las propionibacterias en las glándulas sebáceas adolescentes y adultas pueden convertir sus aminoácidos en ácido propiónico.
- Staphylococcus epidermidis crea olor corporal al romper el sudor en ácido isovalérico (ácido 3-metilbutanoico).[27]
- Bacillus subtilis crea un fuerte olor a pie.[28]
Defensas de la piel[editar]
Péptidos antimicrobianos[editar]
La piel crea péptidos antimicrobianos como las catelicidinas que controlan la proliferación de microbios de la piel. Las catelicidinas no solo reducen el número de microbios directamente, sino que también causan la secreción de liberación de citocinas que induce inflamación, angiogénesis y reepitelización. Condiciones como la dermatitis atópica se han relacionado con la supresión en la producción de catelicidina.[29] En la rosácea, el procesamiento anormal de catelicidina causa inflamación. La psoriasis se ha relacionado con el auto-ADN creado a partir de péptidos de catelicidina que causa autoinflamación. Un factor importante que controla la catelicidina es la vitamina D3.[30]
Acidez[editar]
Las capas superficiales de la piel son naturalmente ácidas (pH 4-4.5) debido al ácido láctico en el sudor y producido por bacterias de la piel.[31] A este pH, la flora mutualista como Staphylococci, Micrococci, Corynebacterium y Propionibacteria crecen pero no bacterias transitorias como las bacterias Gram negativas como Escherichia y Pseudomonas o las Gram positivas como Staphylococcus aureus. Otro factor que afecta el crecimiento de bacterias patológicas es que las sustancias antimicrobianas secretadas por la piel mejoran en condiciones ácidas. En condiciones alcalinas, las bacterias dejan de adherirse a la piel y se eliminan más fácilmente. Se ha observado que la piel también se hincha en condiciones alcalinas y se abre permitiendo el movimiento hacia la superficie.
Sistema inmune[editar]
Si se activa, el sistema inmune en la piel produce inmunidad celular contra microbios como los dermatofitos (hongos de la piel).[32] Una reacción es aumentar el recambio del estrato córneo y eliminar el hongo de la superficie de la piel. Los hongos de la piel como Trichophyton rubrum han evolucionado para crear sustancias que limitan la respuesta inmune a ellos. El desprendimiento de la piel es un medio general para controlar la acumulación de flora sobre la superficie de la piel.
Enfermedades de la piel[editar]
Los microorganismos desempeñan un papel en las enfermedades cutáneas no infecciosas como la dermatitis atópica,[33] rosácea, psoriasis,[34] y el acné[35] piel dañada puede hacer que las bacterias no patógenas se vuelvan patógenas.[36] La diversidad de especies en la piel está relacionada con el desarrollo posterior de dermatitis.[37]
Acné común[editar]
El acné común es una afección cutánea común caracterizada por la producción excesiva de sebo por la unidad pilosebácea y la inflamación de la piel.[38] Las áreas afectadas generalmente están colonizadas por Propionibacterium acnes; miembro de la microbiota comensal incluso en personas sin acné.[39] Las altas poblaciones de P. acnes están relacionadas con el acné vulgar, aunque solo ciertas cepas están fuertemente asociadas con el acné, mientras que otras con una piel sana. La población relativa de P. acnes es similar entre los que tienen acné y los que no.
El tratamiento actual incluye medicamentos antibacterianos tópicos y sistémicos que resultan en una disminución de la colonización y/o actividad de P. acnes.[40] El tratamiento probiótico potencial incluye el uso de Staphylococcus epidermidis para inhibir el crecimiento de P. acnes . S. epidermidis produce ácido succínico que se ha demostrado que inhibe el crecimiento de P. acnes.[41] También se ha demostrado que Lactobacillus plantarum actúa como un antiinflamatorio y mejora las propiedades antimicrobianas de la piel cuando se aplica tópicamente. También se demostró que es eficaz para reducir el tamaño de la lesión de acné.[42]
Dermatitis atópica[editar]
Las personas con dermatitis atópica han mostrado un aumento en las poblaciones de Staphylococcus aureus tanto en piel lesional como no lesional.[39] Los brotes de dermatitis atópica están asociados con una baja diversidad bacteriana debido a la colonización por S. aureus y, después del tratamiento estándar, se ha observado que la diversidad bacteriana aumenta.
Los tratamientos actuales incluyen combinaciones de antibióticos tópicos o sistémicos, corticosteroides y baños de lejía diluidos.[43] Los posibles tratamientos probióticos incluyen el uso de las bacterias cutáneas comensales, S. epidermidis, para inhibir el crecimiento de S. aureus. Durante los brotes de dermatitis atópica, se ha demostrado que los niveles de población de S. epidermidis aumentan como un intento de controlar las poblaciones de S. aureus.[39]
La baja diversidad microbiana intestinal en los bebés se ha asociado con un mayor riesgo de dermatitis atópica.[44] Los bebés con eccema atópico tienen niveles bajos de Bacteroides y niveles altos de Firmicutes. Las bacteroides tienen propiedades antiinflamatorias que son esenciales contra la dermatitis. (Ver microbiota intestinal )
Psoriasis vulgar[editar]
La psoriasis vulgar generalmente afecta los sitios de la piel más secos, como los codos y las rodillas. Las áreas secas de la piel tienden a tener una gran diversidad microbiana y menos poblaciones que los sitios sebáceos.[40] Un estudio que utiliza técnicas de muestreo de hisopos muestra que las áreas ricas en Firmicutes (principalmente Streptococcus y Staphylococcus) y Actinobacteria (principalmente Corynebacterium y Propionibacterium) están asociadas con la psoriasis.[45] Mientras que otro estudio que usa biopsias asocia niveles aumentados de Firmicutes y Actinobacterias con una piel sana.[46] Sin embargo, la mayoría de los estudios muestran que las personas afectadas por la psoriasis tienen una menor diversidad microbiana en las áreas afectadas.
Los tratamientos para la psoriasis incluyen agentes tópicos, fototerapia y agentes sistémicos.[47] La investigación actual sobre el papel de la microbiota de la piel en la psoriasis es inconsistente, por lo tanto, no existen tratamientos probióticos potenciales.
Rosácea[editar]
La rosácea generalmente está conectada a sitios sebáceos de la piel. Los ácaros de la piel Demodex folliculorum producen lipasas que les permiten usar el sebo como fuente de alimento, por lo tanto tienen una gran afinidad por los sitios sebáceos de la piel. Aunque es parte de la microbiota cutánea comensal, los pacientes afectados con rosácea muestran un aumento de D. folliculorum en comparación con individuos sanos, lo que sugiere patogenicidad.[48]
Bacillus oleronius, un microbio asociado a Demodex, no se encuentra típicamente en la microbiota cutánea comensal, pero inicia vías inflamatorias cuyo mecanismo de inicio es similar al de los pacientes con rosácea.[39] Las poblaciones de S. epidermidis también se han aislado de pústulas de pacientes con rosácea. Sin embargo, es posible que Demodex los haya trasladado a áreas que favorecen el crecimiento, ya que Demodex ha demostrado transportar bacterias alrededor de la cara.[49]
Los tratamientos actuales incluyen antibióticos tópicos y orales y terapia con láser.[50] Como la investigación actual aún no ha demostrado un mecanismo claro para la influencia de Demodex en la rosácea, no existen tratamientos probióticos potenciales.
Clínico[editar]
Dispositivos infectados[editar]
Los microbios de la piel son una fuente potencial de dispositivos médicos infectados, como los catéteres.[51]
Higiene[editar]
Es importante tener en cuenta que la piel humana es anfitriona de numerosas especies bacterianas y fúngicas, algunas de las cuales se sabe que son dañinas, algunas son beneficiosas y la gran mayoría no se investigó. El uso de jabones bactericidas y fungicidas conducirá inevitablemente a poblaciones bacterianas y fúngicas que son resistentes a los productos químicos empleados.
Contagio[editar]
La flora de la piel no pasa fácilmente entre las personas: 30 segundos de fricción moderada y contacto seco con las manos dan como resultado una transferencia de solo el 0.07% de la flora natural de las manos desnudas con un mayor porcentaje de guantes.[52]
Eliminación[editar]
El lavado antimicrobiano más efectivo (reducción del 60 al 80%) es con etanol, isopropanol y n-propanol. Los virus son los más afectados por las altas concentraciones (95%) de etanol, mientras que las bacterias son más afectadas por el n-propanol.[53]
Los jabones no medicados no son muy efectivos como lo ilustran los siguientes datos. Los trabajadores de la salud se lavaron las manos una vez con jabón líquido no medicado durante 30 segundos. Los estudiantes / técnicos por 20 veces.[54]
Flora de la piel en dos grupos hospitalarios en unidades formadoras de colonias por mL. condición de piel y manos del grupo no lavado lavado Trabajadores sanitarios sanos 3.47 3.15 Trabajadores de la salud enfermos 3.33 3.29 Estudiantes/técnicos sanos 4.39 3.54 Estudiantes/técnicos enfermos 4.58 4.43
Un uso importante del lavado de manos es prevenir la transmisión de la flora cutánea resistente a los antibióticos que causa infecciones adquiridas en el hospital, como el Staphylococcus aureus resistente a la meticilina. Si bien dicha flora se ha vuelto resistente a los antibióticos debido a los antibióticos, no hay evidencia de que los antisépticos o desinfectantes recomendados seleccionen organismos resistentes a los antibióticos cuando se usan en el lavado de manos.[55] Sin embargo, muchas cepas de organismos son resistentes a algunas de las sustancias utilizadas en jabones antibacterianos como el triclosán.
Una encuesta de jabones en barra en clínicas de dentistas descubrió que todos tenían su propia flora y, en promedio, de dos a cinco géneros diferentes de microorganismos con los que tienen más probabilidades de tener más variedades de especies.[56] Otra encuesta de jabones en los baños públicos encontró aún más flora.[57] Otro estudio encontró que los jabones muy secos no están infectados, mientras que todos descansan en piscinas de agua.[58] Sin embargo, la investigación sobre el jabón que estaba especialmente infectado encontró que la flora del jabón no se transmite a las manos.[59]
Piel dañada[editar]
Lavar la piel repetidamente puede dañar la capa externa protectora y causar pérdida transepidérmica de agua. Esto se puede ver en la aspereza caracterizada por descamación y sequedad, picazón, dermatitis provocada por microorganismos y alérgenos que penetran en la capa corneal y enrojecimiento. Usar guantes puede causar más problemas, ya que produce un ambiente húmedo que favorece el crecimiento de microbios y también contiene irritantes como el látex y el polvo de talco.[60]
El lavado de manos puede dañar la piel porque la capa superior del estrato córneo de la piel consta de 15 a 20 capas de discos de queratina, corneocitos, cada uno de los cuales está rodeado por una película delgada de lípidos de la piel que pueden eliminarse con alcoholes y detergentes.[61]
También se ha encontrado que la piel dañada definida por grietas extensas en la superficie de la piel, enrojecimiento generalizado o sangrado ocasional se coloniza con mayor frecuencia por Staphylococcus hominis y es más probable que sean resistentes a la meticilina.[60] Aunque no está relacionado con una mayor resistencia a los antibióticos, la piel dañada también era más como colonizada por Staphylococcus aureus, bacterias gramnegativas, Enterococos y Candida.
Comparación con otra flora[editar]
La flora de la piel es diferente de la del intestino, que es predominantemente Firmicutes y Bacteroidetes.[62] También hay un bajo nivel de variación entre las personas que no se encuentra en los estudios intestinales.[5] Sin embargo, tanto la flora intestinal como la de la piel carecen de la diversidad que se encuentra en la flora del suelo.[2]
Véase también[editar]
- Enfermedad bacteriana
- Olor corporal
- Flora intestinal
- Flora humana
- Proyecto de microbioma humano
- Microbiología médica
- Ecología microbiana
- Microflora
- Microbiología oral
- Piel
- Flora vaginal
- Zeaspora
Referencias[editar]
- ↑ a b Santamaría González V., Alvarado Delgadillo A. (2013 ). «Microbiota de la piel: el ecosistema cutáneo». Rev Asoc Colomb Dermatol (Revisión, PDF) 21 (2): 147-158.
- ↑ a b c d «Topographical and Temporal Diversity of the Human Skin Microbiome». Science 324 (5931): 1190-1192. 2009. Bibcode:2009Sci...324.1190G. PMC 2805064. PMID 19478181. doi:10.1126/science.1171700.
- ↑ Pappas S. (2009). Your Body Is a Wonderland ... of Bacteria. ScienceNOW Daily News
- ↑ a b c «Skin microbiota: a source of disease or defence?». Br J Dermatol 158 (3): 442-55. 2008. PMC 2746716. PMID 18275522. doi:10.1111/j.1365-2133.2008.08437.x.
- ↑ a b «A diversity profile of the human skin microbiota». Genome Res 18 (7): 1043-50. 2008. PMC 2493393. PMID 18502944. doi:10.1101/gr.075549.107.
- ↑ «Fungal flora of human toe webs». Mycoses 45 (11–12): 488-91. 2002. PMID 12472726. doi:10.1046/j.1439-0507.2002.00796.x.
- ↑ BBC News item
- ↑ Voyles, Jamie; Young, Sam; Berger, Lee; Campbell, Craig; Voyles, Wyatt F.; Dinudom, Anuwat; Cook, David; Webb, Rebecca et al. (23 de octubre de 2009). «Pathogenesis of Chytridiomycosis, a Cause of Catastrophic Amphibian Declines». Science (en inglés) 326 (5952): 582-585. Bibcode:2009Sci...326..582V. ISSN 0036-8075. PMID 19900897. doi:10.1126/science.1176765.
- ↑ Ecological Society of America (4 de agosto de 2011). «Bellybutton microbiomes: Ecological research on the human biome» (Press Release). ScienceDaily. Consultado el 20 de abril de 2013.
- ↑ Nierenberg, Cari (14 de abril de 2011). «New meaning to 'navel-gazing': Scientists study belly button bacteria». Consultado el 29 de septiembre de 2013.
- ↑ a b c d e f Hulcr, Jirir; Andrew M. Latimer; Jessica B. Henley; Nina R. Rountree; Noah Fierer; Andrea Lucky; Margaret D. Lowman; Robert R. Dunn (7 de noviembre de 2012). «A Jungle in There: Bacteria in Belly Buttons are Highly Diverse, but Predictable». PLoS ONE 7 (11): e47712. Bibcode:2012PLoSO...747712H. PMC 3492386. PMID 23144827. doi:10.1371/journal.pone.0047712.
- ↑ «The Wild Life of Your Body». Consultado el 1 de septiembre de 2013.
- ↑ a b Kong, Hiedi (17 de junio de 2011). «Skin microbiome: genomics-based insights into the diversity and role of skin microbes». Trends Mol. Med. 17 (6): 320-8. PMC 3115422. PMID 21376666. doi:10.1016/j.molmed.2011.01.013.
- ↑ a b Grice, Elizabeth; Julia Segre (9 de abril de 2011). «The Skin Microbiome». Nat Rev Microbiol 9 (4): 244-53. PMC 3535073. PMID 21407241. doi:10.1038/nrmicro2537.
- ↑ Kaplan, Karen (1 de junio de 2009). «Study shows you're covered in bacteria - live with it.». The Star. Archivado desde el original el 11 de noviembre de 2013. Consultado el 29 de septiembre de 2013.
- ↑ Grice, Elizabeth; Heidi H. Kong; Sean Conlan; Clayton B. Deming; Joie Davis; Alice C. Young; Gerard G. Bouffard; Robert W. Blakesley et al. (29 de mayo de 2009). «Topographical and Temporal Diversity of the Human Skin Microbiome». Science 324 (5931): 1190-2. Bibcode:2009Sci...324.1190G. PMC 2805064. PMID 19478181. doi:10.1126/science.1171700.
- ↑ a b Parker-Pope, Tara (14 de abril de 2011). «What's in Your Belly Button». Consultado el 29 de septiembre de 2013.
- ↑ a b Nierenberg, Cari. «New meaning to 'navel-gazing': Scientists study Belly Button Bacteria». Consultado el 29 de septiembre de 2013.
- ↑ Callewaert, Chris; Frederiek-Maarten Kerckhof; Michael S. Granitsiotis; Mireille Van Gele; Tom Van de Wiele; Nico Boon (12 de agosto de 2013). «Characterization of Staphylococcus and Corynebacterium Clusters in the Human Axillary Region». PLOS ONE 8 (8): e70538. Bibcode:2013PLoSO...870538C. PMC 3741381. PMID 23950955. doi:10.1371/journal.pone.0070538.
- ↑ Saunders, Chris (12 de julio de 2011). «Navel gazing at NC State leads to important discovery». Red & White for Life :: NC State University Alumni Association. Consultado el 20 de abril de 2013.
- ↑ Aldhous, Peter. «Belly button biome is more than a piece of fluff». Archivado desde el original el 2 de octubre de 2013. Consultado el 29 de septiembre de 2013.
- ↑ «Human microbes». Consultado el 29 de septiembre de 2013.
- ↑ Ahmad, Salar; Shailly Anand; Rup Lal (September 2012). «Skin Commensals Regulate Skin Immunity». Indian J. Microbiol. 52 (3): 517-8. PMC 3460106. PMID 23997352. doi:10.1007/s12088-012-0301-z.
- ↑ Grice, Elizabeth; Julia Segre (6 de junio de 2012). «The Human Microbiome: Our Second Genome». Annu Rev Genom Hum Genet 13 (1): 151-70. PMC 3518434. PMID 22703178. doi:10.1146/annurev-genom-090711-163814.
- ↑ Kerr JR (1994). «Suppression of fungal growth exhibited by Pseudomonas aeruginosa». J Clin Microbiol 32 (2): 525-7. PMC 263067. PMID 8150966. doi:10.1128/JCM.32.2.525-527.1994.
- ↑ «Inhibitory effects of various micro-organisms on the growth of Helicobacter pylori». Lett Appl Microbiol 40 (1): 81-6. 2005. PMID 15613007. doi:10.1111/j.1472-765X.2004.01632.x.
- ↑ «Foot odor due to microbial metabolism and its control». Can. J. Microbiol. 52 (4): 357-64. 2006. PMID 16699586. doi:10.1139/w05-130.
- ↑ «Foot odor due to microbial metabolism and its control». Can J Microbiol 52 (4): 357-64. 2006. PMID 16699586. doi:10.1139/w05-130.
- ↑ Patra, Vijaykumar; Mayer, Gerlinde; Gruber-Wackernagel, Alexandra; Horn, Michael; Lembo, Serena; Wolf, Peter (2018). «Unique profile of antimicrobial peptide expression in polymorphic light eruption lesions compared to healthy skin, atopic dermatitis, and psoriasis». Photodermatology, Photoimmunology & Photomedicine 34 (2): 137-144. PMC 5888155. PMID 29044786. doi:10.1111/phpp.12355.
- ↑ «Antimicrobial peptides and the skin immune defense system». J Allergy Clin Immunol 122 (2): 261-6. 2008. PMC 2639779. PMID 18439663. doi:10.1016/j.jaci.2008.03.027.
- ↑ «Natural skin surface pH is on average below 5, which is beneficial for its resident flora». Int J Cosmet Sci. 28 (5): 359-70. 2006. PMID 18489300. doi:10.1111/j.1467-2494.2006.00344.x.
- ↑ Dahl MV (1993). «Suppression of immunity and inflammation by products produced by dermatophytes». J Am Acad Dermatol 28 (5 Pt 1): S19-S23. PMID 8496406. doi:10.1016/s0190-9622(09)80303-4.
- ↑ Baker BS (2006). «The role of microorganisms in atopic dermatitis». Clin Exp Immunol 144 (1): 1-9. PMC 1809642. PMID 16542358. doi:10.1111/j.1365-2249.2005.02980.x.
- ↑ «Molecular analysis of fungal microbiota in samples from healthy human skin and psoriatic lesions». J Clin Microbiol 44 (8): 2933-41. 2006. PMC 1594634. PMID 16891514. doi:10.1128/JCM.00785-06.
- ↑ «Acne vulgaris: an investigation into the number of anaerobic diphtheroids and members of the Micrococcaceae in normal and acne skin». Br J Dermatol 96 (6): 623-6. 1977. PMID 141301. doi:10.1111/j.1365-2133.1977.tb05206.x.
- ↑ «Microbial ecology of the skin». Annu Rev Microbiol 42 (1): 441-64. 1988. PMID 3144238. doi:10.1146/annurev.mi.42.100188.002301.
- ↑ Williams, Michael R.; Gallo, Richard L. (2017). «Evidence that Human Skin Microbiome Dysbiosis Promotes Atopic Dermatitis». Journal of Investigative Dermatology 137 (12): 2460-2461. PMC 5814121. PMID 29169458. doi:10.1016/j.jid.2017.09.010.
- ↑ Fitz-Gibbon, S; Shuta, T; Bor-Han, C; Nguyen, L; Du, C; Minghsun, L; Elashoff, D; Erfe, MC et al. (2013). «Propionibacterium acnes Strain Populations in the Human Skin Microbiome Associated with Acne». J Invest Dermatol 133 (9): 2152-2160. PMC 3745799. PMID 23337890. doi:10.1038/jid.2013.21.
- ↑ a b c d Grice, EA (2014). «The skin microbiome: potential for novel diagnostic and therapeutic approaches to cutaneous disease». Semin Cutan Med Surg 33 (2): 98-103. PMC 4425451. PMID 25085669. doi:10.12788/j.sder.0087. Archivado desde el original el 11 de abril de 2015. Consultado el 1 de marzo de 2020. Error en la cita: Etiqueta
<ref>no válida; el nombre «Grice2014» está definido varias veces con contenidos diferentes - ↑ a b Hannigan, GD; Grice, EA (2013). «Microbial ecology of the skin in the era of metagenomics and molecular microbiology». Cold Spring Harb Perspect Med 3 (12): a015362. PMC 3839604. PMID 24296350. doi:10.1101/cshperspect.a015362.
- ↑ Muya, S; Wang, Y; Yu, J; Kuo, S; Coda, A; Jiang, Y; Gallo, RL; Huang, CM (2013). «Fermentation of Propionibacterium acnes, a Commensal Bacterium in the Human Skin Microbiome, as Skin Probiotics against Methicillin-Resistant Staphylococcus aureus». PLoS ONE 8 (2): e55380. Bibcode:2013PLoSO...855380S. PMC 3566139. PMID 23405142. doi:10.1371/journal.pone.0055380.
- ↑ Muizzuddin, N; Maher, W; Sullivan, M; Schnittger, S; Mammone, T (2012). «Physiological effect of probiotic on skin.». J Cosmet Sci 63 (6): 385-95. PMID 23286870.
- ↑ Kong, HH; Oh, J; Deming, C; Conlan, S; Grice, EA; Beatson, MA; Nomicos, E; Polley, EC et al. (2012). «Temporal shifts in the skin microbiome associated with disease flares and treatment in children with atopic dermatitis». Genome Res 22 (5): 850-9. PMC 3337431. PMID 22310478. doi:10.1101/gr.131029.111.
- ↑ Abrahamsson, TR; Jakobsson, HE; Andersson, AF; Björkstén, B; Engstrand, L; Jenmalm, MC (2012). «Low diversity of the gut microbiota in infants with atopic eczema». J Allergy Clin Immunol 129 (2): 434-40, 440.e1-2. PMID 22153774. doi:10.1016/j.jaci.2011.10.025.
- ↑ Alekseyenko, AV; Perez-Perez, GI; De Souza, A; Strober, B; Gao, Z; Bihan, M; Li, K; Methé, BA et al. (2013). «Community differentiation of the cutaneous microbiota in psoriasis». Microbiome 1 (1): 31. PMC 4177411. PMID 24451201. doi:10.1186/2049-2618-1-31.
- ↑ Fahlén, A; Engstrand, L; Baker, BS; Powles, A; Fry, L (2012). «Comparison of bacterial microbiota in skin biopsies from normal and psoriatic skin.». Arch Dermatol Res 304 (1): 15-22. PMID 22065152. doi:10.1007/s00403-011-1189-x.
- ↑ Menter, A; Griffiths, CE (2007). «Current and future management of psoriasis.». Lancet 370 (9583): 272-84. PMID 17658398. doi:10.1016/S0140-6736(07)61129-5.
- ↑ Casas, C; Paul, C; Lahfa, M; Livideanu, B; Lejeune, O; Alvarez-Georges, S; Saint-Martory, C; Degouy, A et al. (2012). «Quantification of Demodex folliculorum by PCR in rosacea and its relationship to skin innate immune activation.». Exp Dermatol 21 (12): 906-10. PMID 23171449. doi:10.1111/exd.12030.
- ↑ Jarmuda, S; O'Reilly, N; Zaba, R; Jakubowicz, O; Szkaradkiewicz, A; Kavanagh, K (2012). «Potential role of Demodex mites and bacteria in the induction of rosacea.». J Med Microbiol 61 (Pt 11): 1504-10. PMID 22933353. doi:10.1099/jmm.0.048090-0.
- ↑ Cohen, AF; Tiemstra, JD (2002). «Diagnosis and treatment of rosacea». J Am Board Fam Pract. 15 (3): 214-7. PMID 12038728.
- ↑ «Propionibacterium acnes is a common colonizer of intravascular catheters». J Infect 56 (4): 257-60. 2008. PMID 18336916. doi:10.1016/j.jinf.2008.01.012.
- ↑ «Development of a method to measure bacterial transfer from hands». J Hosp Infect 72 (1): 43-9. 2009. PMID 19282052. doi:10.1016/j.jhin.2009.01.022.
- ↑ «Epidemiologic background of hand hygiene and evaluation of the most important agents for scrubs and rubs». Clin Microbiol Rev 17 (4): 863-93. 2004. PMC 523567. PMID 15489352. doi:10.1128/CMR.17.4.863-893.2004.
- ↑ «Hand washing: changes in the skin flora». Am J Infect Control 35 (6): 417-20. 2007. PMID 17660014. doi:10.1016/j.ajic.2006.07.012.
- ↑ «Use of germicides in the home and the healthcare setting: is there a relationship between germicide use and antibiotic resistance?». Infect Control Hosp Epidemiol 27 (10): 1107-19. 2006. PMID 17006819. doi:10.1086/507964.
- ↑ «Microbial contamination of "in use" bar soap in dental clinics». Indian J Dent Res 17 (2): 70-3. 2006. PMID 17051871. doi:10.4103/0970-9290.29888.
- ↑ «Contamination of bar soaps under "in-use" conditions». J Environ Pathol Toxicol Oncol 5 (4–5): 1-14. 1984. PMID 6394740.
- ↑ «Bacterial flora of commonly used soaps in three hospitals in Nigeria». East Afr Med J 84 (10): 489-95. 2007. PMID 18232270. doi:10.4314/eamj.v84i10.9567.
- ↑ «Washing with contaminated bar soap is unlikely to transfer bacteria». Epidemiol Infect 101 (1): 135-42. 1988. PMC 2249330. PMID 3402545. doi:10.1017/s0950268800029290.
- ↑ a b «Changes in bacterial flora associated with skin damage on hands of health care personnel». Am J Infect Control 26 (5): 513-21. 1998. PMID 9795681. doi:10.1016/s0196-6553(98)70025-2.
- ↑ Kownatzki E (2003). «Hand hygiene and skin health». J Hosp Infect 55 (4): 239-45. PMID 14629966. doi:10.1016/j.jhin.2003.08.018.
- ↑ «Diversity of the human intestinal microbial flora». Science 308 (5728): 1635-8. 2005. Bibcode:2005Sci...308.1635E. PMC 1395357. PMID 15831718. doi:10.1126/science.1110591.
Enlaces externos[editar]
- Esta obra contiene una traducción total derivada de «Skin flora » de Wikipedia en inglés, concretamente de esta versión, publicada por sus editores bajo la Licencia de documentación libre de GNU y la Licencia Creative Commons Atribución-CompartirIgual 4.0 Internacional.
- Celulitis, infección de la piel
- Proyecto de microbioma humano
- Libro de texto en línea de Bacteriología de Todar
