Patescibacteria
| Bacterias ultrapequeñas o CPR | ||
|---|---|---|
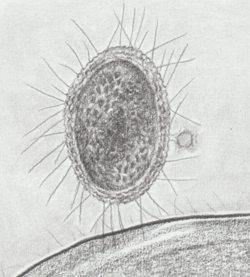 Bacteria ultrapequeña CPR de aguas subterráneas mostrando citoplasma denso, presencia de ribosomas, pili y pared compleja con capa S; además contacto con una bacteria grande y con un bacteriófago. | ||
| Taxonomía | ||
| Dominio: | Bacteria | |
| Filo: |
Patescibacteria Rinke et al. 2013 | |
| Clases | ||
| Sinonimia | ||
| ||
Patescibacteria, también llamado grupo CPR, grupo de las bacterias ultrapequeñas,[1] o, en razón a su tamaño nanométrico: nanobacterias;[2] conforma un amplio grupo de bacterias recientemente descrito (Brown, 2015).[3] Constituye un nuevo filo[4][5][6] de nanoorganismos que se estima pueda incluir desde 26% hasta 50% del total de la diversidad bacteriana,[7] aunque inicialmente se calculó en 15%. Estas bacterias han pasado desapercibidas porque hasta el momento no han podido ser cultivadas, pero recientemente se han identificado mediante análisis genómicos. El análisis de los genomas sugiere que estas bacterias presentan reducidas capacidades metabólicas, lo que ha impedido su cultivo por los procedimientos tradicionales.[8][3]
Probablemente todos los miembros de este grupo tienen células extremadamente pequeñas y genomas reducidos. Por sus capacidades metabólicas limitadas se supone que son simbiontes en las comunidades bacterianas. Hasta el momento, todos los genomas estudiados carecen completamente del ciclo de Krebs y de las cadenas respiratorias y la mayoría tienen una escasa o nula capacidad para sintetizar nucleótidos y aminoácidos. Se desconoce si estas limitaciones son una característica heredada de los primeros organismos o es una consecuencia de una posterior pérdida de capacidades.[3]
Características[editar]
Muchas características, incluyendo las ultraestructurales, están potencialmente relacionadas con la minimización del tamaño de la célula. En general, presentan las siguientes características:[3]
- El genoma siempre es pequeño en relación con las demás bacterias.
- El número de vías biosintéticas es reducido.
- Carecen de factores de biogénesis que fueron considerados universales en las bacterias.
- Tienen intrones y proteínas de autoempalme codificados dentro de sus genes de ARNr, una característica que rara vez se informa en bacterias.
- A todas les falta al menos una proteína ribosómica, la L30.
- Las limitaciones metabólicas son similares, pues todas tienen ciclos incompletos del ácido tricarboxílico y carecen de una compleja cadena de transporte de electrones, incluidas las oxidasas terminales y reductasas.
- Presentan similitudes evolutivas con las nanoarqueas (grupo DPANN de arqueas ultrapequeñas), por su pequeño tamaño, capacidades limitadas, divergencia filogenética profunda según los ribosomas, y porque en su mayoría son simbiontes o episimbiontes de otros miembros de la comunidad microbiana. Aún falta aclarar si este parecido se debe a características ancestrales comunes o a una evolución rápida convergente, o tal vez a una combinación de ambas situaciones.[9]
- Las de vida libre mostrarían un estilo de vida fermentativo. La posible asociación con organismos específicos, incluiría quimioautótrofos involucrados en los ciclos de nitrógeno, azufre y hierro.[10]
- Puede haber relación de contacto con bacteriófagos. El viroma detectado hasta el momento incluye bacteriófagos de cola y bacteriófagos filamentosos.
Tamaño[editar]
Son ultrapequeñas, técnicamente nanobacterias (bacterias de tamaño nanométrico). Es común encontrar diámetros de 250 nm.[2] El volumen es de 0.009 (± 0.002) μm³, pudiendo reducirse aún más cuando hay inanición.[1]
La información microscópica electrónica ha revelado un tamaño similar en arqueas ultrapequeñas.[11] A las bacterias muy pequeñas se les ha llamado "ultramicrobacterias" (UMB), y en este caso, las UMB no solo pertenecen al grupo CPR, sino también hay ejemplos que corresponden a Proteobacteria, Deinococcus-Thermus, Firmicutes, Acidobacteria, Dependentiae y Elusimicrobia; y en todos estos casos los genomas son pequeños, con un tamaño de 0.58 a 3.2 Mb.[12]
Estructura[editar]
Se ha encontrado que presentan:[1]
- Compartimento citoplasmático muy denso, con ADN apretado con estructura en espiral.
- Pocos ribosomas, en un número promedio de 42 (± 9.5) y situados en los extremos celulares en empaquetamientos descritos como polisomas.
- Membrana citoplasmática bacteriana lipídica con ácidos grasos con largas cadenas alquílicas (−CH2-) que tienen solo uno o dos grupos metilo terminales (−CH3). El análisis genómico muestra que no están los componentes necesarios para sintetizar los lípidos de membrana usualmente encontrados en bacterias.
- Pared celular compleja, presentan peptidoglucano y están envueltas en una notable capa S distintiva con una simetría hexagonal.
- Son gramnegativas debido a la capa S externa, sin embargo, la mayoría no presentan membrana externa, por lo que la estructura es monodérmica, básicamente similar a las posibacterias y arqueas.
- Estructuras tipo pili numerosas y discernibles con una variedad de longitudes y grosores, las cuales pasan a través de la pared celular. Ocasionalmente los pili se conectan a otras células grandes.
- Estructuras en forma de anillo enigmáticas asociadas con filamentos internamente.
Hábitat[editar]
Son bacterias muy diversas y están muy extendidas en entornos terrestres y marinos, siendo muy comunes en aguas subterráneas. Hasta ahora (2019) se han encontrado en la microbiota humana, agua potable, acuíferos, sedimento marino, suelo, sedimentos profundos del subsuelo, boca del delfín y otros entornos.[7] Se han encontrado como episimbiontes de otros microorganismos, de los cuales depende debido a su capacidad biosintética limitada. También son importantes en aguas termales, aunque con la mayor temperatura disminuye su abundancia.[13]
Los ambientes de aguas subterráneas han resultado contener una abundancia particularmente alta de estas bacterias, hasta un 38% del total del microbioma. También se encontraron en sedimentos de aguas profundas, permafrost y el subsuelo continental profundo.[10]
Filogenia[editar]

Una filogenia algo consesuada en el GTDB database y el Annotree es la siguiente:[14][15]
| Patescibacteria |
| ||||||||||||||||||||||||||||||||||||||||||||||||
Historia[editar]
Antecedentes[editar]
En varias ocasiones se informó de la presencia de bacterias de tamaño nanométrico. En 1981 se llevó a cabo microcultivos con formación de microcolonias de las llamadas "ultramicrobacterias", identificando por microfotografía bacterias heterótrofas de agua de mar de lento crecimiento.[16] El informe de 1996 de un meteorito de Marte que contendría microbios considerados demasiado pequeños para ser organismos,[17] provocó un largo debate sobre la existencia de nanobacterias. En los años siguientes, varias publicaciones propusieron la existencia de "nanobacterias" en muestras terrestres, a veces con implicaciones médicas, como la posible existencia de Nanobacterium como agente productor de patologías calcificantes;[18] y se postula la existencia de "nanobios" en medios ambientales.[19]
Identificación[editar]
Las bacterias de pequeño tamaño se han mostrado esquivas a la investigación debido a la dificultad de su cultivo. El primer grupo identificado genómicamente se encontró en 1998 en una fuente termal (piscina obsidiana) del parque nacional de Yellowstone pero también en medios mesófilos y se la llamó OP11, conocido ahora como Microgenomatia, que destacaron entre otras muestras por tener una identidad de secuencia muy baja y firmas de secuencia altamente atípicas.[20]
En 2004, se determinó que OP11 (Microgenomatia) es la más diversa de las divisiones bacterianas conocidas, mientras que OD1 (Parcubacteria) es la segunda más diversa. Además se determinó que estos dos grupos, sumando a la bacteria del río sulfuroso SR1 (Absconditabacterales) forman un clado.[21] Este clado fue confirmado combinando métodos de análisis del ARNr SSU y de genes codificadores de proteínas universales procariotas, denominándolo Patescibacteria en 2013, e incluyendo nuevos grupos como WS6 (Dojkabacteria), BH1, GN02 (Gracilibacteria) y TM7 (Saccharimonadia).[22] El nombre Patescibacteria procede del término latino patesco (desnudo, descubierto), haciendo referencia a las limitadas capacidades metabólicas de estas bacterias; sin embargo, este nombre aún no tiene amplio reconocimiento de la comunidad científica y se ha solicitado una consulta comunitaria antes de proponer un nombre formal.[9]
El novedoso método usando proteínas ribosómicas respalda a este supergrupo, lo coloca en posición basal de las bacterias y le denomina CPR.[8]
En 2018 en una propuesta taxonómica para estandarizar la taxonomía bacteriana se encontró que CPR o Patescibacteria comprende un único filo con varios taxones de rango inferior.[4]
Referencias[editar]
- ↑ a b c Luef, B., Frischkorn, K., Wrighton, K. et al. 2015, Diverse uncultivated ultra-small bacterial cells in groundwater. Nat Commun 6, 6372 (2015) doi:10.1038/ncomms7372
- ↑ a b Jeffrey Pommerville, Fundamentals of Microbiology. pag. 87 Jones & Bartlett Learning, LLC, ed. 11°, 2018
- ↑ a b c d Brown, C. T., Hug, L. A., Thomas, B. C., Sharon, I., Castelle, C. J., Singh, A., ... & Banfield, J. F. (2015). Unusual biology across a group comprising more than 15% of domain Bacteria. Nature, 523(7559), 208-211.
- ↑ a b Donovan H. Parks, Maria Chuvochina, David W. Waite, Christian Rinke, Adam Skarshewski, Pierre-Alain Chaumeil, Philip Hugenholtz (2018). A proposal for a standardized bacterial taxonomy based on genome phylogeny. Biorxiv.
- ↑ Genome database. Patescibacteria.
- ↑ Leandro N. Lemos, Julliane D. Medeiros, Francisco Dini-Andreote, Gabriel R. Fernandes, Alessandro M. Varani, Guilherme Oliveira, Victor S. Pylro (2019). Genomic signatures and co-occurrence patterns of the ultra-small Saccharimonadia (phylum CPR/Patescibacteria) suggest a symbiotic lifestyle. Online Library.
- ↑ a b Raphaël Méheust, David Burstein, Cindy J. Castelle & Jillian F. Banfield, 2019, The distinction of CPR bacteria from other bacteria based on protein family content. Nature Communications volume 10, Article number: 4173
- ↑ a b Hug, L. A., Baker, B. J., Anantharaman, K., Brown, C. T., Probst, A. J., Castelle, C. J., ... & Suzuki, Y. (2016). A new view of the tree of life. Nature Microbiology, 1, 16048.
- ↑ a b Cindy J. Castelle & Jillian F. Banfield 2018, Major New Microbial Groups Expand Diversity and Alter our Understanding of the Tree of Life. PERSPECTIVE, VOL 172, Issue 6, P1181-1197, 2018, DOI:https://doi.org/10.1016/j.cell.2018.02.016
- ↑ a b Herrmann Martina, Wegner C-E, Taubert M, Geesink P, Lehmann K, Yan L, Lehmann R, Totsche KU and Küsel K (2019) Predominance of Cand. Patescibacteria in Groundwater Is Caused by Their Preferential Mobilization From Soils and Flourishing Under Oligotrophic Conditions. Front. Microbiol. 10:1407. doi: 10.3389/fmicb.2019.01407
- ↑ Comolli, L. R., Baker, B. J., Downing, K. H., Siegerist, C. E. & Banfield, J. F. Three-dimensional analysis of the structure and ecology of a novel, ultra-small archaeon. ISME J. 3, 159–167 (2009).
- ↑ Liu J, Zhao R, Zhang J, Zhang G, Yu K, Li X and Li B (2018) Occurrence and Fate of Ultramicrobacteria in a Full-Scale Drinking Water Treatment Plant. Front. Microbiol. 9:2922. doi: 10.3389/fmicb.2018.02922
- ↑ Lin-Xing Chen et al. 2019, Candidate Phyla Radiation Roizmanbacteria From Hot Springs Have Novel and Unexpectedly Abundant CRISPR-Cas Systems Front. Microbiol., 03 May 2019 https://doi.org/10.3389/fmicb.2019.00928
- ↑ Mendler, K; Chen, H; Parks, DH; Hug, LA; Doxey, AC (2019). «AnnoTree: visualization and exploration of a functionally annotated microbial tree of life». Nucleic Acids Research 47 (9): 4442-4448. PMC 6511854. PMID 31081040. doi:10.1093/nar/gkz246. Archivado desde el original el 23 de abril de 2021. Consultado el 10 de febrero de 2022.
- ↑ «GTDB release 05-RS95». Genome Taxonomy Database.
- ↑ Francisco Torrella & Richard Y. Morita 1981, Microcultural Study of Bacterial Size Changes and Microcolony and Ultramicrocolony Formation by Heterotrophic Bacteria in Seawater Appl Environ Microbiol. 1981 Feb;41(2):518-27.
- ↑ McKay, D. S. et al. Search for past life on Mars: possible relic biogenic activity in Martian meteorite ALH84001. Science 273, 924–930 (1996)
- ↑ Kumar, C. et al 2011, An Overview on Clinical Implications of Nanobacteria. VL 23, DO 10.5005/jp-journals-10011-1168 Journal of Indian Academy of Oral Medicine and Radiology
- ↑ Uwins, P. J. R., Webb, R. I. & Taylor, A. P. Novel nano-organisms from Australian sandstones. Am. Mineral. 83, 1541–1550 (1998).
- ↑ Philip Hugenholtz 1998, Novel Division Level Bacterial Diversity in a Yellowstone Hot Spring. J Bacteriol. 1998 Jan;180(2):366-76.
- ↑ Harris, J. K., Kelley, S. T., & Pace, N. R. (2004). New perspective on uncultured bacterial phylogenetic division OP11. Applied and Environmental Microbiology, 70(2), 845-849).
- ↑ Rinke, C., Schwientek, P., Sczyrba, A., Ivanova, N. N., Anderson, I. J., Cheng, J. F., ... & Dodsworth, J. A. (2013). Insights into the phylogeny and coding potential of microbial dark matter. Nature, 499(7459), 431-437.
Enlaces externos[editar]
- Robert Sanders, Newfound groups of bacteria are mixing up the tree of life, Berkely News, 15 de junio de 2015.
